2. 上海海洋大学水产与生命学院 上海 201306
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
近10年来,我国和东南亚养殖对虾中普遍出现了多种新发病原的流行和危害,如致急性肝胰腺坏死病弧菌(Vibrio causing APHND, VAHPND)、偷死野田村病毒(covert mortality nodavirus, CMNV)和虾肝肠胞虫(Enterocytozoon hepatopenaei, EHP)等(Tourtip et al, 2009; FAO, 2013; Aranguren et al, 2017; Zhang et al, 2014、2017)。其中,VAHPND主要危害养殖早期的凡纳滨对虾(Litopenaeus vannamei),并导致被感染对虾出现90%以上的死亡率。CMNV感染能导致幼虾生长缓慢以及较高的累积死亡率,EHP感染主要引起生长迟滞(Flegel, 2012; FAO, 2013; Zhang et al, 2017; Fu et al, 2020)。这些新发病原的广泛流行,使我国和东南亚等地对虾养殖业遭受了严重损失(Flegel, 2012; FAO, 2013; Liu et al, 2018)。除上述3种新发病原外,近年来我国养殖对虾还出现了其他新病原感染情况,如2017年报道的虾血细胞虹彩病毒(shrimp hemocyte iridescent virus, SHIV)已引起国内外的广泛关注(Qiu et al, 2017、2018)。
作者课题组在开展对虾新发疫病病原学和流行病研究中发现,我国沿海地区养殖凡纳滨对虾中,一些患病凡纳滨对虾表现出游泳能力下降、生长迟缓等症状。检测发现,这些患病对虾不存在CMNV、EHP及传染性皮下及造血器官坏死病毒(infectious hypodermal and haematopoietic necrosis virus, IHHNV)的感染,但存在一种新型野田村病毒的感染,这种新型野田村病毒的形态和大小与CMNV类似,但其基因组中RNA依赖的RNA聚合酶(RdRp)基因序列与CMNV RdRp基因片段的相似性仅为78% (李小平等, 2019)。为了便于将这类新型的野田村病毒与CMNV区分开来,将其命名为行动障碍野田村病毒(movement disorder nodavirus, MDNV)(李小平等, 2019)。李小平等(2019)利用逆转录环介导等温核酸扩增(RT-LAMP)检测技术,对我国沿海省市养殖虾类中MDNV的流行情况进行初步分析发现,MDNV的流行率呈明显增加的趋势,其传播危害风险值得关注。
目前,MDNV的检测主要依赖RT-LAMP方法。LAMP方法具有便捷、快速和适用于现场检测等优点,但由于方法本身的特性,RT-LAMP在使用中存在对低拷贝起始模板定量困难的问题,使得其使用范围受到限制(王德国等, 2015)。为提高MDNV定量检测的准确性,本研究采用基于MDNV基因设计的探针和引物,开发了MDNV的一步法TaqMan实时荧光定量RT-PCR (TaqMan RT-PCR)检测技术。
1 材料与方法 1.1 对虾样品与实验材料对虾样品于2019年采集自山东、湖南、广西和海南等地的养殖场,共81份,包括凡纳滨对虾、日本对虾(Penaeus japonicus)、斑节对虾(Penaeus monodon)、罗氏沼虾(Macrobrachium rosenbergii)、克氏原螯虾(Procambarus clarkii)和澳洲龙虾(Cherax quadricarinatus)。从养殖池塘中取对虾活体,然后采集对虾的肝胰腺、鳃丝和附肢等组织样本,保存于RNAstore样本保存液(天根, 北京)中,使用低温冰袋运回实验室,冻存于-80℃冰箱备用。本研究用到的白斑综合征病毒(white spot syndrome virus, WSSV)、IHHNV、斑节对虾杆状病毒(Penaeus monodon bacuiovirus, MBV)和肝胰腺细小病毒(hepatopancreatic parvovirus, HPV)等病原阳性核酸来源于作者实验室。
1.2 样品总RNA制备 1.2.1 组织样本的总RNA提取与稀释将上述浸泡在RNAstore中的81份虾类样品,分别切取30 mg组织,放入加有700 μL RNAiso Plus (TaKaRa)的1.5 mL离心管中,充分研磨,然后按照RNAiso Plus使用说明书步骤进行样本组织总RNA提取。总RNA的质检结果OD260 nm/OD280 nm应在1.8~2.1范围内,并将总RNA浓度统一稀释到约200 ng/μL进行后续实验。
1.2.2 MDNV的逆转录套式PCR检测以上述制备的样本组织总RNA为模板,采用TaKaRa一步法逆转录PCR试剂盒对样本进行MDNV的逆转录套式PCR (RT-nPCR)检测,以便确认MDNV阳性核酸样品,用于后续MDNV新方法的建立和应用测试。
第1轮扩增反应的体系组分见表 1,扩增程序:50℃逆转录30 min,94℃变性2 min;94℃变性30 s,52℃退火30 s,72℃延伸1 min,36个循环;72℃延伸10 min;4℃保温。第2轮扩增反应的体系组分见表 2,扩增程序:95℃变性4 min;95℃变性20 s,51℃退火20 s,72℃延伸20 s,35个循环;72℃延伸5 min;4℃保温。MDNV RT-nPCR所用引物序列见表 3。
|
|
表 1 MDNV RT-nPCR的第1轮反应体系 Tab.1 Reaction mixture of the first round MDNV RT-nPCR |
|
|
表 2 MDNV RT-nPCR的第2轮反应体系 Tab.2 Reaction mixture of the second round of MDNV RT-nPCR |
|
|
表 3 MDNV RT-nPCR所用引物序列 Tab.3 Primers used in MDNV RT-nPCR |
第2轮PCR产物先进行1.5%琼脂糖凝胶电泳检测,然后利用Zymo胶回收试剂盒(Sigma-Aldrich, 美国)对目的条带进行纯化,将纯化后的产物连入pMD18-T载体(TaKaRa, 大连)并转化到大肠杆菌DH5α中进行克隆,挑取单克隆菌,送至生工生物工程(上海)股份有限公司测序验证。
1.3 引物和探针的设计选取MDNV的RdRp基因片段,利用Beacon Designer 7.0软件(Premier Biosoft, 美国)设计用于TaqMan RT-PCR检测的寡核苷酸引物和探针。引物包括MDNV-Taq-F1(5'-AATTCGACCCCGAAAGTG G-3')和MDNV-Taq-R1(5'-TCGGTCAAGCAAGCGT C-3'),该引物可扩增产生158 bp的PCR扩增产物;探针序列为5'-FAM TCCATCTTACGACCAGAA- ACCCAACATAMRA-3',探针在5'末端用6-羧基荧光素(FAM)标记,在3'末端用TAMRA淬灭剂标记。在MDNV-Taq-F1引物5'添加T7启动子序列,构建含有T7启动子序列的T7-MDNV-Taq-F1引物(5'-TAATA CGACTCACTATAGGGAATTCGACCCCGAAAGTGG-3'),以便用于RNA标准品构建。正向引物MDNV-Taq-F1、反向引物MDNV-Taq-R1、含有T7启动子序列的T7-MDNV-Taq-F1和探针均由生工生物工程(上海)股份有限公司合成。
1.4 质粒标准品和RNA标准品制备 1.4.1 质粒标准品制备以1.2中检测为MDNV阳性的对虾RNA为模板,利用引物MDNV-Taq-F1和MDNV-Taq-R1进行RT-PCR扩增,获取MDNV靶基因目标片段,将目标片段连入pMD18-T载体(TaKaRa)进行克隆,克隆片段测序验证正确后,用质粒小量制备试剂盒(Omega, 美国)从阳性克隆菌种中制备含有MDNV靶基因片段的pMD18-MDNV重组质粒。用NanoDrop 2000核酸分析仪(Thermo, 美国)测定上述重组质粒的浓度,并通过计算换算成目标基因拷贝数;将pMD18-MDNV重组质粒用无RNA酶的H2O进行10倍的梯度稀释,作为质粒标准品,并置于-80℃保存备用。
1.4.2 RNA标准品制备以pMD18-MDNV质粒为模板,以含有T7启动子序列的引物T7-MDNV- Taq-F1(5'-TAATACGACTCACTATAGGGAATTCGACCCCGAAAGTGG-3')和MDNV-Taq-R1进行PCR扩增,获取目标片段,按照in vitro Transcription T7 kit (for siRNA Synthesis)(TaKaRa)说明书,以表 4的转录体系进行体外转录,获得含有MDNV靶基因的RNA标准品。
|
|
表 4 体外转录RNA体系配制 Tab.4 Preparation of reaction mixtures for RNA transcription |
根据NanoDrop 2000测定转录获得的RNA标准品的浓度,计算出该MDNV RNA标准品的拷贝数,对其进行10倍浓度梯度稀释,用作反应模板。
1.5 TaqMan RT-PCR反应的优化TaqMan PCR反应在实时荧光定量PCR仪(CFX96, Bio-Rad, 美国)中进行,基础反应体系参照Luna® Universal Probe One-Step RT-qPCR kit (New England BioLabs)说明书配制,20 μL反应体系含有试剂1 Luna Universal Probe One-Step Reaction Mix (2×) X μL (X为试剂1的用量,设定范围为7~12 μL),试剂2 Luna WarmStart® RT Enzyme Mix X μL (X为试剂2的用量,设定范围为0.4~1.6 μL),不同终浓度的引物MDNV-TAQ-R1/MDNV-TAQ-F1、探针MDNV-TAQ- P各X μL (X为引物、探针浓度,设定范围各为0.1~ 0.6 μmol/L)。反应程序优化过程:首先将逆转录时间分别设为10、15、20和25 min进行优化;在优化逆转录时间后,再优化扩增程序:95℃预变性,1 min;95℃变性10 s,X℃ (X为退火温度梯度,设定范围为52℃~65℃)退火延伸30 s,共45个循环。优化过程中每组设3个重复,从3次重复实验中选定最佳温度、时间和试剂用量。
1.6 TaqMan RT-PCR方法的分析特异性以WSSV、IHHNV、MBV、HPV和CMNV阳性核酸为特异性分析的模板,同时以MDNV阳性核酸为阳性对照,健康凡纳滨对虾总RNA为阴性对照模板,无RNA酶的水为空白对照模板,进行MDNV TaqMan RT-PCR检测方法的分析特异性(analytical specificity, Asp)测试,每个模板的检测反应设置3个重复。
1.7 TaqMan RT-PCR方法的分析灵敏度采用优化后的TaqMan RT-PCR方法测试10倍浓度梯度稀释的pMD18-MDNV质粒(1.4×1010~ 100 copies/μL)和阴性对照,每个浓度测试设置3个重复,使用Bio-Rad CFX96软件(V6.0.14)绘制扩增Ct值(y)相对于质粒模板起始浓度(x)的标准曲线。基于该标准曲线明确MDNV TaqMan RT-PCR方法以DNA/cDNA为模板时对MDNV的分析灵敏度(analytical sensitivity, Ase)。
采用优化后的TaqMan RT-PCR方法测试10倍连续梯度稀释的RNA标准品模板(5.5×1010~5.5× 100 copies/μL)和阴性对照,使用Bio-Rad CFX96软件(V6.0.14)绘制扩增Ct值(y)相对于RNA模板起始浓度(x)的标准曲线。基于该标准曲线,明确MDNV TaqMan RT-PCR方法以RNA为模板时对MDNV的Ase。
1.8 TaqMan RT-PCR方法的可重复性使用10倍浓度梯度稀释的1.4×109~1.4×102 copies/μL pMD18-MDNV质粒作为模板,每个浓度重复3次,再重复上述实验3次,评估TaqMan RT-PCR检测方法的批内和批间重复性。使用变异系数(CV)评估检测反应的可重复性,所述CV定义为本研究中不同pMD18- MDNV质粒稀释物重复扩增的Ct值的标准偏差(SD)与Ct平均值的百分比;使用SPSS 13进行方差分析(ANOVA)。
1.9 TaqMan RT-PCR方法的应用利用新建立的TaqMan RT-PCR方法对2019年采自我国山东、湖南、广西等地的池塘养殖虾类样品进行MDNV检测,分析MDNV在不同养殖虾类中的流行情况及在这些地区养殖虾类中的流行率,评估该病毒的流行危害风险。
2 结果 2.1 TaqMan PCR反应的条件优化以试剂盒说明书推荐的初始反应体系为基础,经过对反应试剂各组分用量、逆转录时间、引物退火温度等优化,综合考虑最小标准偏差、最小Ct值及经济成本、稳定性等因素,优化后各反应组分用量:20.0 μL反应体系内,试剂1 Luna universal probe one-step reaction mix (2×) 11.0 μL,试剂2 Luna WarmStart® RT enzyme mix 8 μL,0.3 μmol/L引物MDNV-TAQ-R1和MDNV-TAQ-F1分别0.6 μL、探针MDNV-TAQ-P (0.4 μmol/L) 0.8 μL。优化后的逆转录温度为54.5℃,逆转录时间为15 min;优化后的退火温度为60.3℃。
2.2 TaqMan RT-PCR检测方法的特异性分析利用WSSV、MBV、IHHNV、HPV、CMNV和MDNV的阳性核酸为模板进行TaqMan RT-PCR,结果显示,只有MDNV的阳性核酸能够产生特异性的扩增曲线,而其他几种病原的阳性核酸均没有扩散曲线产生(图 1)。表明TaqMan RT-PCR方法能够特异性扩增MDNV核酸,而不与上述虾类常见的5种病原发生交叉反应。

|
图 1 MDNV TaqMan RT-PCR方法的分析特异性 Fig.1 Analytical specificity of MDNV TaqMan RT-PCR assay |
以10倍浓度梯度稀释的RNA标准品为模板,对TaqMan RT-PCR方法的灵敏度进行测试。结果显示,该方法对RNA标准品模板的最低检测限可低至5.5×101 copies/反应。在5.5×1010~5.5×101 copies/反应的起始模板浓度范围内,RNA标准品起始模板浓度对数值和Ct值之间呈线性关系(图 2a、图 2b)。扩增产物阈值循环数Ct值与标准模板起始量(Starting quantity, Sq)的关系曲线为Ct=-3.455 log(Sq)+45.053,R2=0.992。
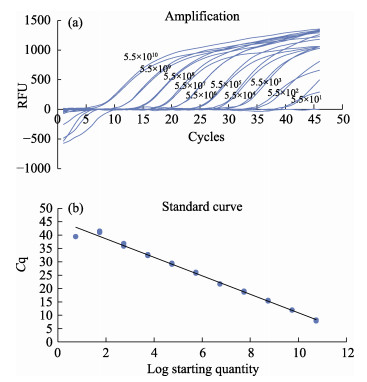
|
图 2 以10倍浓度梯度稀释的RNA标准品进行MDNV TaqMan RT-PCR的扩增曲线(a)和标准曲线(b) Fig.2 The amplification plots (a) and standard curve (b) of the serial 10-fold dilutions RNA standards by MDNV TaqMan RT-PCR |
利用10倍梯度稀释的pMD18-MDNV质粒为模板,对TaqMan RT-PCR方法的灵敏度进行测试。结果显示,TaqMan RT-PCR方法对质粒模板的最低检测限可低至1.4×101 copies/反应。在1.4×1010~1.4×101 copies/反应的起始模板浓度范围内,pMD18-MDNV质粒模板浓度对数值和Ct值之间呈现线性关系(图 3a、图 3b)。扩增产物阈值循环数Ct值与标准模板起始量的关系曲线为Ct=-3.327log(Sq)+42.188,R2=0.988。
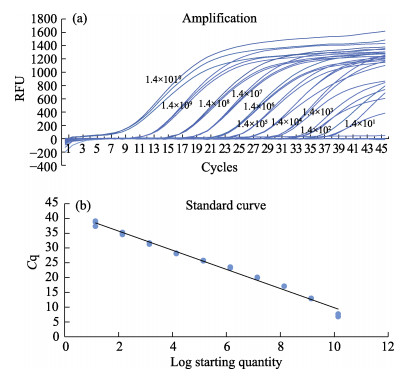
|
图 3 利用10倍浓度梯度稀释质粒标准品进行MDNV TaqMan RT-PCR的扩增曲线(a)和标准曲线(b) Fig.3 The amplification plots (a) and standard curve (b) of the serial 10-fold dilutions plasmids by MDNV TaqMan RT-PCR |
使用pMD18-MDNV质粒标准品为模板,对TaqMan RT-PCR检测方法的可重复性进行评估。结果显示,在起始模板浓度为1.4×109~1.4×102 copies/反应的范围内,TaqMan RT-PCR实验的重现性较好,批内和批间检测反应的Ct值CV分别小于1.27%和1.83% (表 5)。方差分析结果显示,各浓度梯度模板TaqMan RT- PCR反应的批间Ct值均差异不显著(P > 0.05)(表 6)。
|
|
表 5 TaqMan RT-PCR测定法的批内和批间重复性 Tab.5 Intra-assay and inter-assay variability of TaqMan RT-PCR assay |
|
|
表 6 TaqMan RT-PCR分析法的批间变异性 Tab.6 Analysis of variance of inter-assay variability of TaqMan RT-PCR assay |
利用TaqMan RT-PCR对2019年采自我国部分养殖地区的虾类样品进行分析发现(表 7),养殖凡纳滨对虾、罗氏沼虾和澳洲龙虾中均有MDNV阳性检出,其中,凡纳滨对虾和罗氏沼虾中MDNV的阳性检出率分别为23.5% (16/68)和16.6% (1/6),另外,在采自广西的2份澳洲龙虾样品中有1份检出MDNV阳性。从采样地域来看,广西、海南、湖南和山东等地的虾类样品中均有MDNV阳性检出。
|
|
表 7 TaqMan RT-PCR分析方法检测样品中阳性数据 Tab.7 Analysis of variance of inter-assay variability of TaqMan RT-PCR assay |
自1967年第1个野田村病毒(nodamura virus)在日本被分离鉴定后,目前野田村病毒科中已分离鉴定了19种病毒,这些病毒分属于阿尔法野田村病毒属(Alphanodavirus)和贝塔野田村病毒属(Betanodavirus) (King et al, 2011)。从虾类中分离的野田村病毒包括以下5种:罗氏沼虾野田村病毒(Macrobrachium rosenbergii nodavirus, MrNV)、凡纳滨对虾野田村病毒(Penaeus vannamei nodavirus, PvNV)、CMNV、MDNV和桃红对虾野田村病毒(Farfantepenaeus duorarum nodavirus, FdNV)。其中,MrNV主要感染罗氏沼虾,PvNV主要感染凡纳滨对虾,CMNV可感染虾类和鱼类,MDNV主要感染虾类,FdNV分离自健康桃红对虾,不引起宿主发病(Scherer et al, 1967; Tang et al, 2011; King et al, 2011; Ng et al, 2013; Zhang et al, 2014; 李小平等, 2019)。
CMNV被证实是引起养殖凡纳滨对虾发生病毒性偷死病(viral covert mortality disease, VCMD)的病原(Zhang et al, 2014)。2002-2003年前后,该病毒在我国南方省市的高密度养殖对虾中出现,并于2008- 2009年在广西、广东和福建等地养殖对虾中大规模暴发,2010年后开始传播到北方的主要对虾养殖地区(Zhang et al, 2017)。2013-2019年期间,我国沿海地区养殖对虾中CMNV的年度检出率依次为45.93% (130/283)、27.91% (84/301)、20.85% (54/259)、26.8% (68/254)、16.3% (63/387)、29.15% (58/199)和15.3% (28/183),CMNV表现出流行率高、流行范围广、产业危害严重的特点,使我国养殖对虾产业遭受了严重的经济损失(Zhang et al, 2017; 李小平等, 2019)。除感染主要养殖对虾种类外,CMNV还可引起对虾养殖池塘中多种共生生物的感染(Liu et al, 2018)。研究发现,牙鲆(Paralichthys olivaceus)、鲻虾虎鱼(Mugilogobius abei)和鲫鱼(Carassius auratus)等海淡水鱼类中也存在CMNV的自然感染;CMNV是目前已知的19种虾类病原中第1个能跨越物种障碍而感染鱼类的虾类病毒(Zhang et al, 2018; 王崇等, 2018; Wang et al, 2019)。
感染CMNV的对虾主要表现出肝胰腺萎缩、颜色变浅、空肠空胃、生长缓慢等症状,还可见病虾腹部肌肉不透明或局部发白(Huang, 2012; Zhang et al, 2014)。2016年,本实验室在开展对虾流行病研究中发现,沿海省市养殖凡纳滨对虾开始出现MDNV感染和流行;感染MDNV的凡纳滨对虾主要临床症状为游泳能力下降、生长迟缓等,与CMNV感染所致的症状不尽相同。电镜分析发现,MDNV的病毒颗粒大小与CMNV类似,但其RdRp基因序列与CMNV RdRp基因片段的相似性仅为78%,由此判断,MDNV是养殖对虾中出现的一种新型的野田村病毒。本实验室前期开发了MDNV的RT-LAMP检测技术,但在检测实践中发现以逆转录的样品cDNA为模板,当起始模板浓度过低时,MDNV的RT-LAMP方法无法准确定量模板。本研究建立并优化了MDNV的TaqMan RT-PCR方法,可实现对低浓度(1.4×101 copies/反应)模板的准确定量。
基于设计引物应尽可能选择在目标基因的保守区域内,而又不与近缘物种相应基因发生非特异性结合的原则(Rose et al, 2003; Zlateva et al, 2011; Cassar et al, 2017),本研究在依据MDNV RdRp基因设计TaqMan RT-PCR引物和探针时,首先利用多序列比对软件对MDNV RdRp靶基因与MrNV、PvNV和FdNV的RdRp基因进行序列比对分析。结果显示,MDNV RdRp靶基因与后三者RdRp基因的同源性低于25%,MDNV的TaqMan RT-PCR引物和探针与后三者RdRp基因及基因组的同源性低于30%。因此,所设计的引物和探针与MrNV、PvNV和FdNV不存在非特异性结合并产生非特异性扩增的可能性。多序列比对结果显示,MDNV RdRp靶基因与CMNV对应区段的同源性为79.87%,当以CMNV RNA为模板进行TaqMan RT-PCR方法特异性分析时,TaqMan RT- qPCR引物亦未与CMNV核酸发生非特异性反应。因此,本研究所建立的TaqMan RT-PCR方法可用于MDNV的特异性检测。传统的组织病理分析和反转录PCR方法在鉴定病原微生物时耗时较长、效率较低(Zhang et al, 2014; Tang et al, 2007),本研究优化后的MDNV TaqMan RT-PCR方法检测所需时间大约为1.5 h,在较短时间内即可高效地完成逆转录和核酸靶序列扩增,并通过探针荧光信号的变化监测整个反应过程,非常适于开展MDNV大规模检测。本实验室前期建立的MDNV RT-LAMP分析方法最低可检测到浓度为2.61×103 copies/反应的MDNV RNA标准品,当起始模板浓度低于1000 copies/反应时,起始模板浓度与扩增Ct值的相关系数则明显下降(李小平, 2019)。以RNA标准品为模板时,本研究建立的TaqMan RT-PCR最低可检测到5.5×101 copies/反应的RNA。当以质粒模拟cDNA为模板时,具有可检测扩增信号的最低起始模板浓度为1.4×101 copies/反应,说明该方法较前期建立的RT-LAMP方法具有更高的灵敏度。DNA模板浓度在1.4×1010~1.4×101 copies/反应之间、RNA模板浓度在5.5×1010~5.5×101 copies/反应之间时,MDNV TaqMan RT-PCR扩增反应的Ct值与log(Sq)呈良好的线性关系,反映出该方法适合对起始模板浓度范围较广的样品进行准确的定量分析。另外,在起始模板浓度为1.4×109~1.4×102 copies/反应的范围内,本研究建立的TaqMan RT-PCR方法的批内和批间CV分别小于1.27%和1.83%,表明该方法具有良好的稳定性和可重复性。
李小平等(2019)利用RT-LAMP方法对MDNV开展分子流行病学调查的结果显示,2016年我国沿海省市虾类样品中MDNV的阳性检出率为9.4%(24/254)。本研究利用TaqMan RT-PCR方法对2019年采自我国部分地区的养殖虾类样品进行MDNV流行情况分析结果显示,养殖凡纳滨对虾、罗氏沼虾和澳洲龙虾中均有MDNV的阳性检出,其中,凡纳滨对虾中MDNV的阳性检出率高达23.5%(16/68);从采样地域来看,广西、海南、湖南和山东等地的虾类样品均有MDNV阳性检出。上述研究表明,MDNV已扩散到更多虾类养殖地区,且具有较高的流行率,其传播危害风险值得进一步研究。
综上所述,本研究建立的MDNV TaqMan RT-PCR方法具有快速、灵敏和特异性强等特点,可为对虾养殖实践中MDNV的定性、定量检测与监测以及有效防控提供技术支持。养殖对虾中MDNV的高检出率也提示,当前MDNV这一新发病原在我国养殖甲壳类中的流行危害应引起水产管理部门和养殖从业人员的高度关注。
ARANGUREN L F, HAN J E, TANG K F J. Enterocytozoon hepatopenaei (EHP) is a risk factor for acute hepatopancreatic necrosis disease (AHPND) and septic hepatopancreatic necrosis (SHPN) in the Pacific white shrimp Penaeus vannamei. Aquaculture, 2017, 471: 37-42 DOI:10.1016/j.aquaculture.2016.12.038 |
CASSAR O, CHARAVAY F, TOUZAIN F, et al. A novel human T-lymphotropic virus type 1c molecular variant in an indigenous individual from New Caledonia, Melanesia. PLoS Neglected Tropical Diseases, 2017, 11(1): e0005278 DOI:10.1371/journal.pntd.0005278 |
FAO. Report of the FAO/MARD technical workshop on early mortality syndrome (EMS) or acute hepatopancreatic necrosis syndrome (AHPND) of cultured shrimp (under TCP/VIE/ 3304). Hanoi, Viet Nam, 25-27 June 2013. FAO Fisheries and Aquaculture Report No. 1053, 2013, Rome, 54 pp
|
FLEGEL T W. Historic emergence, impact and current status of shrimp pathogens in Asia. Journal of Invertebrate Pathology, 2012, 110(2): 166-173 DOI:10.1016/j.jip.2012.03.004 |
FU S, WEI D, YANG Q, et al. Horizontal plasmid transfer promotes the dissemination of Asian acute hepatopancreatic necrosis disease and provides a novel mechanism for genetic exchange and environmental adaptation. mSystems, 2020, 5(2): e00799-19 |
HUANG J. Experience in EMS/AHPNS from China. In: NACA. The Asia Pacific emergency regional consultation on the emerging shrimp disease: Early mortality syndrome (EMS)/acute hepatopancreatic necrosis syndrome (AHPNS). Bangkok, Thailand, 2012
|
KING A M Q, ADAMS M J, CARSTENS E B, et al. Virus taxonomy: Ninth report of the International Committee on Taxonomy of Viruses. San Diego: Elsevier Academic Press, 2011
|
LI X P, WAN X Y, ZHANG Q L, et al. Molecular epidemiological survey of covert mortality nodavirus (CMNV) in cultured crustaceans in China in 2016~2017. Progress in Fishery Sciences, 2019, 40(2): 65-73 [李小平, 万晓媛, 张庆利, 等. 2016~2017年中国沿海省市虾类偷死野田村病毒(CMNV)分子流行病学调查. 渔业科学进展, 2019, 40(2): 65-73] |
LIU S, WANG X H, XU T T, et al. Vectors and reservoir hosts of covert mortality nodavirus (CMNV) in shrimp ponds. Journal of Invertebrate Pathology, 2018, 154: 29-36 DOI:10.1016/j.jip.2018.03.011 |
NG T F F, ALAVANDI S, VARSANI A, et al. Metagenomic identification of a nodavirus and a circular ssDNA virus in semi-purified viral nucleic acids from the hepatopancreas of healthy Farfantepenaeus duorarum shrimp. Diseases of Aquatic Organisms, 2013, 105(3): 237-242 DOI:10.3354/dao02628 |
QIU L, CHEN M M, WAN X Y, et al. Characterization of a new member of Iridoviridae, shrimp hemocyte iridescent virus (SHIV), found in white leg shrimp (Litopenaeus vannamei). Scientific Reports, 2017, 7(1): 11834 DOI:10.1038/s41598-017-10738-8 |
QIU L, CHEN M M, WANG R Y, et al. Complete genome sequence of shrimp hemocyte iridescent virus (SHIV) isolated from white leg shrimp, Litopenaeus vannamei. Archives of Virology, 2018, 163(3): 781-785 DOI:10.1007/s00705-017-3642-4 |
ROSE T M, HENIKOFF J G, HENIKOFF S. CODEHOP (consensus-degenerate hybrid oligonucleotide primer) PCR primer design. Nucleic Acids Research, 2003, 31(13): 3763-3766 DOI:10.1093/nar/gkg524 |
SCHERER W F, HURLBUT H S. Nodamura virus from Japan: A new and unusual arbovirus resistant to diethyl ether and chloroform. American Journal of Epidemiology, 1967, 86(2): 271-285 DOI:10.1093/oxfordjournals.aje.a120737 |
TANG K F J, PANTOJA C R, REDMAN R M, et al. Development of in situ hybridization and RT-PCR assay for the detection of a nodavirus (PvNV) that causes muscle necrosis in Penaeus vannamei. Diseases of Aquatic Organisms, 2007, 75(3): 183-190 |
TANG K F J, PANTOJA C R, REDMAN R M, et al. Ultrastructural and sequence characterization of Penaeus vannamei nodavirus (PvNV) from Belize. Diseases of Aquatic Organisms, 2011, 94(3): 179-187 DOI:10.3354/dao02335 |
TOURTIP S, WONGTRIPOP S, STENTIFORD G D, et al. Enterocytozoon hepatopenaei sp. nov. (Microsporida: Enterocytozoonidae), a parasite of the black tiger shrimp Penaeus monodon (Decapoda: Penaeidae): Fine structure and phylogenetic relationships. Journal of Invertebrate Pathology, 2009, 102(1): 21-29 DOI:10.1016/j.jip.2009.06.004 |
WANG C, LIU S, LI X P. Infection of covert mortality nodavirus in Japanese flounder reveals host jump of the emerging alphanodavirus. Journal of General Virology, 2019, 100(2): 166-175 DOI:10.1099/jgv.0.001177 |
WANG C, WANG X H, LIU S, et al. Preliminary study on the natural infection of Carassius auratus with covert mortality nodavirus (CMNV). Progress in Fishery Sciences, 2019, 40(2): 25-32 [王崇, 王秀华, 刘爽, 等. 鲫鱼自然感染对虾偷死野田村病毒(CMNV)的初步研究. 渔业科学进展, 2019, 40(2): 25-32] |
WANG D G, WANG Y Z, WANG A P. Study on false-positive amplification of loop-mediated isothermal amplification. Journal of Xuchang University, 2015, 34(5): 81-83 [王德国, 王永真, 王爱萍. 环介导等温扩增技术的假阳性扩增研究. 许昌学院学报, 2015, 34(5): 81-83] |
ZHANG Q L, LIU Q, LIU S, et al. A new nodavirus is associated with covert mortality disease of shrimp. Journal of General Virology, 2014, 95(12): 2700-2709 DOI:10.1099/vir.0.070078-0 |
ZHANG Q L, LIU S, LI J, et al. Evidence for cross-species transmission of covert mortality nodavirus to new host of Mugilogobius abei. Frontiers in Microbiology, 2018, 9: 1447 DOI:10.3389/fmicb.2018.01447 |
ZHANG Q L, XU T T, WAN X Y, et al. Prevalence and distribution of covert mortality nodavirus (CMNV) in cultured crustacean. Virus Research, 2017, 233(2): 113-119 |
ZLATEVA K T, CRUSIO K M, LEONTOVICH A M, et al. Design and validation of consensus-degenerate hybrid oligonucleotide primers for broad and sensitive detection of corona- and toroviruses. Journal of Virological Methods, 2011, 177(2): 174-183 |



