2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071;
3. 荣成楮岛水产有限公司 山东 荣成 264312;
4. 荣成市海洋发展局 山东 荣成 264300
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao, Shandong 266071, China;
3. Rongcheng Chudao Aquaculture Corporation, Rongcheng, Shandong 264312, China;
4. Rongcheng Municipal Marine Development Bureau, Rongcheng, Shandong 264300, China
滤食性贝类是我国海水养殖的主要种类,通过摄食、生物沉积等过程在水层-底栖环境间发挥重要的生物耦连作用(Jones, 1996),其食物来源广泛,包括浮游植物、有机碎屑、浮游动物等(Fukumori et al, 2008)。目前,关于滤食性贝类的食性研究采用显微镜镜检法(Kasim et al, 2009; 乔芮, 2015)、脂肪酸标志法(许强, 2007)和稳定同位素分析(Riera et al, 1996)等方法。显微镜镜检所需样本量较大、费时、对检测人员要求较高,且在采样和固定过程中,可能会忽略一些小的、易碎的和低丰度的物种(Egge et al, 2013);脂肪酸标志法和稳定同位素分析法对食物来源的定性范围较宽,难以具体确定食物组成(许强, 2007; 刘刚等, 2018)。高通量测序技术能一次并行对几十万到几百万条DNA进行序列测定(石琼等, 2015),目前,该技术已被用于海参(Apostichopus japonicus) (Zhang et al, 2016)、海鞘(Pyrosomella verticilliata) (Metfies et al, 2013)和龙虾(Palinuridae) (rorke et al, 2012)等海洋生物的肠道内容物研究。
桑沟湾是我国北方典型的规模化贝类养殖海湾,位于山东半岛东端,北、西、南三面为陆地环抱,湾口朝东,面向黄海(宋云利等, 1996)。20世纪90年代末,桑沟湾开始规模化养殖长牡蛎(Crassostrea gigas)。近年来,长牡蛎已成为该区域主要的养殖贝类(傅明珠等, 2013)。长牡蛎具有很强的滤水能力,利用鳃丝选择和过滤食物(董波等, 2000),研究桑沟湾养殖长牡蛎的食物组成对于了解该区域的饵料状况及贝类的摄食生理响应,进而评估养殖容量具有重要意义。本研究选取真核生物18S rDNA可变区V4区作为标记基因,结合Illumina高通量测序技术,对桑沟湾养殖的长牡蛎和所处养殖水体中真核浮游生物的群落组成、多样性和丰度进行了分析,研究结果可为深入揭示长牡蛎的摄食生态学提供数据支撑。
1 材料与方法 1.1 样品采集2019年8月,在桑沟湾长牡蛎养殖区(37°3'12.42″N,122°33'44.60″E)采集了10个长牡蛎和3个所处养殖水体样品(图 1)。所有采集的长牡蛎均在自然状态下繁殖生长,采集区水温为24.7℃,盐度为31.6,pH为7.88。采集的长牡蛎平均壳高为(113.57±7.50) mm。测量完壳高后的长牡蛎现场进行解剖,打开贝壳后从口腔沿食道剪开,用一次性无菌吸管吸取胃含物,转移至已标号的1.5 mL冻存管中,将冻存管投于液氮罐中带回实验室等待进一步处理。同时,在牡蛎养殖区采集表层(距水面0.5 m)海水1 L,经0.45 μm的醋酸纤维滤膜过滤后,滤膜置于冻存管内液氮保存,带回实验室迅速转移至-80℃备用。
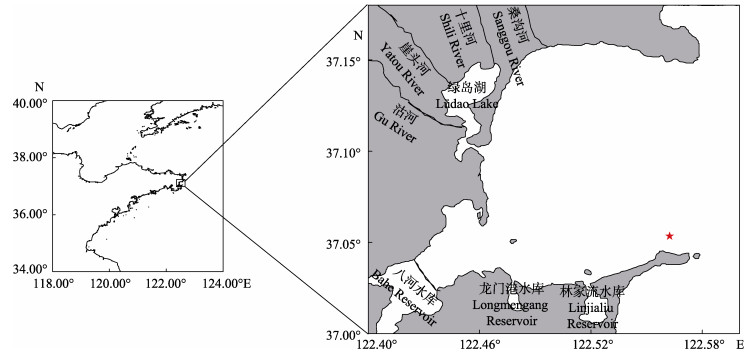
|
图 1 长牡蛎采样位点 Fig.1 Sampling stations of C. gigas |
将上述13个样品分别用灭菌杵棒充分研磨,加入500 μL的DNA裂解液,于恒温水浴锅中55℃水浴48 h,使用试剂盒(Omega M5635-02)提取每个样本的基因组DNA。采用0.8%琼脂糖凝胶电泳检测DNA质量,采用紫外分光光度计对DNA定量。
1.3 18S rDNA的扩增及扩增产物的回收纯化取适量的样品于离心管中,使用无菌水稀释样品至1 ng/μL,以稀释后的基因组DNA为模板,使用通用引物V547(5'-CCAGCASCYGCGGTAATTCC-3')和V4R(5'-ACTTTCGTTCTTGATYRA-3')扩增18S rDNA V4区(Stoeck et al, 2010)。PCR产物经2%琼脂糖凝胶电泳鉴定,并用Axygen凝胶回收试剂盒回收。
1.4 文库构建和上机测序采用Illumina公司的TruSeq Nano DNA LT Library Prep Kit制备测序文库,构建的文库经定量和文库检测,合格后的利用Mi Seq reagent kit V3(600 cycles)进行双端测序。
1.5 数据分析测序得到的原始数据,存在一定比例的干扰数据,为了保证分析结果的准确,首先对原始数据进行拼接、过滤,得到有效数据。基于有效数据按97%的序列相似度进行归并和OTU划分,选取每个OTU中丰度最高的序列作为该OTU的代表序列,与NCBI数据库比对,通过GenBank鉴定OTU代表性序列的微生物分类地位。同时,根据序列数计算样品的Alpha多样性指数,以得到样品内物种丰富度和均匀度信息、不同样品或分组间的共有和特有OTUs信息等。并用Kruskal-Wallis检验展示不同样本组间的alpha多样性差异,通过绘制热图对样品间关系进行展示。
2 结果与分析 2.1 测序数据及样品丰度及多样性分析对长牡蛎胃含物及所处养殖水体中浮游生物18S rDNA进行Illumina高通量测序,平均得到128, 789条原始序列,质量过滤平均得到111, 359条高质量序列,其中,长牡蛎胃含物的原始序列平均为128, 413条,高质量序列平均为113, 376条;长牡蛎养殖水体的原始序列平均为130, 044条,高质量序列平均为104, 638条。在97%相似性水平上,对111, 359条高质量序列划分OTUs,一共得到1345个OTUs,其中,长牡蛎胃含物的OTUs数平均为773个,养殖区水样OTUs平均为714个(表 1)。把得到的OTUs代表序列与NCBI数据库进行比对,注释得到每个OTU对应的物种分类信息。除未能归属到任何已知分类单元的OTU数目外,只能分类至门、纲、属、种的OTU分别为45、51、40和17个,分别占OTU总数的18.83%、21.34%、16.74%和7.11%。
|
|
表 1 不同样品的原始序列数、高质量序列数和OTUs数 Tab.1 The raw reads, high quality reads and OTUs in different samples |
通过对长牡蛎胃含物及所处养殖水体中真核生物的群落多样性进行分析,发现所处养殖水体具有较高的群落多样性指数(表 2),且所处养殖水体与长牡蛎胃含物中的Chao I指数、Observed species指数、Shannon指数和Simpson指数等差异显著(P < 0.05) (图 2),表明所处养殖水体中真核浮游生物的丰富度和多样性显著高于长牡蛎胃含物。2个样品的Good's覆盖率均高于99.9%,且随着序列数增加,稀释曲线趋于平稳(图 3),说明现有的测序深度足以反映样品中绝大多数微型浮游生物信息。
|
|
表 2 不同样品的真核生物群落多样性指数 Tab.2 The diversity index for eukaryote community in different samples |

|
图 2 Alpha多样性指数的分组箱线图 Fig.2 Box-plot of the Alpha diversity index S:长牡蛎所处养殖水体;W:长牡蛎胃含物。下同 S: Seawater of aquaculture area; W: Stomach contents of C. gigas. The same as below |
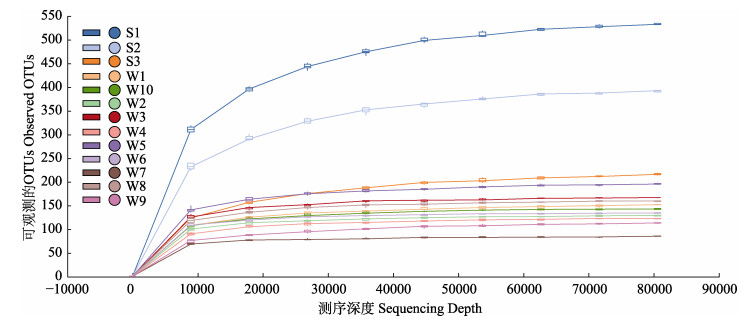
|
图 3 样品稀疏曲线 Fig.3 Sample rare faction curves |
从13个样品中共鉴定出45个门类,其中,长牡蛎胃含物中真核生物分属于34个门,所处养殖水体分属于37个门,每个样品中未鉴定出的真核生物比例为0.12%~1.46%。如图 4所示,绿藻门(Chlorophyta)是长牡蛎胃含物和所处养殖水体中整体丰度最高的门类,分别占总真核生物的(36.79±9.51)%和(55.38±0.76)%。甲藻门(Pyrrophyta)是长牡蛎胃含物中真核生物的第二大门类,占总真核生物的(31.04±12.48)%;而在所处养殖水体中,脊索动物门(Chordata)占总真核生物的(32.58±2.37)%,是真核生物的第二大门类。

|
图 4 所处养殖水体及长牡蛎胃含物中真核生物的物种组成 Fig.4 Eukaryote composition in aquaculture area and stomach of C. gigas |
在长牡蛎胃含物中,前10个丰度最高的门类占总体的87.27%~96.79%,平均占比94.07%。如图 5a所示,长牡蛎胃含物中真核生物在门水平上的丰度差异显著,其中,绿藻门、甲藻门、链型植物(Streptophytina)、硅藻门(Bacillariophyta)和原生动物(Protozoa)为主要类群,相对丰度分别为37.94%、36.11%、10.43%、8.48%和4.11%。绿藻门中的主要类群为共球藻纲(Trebouxiophyceae)、Picochlorum属的微微型单细胞藻类;甲藻门中的主要类群分属于横裂甲藻纲(Dinophyceae)、共生藻属(Symbiodinium);链型植物主要为菊亚目(Asterales)、菊科(Asteraceae)中的一些种类;硅藻门的主要类群为中心硅藻纲(Coscinodiscophyceae)、海链藻属(Thalassiosira)的浮游植物;原生动物主要为顶复门(Apicomplexa)、纤毛门(Ciliophora)和微型鞭毛虫类(Microstrichinella)等。在所处养殖水体中,前10个丰度最高的门类占总体的98.00%,如图 5b所示,绿藻门、脊索动物门、节肢动物门、甲藻门和硅藻门为其主要类群,相对丰度分别为56.49%、33.31%、5.19%、1.59%和1.41%,绿藻门和脊索动物门的占比非常大,占到了总真核生物的89.80%。绿藻门中的主要类群是Picochlorum属和Micromonas属的微微型单细胞藻类;脊索动物门中的主要类群为尾索动物亚门(Chordata)海鞘纲(Ascidiacea)中的浮游动物;节肢动物门中的类群主要分属于六肢幼虫纲(Hexanauplia)拟哲水蚤属(Paracalanus);甲藻门的主要类群为橫裂甲藻纲共生藻属中的浮游植物;硅藻门主要分属于中心硅藻纲海链藻属。
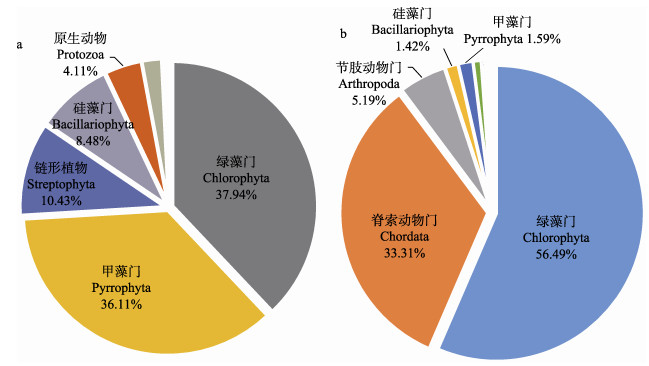
|
图 5 长牡蛎胃含物(a)及所处养殖水体中(b)10个丰度最高门类相对丰度 Fig.5 Relative abundance of 10 most abundant phyla in stomach content (a) of C. gigas and aquaculture area (b) |
为展示各样本的物种丰度分布趋势,使用平均丰度前50位的属水平丰度数据绘制热图,进行物种组成分析(图 6)。结果显示,样品分为2组,一组是包含长牡蛎胃含物中的10个样品,另一组包含所处养殖水体的3个样品。利用Primer进行相似性百分比分析发现,所处养殖水体组内相似性为73.68%,长牡蛎胃含物组内相似性为67.98%,二者差异性为59.09%。表明长牡蛎胃含物和所处养殖水体中的真核生物存在一定差异性,且长牡蛎个体间摄食具有差异性。为更直观地体现长牡蛎胃含物与所处养殖水体中群落组成的差异性和相似性,构建韦恩图(图 7)。长牡蛎胃含物和所处养殖水体中真核生物共有1345个OTUs,其中,胃含物中特有的OTUs有572个,约占总OTUs的42.53%;所处养殖水体中特有的OTUs为631个,占总OTUs的46.91%;2个样品共有OTUs 142个,约占总OTUs的10.56%。共有物种中,在门水平上相对丰度较高的物种主要分属于脊索动物门、绿藻门和甲藻门;在属水平上主要是微微型绿藻、共生藻属、海链藻属中的浮游植物。
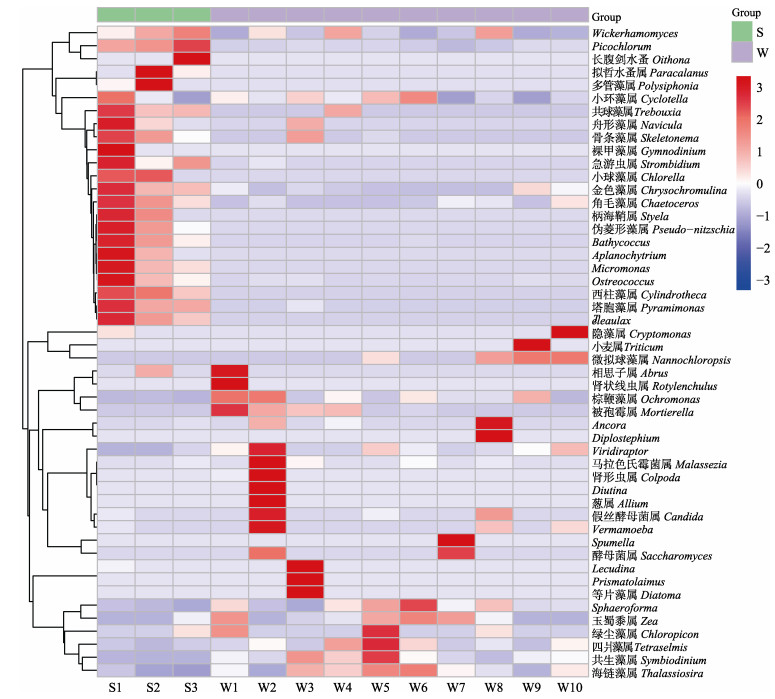
|
图 6 不同样品真核生物组成聚类热图 Fig.6 Hierarchically clustered heatmap of the eukaryotic composition of different samples |

|
图 7 不同样品中OTUs分布韦恩图 Fig.7 Venn diagram showing the unique and shared OTUs in samples |
本研究运用高通量测序技术对长牡蛎所处养殖水体中的真核生物进行了采集和优势种分析。经鉴定,所处养殖水体中共鉴定出真核生物37门155属165种,其中,绿藻门18属21种,主要以Picochlorum属的微微型单细胞藻为主,是第一大类群,占总浮游生物的56.49%,这与王晓敏(2017)对桑沟湾浮游植物研究有一定的差异,其调查结果为硅藻类浮游植物占优势。造成这种现象的原因推测一方面是鉴定方法的不同。王晓敏(2017)使用的是传统的显微镜技术,光学显微镜很难对粒径较小的浮游植物(如微微型浮游植物和部分微型浮游植物)分类鉴定(Mackey et al, 1996; 邓春梅等, 2010),此次调查的优势种——Picochlorum属微藻是一种粒径 < 2 μm的单细胞海洋绿藻(Hironaka, 2000),传统的显微镜技术可能会忽视它的存在。另一方面是采样位置的差异。王晓敏(2017)通过对全湾的调查得出硅藻为桑沟湾的优势物种,本实验仅研究了牡蛎养殖区的主要种类。有研究表明,由于滤食性贝类的摄食机制,长期高密度养殖滤食性贝类的海域容易出现浮游植物小型化的趋势,个体较大的优势种类逐渐被个体较小的种类代替(Nakamura et al, 2000; Frau et al, 2016),徐文琦等(2016)及李凤雪等(2020)对桑沟湾的研究也验证了这点。由此可见,滤食性贝类的养殖对浮游植物群落结构造成了一定影响。脊索动物门6属6种,主要以海鞘纲生物为主,为第二大类群。海鞘纲营固着生活(齐占会等, 2010),是有害的污损生物(韩帅帅等, 2018; 周斌等, 2016),桑沟湾大规模的贝藻养殖活动为海鞘提供大量的附着基,8月,玻璃海鞘(Ciona intestinalis)和柄海鞘(Styela clava)达到了最高峰(王光花等, 2005),严重影响了贝类养殖活动。海鞘虽是固着性生物,但其受精卵会首先变成可自由游泳的幼体,之后经过变态才进入固着生活的阶段(Dybern, 1965; Svane et al, 1993),适宜的温度和旺盛的繁殖力使其成为牡蛎养殖区第二大类真核浮游生物。玻璃海鞘身体呈透明态,传统的显微镜研究往往会忽视其存在,从而影响了对浮游生物优势种类的判断。高通量测序技术通过对物种DNA分子进行序列测定和比对,从而弥补了这一不足。
滤食性贝类能摄取环境中所有可以利用的有机质,并通过颗粒选择将可利用的食物颗粒分选出来以最大程度地获得能量(Ward et al, 2004)。近年来,很多学者运用不同方法研究了长牡蛎的食物来源。Riera等(1996)运用稳定碳同位素分析法研究了法国Marennes-Oléron海湾长牡蛎的食物来源,表明陆源有机质和底栖硅藻对长牡蛎有巨大贡献;Kasim等(2009)运用镜检法分析了日本Akkeshi-ko河口长牡蛎的胃含物,表明底栖硅藻是其主要的食物来源,其次是浮游硅藻和甲藻;许强(2007)运用脂肪酸标记法分析了潮间带长牡蛎的食物来源,表明长牡蛎的饵料主要来源于硅藻,其次是鞭毛藻和大型绿藻床;王先锋等(2017)利用18S rDNA方法分析了中国海州湾平岛太平湾长牡蛎的摄食情况,发现链型植物是长牡蛎胃含物中整体丰度最高的门类,其次是绿藻门和囊泡虫门(Alveolata)。本研究在长牡蛎胃含物中发现真核生物34门144属153种,绿藻门和甲藻门是其主要的摄食类群,分别占总食物的37.94%和36.11%。有研究表明,长牡蛎对2 μm颗粒物的保留效率仅为50% (Riisgard, 1988),但本研究却在长牡蛎胃含物中发现了大量 < 2 μm的共球藻纲Picochlorum属单细胞海洋绿藻。造成这种现象的原因推测有两点,一是王芳等(1998)通过扫描电镜观察了太平洋牡蛎的鳃结构,发现它的过滤器官孔隙直径 < 1 μm,可以有效地滤取大多数浮游植物细胞。作为长牡蛎适口饵料的硅藻类浮游植物在该区域含量较低,仅占总真核浮游生物的8.48%,在这种情况下,占比56.49%的绿藻门可通过长牡蛎的多次滤取成为了其补充碳源,有研究表明,贝类的饵料结构与海区浮游植物种类和数量明显相关(郭皓等, 1999)。二是绿藻分泌大量的胞外聚合物,使藻表面带有负电荷和具有粘附性(Gutnick et al, 2000; Pal et al, 2008),藻细胞通过静电及吸附作用形成藻-菌、藻-藻聚合体(许荔萍, 2017),增大了微藻粒径,从而增加了长牡蛎对其的保留效率。长牡蛎的胃中除含有大量海洋性天然饵料如绿藻、甲藻等外,还有相当数量的链型植物(主要为菊亚目菊科中的类群)。可能的解释是长牡蛎的采样区域位于荣成桑沟湾靠近楮岛村的牡蛎养殖区,楮岛村的马路两边及田地附近栽种了大量的剑叶金鸡菊(Coreopsis lanceolata),花期为5—9月,为入侵种类,繁殖力很强,通过“风传媒”方式传播(曾建军等, 2012)。一方面,剑叶金鸡菊的花粉随海风传播到海水中,经过海流的传播,这些颗粒可能被长牡蛎滤食;另一方面,其叶片等组织脱落腐烂后,随着雨水流入海水中被长牡蛎摄食。这在一定程度上也说明了长牡蛎的营养可塑性,表明长牡蛎的食物源不仅仅来源于海洋,而且还摄食链型植物。
在长牡蛎胃含物中,还发现了相当比例的原生动物(其主要为顶复门、纤毛门和微型鞭毛虫类等),占总真核生物的4.11%。原生动物消耗细菌和微微型浮游生物(Leakey, 1992),并且被众多中型浮游动物(尤其是桡足类)所摄食(Hartmann et al, 2011),它们被认为是微微型浮游生物与微型或者大型浮游生物之间的主要营养纽带,在海洋食物网中起着承上启下的作用(Stoecker et al, 1990)。在长牡蛎胃中发现了4.11%的原生动物,这也说明了原生动物在微微型浮游生物与滤食性贝类之间也充当着重要的桥梁作用,在浮游植物匮乏甚至充足时,原生动物可作为滤食性贝类的补充碳源。有研究表明,海洋中超过50%的初级生产力是由 < 3 μm的单细胞浮游植物所贡献(Benovic, 1986),在本研究区域更是占海水中总真核生物的56.49%。这部分浮游植物虽不能被滤食性贝类有效保留,但它们可以通过原生动物实现对其的间接利用,微微型浮游植物通过这种“打包”作用将能量传递到滤食性贝类中。以微微型浮游植物为起点的微型生物食物网在浅海贝类养殖生态系统中占有十分重要的地位。在所处养殖水体中,Picochlorum属、海鞘纲、哲水蚤属、共生藻属和海链藻属中的浮游生物为主要类群;在长牡蛎胃含物中,Picochlorum属、共生藻属、菊科和海链藻属中的生物为其主要类群。通过对长牡蛎胃含物内和所处养殖水体中物种组成和相对丰度百分比相似性分析可知,长牡蛎可滤食的物种平均占所在海域中物种的一半以上,但主要摄食的种类只有有限的几类,这与张雪(2013)对虾夷扇贝(Patinopecten yessoensis)的食性研究结果相一致。调查期间,长牡蛎倾向于摄食占调查区域真核浮游生物丰度比例较高的微微型绿藻、共生藻属和海链藻属中的浮游植物,说明长牡蛎对食物具有选择性且受海域可摄食浮游生物丰度的影响,可摄食浮游生物丰度的变化可引起长牡蛎的食物组成及相对丰度的变动。
本研究发现,长牡蛎胃含物中含有水体中不存在的OTUs 572个,造成这种现象的原因推测有以下几点:一方面长牡蛎胃含物特有OTUs比对归类后主要为顶复门艾美球虫科(Eimeridae)、壶菌门(Chytridiomycota)壶菌纲(Chytridiomycetes)和链型植物豆科中的生物,艾美球虫科和壶菌纲中的一些种类可以寄生在贝类的肠道中(周永灿, 2000; 陈皓文等, 2007),而水体中的含量较低,达不到检测限度;豆科植物(Leguminosae)来自于陆地,并不是海洋中的常驻物种,被长牡蛎摄食后在消化道中需一定时间才可以被完全消化吸收,推测在此期间,豆科植物的腐烂组织可能已随海流飘走或是沉入海底,采集的水样具有偶然性,没有采集到,后续会增加采样数量以消除偶然性误差。另一方面,长牡蛎胃含物中特有的物种在海水中的占比非常低,低于检测限度(0.5 ng/μL),无法对其DNA进行扩增,但通过生物富集作用这些较低的物种在长牡蛎的胃含物中进行积累,从而在胃含物中检测到。
研究表明,长牡蛎胃含物中真核生物成分复杂,食物来源丰富,不仅含有浮游植物等初级生产者,还包括原生动物等次级消费者。本研究采用高通量测序技术成功识别出夏季长牡蛎的食物组成,但海洋中的浮游生物随季节变化,这也在很大程度上左右着滤食性贝类的饵料结构组成,后续将尝试将时间步长加密到季节尺度,为系统掌握不同季节长牡蛎的食谱组成“快照”奠定基础。
BENOVIC A. Contribution of Synechococcus spp. to size- fractionated primary productivity in three water masses in the northwest Atlantic Ocean. Deep Sea Research Part B Oceanographic Literature Review, 1986, 33(12): 1023-1038 |
CHEN H W, CHEN Y. Diseases of cultured oysters caused by pathogenetic microbes. Fisheries Science, 2007, 26(9): 531-534 [陈皓文, 陈阳. 牡蛎的微生物疾病. 水产科学, 2007, 26(9): 531-534 DOI:10.3969/j.issn.1003-1111.2007.09.015] |
DENG C M, YAO P, LIU S X, et al. Supplier selection strategy based on cache state for proxy assistant P2P-VoD system. Periodical of Ocean University of China (Natural Science), 2010, 40(4): 91-98, 104 [邓春梅, 姚鹏, 刘淑霞, 等. 海洋浮游藻色素分析和化学分类研究进展. 中国海洋大学学报(自然科学版), 2010, 40(4): 91-98, 104] |
DONG B, XUE Q Z, LI J. Advances in studies on the feeding physiology of suspension-feeding of luscs. Marine Sciences, 2000, 24(7): 31-34 [董波, 薛钦昭, 李军. 滤食性贝类摄食生理的研究进展. 海洋科学, 2000, 24(7): 31-34 DOI:10.3969/j.issn.1000-3096.2000.07.012] |
DYBERN B I. The life cycle of Ciona intestinalis (L.) f. typica in relation to the environmental temperature. Oikos, 1965, 16(1/2): 109-131 |
EGGE E, BITTNER L, ANDERSEN T, et al. 454 pyrosequencing to describe microbial eukaryotic community composition, diversity and relative abundance: A test for marine haptophytes. PLoS One, 2013, 8(9): e74371 DOI:10.1371/journal.pone.0074371 |
FRAU D, MOLINA F R, MAYORA G. Feeding selectivity of the invasive mussel Limnoperna fortunei (Dunker, 1857) on a natural phytoplankton assemblage: What really matters?. Limnology, 2016, 17(1): 47-57 DOI:10.1007/s10201-015-0459-2 |
FU M Z, PU X M, WANG Z L, et al. Integrated assessment of mariculture ecosystem health in Sanggou Bay. Acta Ecologica Sinica, 2013, 33(1): 238-248 [傅明珠, 蒲新明, 王宗灵, 等. 桑沟湾养殖生态系统健康综合评价. 生态学报, 2013, 33(1): 238-248] |
FUKUMORI K, OI M, DOI H, et al. Food sources of the pearl oyster in coastal ecosystems of Japan: evidence from diet and stable isotope analysis. Estuarine Coastal and Shelf Science, 2008, 76(3): 704-709 DOI:10.1016/j.ecss.2007.07.046 |
GUO H, CAO L. The relationship between food structure of raft for cultivated shellfish and phytoplankton in Changjiang'ao, Dalian. Journal of Shanghai Fisheries University, 1999, 8(2): 112-118 [郭皓, 曹丽. 大连常江澳浮筏养殖贝类的饵料结构与浮游植物的关系. 上海海洋大学学报, 1999, 8(2): 112-118] |
GUTNICK D L, BACH H. Engineering bacterial biopolymers for the biosorption of heavy metals; new products and novel formulations. Applied Microbiology and Biotechnology, 2000, 54(4): 451-460 DOI:10.1007/s002530000438 |
HAN S S, CAO W H, CHEN D, et al. Review of fouling ascidians in the coastal waters of China. Ecological Science, 2018, 37(1): 186-191 [韩帅帅, 曹文浩, 陈迪, 等. 中国沿海污损性海鞘生态特点及研究展望. 生态科学, 2018, 37(1): 186-191] |
HARTMANN H J, TALEB H, ALEYA L, et al. Predation on ciliates by the suspension-feeding calanoid copepod Acanthodiaptomus denticornis. Canadian Journal of Fisheries and Aquatic Sciences, 2011, 50(7): 1382-1393 |
HIRONAKA J L. Characterization of a unicellular coccoid green alga collected from the salt plains national wildlife refuge, Oklahoma. Masterxs Thesis of Oklahoma State University, 2000
|
JONES C G. Organisms as ecosystem engineers. New York: Springer Press, USA, 1996: 130-147
|
KASIM M, MUKAI H. Food sources of the oyster (Crassostrea gigas) and the clam (Ruditapes philippinarum) in the Akkeshi-ko estuary. Plankton and Benthos Research, 2009, 4(3): 104-114 DOI:10.3800/pbr.4.104 |
LEAKEY R J G. Planktonic ciliates in southampton water: Abundance, biomass, production, and role in pelagic carbon flow. Marine Biology, 1992, 114(1): 67-83 |
LI F X, JIANG Z J, GAO Y P, et al. Distribution of size-fractionated phytoplankton and its relationship with environmental variables in Sanggou Bay. Progress in Fishery Sciences, 2020, 41(1): 31-40 [李凤雪, 蒋增杰, 高亚平, 等. 桑沟湾浮游植物粒径结构及其与环境因子的关系. 渔业科学进展, 2020, 41(1): 31-40] |
LIU G, NING Y, XIA X F, et al. The application of high-throughput sequencing technologies to wildlife diet analysis. Acta Ecologica Sinica, 2018, 38(9): 359-368 [刘刚, 宁宇, 夏晓飞, 等. 高通量测序技术在野生动物食性分析中的应用. 生态学报, 2018, 38(9): 359-368] |
MACKEY M D, MACKEY D J, HIGGINS H W, et al. CHEMTAX - A program for estimating class abundances from chemical markers: application to HPLC measurements of phytoplankton. Marine Ecology Progress, 1996, 144(1): 265-283 |
METFIES K, NICOLAUS A, VON HARBOU L, et al. Molecular analyses of gut contents: Elucidating the feeding of co-occurring salps in the Lazarev Sea from a different perspective. Antarctic Science, 2014, 26(5): 545-553 DOI:10.1017/S0954102014000157 |
NAKAMURA Y, KERCIKU F. Effects of filter-feeding bivalves on the distribution of water quality and nutrient cycling in a eutrophic coastal lagoon. Journal of Marine Systems, 2000, 26(2): 209-221 DOI:10.1016/S0924-7963(00)00055-5 |
O'RORKE R, LAVERY S, CHOW S, et al. Determining the diet of larvae of western rock lobster (Panulirus cygnus) using high-throughput DNA sequencing techniques. PLoS One, 2012, 7(8): e42757 DOI:10.1371/journal.pone.0042757 |
PAL A, PAUL A K. Microbial extracellular polymeric substances: Central elements in heavy metal bioremediation. Indian Journal of Microbiology, 2008, 48(1): 49-64 DOI:10.1007/s12088-008-0006-5 |
QIAO R. Microscope and phytoplankton pigments analysis of the shellfish feeding habits. Master's Thesis of Shanghai Ocean University, 2015 [乔芮. 基于镜检和浮游植物色素分析的贝类食性研究. 上海海洋大学硕士研究生学位论文, 2015]
|
QI Z H, FANG J G, ZHANG J H, et al. Seasonal succession of fouling communities in the poly-culture area of scallop Chlamys farreri and kelp Laminaria japonica in Sanggou Bay. Progress in Fishery Sciences, 2010, 31(4): 72-77 [齐占会, 方建光, 张继红, 等. 桑沟湾贝藻养殖区附着生物群落季节演替研究. 渔业科学进展, 2010, 31(4): 72-77 DOI:10.3969/j.issn.1000-7075.2010.04.010] |
RIERA P, RICHARD P. Isotopic determination of food sources of Crassostrea gigas along a trophic gradient in the estuarine bay of Marennes-Oléron. Estuarine Coastal and Shelf Science, 1996, 42(3): 347-360 DOI:10.1006/ecss.1996.0023 |
RⅡSGARD H U. Efficiency of particle retention and filtration rate in 6 species of Northeast American bivalves. Marine Ecology Progress Series, 1988, 45(3): 217-223 |
SHI Q, SUN Y. Introduction to marine genomics. Guangzhou: Sun Yat-sen University Press, 2015 [石琼, 孙颖. 海洋生物基因组学概. 广州: 中山大学出版社, 2015]
|
SONG Y L, CUI Y, SUN Y, et al. Study on nutrient state and influencing factors in Sanggou Bay. Marine Fisheries Research, 1996, 17(2): 41-51 [宋云利, 崔毅. 桑沟湾养殖海域营养状况及其影响因素分析. 海洋水产研究, 1996, 17(2): 41-51]
|
STOECKER D K, MCDOWELL C J. Predation on protozoa: its importance to zooplankton. Journal of Plankton Research, 1990, 12(5): 891-908 DOI:10.1093/plankt/12.5.891 |
STOECK T, BASS D, NEBEL M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water. Molecular Ecology, 2010, 19(S1): 21-31 |
SVANE I, HAVENHAND JN. Spawning and dispersal in Ciona intestinalis (L.). Marine Ecology, 2008, 14(1): 53-66 |
WANG F, DONG S L, FAN R Q, et al. The observation of the gills of four species filtering-feeding bivalve using scanning electron microscope. Periodical of Ocean University of China (Natural Science), 1998, 28(2): 240-244 [王芳, 董双林, 范瑞青, 等. 四种滤食性贝类滤食器官鳃的扫描电镜观察. 中国海洋大学学报(自然科学版), 1998, 28(2): 240-244] |
WANG G H, YUE B. Fouling development and its effect on the growth and livability of Chlamys ferreri. Shandong Fisheries, 2005, 22(7): 5-7 [王光花, 岳斌. 附着生物对栉孔扇贝生长及存活的影响. 齐鲁渔业, 2005, 22(7): 5-7] |
WANG X F, LIN C G, XU Q, et al. Impact of enteromorpha prolifera green tide on oyster feeding using 18S rDNA molecular method. Oceanologia et Limnologia Sinica, 2017, 48(6): 1362-1370 [王先锋, 林承刚, 许强, 等. 利用18S rDNA分子方法分析浒苔绿潮过境期间长牡蛎摄食情况的变化. 海洋与湖沼, 2017, 48(6): 1362-1370] |
WANG X M. Studies on phytoplankton community under integrated multi-trophic aquaculture modes in Sanggou Bay. Master's Thesis of Jinan University, 2017 [王晓敏. 桑沟湾多营养级综合养殖模式下浮游植物群落结构研究. 暨南大学硕士研究生学位论文, 2017]
|
WARD J E, SHUMWAY S E. Separating the grain from the chaff: Particle selection in suspension- and deposit-feeding bivalves. Journal of Experimental Marine Biology and Ecology, 2004, 300(1/2): 83-130 |
XU L P. The effects of extracellular polymeric substances on algal interface and toxicity of arsenic to algae. Master's Thesis of Zhejiang University, 2017 [许荔萍. 胞外聚合物界面作用及其对绿藻砷毒的影响. 浙江大学硕士研究生学位论文, 2017]
|
XU Q. Evaluation of food sources of bivalve in seaweed and filter-feeding bivalve polyculture ecosystem. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2007 [许强. 贝藻混养系统中贝类食物来源的定量分析. 中国科学院海洋研究所博士研究生学位论文, 2007]
|
XU W Q, LIU S M. Phytoplankton responses to phosphorus in Sanggou Bay in spring and autumn. Advances in Marine Sciences, 2016(2): 26-37 [徐文琦, 刘素美. 春季和秋季桑沟湾浮游植物对磷的响应. 海洋科学前沿, 2016(2): 26-37] |
ZENG J J, XIAO Y A, ZHOU X J. Clonal reproductive property and community characteristic of invasive species Coreopsis lanceolata. Bulletin of Botanical Research, 2012, 32(2): 147-150 [曾建军, 肖宜安, 周小军. 入侵植物剑叶金鸡菊克隆繁殖特性及群落特征. 植物研究, 2012, 32(2): 147-150] |
ZHANG H, XU Q, ZHAO Y, et al. Sea cucumber (Apostichopus japonicus) eukaryotic food source composition determined by 18s rDNA barcoding. Marine Biology, 2016, 163(7): 153 DOI:10.1007/s00227-016-2931-x |
ZHANG X. Study on phytoplankton community structure and the relationship with feeding habits of Patinopecten yessoensis in the waters adjacent to Zhangzidao Island. Master's Thesis of Shanghai Ocean University, 2013 [张雪. 獐子岛邻近水域浮游植物群落结构及其与虾夷扇贝(Patinopecten yessoensis)食性关系的研究. 上海海洋大学硕士研究生学位论文, 2013]
|
ZHOU B, FENG C H, LIU W, et al. Ecological research of fouling organisms on the offshore platform in Bohai Sea. Progress in Fishery Sciences, 2016, 37(3): 9-13 [周斌, 冯春辉, 刘伟, 等. 渤海石油平台污损生物生态研究. 渔业科学进展, 2016, 37(3): 9-13] |
ZHOU Y C. Research progress on diseases of marine shellfish. Natural Science Journal of Hainan University (Natural Science), 2000, 18(2): 207-212 [周永灿. 海洋贝类病害及其研究进展. 海南大学学报(自然科学版), 2000, 18(2): 207-212 DOI:10.3969/j.issn.1004-1729.2000.02.024] |



