2. 中国水产科学研究院黄海水产研究所 农业农村部海水养殖病害防治重点实验室 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071;
3. 青岛市精神卫生中心 山东 青岛 266034;
4. 中国海洋大学海洋生命学院 山东 青岛 266003
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Key Laboratory of Maricultural Organism Disease Control, Ministry of Agriculture and Rural Affairs, Qingdao, Shandong 266071, China;
3. Qingdao Mental Health Center, Qingdao, Shandong 266034, China;
4. College of Marine Life Sciences, Ocean University of China, Qingdao, Shandong 266003, China
褐藻生物量巨大,生长速度较快,被视为最有前景的生物资源之一(Flórez-Fernández et al, 2019)。褐藻含有多种生物活性物质,如褐藻胶、岩藻聚糖、岩藻黄质、甘露醇、多酚和植物激素等(Li et al, 2019a; Sharma et al, 2014、2016)。其中,褐藻胶约占褐藻干物质的22%~44% (w/w),是褐藻细胞壁的主要成分(Wargacki et al, 2012)。
褐藻胶是一种线性聚合物,由β-d-甘露糖醛酸(M)和α-1-古罗糖醛酸(G)作为单体单元组成。单体通过1, 4-糖苷键以不同顺序结合,构成聚古罗糖醛酸片段(PolyG)、聚甘露糖醛酸酯片段(PolyM)和杂聚物片段(PolyMG)(Wong et al, 2000)。褐藻胶裂解酶能切断褐藻胶的1, 4-糖苷键,在相对温和可控的条件下,将褐藻胶水解成褐藻寡糖(alginate oligosaccharides, AOS) (Li et al, 2019b)。AOS具有抗氧化、抗肿瘤和抗凝血等生理活力,在医疗健康相关领域具有潜在的应用(孙丽萍等, 2005; Iwamoto et al, 2003; 辛现良等, 2001; Lin et al, 2007)。此外,AOS对植物根系发育具有显著的促进作用(Xu et al, 2003; Zhang et al, 2014)。
多种海洋细菌可以产生褐藻胶裂解酶,包括链霉菌(Streptomycete sp.)、假单胞菌(Pseudomonas sp.)、沙门氏菌(Salmonella sp.)、黄杆菌(Flavobacterium sp.)、芽孢杆菌(Bacillus sp.)和弧菌(Vibrio sp.)等(Chen et al, 2016; Inoue et al, 2016; Jagtap et al, 2014; Li et al, 2015; Schiller et al, 1993; Thomas et al, 2013)。为了提高褐藻胶裂解酶的活力,研究者克隆了多种褐藻胶裂解酶的编码基因并进行重组表达(He et al, 2018; Zhang et al, 2020)。其中,低温褐藻胶裂解酶受到广泛关注。在低温条件下,植物激素等活性物质的不稳定性能够得到控制,并且低温催化具有低污染风险和低能耗的优势(Gao et al, 2018)。但由于低温酶通常稳定性差且活力低,已报道的褐藻胶裂解酶并不能满足工业要求。因此,需要筛选出符合工业应用需求的、具有鲁棒性的低温褐藻胶裂解酶。
本研究筛选到的海洋细菌Yangia sp. SJ-H-12能在低温条件下分泌褐藻胶裂解酶,克隆褐藻胶裂解酶编码基因Alyya并在食品级真核宿主解脂耶氏酵母(Yarrowia lipolytica)中表达,分泌的褐藻胶裂解酶ALYYA具有较好的温度稳定性和pH稳定性,可以适应不同盐度,并可制备较低分子量的寡糖,说明ALYYA可以成为褐藻胶降解的有效工具。
1 材料与方法 1.1 菌株和培养基褐藻胶购自明月海藻集团(青岛)。PolyM和PolyG购自青岛博智汇力生物技术有限公司。分泌表达采用解脂耶氏酵母系统,URA–菌株和表达载体pINA1312由中国海洋大学池振明教授提供。在以褐藻胶为单一碳源的ASC(褐藻酸唯一碳源培养基,alginate as sole carbon source)固体培养基中筛选和培养产褐藻胶裂解酶的菌株。ASC培养基包含10 g/L褐藻胶钠、5.0 g/L (NH4)2SO4、1.0 g/L MgSO4·7H2O和0.1 g/L FeSO4,海水配制。在含有1.7 g/L酵母基础氮源、10.0 g/L葡萄糖、5.0 g/L (NH4)2SO4和25.0 g/L琼脂的YNB板上筛选解脂耶氏酵母转化子(Madzak et al, 2015)。GPPB培养基用于重组酶生产,包含30.0 g/L葡萄糖、1.0 g/L (NH4)2SO4、2.0 g/L酵母提取物、2.0 g/L KH2PO4、3.0 g/L K2HPO4和0.1 g/L MgSO4·7H2O,pH为6.8。2216E培养基包含1.0 g/L酵母提取物、5.0 g/L蛋白胨、0.01 g/L磷酸铁和20.0 g/L琼脂,海水配制。
1.2 产褐藻胶裂解酶菌株的筛选对采集的绿烂海带(Saccharina japonica)样品使用无菌海水清洗3次,加0.5 mL无菌海水研磨成匀浆液,稀释涂布在ASC平板,放置于20℃培养7 d。平板上分离得到的菌株转移至ASC液体培养基中,并在20℃下培养48 h,发酵液以5000×g离心。以0.5%(w/v)褐藻胶溶液作为底物,采用DNS(3, 5-二硝基水杨酸)法检测褐藻胶裂解酶活力,1 U酶活力定义为1 min产生1 μmol还原糖所需酶量(褚洪蕊等, 2008)。分离得到的菌株在含30%甘油的2216E保种液中–80℃保藏。
1.3 菌种鉴定产褐藻胶裂解酶菌株于2216E液体培养基中培养2~3 d。从每个培养基取1 mL菌液离心,取菌体,95℃保温5 min,破碎细胞释放细菌DNA,作为PCR模板DNA,用通用引物27F (5′-AGAGTTTGATCCTGG CTCAG-3′)和1492R (5′-TACGGCTACCTTGTTACGA CTT-3′)扩增细菌的16S rDNA。对菌株的16S rDNA进行测序。通过BLAST,与其他16S rDNA序列进行比较。使用MEGA 7.0软件根据Neighbor-Joining的方法构建系统发育树。
1.4 褐藻胶裂解酶ALYYA的生物信息学分析SJ-H-12菌株基因组DNA已完成测序和注释(诺禾致源)。序列分析显示,基因组中存在1个潜在的褐藻胶裂解酶基因,大小为1113 bp。使用SignalIP 4.1服务器(http://www.cbs.dtu.dk/services/SignalP-4.1/)分析信号肽;在保守域数据库(https://www.ncbi.nlm.nih.gov/cdd)中进行结构域分析;在线预测理论pI和MW(http://web.expasy.org/compute_pi/)。基于报道的褐藻胶裂解酶,使用MEGA 7.0软件根据Neighbor- Joining的方法构建系统发育树。
1.5 ALYYA的分泌表达与纯化对Alyya基因进行密码子优化,连接XPR2信号肽基因(Synbio Technologies, 中国)。合成的DNA片段连接pINA1312质粒,用于转化URA–菌株。在GPPB (glucose in phosphate buffer)液体培养基中30℃培养84 h,检测到阳性转化子的褐藻胶裂解酶活力。将菌株M34上清液调节pH为7.5,然后加至Ni-NTA琼脂糖凝胶柱(GE Healthcare, 美国)。将ALYYA酶连接至凝胶,用咪唑溶液洗涤。用12%(w/v)凝胶SDS- PAGE验证ALYYA的纯度和分子量。以polyM、polyG和褐藻胶为底物,研究ALYYA的底物偏好性。
1.6 温度和pH对ALYYA活力和稳定性的影响在温度为10℃~60℃、pH=8.0的10 mmol/L甘氨酸–NaOH缓冲液中进行ALYYA催化水解反应。检测活力以确定最佳反应温度。为评估ALYYA的热稳定性,将纯化的ALYYA在10℃~60℃下孵育12 h,然后在35℃下测量剩余活力。在不同pH的10 mmol/L缓冲液[Na2HPO4–柠檬酸(pH 3.0~8.0),甘氨酸–NaOH (pH 8.5~11.0)]中,制备褐藻胶溶液作为底物。使用这些褐藻胶溶液测定酶活力以确定最佳反应pH。通过估计在不同pH的缓冲液中于4℃孵育12 h后剩余的酶活力来检测pH的稳定性。所有反应均重复3次。
1.7 NaCl对ALYYA活力的影响配制0.5%(w/v)的褐藻胶溶液(pH 8.0),分别加入不同量的NaCl。以不同盐度的褐藻胶溶液为底物,加入等量的ALYYA,在35℃下测定酶活力,研究NaCl对ALYYA活力的影响。
1.8 酶解产物的分析将纯化的ALYYA (40 U)加入10 mL、pH 8.0的0.5%(w/v)褐藻胶溶液中,35℃下反应40 min。每5 min检测黏度和235 nm的吸光度。当黏度和吸光度均稳定时,将降解产物脱盐并通过ESI-MS确定聚合度(DP)。
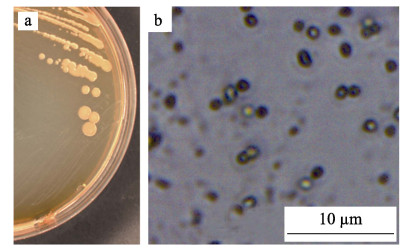
2 结果与讨论 2.1 产低温褐藻胶裂解酶菌株的筛选从绿烂海带中共分离得到46株海洋细菌。其中,7个菌株20℃下能在ASC平板上生长,说明这些菌株能够产生低温褐藻胶裂解酶。SJ-H-12菌株在液体ASC培养基中能分泌最高活力的褐藻胶裂解酶,达到2.44 U/mL。在2216E平板上,菌株SJ-H-12的菌落光滑且不透明,呈乳黄色,圆形菌落边缘整齐(图 1a);显微镜下,该菌株呈不规则球形(图 1b)。Blast比对分析发现,其16S rDNA序列与海洋细菌Yangia属相似度最大。聚类分析表明,菌株SJ-H-12与Yangia sp. CDJ15B-A02在同一分支上(图 2)。综合Blast和系统发育树的结果,认为SJ-H-12属于Yangia属,命名为Yangia sp. SJ-H-12。
|
图 1 SJ-H-12菌株的平板菌落形态(a)和细胞形态(b) Fig.1 Plate colony (a) and cell (b) morphology of SJ-H-12 strain |

|
图 2 基于16S rDNA序列以Neighbor-Joining的方法构建系统发育树(比例尺表示0.020的遗传距离) Fig.2 Neighbor-Joining phylogenetic tree based on 16S rDNA sequence (The scale bar indicated a genetic distance of 0.020) |
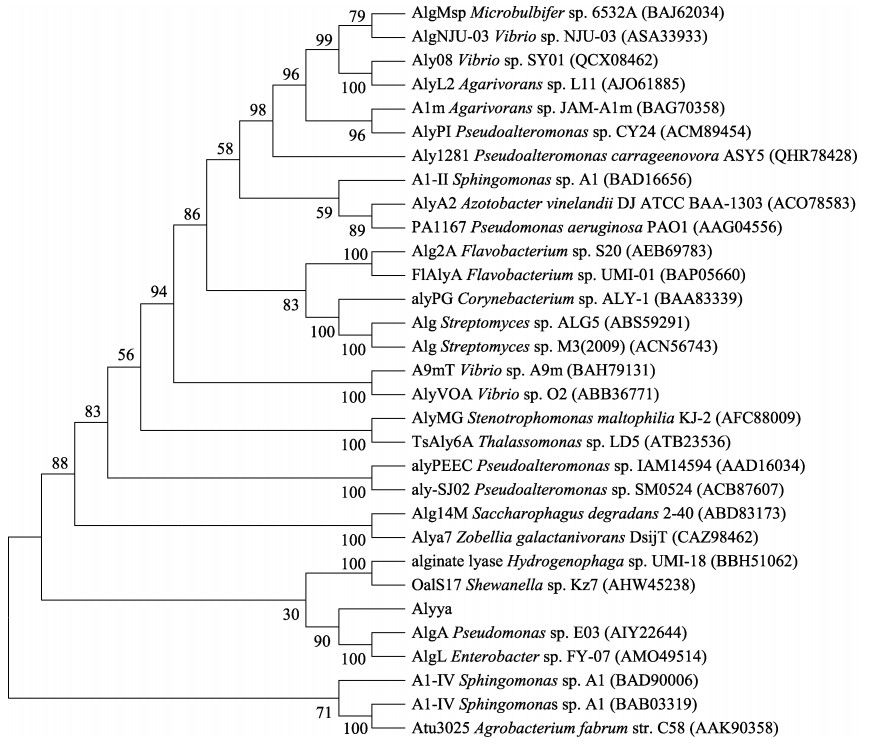
菌株SJ-H-12的基因组中存在1个潜在的褐藻胶裂解酶编码基因。开放阅读框由1113 bp组成,编码370个氨基酸的蛋白质(MT533612)。该酶被命名为ALYYA,并进行进一步的生物信息学分析。ALYYA的前23个氨基酸是信号肽,与分泌特征一致。去掉信号肽后,成熟酶的理论等电点(pI)为4.00,分子量(MW)为38.1 kDa。在NCBI上分析发现,ALYYA具有单个保守结构域。为确定ALYYA的进化位置,根据氨基酸序列和其他已报道的褐藻胶裂解酶构建了系统进化树。如图 3所示,ALYYA与Pseudomonas sp. E03的AlgA和Enterobacter sp. FY-07的AlgL位于同一分支。已报道的多数褐藻胶裂解酶属于多糖水解酶7家族(PL7)和17家族(PL17),而AlgA和AlgL均属于5家族(PL5),具有polyM片段偏好性(Inoue et al, 2016; Qin et al, 2018; Uchimura et al, 2010)。ALYYA的发现丰富了PL5家族褐藻胶裂解酶的研究,推测具有较强的polyM片段偏好性。
|
图 3 基于褐藻胶裂解酶氨基酸序列构建的Bootstrap consensus系统发育树 Fig.3 Bootstrap consensus phylogenetic tree based on alginate lyase amino acids sequence |
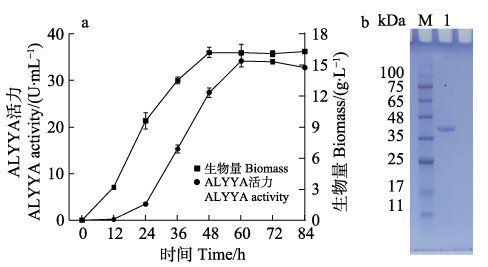
在以往研究中,大多数褐藻胶裂解酶在大肠杆菌(Escherichia coli)中表达。然而,由于大肠杆菌分泌能力弱、合成内毒素且具有细胞壁抗原物质,大肠杆菌表达褐藻胶裂解酶在工业生产中受到了限制(Miyamoto et al, 2009)。本研究在食品级宿主解脂耶氏酵母中表达了ALYYA,这是一种广泛使用的异源宿主,具有出色的分泌能力(Madzak, 2015)。重组菌株M34显示出最高的细胞外活力。如图 4a所示,在GPPB培养基中培养60 h后,重组菌株M34中的ALYYA活力达到34.2 U/mL,生物量为16.2 g/L。胞外酶的活力是野生菌株(2.44 U/mL)的14倍。从上清液中纯化ALYYA蛋白,并通过SDS-PAGE分析,如图 4b所示,在泳道上具有单一条带,分子量约为39.0 kDa,与去掉信号肽的氨基酸序列加上有His标签得到的理论分子量接近。纯化的ALYYA对褐藻胶的比活力为676.4 U/mg,而对polyG片段和polyM片段的比活力分别为213.2 U/mg和913.6 U/mg。此结果验证了生物信息学分析ALYYA对polyM偏好的预测。
|
图 4 分泌到培养基中的ALYYA活力的时间曲线(a)和纯化ALYYA的SDS-PAGE分析(b) Fig.4 Time curve of ALYYA activity secreted into the culture medium (a) and SDS-PAGE analysis of purified ALYYA (b) 数据均基于3个平行;M泳道表示预染色蛋白梯;泳道1表示纯化的ALYYA Data were based on 3 parallels; Lane M represents pre-stained protein ladder; Lane 1 represents purified ALYYA |
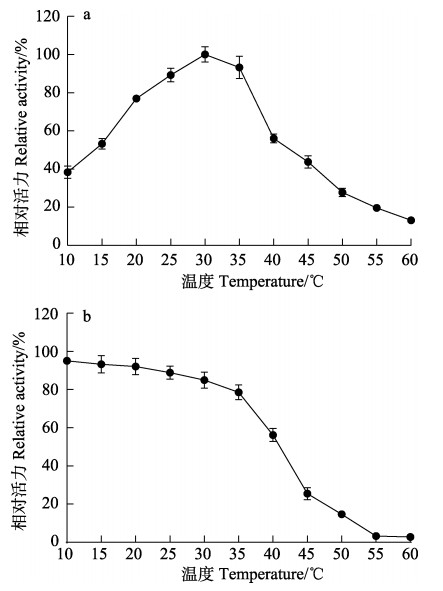
在酶纯化的基础上,研究了ALYYA的酶学性质。如图 5a所示,ALYYA的催化活力在30℃时最高;在25℃~35℃之间检测到80%以上的最高活力。当温度升至40℃以上时,活力急剧下降。在较低温度10℃和20℃下的催化活力分别为最高值的53.2%和38.4%。低于35℃时,ALYYA非常稳定;在40℃下孵育2 h后,剩余约60%的活力。高于45℃时,ALYYA丧失大部分活力。多数褐藻胶裂解酶在约40℃时具有最高活力,而低温褐藻胶裂解酶在低于35℃时具有最高的催化活力,在20℃时具有最高活力的50%以上。当前,尚无能应用于工业生产的商品化褐藻胶裂解酶,且报道的褐藻胶裂解酶多数为中温酶。与已经报道的少数低温褐藻胶裂解酶相比,ALYYA在20℃时的活力更高,并具有更好的热稳定性(图 5b)。ALYYA可作为降解褐藻胶的新型工具酶。
|
图 5 不同温度对ALYYA活力(a)和稳定性(b)的影响 Fig.5 Effects of different temperature on the activity (a) and stability (b) of ALYYA |
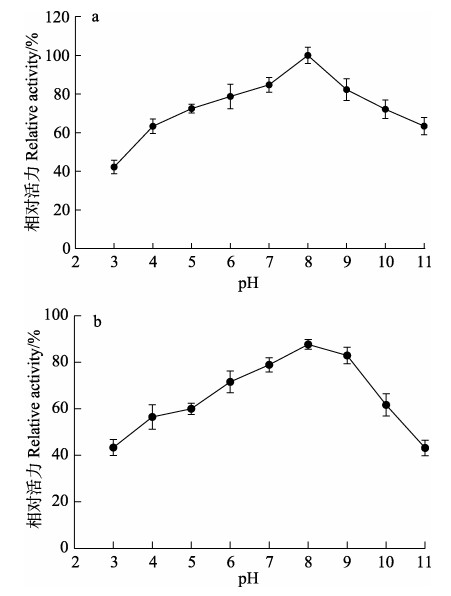
如图 6a所示,ALYYA活力在pH 8.0时最高,且在pH 6.0~9.0时超过80%。此外,在孵育12 h后,ALYYA在pH 5.0~10.0的范围内保留了超过60%的活力。在本研究的整个pH范围(3.0~11.0),孵育12 h后均保留了40%以上的活力(图 6b)。而细菌来源的褐藻胶裂解酶多数倾向于在中性附近催化水解反应(Mochizuki et al, 2015)。与其他低温褐藻胶裂解酶相比,ALYYA在更大的pH范围内表现出催化活力。例如,TsAly6A和AlgNJU-03的pH稳定范围分别仅为6.60~8.95和6.0~9.0 (Zhuang et al, 2018; Zhu et al, 2017)。实际上,ALYYA的pH稳定范围甚至比中温褐藻胶裂解酶AlgNJ04的还要大(Zhu et al, 2018)。这种独特的pH稳定性使ALYYA可以在不同催化环境下完成褐藻中活性物质的提取和转化。另外,Pseudomonas sp. E03的AlgA同样属于PL5家族,最适反应温度为30℃,最适反应pH为8,与本研究中ALYYA的pH和温度特性类似(Zhu et al, 2015)。
|
图 6 不同pH对ALYYA活力(a)和稳定性(b)的影响 Fig.6 Effects of different pH on the activity (a) and stability (b) of ALYYA |
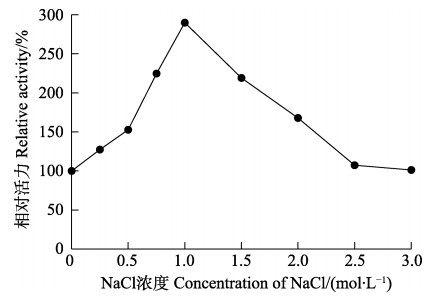
如图 7所示,NaCl在0~2.0 mol/L的浓度下能够明显激活ALYYA的活力。在0.75 mol/L NaCl浓度下,ALYYA的活性达到最高(290.0%)。NaCl对大多数褐藻胶裂解酶的活化是必不可少的。在1 mol/L NaCl中,AlgM4活力比无NaCl时增加了7倍左右(Huang et al, 2015)。在0.3 mol/L NaCl中的Aly08活力是无NaCl时的8倍左右(Wang et al, 2019)。相比之下,ALYYA的活力较为稳定,对NaCl的依赖较弱,并具有耐盐性。不同来源、品种和加工方法得到的褐藻的成分和盐度均有所区别。因此,ALYYA是一种可以适应不同盐度的酶,能满足各种加工需求。
|
图 7 不同浓度NaCl对ALYYA活力的影响 Fig.7 The effect of different concentration of NaCl on activity of ALYYA |
|
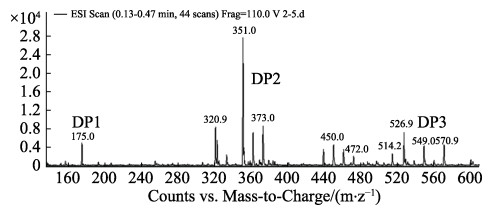
图 8 通过ESI-MS分析ALYYA的降解产物 Fig.8 Analysis of degradation products of ALYYA by ESI-MS |
使用ALYYA水解褐藻胶溶液40 min,当黏度不再下降时,通过负离子ESI-MS检测终产物。如图 7所示,产物主要为二糖(351.05 m/z),另有少部分单糖(175.00 m/z)和三糖(526.90 m/z)。因此,ALYYA是一种内切褐藻胶裂解酶。而低温褐藻胶裂解酶Algb和AlgNJU-03产生的寡糖主要是DP2~DP5 (Zhu et al, 2017)。ALYYA可制备较低分子量的寡糖,具有潜在的药物应用前景。
3 结论综上所述,本研究克隆了一种新型的低温褐藻胶裂解酶的编码基因,并对其进行了胞外分泌表达和性质研究。ALYYA在温度为25℃~35℃时表现出80%以上的活力,且在30℃时表现出最高活力,是典型的低温褐藻胶裂解酶;此外,ALYYA在pH为4.0~10.0的范围内保持了70%以上的活力,在pH为3.0~11.0的范围内保持了40%以上的活力,它的pH稳定范围比中温褐藻胶裂解酶大,表现出优异的pH稳定性;研究发现,ALYYA是一种可以适应不同盐度的酶,并可制备较低分子量的寡糖。因此,ALYYA可以成为工业应用的有效工具。
CHEN X, DONG S, XU F, et al. Characterization of a new cold- adapted and salt-activated polysaccharide lyase family 7 alginate lyase from Pseudoalteromonas sp.SM0524. Frontiers in Microbiology, 2016, 7: 1120-1129 |
CHU H R, TANG J C. Study on fermentation and enzyme production conditions of alginic acid degrading bacteria A7. Marine Science, 2008, 32(11): 93-96 [褚洪蕊, 唐景春. 褐藻酸降解菌A7的发酵及产酶条件研究. 海洋科学, 2008, 32(11): 93-96] |
FLÓREZ-FERNÁNDEZ N, TORRES M D, GONZÁLEZ- MUÑOZ M J, et al. Recovery of bioactive and gelling extracts from edible brown seaweed Laminaria ochroleuca by non-isothermal autohydrolysis. Food Chemistry, 2019, 277: 353-361 DOI:10.1016/j.foodchem.2018.10.096 |
GAO S, ZHANG Z, LI S. Characterization of a new endo-type polysaccharide lyase (PL) family 6 alginate lyase with cold-adapted and metal ions-resisted property. International Journal of Biological Macromolecules, 2018, 120: 729-735 DOI:10.1016/j.ijbiomac.2018.08.164 |
HE M, GUO M, ZHANG X, et al. Purification and characterization of alginate lyase from Sphingomonas sp.ZH0. Journal of Bioscience and Bioengineering, 2018, 126(3): 310-316 DOI:10.1016/j.jbiosc.2018.01.017 |
HUANG G, WANG Q, LU M, et al. AlgM4: A new salt-activated alginate lyase of the PL7 family with endolytic activity. Marine Drugs, 2018, 16(4): 120-133 DOI:10.3390/md16040120 |
INOUE A, ANRAKU M, NAKAGAWA S, et al. Discovery of a novel alginate lyase from Nitratiruptor sp.SB155-2 thriving at deep-sea hydrothermal vents and identification of the residues responsible for its heat stability. Journal of Biological Chemistry, 2016, 291(30): 15551-15563 DOI:10.1074/jbc.M115.713230 |
JAGTAP S, HEHEMANN J, POLZ M, et al. Comparative biochemical characterization of three exolytic oligoalginate lyases from Vibrio splendidus reveals complementary substrate scope, temperature, and pH adaptations. Applied and Environmental Microbiology, 2014, 80(14): 4207-4214 DOI:10.1128/AEM.01285-14 |
LI S Y, HE N N, WANG L N. Efficiently anti-obesity effects of unsaturated alginate oligosaccharides (UAOS) in high-fat diet (HFD)-fed mice. Marine Drugs, 2019a, 17(9): 540 DOI:10.3390/md17090540 |
LI S Y, WANG Z P, WANG L N, et al. Combined enzymatic hydrolysis and selective fermentation for green production of alginate oligosaccharides from Laminaria japonica. Bioresource Technology, 2019b, 281: 84-89 DOI:10.1016/j.biortech.2019.02.056 |
LI S, YANG X, BAO M, et al. Family 13 carbohydrate-binding module of alginate lyase from Agarivorans sp.L11 enhances its catalytic efficiency and thermostability, and alters its substrate preference and product distribution. FEMS Microbiology Letters, 2015, 362(10): 54-61 |
LIN C Z, GUAN H S, LI H H, et al. The influence of molecular mass of sulfated propylene glycol ester of low-molecular- weight alginate on anticoagulant activities. European Polymer Journal, 2007, 43(7): 3009-3015 DOI:10.1016/j.eurpolymj.2007.04.015 |
MADZAK C. Yarrowia lipolytica: Recent achievements in heterologous protein expression and pathway engineering. Applied Microbiology and Biotechnology, 2015, 99(11): 4559-4577 DOI:10.1007/s00253-015-6624-z |
MIYAMOTO T, OKANO S, KASAI N. Inactivation of Escherichia coli endotoxin by soft hydrothermal processing. Applied and Environmental Microbiology, 2009, 75(15): 5058-5063 DOI:10.1128/AEM.00122-09 |
MOCHIZUKI S, NISHIYAMA R, INOUE A, et al. A novel aldo-keto reductase HdRed from the Pacific abalone Haliotis discus hannai, which reduces alginate-derived 4- deoxy-L-erythro- 5-hexoseulose uronic acid to 2-keto- 3-deoxy-D-gluconate. Journal of Biological Chemistry, 2015, 290(52): 30962-30974 DOI:10.1074/jbc.M115.686725 |
QIN H M, MIYAKAWA T, INOUE A, et al. Structural basis for controlling the enzymatic properties of polymannuronate preferred alginate lyase FlAlyA from the PL-7 family. Chemical Communication, 2018, 54: 555-558 DOI:10.1039/C7CC06523J |
SCHILLER N, MONDAY S, BOYD C, et al. Characterization of the Pseudomonas aeruginosa alginate lyase gene (algL): Cloning, sequencing, and expression in Escherichia coli. Journal of Bacteriology, 1993, 175(15): 4780-4789 DOI:10.1128/jb.175.15.4780-4789.1993 |
SHARMA H S S, FLEMING C, SELBY C, et al. Plant biostimulants: A review on the processing of macroalgae and use of extracts for crop management to reduce abiotic and biotic stresses. Journal of Applied Phycology, 2014, 26(1): 465-490 DOI:10.1007/s10811-013-0101-9 |
SHARMA S, HORN S J. Enzymatic saccharification of brown seaweed for production of fermentable sugars. Bioresource Technology, 2016, 213: 155-161 DOI:10.1016/j.biortech.2016.02.090 |
SUN L P, XUE C H, XU J C, et al. A study of the antioxidant abilities of alginate oligosaccharides. Periodical of Ocean University of China (Natural Sciences), 2005, 35(5): 811-814 [孙丽萍, 薛长湖, 许家超, 等. 褐藻胶寡糖体外清除自由基活性的研究. 中国海洋大学学报(自然科学版), 2005, 35(5): 811-814] |
THOMAS F, LUNDQVIST L, JAM M, et al. Comparative characterization of two marine alginate lyases from Zobellia galactanivorans reveals distinct modes of action and exquisite adaptation to their natural substrate. Journal of Biological Chemistry, 2013, 288: 23021-23037 DOI:10.1074/jbc.M113.467217 |
UCHIMURA K, MIYAZAKI M, NOGI Y, et al. Cloning and sequencing of alginate lyase genes from deep-sea strains of Vibrio and Agarivorans and characterization of a new Vibrio enzyme. Marine Biotechnology, 2010, 12: 526-533 DOI:10.1007/s10126-009-9237-7 |
WANG Y, CHEN X, BI X, et al. Characterization of an alkaline alginate lyase with pH-stable and thermo-tolerance property. Marine Drugs, 2019, 17(5): 308-322 DOI:10.3390/md17050308 |
WARGACKI A J, LEONARD E, WIN M N, et al. An engineered microbial platform for direct biofuel production from brown macroalgae. Science, 2012, 335(6066): 308-313 DOI:10.1126/science.1214547 |
WONG T Y, PRESTON L A, SCHILLER N L. Alginate lyase: Review of major sources and enzyme characteristics, structure-function analysis, biological roles, and applications. Annual Review of Microbiology, 2000, 54(1): 289-340 DOI:10.1146/annurev.micro.54.1.289 |
XIN X L, GENG M Y, QI X, et al. Study on the action mechanism of a new type anti-cerebral ischemia marine drug 989. Periodical of Ocean University of China (Natural Sciences), 2001, 31(4): 535-536 [辛现良, 耿美玉, 戚欣, 等. 新型抗脑缺血海洋新药989作用机理的初步探讨. 青岛海洋大学学报(自然科学版), 2001, 31(4): 535-536] |
XU X, YOSHIKO I, YOSHIE K, et al. Root growth-promoting activity of unsaturated oligomeric uronates from alginate on carrot and rice plants. Bioscience Biotechnology and Biochemistry, 2003, 67(9): 2022-2025 DOI:10.1271/bbb.67.2022 |
YOSHIKO I, XU X, TADASHI T, et al. Enzymatically depolymerized alginate oligomers that cause cytotoxic cytodine production in human mononuclear cells. Bioscience Biotechnology and Biochemistry, 2003, 67(2): 258-263 DOI:10.1271/bbb.67.258 |
ZHANG Y, YIN H, ZHAO X, et al. The promoting effects of alginate oligosaccharides on root development in Oryza sativa L. mediated by auxin signaling. Carbohydrate Polymers, 2014, 113: 446-454 DOI:10.1016/j.carbpol.2014.06.079 |
ZHANG Z, TANG L, BAO M. Functional characterization of carbohydrate-binding modules in a new alginate lyase, TsAly7B, from Thalassomonas sp.LD5. Marine Drugs, 2020, 18(1): 25-35 |
ZHU B W, HUANG L S X, TAN H D, et al. Characterization of a new endo-type polyM-specific alginate lyase from Pseudomonas sp. Biotechnology Letters, 2015, 37(2): 409-415 DOI:10.1007/s10529-014-1685-0 |
ZHU B, NI F, NING L, et al. Cloning and characterization of a new pH-stable alginate lyase with high salt tolerance from marine Vibrio sp.NJ-04. International Journal of Biological Macromolecules, 2018, 108: 1063-1070 DOI:10.1016/j.ijbiomac.2017.11.018 |
ZHU B, SUN Y, NI F, et al. Characterization of a new endo-type alginate lyase from Vibrio sp.NJU-03. International Journal of Biological Macromolecules, 2017, 164: 1140-1147 |
ZHUANG J, ZHANG K, LIU X, et al. Characterization of a novel polyM-preferred alginate lyase from Marine Vibrio splendidus OU02. Marine Drugs, 2018, 16(9): 295-230 DOI:10.3390/md16090295 |



