2. 湖南师范大学 省部共建淡水鱼类发育生物国家重点实验室 湖南 长沙 410081;
3. 贵州大学动物科学学院 贵州 贵阳 550025
2. State Key Laboratory of Developmental Biology of Freshwater Fish, School of Life Sciences, Hunan Normal University, Changsha, Hu'nan 410081, China;
3. School of Animal Science, Guizhou University, Guiyang, Guizhou 550025, China
褪黑激素(melatonin)是一种广泛分布于动物、植物、细菌和真菌中的天然小分子物质(Xu et al, 2017)。在哺乳动物中,褪黑激素主要由下丘脑中的松果体在夜间分泌,在性腺、骨髓、视网膜、晶状体、肾脏和肝脏等组织中也有发现(Carpentieri et al, 2012; Acuña-Castroviejo et al, 2014)。褪黑激素不仅具有调节昼夜节律、清除自由基的作用(Han et al, 2017),还能通过内分泌、旁分泌和自分泌方式保护细胞抗氧化和炎症损伤、改善胰岛素抵抗(Heo et al, 2018)、调控生殖发育(Maitra et al, 2016)、调节脂质代谢(Yang et al, 2017)和控制血压(de Berardis et al, 2013)。褪黑激素还能参与维持神经元的内稳态(Hermoso et al, 2016)和保护机体免疫系统的功能(艾克拜尔·热合曼等, 2018; Zhang et al, 2019)。
褪黑激素的功能主要通过与其高亲和力的特异性褪黑激素受体(melatonin receptor, Mtnr1)结合,再进一步启动信号级联反应,参与细胞内各种生理过程。Mtnr1属于G蛋白偶联受体超级家族(Brydon et al, 1999),与其他G蛋白偶联受体结构相似,包括7个跨膜域、1个G蛋白结合位点、多个磷酸化以及糖基化位点(Klosen et al, 2019)。另外,第3跨膜域的下游含有NRY结构域和C(C/Y) ICHS结构域,第7跨膜域中存在NPXXY结构域(Reppert et al, 1995b; Klosen et al, 2019)。Mtnr1在脊椎动物中存在3种不同亚型,分别为Mtnr1A (Mel 1a或MT1)、Mtnr1B (Mel 1b或MT2)和Mtnr1C (Mel 1c或GPR50) (Reppert et al, 1995a),而哺乳动物中仅有前2种亚型:Mtnr1A和Mtnr1B (Gaildrat et al, 2000),仅在鱼类等低等脊椎动物中发现第3种亚型Mtnr1C (Dufourny et al, 2008)。此外,鱼类特有基因组加倍事件导致Mtnr1A和Mtnr1B存在多个不同亚型,如斑马鱼(Danio rerio) Mtnr1Aa和Mtnr1Ab (Reppert et al, 1995b);虹鳟(Oncorhynchus mykiss) Mtnr1Aa、Mtnr1Ab和Mtnr1B (Mazurais et al, 1999);星点东方鲀(Takifugu niphobles) Mel1a1.4、Mel1a1.7和Mel1b (Ikegami et al, 2009b);大弹涂鱼(Boleophthalmus pectinirostris) Mel1a1.4、Mel1a1.7和Mel1b (朱文博等, 2012; Hong et al, 2014);大西洋鲑(Salmo salar) Mtnr1Aaα、Mtnr1Aaβ、Mtnr1Ab、Mtnr1al和Mtnr1B (Ciani et al, 2019);金鱼(Carassius auratus)Mel1a1.4、Mel1a1.7和Mel1b (Ikegami et al, 2009a)。值得注意的是,Mtnr1广泛参与机体神经调节(Klosen et al, 2019)、节律同步(Ikegami et al, 2009b)、内分泌激素合成(Ciani et al, 2019)和生长繁殖(Ikegami et al, 2009b; Hong et al, 2014)等生理活动。例如,敲除Mtnr1A基因后的小鼠表现出焦虑、类似抑郁症,增加了奖励和上瘾倾向,降低了快速眼动睡眠(Comai et al, 2019),生理节律(包括年生殖节律)也发生改变(Ng et al, 2017)。另外,Mtnr1在机体的细胞和体液免疫反应过程发挥积极作用,如在人(Homo sapiens)淋巴细胞(Jurkat)和单核细胞(U937)研究中发现,褪黑激素能通过Mtnr1增强其白细胞介素IL-2和IL-6分泌(García-Mauriño et al, 2000),添加MtnrB抑制剂Luzindole后,不仅降低了人淋巴细胞中IL-2和IL-2受体的分泌(Carrillo-Vico et al, 2004),还增强了前列腺素E2对IL-2的抑制效果(Carrillo-Vico et al, 2005)。此外,褪黑激素能促进野生型和MtnrA敲除型小鼠脾淋巴细胞增殖,而抑制MtnrA蛋白后增殖现象消失(Drazen et al, 2001)。这些研究证实了Mtnr1参与哺乳动物神经活动和免疫过程,但有关硬骨鱼类Mtnr1的研究仍处于起步阶段。
草鱼(Ctenopharyngodon idella)是我国重要的淡水经济鱼类,其2018年产量达到5.5万t (农业农村部渔业渔政管理局等, 2019)。草鱼疾病的暴发与其免疫调节能力密切相关,褪黑激素在机体免疫过程中具有重要作用,Mtnr1是褪黑激素发挥生理功能的关键受体分子,因此,解析Mtnr1基因在草鱼免疫调节中的功能对草鱼疾病的免疫防治具有重要意义。本研究以草鱼为研究对象,克隆得到Mtnr1基因6种异构性的全长cDNA序列,通过生物信息学技术,研究不同亚型的序列和结构特征,同时,采用实时荧光定量PCR (RT-qPCR)检测6种亚型在不同组织/器官中的分布和表达模式,研究结果可为Mtnr1在脊椎动物中的进化补充数据,同时,可为后续深入研究其基因功能以及揭示Mtnr1在草鱼免疫过程中的作用奠定分子基础。
1 材料与方法 1.1 实验材料实验草鱼购自湖北荆州水产市场,大小规格为(1.00±0.25) kg,暂养在实验室循环养殖系统2周。水温为(24±2)℃。饱食投喂2次/d (09:00和17:00)。暂养结束后,饥饿24 h取样,用MS-222 (10 mg/L)麻醉鱼体,在冰上迅速取出其肝脏、心脏、鳃、脑、肌肉、前肠、中肠、后肠和肾脏9个组织,液氮速冻后,存于–80℃冰箱备用。
1.2 实验方法 1.2.1 总RNA的提取和cDNA的合成采用Total RNA kit I提取试剂盒(Omega, 美国)提取总RNA,使用1%的琼脂糖凝胶电泳和超微量核酸蛋白测定仪(Thermo Scientific, 美国)检测RNA完整性和浓度。利用MonScriptTM RTⅢ Super Mix with dsDNase (two-step) (莫纳, 苏州)合成cDNA,严格按照试剂盒说明书操作,PCR产物保存于–20℃。
1.2.2 草鱼Mtnr1基因全长cDNA克隆利用斑马鱼、鲤鱼(Cyprinus carpio)、鲫鱼(Carassius auratus)以及哺乳动物人类、褐家鼠(Rattus norvegicus)等已公布Mtnr1A、Mtnr1B和Mtnr1C基因序列,比对保守区域后设计引物(表 1),进行扩增实验。再基于获得的部分cDNA序列,分别利用RACE技术,按照RACE试剂盒(TaKaRa)对Mtnr1的5′和3′端进行扩增。扩增产物测序正确后,通过DNAStar软件拼接获得完整的cDNA序列。
|
|
表 1 草鱼褪黑激素受体Mtnr1基因克隆扩增引物 Tab.1 Amplification primers used for cloning Mtnr1 genes from C. idella |
利用NCBI中Blast (http://blast.ncbi.nlm.nih.gov/)分析获得的Mtnr1不同亚型cDNA序列的正确性。通过NCBI网站的ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/)查找Mtnr1不同亚型的ORF框。使用ExPASy (http://web.expasy.org/translate/)翻译Mtnr1不同亚型cDNA编码蛋白序列。利用ProtParam在线软件预测Mtnr1不同亚型的蛋白基本理化特性(https://web.expasy.org/protparam/)。通过Tmpred在线软件对Mtnr1不同亚型的跨膜域进行预测(http://www.cbs.dtu.dk/services/ TMHMM/#opennewwindow)。并采用Singal4.1软件预测其信号肽。采用Phyre2软件对Mtnr1不同亚型蛋白质的三级结构进行预测(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)。利用Cell-PLoc2.0在线软件(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)预测Mtnr1的亚细胞定位。通过NetPhos和NetNGlyc软件分别对蛋白磷酸化位点(http://www.cbs.dtu.dk/services/NetPhos/)和糖基化位点(http://www.cbs.dtu.dk/services/NetNGlyc/)进行分析。采用ClustalW软件对Mtnr1不同亚型不同物种间进行序列比对和一致性分析。最后利用MEGA 5.2软件,以邻接法(NJ)对Mtnr1构建系统进化树,设定bootstrap值为1000。
1.2.4 草鱼Mtnr1基因的表达模式随机选取4条暂养后的草鱼,分别提取9个不同组织(肝脏、心脏、鳃、脑、肌肉、前肠、中肠、后肠和肾脏)的总RNA,设置4个生物学重复。以总RNA为模板,按照MonScriptTM RTⅢ Super Mix with dsDNase (莫纳,苏州)说明书合成cDNA,并将cDNA稀释10倍作为RT-qPCR模板,保存于–20℃备用。Mtnr1基因的组织表达模式测定方法参考侯吉伦等(2019):依据克隆获得的草鱼Mtnr1基因序列,利用Primer 5.0软件,设计Mtnr1基因RT-qPCR定量引物RT-F和RT-R,并以β-actin为内参基因(表 1)。根据说明书设置定量反应体系:SYBRⓇ fast qPCR master mixⅡ20 μL,cDNA模板4 μL,引物各0.5 μL。RT-qPCR反应程序:95℃ 5 min;95℃ 15 s,60℃ 30 s,72℃ 20 s,共40个循环。利用2–ΔΔCt方法(Livak et al, 2002)检测Mtnr1不同亚型在不同组织中的表达模式。
1.3 统计分析实验结果均采用平均值±标准误(Mean±SE)表示,为确保实验过程中数据的准确性,设置4个生物学重复,每个生物学重复设置3个技术重复。采用SPSS 18.0进行统计分析,采用单因素方差分析(one-way ANOVA)的Tukey法对Mtnr1不同亚型的相对表达量进行两两比较,显著性水平设置为0.05。
2 结果与分析 2.1 Mtnr1基因亚型cDNA序列分析利用DNAStar将克隆获得的cDNA进行拼接,并通过NCBI中的Blast对获取的序列进行正确性验证,本实验克隆得到草鱼6个Mtnr1 cDNA序列,分别为Mtnr1Aa、Mtnr1Ab、Mtnr1Alike、Mtnr1Ba、Mtnr1Bb和Mtnr1C,并将其提交至NCBI的GenBank,序列号分别为MT883605、MT883607、MT883608、MT883606、MT883609和MT883610,其全长分别为2045、2036、2031、2799、2535和2477 bp (表 2)。ProtParam软件预测发现,草鱼Mtnr1基因编码蛋白的氨基酸数、分子量和等电点分别见表 2。Mtnr1Aa、Mtnr1Ab、Mtnr1Alike、Mtnr1Ba、Mtnr1Bb和Mtnr1C基因编码蛋白均由20种氨基酸组成,含量最高的氨基酸分别为缬氨酸(12.6%)、缬氨酸(11.3%)、亮氨酸(11.6%)、亮氨酸(13.0%)、亮氨酸(11.6%)和亮氨酸(12.5%),含量最低的氨基酸分别为色氨酸(1.7%)、组氨酸(1.4%)、谷氨酰胺(1.5%)、组氨酸(1.1%)、组氨酸(1.2%)和组氨酸(1.4%)。带正(负)电氨基酸残基数分别为28(17)、18(32)、19(33)、17(35)、16(30)和11(31)。原子组成分别为C1853H2853N461O475S18、C1877H2905N463O479S17、C1836H2845N463O458S15、C1820H2883N489O470S18、C1819H2825N463O463S13和C1839H2885N481O484S20。蛋白质不稳定系数分别为35.19、35.32、30.93、38.67、35.74和34.75,均属于稳定蛋白。总平均亲水性分别为0.560、0.560、0.556、0.490、0.675和0.502,均为亲水性蛋白。脂肪酸指数分别为113.77、116.36、120.90、115.24、119.28和109.58。
|
|
表 2 草鱼褪黑激素受体Mtnr1 cDNA基本信息 Tab.2 The information of Mtnr1 cDNA of C. idella |
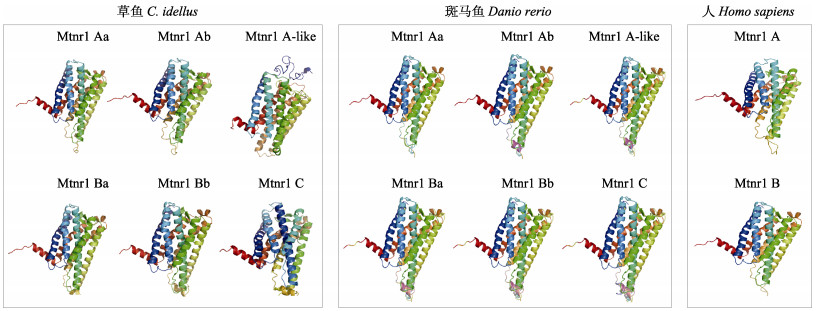
利用TMpred在线软件对草鱼Mtnr1的6种亚型蛋白跨膜域进行分析。结果显示,草鱼Mtnr1的6种亚型均存在7个跨膜结构,包括N端、跨膜区域、细胞膜内和膜外区域以及C端,表明其为非胞内蛋白。通过Cell-PLoc 2.0在线软件预测Mtnr1的6种亚型编码蛋白亚细胞定位,显示均定位于细胞膜。通过SignalP4.1预测草鱼Mtnr1的6种基因编码蛋白质的氨基酸序列,均未发现信号肽序列,说明6种亚型编码蛋白均为非分泌型蛋白。利用Phyre2在线软件构造草鱼Mtnr1的6种模拟蛋白3D亚型与人Mtnr1A和Mtnr1B蛋白以及斑马鱼的6种模拟结构对比,结果显示,蛋白结构较为相似(图 1)。此外,通过多重氨基酸序列比较可知,Mtnr1的6种亚型具有NRY结构域、CYICHS结构域、NAXXY结构域和G蛋白偶联受体结合位点(图 2)。利用NetNGlyc 1.0和NetPhos 3.1预测功能位点,结果发现,6种亚型受体的糖基化位点和磷酸化位点数分别为Mtnr1Aa (Asn4、Asn9和Asn117/22、Mtnr1Ab (Asn7、Asn13和Asn121)/28、Mtnr1Alike (Asn37和Asn144)/17、Mtnr1Ba (Asn4、Asn10和Asn122)/24、Mtnr1Bb(Asn10和Asn121)/25和Mtnr1C (Asn7、Asn25和Asn121)/30。
|
图 1 草鱼、斑马鱼和人的基于Mtnr1亚型氨基酸序列预测的蛋白3D结构 Fig.1 Protein structure prediction of C. idellus, Danio rerio and Homo sapiens based on sequences of Mtnr1 |

|
图 2 草鱼不同Mtnr1亚型氨基酸序列多重比对分析结果 Fig.2 Multiple alignment of the deduced amino acid sequence of grass carp Mtnr1 subtypes 利用DNAMAN (V9.0)对亚型氨基酸序列进行多重比对,保守区域用黑色表示。红色实线框中分别为7个跨膜域(编号Ⅰ~Ⅶ);黑色虚线框中为G-蛋白偶联受体结合位点;绿色实线框中为NRY结构域;紫色实线框中为NAXXY结构域;红色虚线框为糖基化位点。黑色五角星为半胱氨酸 Multiple alignments were generated using DNAMAN 9.0, and conserved amino acids shaded in black. Red boxes show seven transmembrane domain (TMD) Ⅰ~Ⅶ. The dotted boxes show the binding site of G-protein coupled receptors. The green box shows conserved NRY motif. The purple box shows the NAXXY motif. The red dot boxes show the NetNGlyc sites. The pentagrams show cysteines |
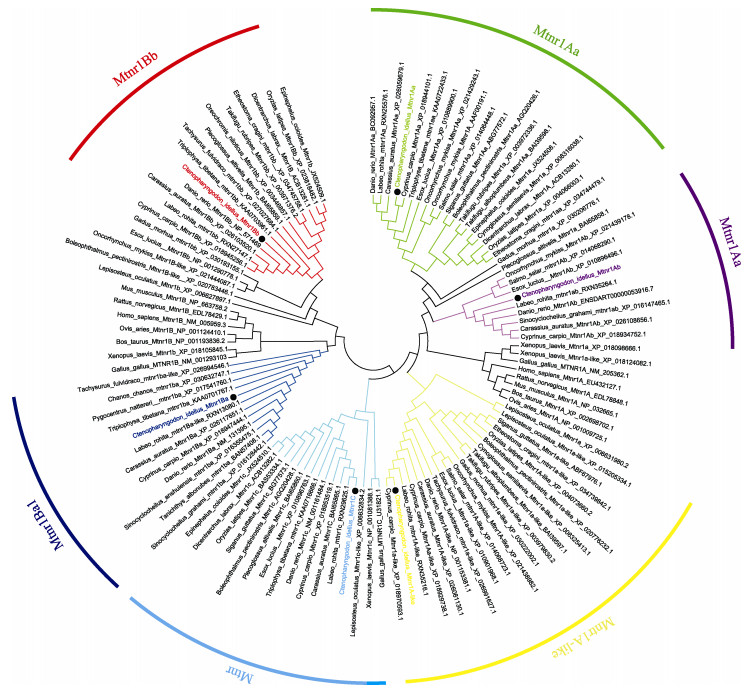
基于ClustalW,对草鱼Mtnr1的6种不同亚型编码的氨基酸序列同已有物种的Mtnr1序列同源性进行比较分析,结果见表 3。6种亚型与其他鱼对应的亚型同源性很高,分别为93.1%~99.4%、84.8%~ 95.2%、82.8%~96.8%、90.1%~97.5%、79.7%~98.3%和90.6%~95.6%。另外,运用MEGA 5.2软件构建草鱼Mtnr1的6种亚型氨基酸序列的系统进化树(图 3),Bootstrap重复次数设为1000次迭代。结果表明,构建的进化树与氨基酸的多重比较结果较一致。草鱼Mtnr1的6种亚型均与其他鱼类的对应亚型聚为一支,且与鲤鱼、鲫鱼和斑马鱼亲缘关系最近。
|
|
表 3 草鱼Mtnr1氨基酸序列与其他物种对应氨基酸序列的同源性 Tab.3 Homologies of deduced amino acid sequences of six Mtnr1 isoforms gene of C. idellus and other species |
|
图 3 采用MEGA 5.2软件构建的基于草鱼Mtnr1氨基酸序列的NJ系统进化树 Fig.3 Phylogenetic tree based on the protein sequences of Mtnr1 from C. idellus and other vertebrate species using the neighbor-joining (NJ) method in MGEA 5.2 |
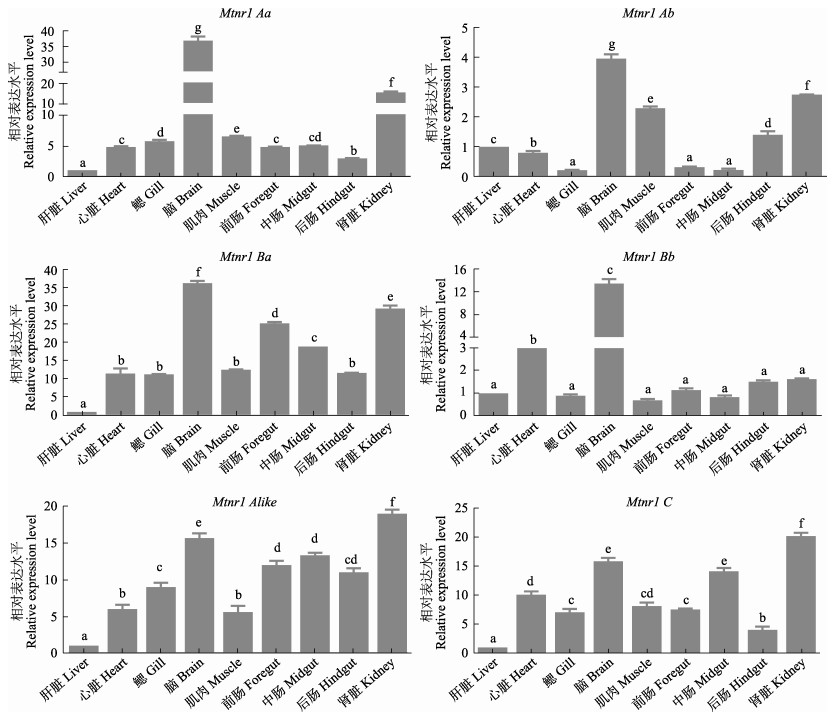
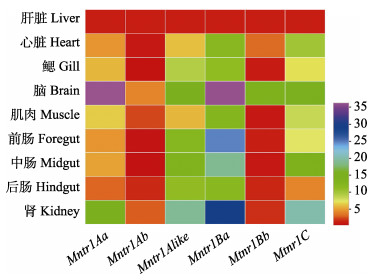
采用RT-qPCR技术检测了Mtnr1基因在草鱼肝脏、心脏、鳃、脑、肌肉、前肠、中肠、后肠和肾脏9个组织的表达情况。结果显示,Mtnr1的6种亚型在所检测的组织中均有表达(图 4)。其中,Mtnr1Aa基因在脑组织中表达量最高(P < 0.05),肾脏、鳃、中肠、心脏次之,而在肝脏组织中表达量最低(P < 0.05)。Mtnr1Ab基因在脑组织中表达量最高(P < 0.05),然后依次为肾脏、肌肉、后肠、肝脏和心脏,在鳃、前肠及中肠中表达量最低(P < 0.05),而鳃、前肠以及中肠之间表达量无显著差异(P > 0.05)。Mtnr1Alike基因在肾脏中表达量最高(P < 0.05),然后依次为脑、前肠、后肠和中肠,而在肝脏中表达量最低(P < 0.05)。Mtnr1Ba基因mRNA水平在脑中表达量最高,其次在肾脏、前肠和中肠表达量居中,随后心脏、鳃、肌肉和后肠中表达量次之,且它们之间无显著性差异(P > 0.05),在肝脏中表达量最低(P < 0.05)。Mtnr1Bb在脑中含量最高,其次为心脏,在其余组织中含量最低且它们之间无显著性差异(P > 0.05)。Mtnr1C基因表达量在肾脏组织中最高(P < 0.05),其次为脑、中肠和心脏,在肝脏组织中含量为最低(P < 0.05)。此外,从对亚型表达热图可知(图 5),6种亚型均在脑和肾脏组织中大量表达,其次为肠道组织,进一步发现,在脑组织中Mtnr1Aa基因表达量最高(P < 0.05),其次为Mtnr1Ba、Mtnr1C、Mtnr1Alike和Mtnr1Bb,而Mtnr1Ab基因表达量最低(P < 0.05) (图 5)。在肾脏组织中发现Mtnr1Ba表达量最高,其次为Mtnr1C、Mtnr1Alike、Mtnr1Aa和Mtnr1Ab,而Mtnr1Bb基因表达量最低(P < 0.05) (图 5)。另外,在前肠中发现Mtnr1Ba基因表达量最高(P < 0.05),其次为Mtnr1Alike、Mtnr1C、Mtnr1Aa和Mtnr1Bb,而Mtnr1Ab表达量最低(P < 0.05)。中肠中Mtnr1Ba基因表达量最高(P < 0.05),其次为Mtnr1C、Mtnr1Alike、Mtnr1Aa和Mtnr1Bb,而Mtnr1Ab表达量最低(P < 0.05)。后肠中Mtnr1Ba和Mtnr1Alike基因表达量最高(P < 0.05),且二者之间无显著差异(P > 0.05),其次为Mtnr1C、Mtnr1Aa和Mtnr1Ab,而Mtnr1Bb表达量最低(P < 0.05) (图 5)。
|
图 4 草鱼Mtnr1基因6种亚型在不同组织中的表达模式 Fig.4 Expression of 6 isoforms of Mtnr1 mRNA in different tissues of C. idellus 值为平均值±标准误(n=4),以β-actin基因表达量作为内参基因,设置3个技术重复。不同小写字母表示差异显著(P < 0.05) Data represent Mean±SE (n=4), expressed relative to expression of the housekeeping gene β-actin. Three technological repetitions were performed for each sample. Different letters indicate significant differences among the different tissues (P < 0.05) |
|
图 5 Mtnr1亚型的组织表达热图 Fig.5 Heatmap of expression pattern of the Mtnr1 family mRNA in different tissues of C. idellus 基因表达水平从高到低依次为紫色到红色 The levels of gene expression in the heatmap from high to low: purple to red |
本研究成功克隆得到草鱼Mntr1基因6个亚型的cDNA全长序列,全长序列长度为2031~2799 bp,编码的氨基酸个数为344~361,无信号肽序列,均属于非分泌型蛋白。Mtnr1的6种亚型由7个跨膜结构(Ⅰ~Ⅶ)构成,属于典型的G蛋白偶联受体(Shiu et al, 1998)。多序列比对结果显示,草鱼Mtnr1存在NRY结构域、CYICHS结构域、NAXXY结构域和G蛋白偶联受体结合位点以及多个糖基化和磷酸化位点,这与大西洋鲑和大弹涂鱼的Mtnr1基因研究结果一致(Hong et al, 2014; Ciani et al, 2019; Klosen et al, 2019),暗示草鱼Mtnr1在进化中具有较高的保守性。另外,6种亚型的3D结构与斑马鱼和人的Mntr1空间结构相似,进一步证实其进化保守性。同时,同源性比较发现,草鱼Mtnr1基因6种亚型(Mtnr1Aa、Mtnr1Ab、Mtnr1Alike、Mtnr1Ba、Mtnr1Bb和Mtnr1C)与其他鱼对应的Mntr1亚型同源性很高,分别为93.1%~99.4%、84.8%~95.2%、82.8%~96.8%、90.1%~ 97.5%、79.7%~98.3%、和90.6%~95.6%。系统进化树结果显示,草鱼Mtnr1基因6种亚型与鲤鱼、鲫鱼和斑马鱼等鲤科(Cyprinidae)鱼类的各亚型聚为一支,表明草鱼Mtnr1与鲤科鱼类亲缘关系较近,这与它们的进化地位一致。
Mtnr1的6种亚型基因在草鱼脑、肝脏、心脏、鳃、肌肉、前肠、中肠、后肠和肾脏9个组织中均有表达,表明Mntr1可能广泛参与机体各种生理活动(Hong et al, 2014)。本研究发现,Mtnr1Aa、Mtnr1Ab、Mtnr1Ba和Mtnr1Bb 4种亚型集中在脑组织中表达,暗示其主要参与神经调节相关的活动(Klosen et al, 2019)。这与大西洋鲑(Ciani et al, 2019)、星点东方鲀(Ikegami et al, 2009b)和金鱼(Ikegami et al, 2009a)中的研究相似。如Ikegami等(2009b)研究发现,Mel1a 1.4、Mel1a 1.7和Mel1b均在星点东方鲀脑组织、视网膜、脑垂体和外围组织中表达,且在脑组织中表达量最高。另外,褪黑激素在生物钟网络中既是视交叉上核(suprachiasmatic nuclei, SCN)的时钟输出,又是内部同步器,表明它的主要功能是充当时钟输如“time giver” (Pevet et al, 2011)。而褪黑激素功能发挥依赖于Mtnr1的表达,Mtnr1在脑组织中的高表达,间接证实褪黑激素在草鱼机体神经调节过程中具有重要作用。
此外,本研究显示,Mtnr1Ba基因在草鱼前肠、中肠和后肠中均高表达,Mtnr1Alike在后肠中高表达,Mtnr1的表达呈现组织差异性。这与哺乳动物和水禽类中的研究类似,如大鼠MT2在其结肠中mRNA水平最高,而MT1基因在十二指肠内表达量最高,在回肠和空肠中表达低(Stebelová et al, 2010);MT1和MT2在人胃肠道肌间神经丛、肠上皮和胃肠血管中均有表达,且在大肠上皮表达量最高(Söderquist et al, 2015);鸭肠道2-125碘褪黑素结合位点密度大小依次为回肠、空肠 > 十二指肠、结肠 > 盲肠(Lee et al, 1993)。另外,研究表明,Mtnr1介导褪黑激素作用肠道粘膜肌层和肠肌间神经丛发挥免疫功能,抵抗各种病原微生物的侵袭(Esteban-Zubero et al, 2017)。本研究中,Mtnr1基因在肠道不同部位表达量存在差异,暗示Mtnr1在草鱼肠道不同部位可能发挥不同的免疫功能,但尚需进一步研究。
值得注意的是,本研究发现,除Mtnr1Alike和Mtnr1C亚型外,Mtnr1Aa、Mtnr1Ab和Mtnr1Ba也在肾脏中高表达,这与金鱼中的研究结果类似(Ikegami et al, 2009a),而与欧洲鲈鱼(Dicentrarchus labrax) (Sauzet et al, 2008)鳃、星点东方鲀(Ikegami et al, 2009b)神经和垂体表达相异,究其原因可能是亚型基因表达存在物种和组织的特异性。肾脏作为鱼体主要的免疫器官,免疫功能相关基因在该组织中呈现高表达(侯吉伦等, 2019),在哺乳动物小鼠的胸腺和肾脏免疫器官中已经证实,Mtnr1mRNA表达量较高(Carrillo-Vico et al, 2003)。本研究结果显示,草鱼Mntr1亚型mRNA在肾脏中表达量高,暗示Mtnr1可能与免疫相关。而褪黑激素主要通过与高亲和力的特异性Mntr1蛋白结合,随后启动信号级联反应在靶细胞上介导各种生理活动(Klosen et al, 2019)。另外,褪黑激素可以直接作用于机体的免疫活性过程,如Cuesta等(2008)研究发现,金头鲷(Sparus aurata)注射褪黑激素后,血液中过氧化物酶、吞噬作用和活性氧中间体等活性以及干扰素-1β、主要组织相容性复合体、病毒相关效应和淋巴标记等基因表达显著上升。同样地,相对于对照组,注射褪黑激素后中华绒螯蟹(Eriocheir sinensis)血淋巴中超氧化物歧化酶活性显著增加,丙二醛的含量显著下调(She et al, 2019)。目前,尽管本研究Mntr1基因在肾脏中高表达,但仅推测Mntr1亚型介导了草鱼的免疫反应,而草鱼受到病原或褪黑激素刺激后,Mntr1亚型的表达和免疫效应情况并不清楚,需要后续开展实验进一步探究。
综上所述,本研究首次在草鱼组织中成功克隆得到Mntr1的6种亚型的cDNA序列,并对其序列结构进行了分析,通过对6种亚型在组织表达模式的研究,初步表明Mtnr1Aa和Mtnr1Ba基因与草鱼神经调节和免疫反应有关,为今后深入研究Mtnr1Aa和Mtnr1Ba基因在草鱼神经调节和免疫功能方面提供了理论依据。
ACUÑA-CASTROVIEJO D, ESCAMES G, VENEGAS C, et al. Extrapineal melatonin: Sources, regulation, and potential functions. Cellular and Molecular Life Sciences, 2014, 71(16): 2997-3025 DOI:10.1007/s00018-014-1579-2 |
BRYDON L, PETIT L, DE COPPET P, et al. Polymorphism and signalling of melatonin receptors. Reproduction, Nutrition, and Development, 1999, 39(3): 315-324 DOI:10.1051/rnd:19990304 |
Bureau of Fisheries, Ministry of Agriculture and Rural Affairs, National Fisheries Technology Extension Center, China Society of Fisheries. China fishery statistical yearbook 2019. Beijing: China Agriculture Press, 2019 [农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 2019中国渔业统计年鉴. 北京: 中国农业出版社, 2019]
|
CARPENTIERI A, DÍAZ DE BARBOZA G, ARECO V, et al. New perspectives in melatonin uses. Pharmacological Research, 2012, 65(4): 437-444 DOI:10.1016/j.phrs.2012.01.003 |
CARRILLO-VICO A, CALVO J R, ABREU P, et al. Evidence of melatonin synthesis by human lymphocytes and its physiological significance: Possible role as intracrine, autocrine, and/or paracrine substance. FASEB Journal, 2004, 18(3): 537-539 DOI:10.1096/fj.03-0694fje |
CARRILLO-VICO A, GARCIA-PERGANEDA A, NAJI L, et al. Expression of membrane and nuclear melatonin receptor mRNA and protein in the mouse immune system. Cellular and Molecular Life Sciences, 2003, 60(10): 2272-278 DOI:10.1007/s00018-003-3207-4 |
CARRILLO-VICO A, LARDONE P J, FERNANDEZ-SANTOS J M, et al. Human lymphocyte-synthesized melatonin is involved in the regulation of the interleukin-2/interleukin-2 receptor system. Journal of Clinical Endocrinology and Metabolism, 2005, 90(2): 992-1000 DOI:10.1210/jc.2004-1429 |
CIANI E, FONTAINE R, MAUGARS G, et al. Melatonin receptors in Atlantic salmon stimulate cAMP levels in heterologous cell lines and show season-dependent daily variations in pituitary expression levels. Journal of Pineal Research, 2019, 67(3): e12590 |
COMAI S, LOPEZ-CANULA M, DE GREGORIO D, et al. Melatonin MT1 receptor as a novel target in neuropsychopharma-cology: MT1 ligands, pathophysio- logical and therapeutic implications, and perspectives. Pharmacological Research, 2019, 144: 343-356 DOI:10.1016/j.phrs.2019.04.015 |
CUESTA A, CEREZUELA R, ESTEBAN M A, et al. In vivo actions of melatonin on the innate immune parameters in the teleost fish gilthead seabream. Journal of Pineal Research, 2008, 45(1): 70-78 DOI:10.1111/j.1600-079X.2008.00557.x |
DE BERARDIS D, MARINI S, FORNARO M, et al. The melatonergic system in mood and anxiety disorders and the role of agomelatine: Implications for clinical practice. International Journal of Molecular Sciences, 2013, 14(6): 12458-12483 DOI:10.3390/ijms140612458 |
DRAZEN D L, BILU D, BILBO S D, et al. Melatonin enhancement of splenocyte proliferation is attenuated by luzindole, a melatonin receptor antagonist. American Journal of Physiology-Regulatory Integrative and Comparative Physiology, 2001, 280(5): 1476-1482 DOI:10.1152/ajpregu.2001.280.5.R1476 |
DUFOURNY L, LEVASSEUR A, MIGAUD M, et al. GPR50 is the mammalian ortholog of Mel1c: Evidence of rapid evolution in mammals. BMC Evolutionary Biology, 2008, 8: 105 DOI:10.1186/1471-2148-8-105 |
ESTEBAN-ZUBERO E, LÓPEZ-PINGARRÓN L, ALATORRE-JIMÉNEZ M A, et al. Melatonin's role as a co-adjuvant treatment in colonic diseases: A review. Life Science, 2017, 170: 72-81 DOI:10.1016/j.lfs.2016.11.031 |
GAILDRAT P, FALCÓN J. Melatonin receptors in the pituitary of a teleost fish: mRNA expression, 2-[125I]iodomelatonin binding and cyclic AMP response. Neuroendocrinology, 2000, 72: 57-66 DOI:10.1159/000054571 |
GARCÍA-MAURIÑO S, POZO D, CALVO J R, et al. Correlation between nuclear melatonin receptor expression and enhanced cytokine production in human lymphocytic and monocytic cell lines. Journal of Pineal Research, 2000, 29(3): 129-137 DOI:10.1034/j.1600-079X.2000.290301.x |
HAN L S, WANG H C, LI L, et al. Melatonin protects against maternal obesity-associated oxidative stress and meiotic defects in oocytes via the SIRT3-SOD2-dependent pathway. Journal of Pineal Research, 2017, 63(3): e12431 DOI:10.1111/jpi.12431 |
HEO J I, YOON D W, YU J H, et al. Melatonin improves insulin resistance and hepatic steatosis through attenuation of alpha-2-HS-glycoprotein. Journal of Pineal Research, 2018, 65(2): e12493 DOI:10.1111/jpi.12493 |
HERMOSO D A M, SHIMADA L B C, GILGLIONI E H, et al. Melatonin protects female rats against steatosis and liver oxidative stress induced by oestrogen deficiency. Life Sciences, 2016, 157: 178-186 DOI:10.1016/j.lfs.2016.05.044 |
HONG L Y, HONG W S, ZHU W B, et al. Cloning and expression of melatonin receptors in the mudskipper Boleophthalmus pectinirostris: Their role in synchronizing its semilunar spawning rhythm. General and Comparative Endocrinology, 2014, 195: 138-150 DOI:10.1016/j.ygcen.2013.11.004 |
HOU J L, GUO Y N, FU Y S, et al. Cloning and expression analysis of efhd2 and tbc1d25 gene in Japanese flounder (Paralichthys olivaceus). Progress in Fishery Sciences, 2019, 40(3): 57-68 [侯吉伦, 郭亚男, 付元帅, 等. 牙鲆efhd2和tbc1d25基因的克隆和表达分析. 渔业科学进展, 2019, 40(3): 57-68] |
IKEGAMI T, AZUMA K, NAKAMURA M, et al. Diurnal expressions of four subtypes of melatonin receptor genes in the optic tectum and retina of goldfish. Comparative Biochemistry and Physiology Part A, Molecular and Integrative Physiology, 2009a, 152(2): 219-224 DOI:10.1016/j.cbpa.2008.09.030 |
IKEGAMI T, MOTOHASHI E, DOI H, et al. Synchronized diurnal and circadian expressions of four subtypes of melatonin receptor genes in the diencephalon of a puffer fish with lunar-related spawning cycles. Neuroscience Letters, 2009b, 462(1): 58-63 DOI:10.1016/j.neulet.2009.06.076 |
KLOSEN P, LAPMANEE S, SCHUSTER C, et al. MT1 and MT2 melatonin receptors are expressed in nonoverlapping neuronal populations. Journal of Pineal Research, 2019, 67(1): e12575 |
LEE P P, PANG S F. Melatonin and its receptors in the gastrointestinal tract. Neurosignals, 1993, 2(4): 181-193 DOI:10.1159/000109491 |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR. Methods, 2002, 25(4): 402-408 |
MAITRA S K, HASAN K N. The role of melatonin as a hormone and an antioxidant in the control of fish reproduction. Frontiers in Endocrinology, 2016, 7: 38 |
MAZURAIS D, BRIERLEY I, ANGLADE I, et al. Central melatonin receptors in the rainbow trout: Comparative distribution of ligand binding and gene expression. Journal of Comparative Neurology, 1999, 409(2): 313-324 DOI:10.1002/(SICI)1096-9861(19990628)409:2<313::AID-CNE11>3.0.CO;2-1 |
NG K Y, LEONG M K, LIANG H, et al. Melatonin receptors: Distribution in mammalian brain and their respective putative functions. Brain Structure and Function, 2017, 222(7): 2921-2939 DOI:10.1007/s00429-017-1439-6 |
PEVET P, CHALLET E. Melatonin: Both master clock output and internal time-giver in the circadian clocks network. Journal of Physiology, 2011, 105(4/5/6): 170-182 |
REHEMAN A, FENG X W. The distribution of melatonin receptors and its influence on the immune system. Contemporary Animal Husbandry, 2018(12): 48-52 [艾克拜尔·热合曼, 冯昕炜. 褪黑激素受体的分布及其对免疫系统的作用. 当代畜牧, 2018(12): 48-52] |
REPPERT S M, GODSON C, MAHLE C D, et al. Molecular characterization of a second melatonin receptor expressed in human retina and brain: the Mel1b melatonin receptor. Proceedings of the National Academy of Sciences of the United States of America, 1995a, 92(19): 8734-8738 DOI:10.1073/pnas.92.19.8734 |
REPPERT S M, WEAVER D R, CASSONE V M, et al. Melatonin receptors are for the birds: Molecular analysis of two receptor subtypes differentially expressed in chick brain. Neuron, 1995b, 15(5): 1003-1015 DOI:10.1016/0896-6273(95)90090-X |
SAUZET S, BESSEAU L, HERRERA P P, et al. Cloning and retinal expression of melatonin receptors in the European sea bass, Dicentrarchus labrax. General and Comparative Endocrinology, 2008, 157(2): 186-195 DOI:10.1016/j.ygcen.2008.04.008 |
SHE Q, HAN Z, LIANG S, et al. Impacts of circadian rhythm and melatonin on the specific activities of immune and antioxidant enzymes of the Chinese mitten crab (Eriocheir sinensis). Fish and Shellfish Immunology, 2019, 89: 345-353 DOI:10.1016/j.fsi.2019.04.011 |
SHIU S Y W, PANG S F. An updated phylogenetic analysis of vertebrate melatonin receptor sequences: Reflection on the melatonin receptor nomenclature by the Nomenclature Subcommittee of the International Union of Pharmacology. Biological Signals and Receptors, 1998, 7: 244-248 DOI:10.1159/000014549 |
SÖDERQUIST F, HELLSTRÖM P M, CUNNINGHAM J L. Human gastroenteropancreatic expression of melatonin and its receptors MT1 and MT2. PLoS One, 2015, 10(3): e0120195 DOI:10.1371/journal.pone.0120195 |
STEBELOVÁ K, ANTTILA K, MÄNTTÄRI S, et al. Immunohistochemical definition of MT2 receptors andmelatonin in the gastrointestinal tissues of rat. Acta Histochemica, 2010, 112(1): 26-33 DOI:10.1016/j.acthis.2008.03.004 |
XU P F, WANG J L, HONG F, et al. Melatonin prevents obesity through modulation of gut microbiota in mice. Journal of Pineal Research, 2017, 62(4): e12399 DOI:10.1111/jpi.12399 |
YANG W, TANG K, WANG Y, et al. Melatonin promotes triacylglycerol accumulation via MT2 receptor during differentiation in bovine intramuscular preadipocytes. Scientific Reports, 2017, 7: 15080 DOI:10.1038/s41598-017-12780-y |
ZHANG C, ZHANG Q, PANG Y, et al. The protective effects of melatonin on oxidative damage and the immune system of the Chinese mitten crab (Eriocheir sinensis) exposed to deltamethrin. Science of Total Environment, 2019, 653(25): 1426-1434 |
ZHU W B, LIAN H B, HONG W S, et al. Localization of melatonin receptor Mel1a in mudskipper (Boleophthalmus pectinirostris). Journal of Xiamen University (Natural Science), 2012, 51(6): 1092-1096 [朱文博, 连惠波, 洪万树, 等. 褪黑激素受体亚型Mel_(1a)在大弹涂鱼体内的分布. 厦门大学学报(自然科学版), 2012, 51(6): 1092-1096] |




