2. 高邮市水产技术指导站 江苏 高邮 225600
2. Aquatic Product Technology Extension Station of Gaoyou, Gaoyou, Jiangsu 225600, China
近年来,对虾养殖已成为我国水产行业的重要组成部分(农业农村部渔业渔政管理局等, 2020)。经过多年的发展,江苏沿海地区形成了多种凡纳滨对虾(Litopenaeus vannamei)养殖模式,包括池塘鱼虾混养、小型温棚养殖等,经济效益显著。
随着对虾养殖业的迅猛发展,各种病害频发(Lee et al, 2015)。为推进水产养殖绿色发展,农业农村部大力开展用药减量行动,益生菌(Chumpol et al, 2017; 刘文亮等, 2017)、中草药(朱璐丹等, 2019; Zhang et al, 2021)等新型无抗化防病技术不断被运用到生产实践中。目前,国务院兽医行政管理部门批准的水产养殖用抗菌药有12种(农业农村部, 2020)。研究表明,抗生素在水产养殖上使用后部分被养殖对象吸收,不能吸收的部分则被排出体外(张骞月等, 2015),残留在土壤、水源中(D'costa et al, 2006),对动物体内以及环境中的细菌耐药性产生定向选择作用(Österblad et al, 2001),诱导耐药基因(antibiotic resistance genes, ARGs)的产生(Fridman et al, 2014; Frimodt-Møller et al, 2019)与传递(Frost et al, 2005; Mazel, 2006),改变环境和生物体内微生态结构,最终威胁人类健康与安全。
本课题组前期调研发现,江苏地区凡纳滨对虾养殖过程多使用微生态制剂来调节水质,为避免破坏池塘菌群结构,生产中很少施用抗生素。本研究选择4种不同养殖模式的凡纳滨对虾虾苗和成虾作为研究对象,利用K-B纸片法研究其肠道可培养细菌对6大类12种抗生素(四环素、多西环素、氟苯尼考、氯霉素、磺胺异恶唑、复方新诺明、新霉素、庆大霉素、环丙沙星、诺氟沙星、头孢曲松和头孢噻肟)的耐药性,并通过qRT-PCR技术检测肠道细菌总DNA中5大类9种ARGs (tetA、tetG、qnrA、qnrS、cmle3、floR、aadA、sul1、sul2)的丰度,探究江苏地区常见4种养殖模式下凡纳滨对虾不同生长阶段肠道耐药菌(antibiotics resistant bacteria, ARB)和ARGs污染的变化情况,为后续防控细菌耐药性产生以及水产养殖科学用药提供参考依据。
1 材料与方法 1.1 样品采集本研究选取江苏地区12家养殖场凡纳滨对虾4种主要养殖模式苗种投放前和成虾上市前2个时间节点的对虾为研究对象,苗种来源于省外不同虾苗生产企业,每种模式苗种、成虾样品各3份,共8组24个样品。4种养殖模式分别为:模式1 [M1,土塘养殖:斑点叉尾 

|
|
表 1 样品信息 Tab.1 Sample information (n=10; Mean±SD) |
对虾体表用75%酒精擦拭消毒,并用灭菌生理盐水漂洗后再进行肠道细菌分离培养。每组成虾样品取10只成虾的肠道,混匀称重后,加入1 mL灭菌生理盐水充分研磨,取100 μL适当稀释后的研磨液于2216E平板上均匀涂布,每个样品做3个重复,28℃培养过夜后进行计数。每组虾苗样品取10只虾苗重复上述操作。取均值作为各组凡纳滨对虾肠道可培养细菌含量。在平板上挑取形态不同的单菌落纯化培养2代后,利用细菌16S rDNA通用引物27F和1492R进行PCR扩增,产物经生工生物工程(上海)股份有限公司测序后,通过Blast检索系统对细菌序列同源性进行分析。
1.3 可培养细菌耐药性检测以大肠埃希氏菌(Escherichia coli)标准菌株(ATCC 25922)为质控菌,利用K-B纸片法测定上述分离纯化的细菌对四环素类、氯霉素类、磺胺类、氨基糖苷类、喹诺酮类和β-内酰胺类6大类12种抗生素的耐药性,每株菌做3个重复。药物敏感性实验方法及判读标准参考美国临床与实验室标准化研究所(CLSI)操作手册(Clinical and Laboratory Standards Institute, 2017)和《全国临床检验操作规程》(尚红等,2015)。药敏纸片和MH培养基购于杭州微生物试剂有限公司。耐药菌占比计算公式如下:
| $ \begin{array}{l}虾苗(成虾)耐药菌占比(\text{%})=\\ \;\;\;\;\;\;\frac{虾苗(成虾)肠道可培养细菌中耐药菌株数}{虾苗(成虾)肠道可培养细菌总株数}\end{array} $ |
利用ARGs引物(目的基因信息详见表 2),对本研究室储存的对虾源细菌DNA样品进行PCR扩增,琼脂糖凝胶电泳法检测目的条带。挑选出强阳性样本,用TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.4.0 (TaKaRa)回收目的条带。用pMD18-T Vector Cloning Kit (TaKaRa)对目的基因进行克隆。利用蓝白斑筛选、通用引物M13普通PCR扩增以及菌液测序的方法来验证克隆成功与否。用TaKaRa MiniBEST Plasmid Purification Kit Ver.4.0 (TaKaRa)提取克隆成功菌液的质粒,并以此质粒为标准品模板,利用Applied Biosystems Step One Plus Real-Time PCR System进行qRT-PCR分析,绘制标准曲线。
|
|
表 2 目的基因信息 Tab.2 Target gene information |
目的基因PCR程序:95℃ 5 min;95℃ 30 s,Tm 45 s,72℃ 1 min,35个循环72℃ 10 min。qRT-PCR程序:95℃ 30 s;95℃ 5 s,60℃ 30 s,40个循环。溶解曲线程序:95℃ 15 s,60℃ 15 s,95℃ 15 s。20 L反应体系:10 μL TB Green Premix Ex Taq (TaKaRa),上、下游引物各0. 4 μL,ROX 0.4 μL,模板0.2 μL。
1.4.2 样品DNA提取及qRT-PCR检测ARGs丰度用3S DNA Isolation Kit for Environment Samples (上海博彩生物)提取样品肠道细菌DNA(每组成虾样品取10只成虾的肠道研磨混匀后提取,每组虾苗样品取10只虾苗研磨混匀后提取),1.0%琼脂糖凝胶电泳和Thermo Nanodrop 2000检验DNA质量,以合格的DNA为模板,进行qRT-PCR检测,条件与反应体系同1.4.1。根据标准曲线计算各基因拷贝数的绝对含量,以16S rDNA为内参基因作归一化处理,计算各ARGs的相对含量,并以该相对含量进行下一步数据统计和分析。
1.5 数据统计方法采用Excel 2019进行可培养细菌数量统计、药物敏感性分析、ARGs标准曲线绘制以及ARGs样品Ct值归一化处理等。用TBtools对基因相对含量进行聚类热图分析(Chen et al, 2020)。
2 结果 2.1 可培养细菌平板计数及分类鉴定结果苗种样品中可培养细菌总数最小值为33 CFU/g,最大值为2.73×104 CFU/g;成虾样品可培养细菌总数最小值为6.33×104 CFU/g,最大值为1.12×107 CFU/g。按不同养殖模式分类,4种养殖模式苗期至成虾期细菌总数分别上升1832.1 (M1)、206.1 (M2)、610.0 (M3)和81.8 (M4)倍。各组对虾肠道可培养细菌含量对数值见图 1。
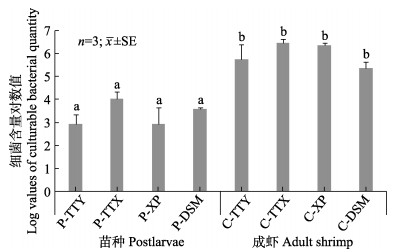
|
图 1 各组对虾肠道可培养细菌含量对数值 Fig.1 Log values of culturable bacterial quantity in shrimp intestinal tract of different groups 柱形图上方不同字母表示差异显著(P < 0.05).纵坐标为对应组别3个样品可培养细菌含量对数值均值 Different letters on the column indicate significant difference (P < 0.05). The ordinate represents mean log values of three samples' culturable bacterial quantity in the corresponding group |
共分离纯化得到17种、49株可培养细菌,来源于8个不同属,分别为弧菌属(Vibrio)、发光杆菌属(Photobacterium)、气单胞菌属(Aeromonas)、西瓦氏菌属(Shewanella)、芽孢杆菌属(Bacillus)、微小杆菌属(Exiguobacterium)、盐芽孢杆菌属(Halobacillus)、假交替单胞菌属(Pseudoalteromonas)(表 3)。其中,数量最多的为弧菌属,共计30株,占61.2%。弧菌属主要种类为溶藻弧菌(V. alginolyticus)和副溶血弧菌(V. parahaemolyticus),分别为11株和9株,占比为36.7%和30%。苗种样品中分离到3属,10种,21株细菌。其中,数量最多的为溶藻弧菌(9株),占42.9%。成虾样品中共分离到8属,12种,28株细菌,其中副溶血弧菌8株,占30.8%。
|
|
表 3 可培养细菌分类及数量 Tab.3 Classification and quantity of culturable dominant bacteria |
对虾肠道耐药菌占比结果见表 4。虾苗和成虾肠道耐药菌占比均较低,对氯霉素、庆大霉素、头孢曲松等药物的耐药菌占比都为0,对四环素类和磺胺类抗生素耐药的菌株占比稍高,但最大也只有19.05%。同时,成虾肠道耐药菌占比均低于虾苗。
|
|
表 4 对虾肠道耐药菌占比 Tab.4 Antibiotics resistant bacteria (ARB) proportion in shrimp intestinal tract |
以ARGs拷贝数对数值为横坐标,样品Ct值为纵坐标绘制标准曲线,结果见表 5。各ARGs标准曲线斜率(a)介于–3.388 6~–3.142 8之间,截距(b)介于40.647~46.469之间,相关系数(R2)介于0.991 3~0.998 5之间,引物扩增效率(E%)介于97.3~108.1之间,溶解曲线无杂峰。结果表明,标准曲线线性良好,且引物扩增效率高,能够用于相对拷贝数的计算。
|
|
表 5 抗生素ARGs标准曲线 Tab.5 Standard curves of drug antibiotic resistance genes |
结果显示,所有耐药基因丰度在凡纳滨对虾肠道中表现较低,以样品P-TTY-2中cmle3丰度稍高,为84.6;tetA在C-TTY-1和C-DSM-1两份样品中未被检出;qnrA在C-TTX-1、C-XP-1、C-XP-2、C-XP-3、C-DSM-1、C-DSM-2和C-DSM-3七份样品中未被检出。sul2在样品C-DSM-1中未被检出。样品中9种ARGs相对含量范围见表 6。
|
|
表 6 9种ARGs相对含量范围 Tab.6 Relative contents range of 9 antimicrobial resistant genes/(copies/16S rDNA) |
ARGs丰度检测结果见图 2。由ARGs丰度聚类分析结果可见,苗种和成虾样品聚在不同的分支,养成后的对虾中的ARGs丰度均较养殖前虾苗中低,表明经过1个养殖周期,ARGs丰度降低。氯霉素类(cmle3、floR)、四环素类(tetG)和磺胺类(sul2) 4种ARGs在虾苗样品中出现。

|
图 2 9种ARGs的丰度热图 Fig.2 Heat map of abundance of 9 antimicrobial resistant genes |
张盛静等(2015)研究表明,凡纳滨对虾在苗种阶段肠道内可培养的细菌属包括弧菌属、发光杆菌属、芽孢杆菌属和盐单胞菌属(Halomonas)等,其中弧菌属占绝对优势,养成阶段肠道内可培养的细菌属包括乳球菌属(Lactococcus)、弧菌属、芽孢杆菌属、发光杆菌属、希瓦氏菌属、节杆菌属(Arthrobacter)、微杆菌属(Microbacterium)等。本研究结果与其相似,部分种属之间的差异可能与养殖过程中微生态制剂使用较多(吴定心, 2016)和抗生素使用较少有关。
3.2 可培养细菌抗生素耐药性目前,国内已批准的水产养殖用抗微生物药物有12种(农业农村部, 2020),在鱼类细菌病的防控中起着重要作用。在我国部分养殖区(王志芳等, 2019; 李兆新等, 2018)的环境中能检出喹诺酮类、磺胺类、四环素类等抗生素残留,且距离养殖区越近浓度越高,呈现明显的时空分布特征(连璐璐, 2016)。持续性的低剂量抗生素胁迫,会增强细菌耐受性和耐药性,降低抗生素治疗效率。研究显示,间歇性将E. coli群体暴露于氨苄青霉素(ampicillin)中,其群体会对氨苄青霉素产生耐受性(Fridman et al, 2014),诱发菌株突变产生耐药性(Frimodt-Møller et al, 2019)。给日本沼虾(Macrobrachium nipponense)长期投饲含安全剂量抗生素的日粮后,会增加其体内细菌耐药性选择压力(Sun et al, 2020)。因此,必须重视细菌的耐药性。
本研究分离所得可培养细菌中对常见抗生素的耐药性均较低,仅对四环素类和磺胺类抗生素存在一定的耐药性,这与现实环境抗生素残留污染趋势类似(王志芳等, 2019; 李兆新等, 2018; 连璐璐, 2016),提示,这些细菌的耐药能力可能与自然环境中抗生素污染情况有关。另外,成虾耐药菌占比较虾苗降低,表明成虾肠道耐药菌污染程度较虾苗轻。
3.3 ARGs丰度差异抗生素使用可增加ARGs丰度和多样性(Fridman et al, 2014; Frimodt-Møller et al, 2019)。已有文献报道在对虾体内、肠道及养殖环境中检测出多种抗生素ARGs (Su et al, 2017; 洪斌等, 2019),整合子等一些移动基因原件(mobile genetic element, MGE)可促进ARGs在细菌间的传播(Mazel, 2006; Frost et al, 2005)。天津地区水产养殖环境中,磺胺类ARGs sul1和sul2丰度较高(Gao et al, 2012);对虾体内和养殖水环境中优势ARGs分别为sulⅠ、qnrD、cmlA、floR (Su et al, 2017)。本研究结果与前人研究结果类似,其中,floR和cmle3丰度稍高,可能与动物专用抗菌药氟苯尼考的使用有关,而磺胺类和四环素类ARGs则可能来源于环境污染。另外,研究还发现,成虾样品的ARGs丰度均显著低于苗种样品(P < 0.05),提示经过1个养殖周期,对虾体内ARGs污染程度下降。
3.4 细菌耐药性和ARGs关系分析研究结果显示,菌株对各大类抗生素的敏感性与其所对应的ARGs丰度并不能呈现完全一一对应的关系,这与闫倩倩等(2020)、牛丽等(2019)和赵姝等(2019)等研究结果类似。如氟苯尼考和氯霉素的耐药菌占比都很低,但floR和cmle3丰度高。这可能是因为对虾肠道可培养细菌占肠道菌群的比例低,且本研究仅针对肠道中可培养细菌展开耐药性研究,因此二者结果存在差异。又如,新霉素耐药菌占比较高,但aadA丰度较低。这可能是因为细菌受到脂质层(Lambert, 2002)和抗渗屏障(Mcdonnell et al, 1999)等因素的影响而表现出较强的耐药性,或者该耐药能力由氨基糖苷类其他的ARGs所赋予。
研究发现,多数细菌耐药性和ARGs丰度呈现出对应关系。如磺胺异恶唑、复方新诺明的耐药菌占比和sul2丰度表现相一致,其来源可能与自然环境污染有关。环丙沙星、诺氟沙星耐药菌占比和qnrS、qnrA丰度都较低,提示本地区凡纳滨对虾养殖业中,这2类药物可能使用较少,符合国家禁用这2种抗生素的规定,也表明我国自2017年开展的水产养殖用药减量行动的效果逐步显现。
4 结论本研究阐述了江苏地区4种养殖模式下,凡纳滨对虾肠道细菌耐药性和ARGs丰度与不同生长阶段的关系。研究表明,江苏地区4种养殖模式凡纳滨对虾肠道可培养细菌属为弧菌属;成虾肠道可培养细菌种类和数量较虾苗显著上升(P < 0.05),耐药菌占比降低,ARGs丰度显著降低(P < 0.05);各模式之间未发见明显规律。研究结果显示,各模式下成虾肠道细菌耐药性污染程度均小于虾苗,提示,科学、合理的养殖方法可能会降低凡纳滨对虾肠道可培养细菌的耐药性。
BACH H J, TOMANOVA J, SCHLOTER M, et al. Enumeration of total bacteria and bacteria with genes for proteolytic activity in pure cultures and in environmental samples by quantitative PCR mediated amplification. Journal of Microbiological Methods, 2002, 49(3): 235-245 DOI:10.1016/S0167-7012(01)00370-0 |
Bureau of Fisheries, Ministry of Agriculture and Rural Affairs, National Fisheries Technology Extension Center, China Society of Fisheries. China fishery statistical yearbook 2020. Beijing: China Agriculture Press, 2020: 21-27 [农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 2020中国渔业统计年鉴. 北京: 中国农业出版社, 2020: 21-27]
|
CHEN C, CHEN H, ZHANG Y, et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202 DOI:10.1016/j.molp.2020.06.009 |
CHUMPOL S, KANTACHOTE D, RATTANACHUAY P, et al. In vitro and in vivo selection of probiotic purple nonsulphur bacteria with an ability to inhibit shrimp pathogens: Acute hepatopancreatic necrosis disease-causing Vibrio parahaemolyticus and other vibrios. Aquaculture Research, 2017, 48(6): 3182-3197 DOI:10.1111/are.13149 |
Clinical and Laboratory Standards Institute (CLSI). Performance standards for antimricrobial susceptibility testing. 2017, 20–36
|
D′COSTA V M, MCGRANN K M, HUGHES D W, et al. Sampling the antibiotic resistome. Science (New York, NY), 2006, 311(5759): 374-377 DOI:10.1126/science.1120800 |
FRIDMAN O, GOLDBERG A, RONIN I, et al. Optimization of lag time underlies antibiotic tolerance in evolved bacterial populations. Nature, 2014, 513(7518): 418-421 DOI:10.1038/nature13469 |
FRIMODT-MØLLER J, LØBNER-OLESEN A. Efflux-pump upregulation: From tolerance to high-level antibiotic resistance?. Trends in Microbiology, 2019, 27(4): 291-293 DOI:10.1016/j.tim.2019.01.005 |
FROST L S, LEPLAE R, SUMMERS A O, et al. Mobile genetic elements: The agents of open source evolution. Nature Reviews Microbiology, 2005, 3(9): 722-732 DOI:10.1038/nrmicro1235 |
GAO P, MAO D, LUO Y, et al. Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment. Water Research, 2012, 46(7): 2355-2364 DOI:10.1016/j.watres.2012.02.004 |
HE L Y, LIU Y S, SU H C, et al. Dissemination of antibiotic resistance genes in representative broiler feedlots environments: Identification of indicator ARGs and correlations with environmental variables. Environmental Science and Technology, 2014, 48(22): 13120-13129 DOI:10.1021/es5041267 |
HE X L, XU Y B, CHEN J L, et al. Evolution of corresponding resistance genes in the water of fish tanks with multiple stresses of antibiotics and heavy metals. Water Research, 2017, 124: 39-48 DOI:10.1016/j.watres.2017.07.048 |
HONG B, NIU B, CHEN P, et al. Diversity of gut microbiota and antibiotic resistance genes in Litopenaeus vannamei and Macrobrachium rosenbergii. Journal of Fisheries of China, 2019, 43(5): 1347-1358 [洪斌, 牛犇, 陈萍, 等. 凡纳滨对虾和罗氏沼虾肠道微生物及抗生素抗性基因多样性分析. 水产学报, 2019, 43(5): 1347-1358] |
LAMBERT P A. Cellular impermeability and uptake of biocides and antibiotics in Gram-positive bacteria and mycobacteria. Journal of Applied Microbiology, 2002, 92(S1): 46S-54S |
LEE C, CHEN I, YANG Y, et al. The opportunistic marine pathogen Vibrio parahaemolyticus becomes virulent by acquiring a plasmid that expresses a deadly toxin. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(34): 10798-10803 DOI:10.1073/pnas.1503129112 |
LI Y, QU L Y, ZHU P F, et al. Distribution characteristics of antibiotic resistance bacteria and related resistance genes in mariculture area of Shandong. Marine Environmental Science, 2016, 35(1): 55-62 [李壹, 曲凌云, 朱鹏飞, 等. 山东地区海水养殖区常见抗生素耐药菌及耐药基因分布特征. 海洋环境科学, 2016, 35(1): 55-62] |
LI Z X, DONG X, WU M M, et al. Quinolone residues in seawater of aquaculture area, Sanggou Bay, Yellow Sea, China. Marine Environmental Science, 2018, 37(2): 182–186, 192 [李兆新, 董晓, 吴蒙蒙, 等. 黄海桑沟湾养殖区海水中喹诺酮类抗生素的残留状况. 海洋环境科学, 2018, 37(2): 182–186, 192] |
LIAN L L. Distribution of antibiotics in surface and core sediments from aquaculture areas. Master´s Thesis of Dalian University of Technology, 2016, 28–61 [连璐璐. 抗生素在滨海养殖区表层及柱状沉积物中的分布特征. 大连理工大学硕士研究生学位论文, 2016, 28–61]
|
LIU W L, XU H, TANG Y, et al. The effect of diet with Bacillus cereus biofilm on the growth rate, disease resistance and intestinal microflora of Litopenaeus vannamei. Progress in Fishery Sciences, 2017, 38(4): 87-95 [刘文亮, 许华, 唐杨, 等. 饲料中补充蜡样芽孢杆菌(Bacillus cereus)生物膜对凡纳滨对虾(Litopenaeus vannamei)生长、抗病力及其肠道微生物组成的影响. 渔业科学进展, 2017, 38(4): 87-95] |
MAZEL D. Integrons: Agents of bacterial evolution. Nature Reviews Microbiology, 2006, 4(8): 608-620 DOI:10.1038/nrmicro1462 |
MCDONNELL G, RUSSELL A D. Antiseptics and disinfectants: Activity, action, and resistance. Clinical Microbiology Reviews, 1999, 12(1): 147-179 DOI:10.1128/CMR.12.1.147 |
Ministry of Agriculture and Rural Affairs. Guidance on aquaculture drug application, No. 1, No. 2, 2020. 2020, 3–4 [农业农村部. 水产养殖用药明白纸2020年1、2号. 2020, 3–4]
|
NIU L, BA Y B, BAI F J, et al. Comparison of antimicrobial resistance of Vibrio parahaemolyticus isolated from different sources. Journal of Food Science and Biotechnology, 2019, 38(12): 9-16 [牛丽, 巴永兵, 白凤佳, 等. 不同来源副溶血性弧菌耐药情况比较. 食品与生物技术学报, 2019, 38(12): 9-16 DOI:10.3969/j.issn.1673-1689.2019.12.002] |
ÖSTERBLAD M, NORRDAHL K, KORPIMÄKI E, et al. How wild are wild mammals?. Nature, 2001, 409: 37-38 |
PEI R, KIM S C, CARLSON K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG). Water Research, 2006, 40(12): 2427-2435 DOI:10.1016/j.watres.2006.04.017 |
SHANG H, WANG Y S, SHEN Z Y. National guide for clinical laboratory procedures. Beijing: People's Medical Publishing House, 2015: 21-33 [尚红, 王毓三, 申子瑜. 全国临床检验操作规程. 北京: 人民卫生出版社, 2015: 21-33]
|
SU H C, LIU S, HU X J, et al. Occurrence and temporal variation of antibiotic resistance genes (ARGs) in shrimp aquaculture: ARGs dissemination from farming source to reared organisms. Science of the Total Environment, 2017, 607-608: 357-366 DOI:10.1016/j.scitotenv.2017.07.040 |
SUN S, KORHEINA D K A, FU H, et al. Chronic exposure to dietary antibiotics affects intestinal health and antibiotic resistance gene abundance in oriental river prawn (Macrobrachium nipponense), and provokes human health risk. Science of the Total Environment, 2020, 720: 137478 DOI:10.1016/j.scitotenv.2020.137478 |
WANG H P, YAN H, ZHAO J R, et al. Quantitative detection of six classes of antibiotic resistance and class Ⅰ integrin genes in aquatic products. Modern Food Science and Technology, 2017, 33(5): 270-276 [王慧平, 闫鹤, 赵俊仁, 等. 水产品中6类抗生素抗性基因和Ⅰ类整合子的定量检测. 现代食品科技, 2017, 33(5): 270-276] |
WANG Z F, LEI Y, XIAO J, et al. Residue status of antibiotics in aquaculture ponds of main tilapia aquaculture areas in Guangxi. Journal of Southern Agriculture, 2019, 50(4): 891-897 [王志芳, 雷燕, 肖俊, 等. 广西罗非鱼主产区养殖池塘抗生素残留状况分析. 南方农业学报, 2019, 50(4): 891-897 DOI:10.3969/j.issn.2095-1191.2019.04.29] |
WU D X. Studies of effects on Litopenaeus vannamei bacterial ecology caused by probiotics application and mechanism of interactin between microalgae and probiotics. Doctoral Dissertation of Huazhong Agricultural University, 2016, 40–72 [吴定心. 微生物制剂对南美白对虾养殖体系微生态的影响及其与藻类关系的研究. 华中农业大学博士研究生学位论文, 2016, 40–72]
|
YAN Q Q, LI B, LIAO M J, et al. Distribution characteristics of antibiotic resistant bacteria and antimicrobial resistant genes in the intestine of cultured sea cucumber (Apostichopus japonicus) seedlings in Shandong Province. Progress in Fishery Sciences, 2020, 41(4): 134-143 [闫倩倩, 李彬, 廖梅杰, 等. 山东主要刺参养殖区幼参肠道抗生素耐药菌及耐药基因分布特征. 渔业科学进展, 2020, 41(4): 134-143] |
ZHANG Q Y, ZHAO W W, WU W. Antibiotics resistance gene pollution and its research progress acheived in aquaculture environment. Journal of Agricultural Science and Technology, 2015, 17(6): 125-134 [张骞月, 赵婉婉, 吴伟. 水产养殖环境中抗生素抗性基因污染及其研究进展. 中国农业科技导报, 2015, 17(6): 125-134] |
ZHANG S J, ZHAO X J, SONG X L, et al. Analysis of the culturable bacteria's quantity and composition in the intestinal tract of cultivation shrimp. Journal of Shanghai Ocean University, 2015, 24(2): 211-218 [张盛静, 赵小金, 宋晓玲, 等. 人工养殖对虾肠道内可培养细菌数量及组成分析. 上海海洋大学学报, 2015, 24(2): 211-218] |
ZHANG Z, YANG Z Y, WANG Y G, et al. Effectiveness of garden burnet, Sanguisorba officinalic L., in controlling acute hepatopancreatic necrosis disease caused by infection of Vibrio parahaemolyticus in shrimp farming. Aquaculture, 2021, 531: 735875 DOI:10.1016/j.aquaculture.2020.735875 |
ZHAO S, LI J, MA L C, et al. Analysis of phenotype and genetype in quinolone resistance in Vibrio from mairculture. Marine Fisheries, 2019, 41(4): 463-471 [赵姝, 李健, 马立才, 等. 海水养殖动物源弧菌喹诺酮类药物耐药表型与基因型分析. 海洋渔业, 2019, 41(4): 463-471 DOI:10.3969/j.issn.1004-2490.2019.04.008] |
ZHU L D, CHEN K, XI B W, et al. In vitro antibacterial effect of fraxetin on pathogenic Aeromonas hydrophila. Journal of Fishery Sciences of China, 2019, 26(5): 984-992 [朱璐丹, 陈凯, 习丙文, 等. 秦皮素的抑菌作用及其对嗜水气单胞菌毒力的影响. 中国水产科学, 2019, 26(5): 984-992] |



