2. 南方海洋科学与工程广东省实验室(湛江) 广东 湛江 524025
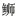 (Seriola dumerili) dnd的一致性最高(72.3%)。系统进化分析表明,Rcdnd与高体
(Seriola dumerili) dnd的一致性最高(72.3%)。系统进化分析表明,Rcdnd与高体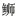 的同源蛋白亲缘关系最接近。基于半定量RT-PCR的组织表达分布分析结果显示,Rcdnd在性腺中特异表达,在其他组织中均无表达。实时荧光定量PCR(qRT-PCR)结果显示,在精巢发育过程(Ⅱ~Ⅴ期),Rcdnd的表达水平呈逐渐上升趋势;在卵巢发育过程(Ⅰ~Ⅲ期),Rcdnd的表达水平先显著升高后趋于稳定,在150 dph (Ⅱ期)时的表达量最高。原位杂交结果显示,不同发育时期性腺组织中,Rcdnd mRNA主要在生殖细胞中表达。在精巢中,Rcdnd mRNA集中表达于精原细胞和初级精母细胞的周缘,次级精母细胞中的杂交信号明显减弱,而精细胞和成熟精子中几乎检测不到杂交信号。在卵巢中,Rcdnd mRNA在卵原细胞中的表达最强,在初级卵母细胞中的表达较弱,其中,Ⅰ、Ⅱ和Ⅲ时相卵母细胞检测到的杂交信号强度无显著差异,且杂交信号均匀分布于细胞质中。研究结果表明,Rcdnd基因可能参与军曹鱼性腺发育过程,上述结果可为揭示军曹鱼两性生殖细胞发生发育的调控机理提供理论依据。
的同源蛋白亲缘关系最接近。基于半定量RT-PCR的组织表达分布分析结果显示,Rcdnd在性腺中特异表达,在其他组织中均无表达。实时荧光定量PCR(qRT-PCR)结果显示,在精巢发育过程(Ⅱ~Ⅴ期),Rcdnd的表达水平呈逐渐上升趋势;在卵巢发育过程(Ⅰ~Ⅲ期),Rcdnd的表达水平先显著升高后趋于稳定,在150 dph (Ⅱ期)时的表达量最高。原位杂交结果显示,不同发育时期性腺组织中,Rcdnd mRNA主要在生殖细胞中表达。在精巢中,Rcdnd mRNA集中表达于精原细胞和初级精母细胞的周缘,次级精母细胞中的杂交信号明显减弱,而精细胞和成熟精子中几乎检测不到杂交信号。在卵巢中,Rcdnd mRNA在卵原细胞中的表达最强,在初级卵母细胞中的表达较弱,其中,Ⅰ、Ⅱ和Ⅲ时相卵母细胞检测到的杂交信号强度无显著差异,且杂交信号均匀分布于细胞质中。研究结果表明,Rcdnd基因可能参与军曹鱼性腺发育过程,上述结果可为揭示军曹鱼两性生殖细胞发生发育的调控机理提供理论依据。2. Southern Marine Science and Engineering Guangdong Laboratory (Zhanjiang), Zhanjiang, Guangdong 524025, China
dnd基因最初鉴定于斑马鱼(Danio rerio)中(Weidinger et al, 2003),可编码一种进化上保守的RNA结合蛋白,作为脊椎动物生殖质的重要组分之一,特异表达于生殖系细胞中,并在生殖细胞的发生及发育过程中发挥着重要作用(Gross-Thebing et al, 2017)。基于此特性,dnd基因常作为分子标记物,广泛应用于鱼类生殖细胞的相关研究(程琳等, 2020)。目前,已在多种脊椎动物中克隆和鉴定出dnd的同源基因,包括小鼠(Mus musculus) (Bhattacharya et al, 2007)、红原鸡(Gallus gallus) (Aramaki et al, 2009)、非洲爪蟾(Xenopus laevis) (Horvay et al, 2006)、大西洋鳕鱼(Gadus morhua) (Škugor et al, 2014)、大西洋鲑(Salmo salar) (Wargelius et al, 2016)、泥鳅(Misgurnus anguillicaudatus) (Fujimoto et al, 2010)、银鲫(Carassius gibelio) (Li et al, 2016)等。
据报道,dnd基因的表达模式在不同物种中存在明显的差异,如dnd基因仅在小鼠精巢中特异表达(Bhattacharya et al, 2007);在非洲爪蟾中,dnd基因则特异表达于卵巢(Horvay et al, 2006);而大黄鱼(Larimichthys crocea) (陈仕海等, 2015)、大菱鲆(Scophthalmus maximus) (Lin et al, 2013)和牙鲆(Paralichthys olivaceus) (Wang et al, 2015) dnd基因均在性腺中特异表达,但在卵巢中的表达水平要高于精巢。关于dnd基因功能方面,小鼠dnd基因发生突变后,将抑制原始生殖细胞(PGCs)的产生,导致部分特定遗传背景的个体产生睾丸生殖细胞瘤(Youngren et al, 2005);抑制非洲爪蟾胚胎发育时期dnd基因的表达将直接导致其PGCs的缺失(Horvay et al, 2006);敲除斑马鱼dnd基因可引起PGCs的迁移发生异常,随后调亡,部分雌性个体甚至出现性逆转的现象(Weidinger et al, 2003)。以上研究结果表明,dnd基因在PGCs发生、迁移及性别分化中发挥着重要的调节作用。
军曹鱼(Rachycentron canadum)隶属鲈形目(Perciformes)、军曹鱼科(Rachycentridae)、军曹鱼属(Rachycentron),为广盐暖水性海水鱼类,俗称海鲡、海龙鱼,广泛分布于热带和亚热带海域(东太平洋除外) (Castellanos-Galindo et al, 2016),在中国南海部分海域亦有少量分布(陈刚等, 2004)。由于军曹鱼具有生长速率快、易于驯化、抗病力强、肉厚质地细腻等特点(Hamilton et al, 2013),近年来在南方沿海地区的养殖发展迅猛,随着人工繁殖和大规模育苗技术的突破,军曹鱼已成为我国极具前景的海水网箱养殖鱼类之一。
本研究克隆分析军曹鱼dnd (Rcdnd)基因cDNA全长序列,利用半定量RT-PCR和实时荧光定量PCR技术(qRT-PCR)检测Rcdnd的组织表达分布及其在不同发育期性腺组织中的表达水平,利用原位杂交技术初步分析Rcdnd mRNA在配子形成过程中的表达定位分布,旨在从分子水平阐明dnd基因在军曹鱼配子发生过程中的作用,同时也为进一步研究鱼类PGCs发生、迁移和分化的分子机理提供基础理论资料。
1 材料与方法 1.1 实验材料及处理实验用军曹鱼于2019年6月—2020年4月采自广东省茂名市电白县某室内养殖基地,军曹鱼幼鱼饲养于直径9 m、水深2.5 m的金属圆形水池内,流水充气,养殖盐度27.0~30.5,温度25.0℃~30.0℃,分别对90、120、150、185、210和360 dph体质良好的军曹鱼进行采样,其中,实验用雌鱼共23尾,体重215.0~5050.0 g,体长29.8~68.5 cm;雄鱼共18尾,体重230.0~4225.0 g,体长29.2~64.5 cm。
对随机采集的军曹鱼活体进行形态学指标的测量,随后立即解剖,取出性腺,鉴定其性别,将同一尾鱼的两叶性腺分开保存,其中一叶性腺于4℃在RNA Later中静置过夜,最后转移至–80℃超低温冰箱保存,用于qRT-PCR实验;另外一叶性腺从中间剪为两段,分别放入4%多聚甲醛(PFA)中,固定24 h后转入焦碳酸二乙酯(DEPC)配制的70%乙醇中,用于切片原位杂交实验。此外,选取150 dph军曹鱼雌雄各1尾,将其脑、肌肉、鳃、肝、肠、胃、脾、体肾、心、皮肤、眼睛、精巢和卵巢分离,经DEPC水冲洗后放入RNA Later中,–80℃保存待用。
1.2 Rcdnd基因全长cDNA的克隆将采集的军曹鱼卵巢放入液氮中研磨,采用Trizol法(Invitrogen)提取总RNA。通过SimpliNano超微量核酸蛋白测定仪检测总RNA的浓度和纯度,并以1.5%琼脂糖凝胶电泳检测其完整性。参考EasyScript One-step cDNA Synthesis试剂盒(TransGen)说明书,将2 μg总RNA反转录合成第一链cDNA,另取2 μg总RNA,按照SMARTer® RACE 5′/3′试剂盒(Clontech)说明书,合成5′/3′ RACE-Ready cDNA。
从课题组前期获得的军曹鱼全基因组数据库(暂未上传至NCBI数据库)中提取dnd基因的CDS序列信息,分别在序列两端设计上下游特异性引物Rcdnd-F和Rcdnd-R(表 1),PCR反应条件:95℃ 5 min;95℃ 30 s,67℃ 30 s,72℃ 1 min 20 s,共35个循环;72℃ 10 min,PCR产物经1.5%琼脂糖凝胶电泳检测,确定为目的片段后用胶回收试剂盒(TransGen)纯化回收,并将纯化产物连接到pMD-18T (TaKaRa)载体,进而转化至DH-5α感受态细胞,筛选挑取单克隆阳性菌落由生工生物工程(上海)股份有限公司测序。根据测序片段,分别在靠近序列5′和3′末端各设计2条RACE引物Rcdnd 5′-R1/R2和Rcdnd 3′-F1/F2 (表 1),采用巢式PCR获取Rcdnd基因5′和3′末端序列。
|
|
表 1 本研究所用引物 Tab.1 Primers used in this study |
使用NCBI数据库上的BLASTP对克隆所得Rcdnd氨基酸序列进行比对,下载其他硬骨鱼类及高等脊椎动物的dnd氨基酸序列,利用GenDoc进行氨基酸序列多重比对分析。利用MEGA 5.0软件,以邻接法(neighbour-joining, NJ)构建系统进化树,针对进化树各分支结点均进行1000次重复计算检验。
1.3 Rcvasa基因在各组织及性腺发育过程中的表达通过组织学观察并参照国内学者(刘筠, 1993)对鱼类性腺的分期方法,依据性腺中生殖细胞的种类、成熟度、数量占比及排列方式进行发育分期的划分,提取不同发育时期军曹鱼的性腺以及150 dph军曹鱼个体各组织的总RNA,按照反转录试剂盒说明书合成cDNA。基于测序获得的Rcdnd基因序列,设计一对特异性扩增引物Rcdnd-F1/R1,并以军曹鱼β-actin作为内参基因(表 1)。PCR反应条件:94℃ 5 min;94℃ 30 s,62℃ 30 s,72 ℃ 20 s,35个循环;72℃ 10 min。通过琼脂糖凝胶电泳检测Rcdnd在不同组织中的半定量RT-PCR结果,利用Tanon 4100凝胶成像系统采集图像。qRT-PCR实验流程按照SYBR® Premix Ex TaqTM试剂盒(TaKaRa)说明书进行操作,使用ABI 7500型实时荧光定量PCR仪检测Rcdnd在不同发育时期精巢和卵巢中的表达,每个实验样品设置3个重复。根据测得的Ct值,采用2–ΔΔCt法(Schmittgen et al, 2008)计算Rcdnd的相对表达量。所得数据结果均以平均值±标准差(Mean±SD, n=3)表示,利用统计学软件SPSS 19.0进行单因素方差分析(one-way ANOVA)及Duncan´s多重比较,P < 0.05表示有显著性差异。
1.4 性腺组织原位杂交根据已获得的Rcdnd基因序列,利用Primer Premier 6.0软件设计具有特异性的寡核苷酸探针序列(表 1),探针的5′和3′端用地高辛(DIG)标记,由武汉赛维尔生物科技有限公司合成。
将石蜡包埋的120、210和360 dph军曹鱼精巢和卵巢样品进行组织切片(5~6 μm),具体杂交过程综合参考相关研究中的方法(李晓妮等, 2017; 史宝等, 2017)。滴加BCIP/NBT显色液进行化学显色,显微观察阳性后再滴加核固红染核,中性树胶封片,最后显微镜检,采集图像分析。
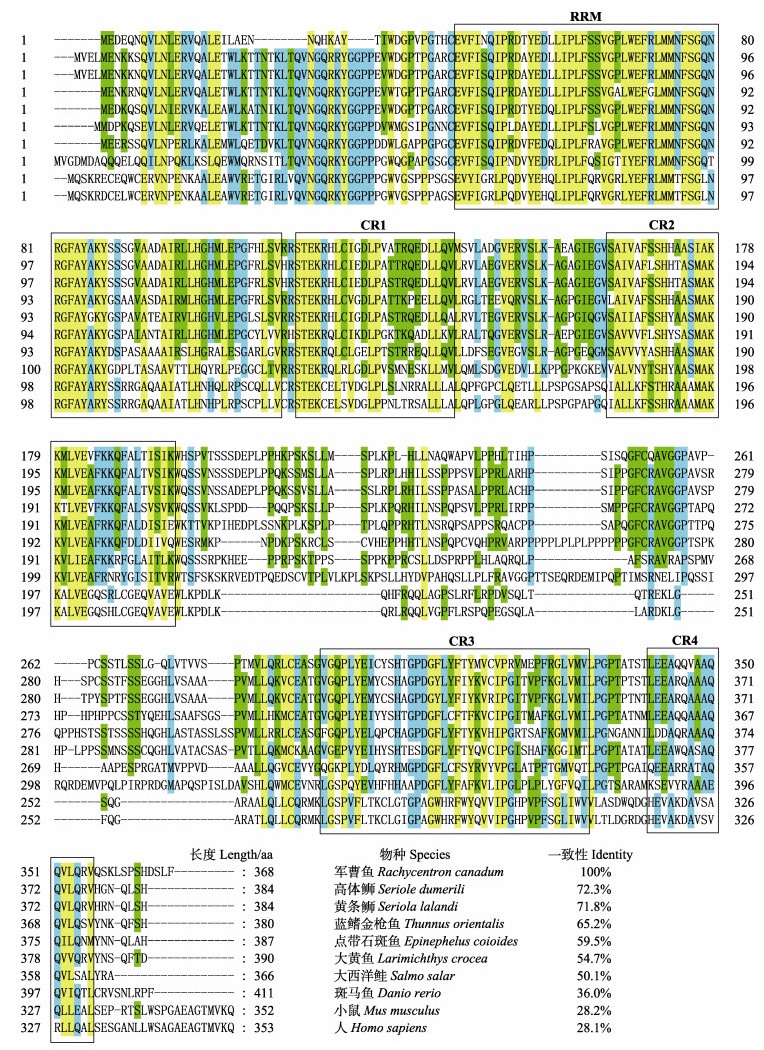
2 结果与分析 2.1 Rcdnd cDNA全长及序列分析实验克隆所得的Rcdnd序列全长1339 bp,其中,5′非编码区59 bp,3′非编码区173 bp,开放阅读框(ORF) 1107 bp,共编码368个氨基酸,已提交NCBI GenBank (登录号: MW436697);预测的编码蛋白的分子质量为40.36 kDa,等电点(pI)为6.30。蛋白结构域预测结果显示,在推导的Rcdnd氨基酸序列中,42~114 aa为一段RNA结合蛋白保守的RNA识别基序(RRM) (图 1)。此外,通过氨基酸序列多重比对分析发现,Rcdnd氨基酸序列还具有4个C端保守结构域(CR1~4),同时发现,军曹鱼dnd氨基酸序列与鲹科(Carangidae)鱼类的一致性最高,如高体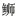
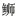

|
图 1 军曹鱼dnd cDNA全长序列和氨基酸序列分析 Fig.1 Complete sequence of R. canadum dnd and analysis of deduced amino acid sequence 下划线指示起始密码子和终止密码子,粗体斜体指示加尾信号,灰色阴影指示RRM识别基序 The initiation codon ATG and stop codon TAA are underlined. The polyadenylation signal AATTAAA are in bold and italic. The RNA recognition motif (RRM) are highlighted in grey shadow |
|
图 2 军曹鱼dnd氨基酸序列的多重比对 Fig.2 Multiple alignment of R. canadum dnd deduced amino acid sequence 方框表示5个氨基酸特异性保守结构域 The frame regions indicate the five amino acid specific conserved regions |
基于军曹鱼与GenBank数据库中已发表的其他物种dnd氨基酸序列所构建的系统进化树中(图 3),硬骨鱼类均聚为一簇,而两栖类、鸟类和哺乳类等高等脊椎动物则聚为另一簇,其中,军曹鱼与高体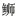

|
图 3 基于dnd氨基酸序列构建的系统进化树(NJ树) Fig.3 Phylogenetic tree of dnd amino acid sequences based on neighbor-joining (NJ) method Bootstrap检验的重复次数为1000次,标尺0.05为进化距离 The tree is based on a 1000 bootstrap procedure, the scale bar 0.05 in terms of genetic distance is indicated below the tree |
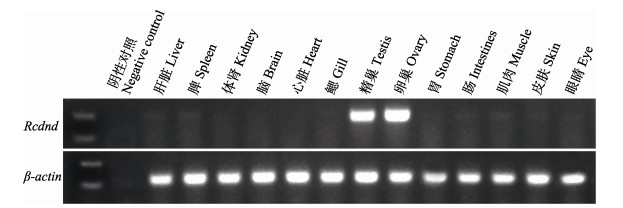
采用半定量RT-PCR检测了Rcdnd在150 dph军曹鱼各组织中的表达水平,结果表明,Rcdnd在性腺中特异表达,且卵巢和精巢中均检测到较高的表达水平,在性腺外的其他组织中均无表达(图 4)。
|
图 4 RT-PCR分析Rcdnd在军曹鱼不同组织中的表达 Fig.4 Tissue distribution of dnd expression in R. canadum by RT-PCR |
在精巢发育过程中,Rcdnd的表达水平呈逐渐上升趋势,其在90 dph (Ⅱ期)的表达量最低,120 dph (Ⅱ~Ⅲ期)时表达量无显著变化,而在150 dph (Ⅲ期)~ 360 dph (Ⅴ期)的表达量均出现显著升高,其中,185 dph (Ⅲ期)时表达量上升至90 dph的2.68倍,210 dph (Ⅳ期)的表达量与185 dph (Ⅲ期)无显著差异,而360 dph (Ⅴ期)时表达量进一步显著升高并达到最大值,约为90 dph的3.33倍(图 5A)。

|
图 5 军曹鱼性腺周年发育过程中Rcdnd mRNA的表达水平 Fig.5 Rcdnd mRNA expression level in annual gonadal development of R. canadum 上标不同字母表示差异显著(P < 0.05) Different superscripts indicate significant difference (P < 0.05) |
卵巢发育过程中,Rcdnd的表达水平先显著升高后趋于平稳,90 dph (Ⅰ期)时的表达量最低,随后在120 dph (Ⅰ~Ⅱ期)~360 dph (Ⅲ期)的表达量均显著高于90 dph,其中,150 dph表达量上升至最大值,约为90 dph的2.25倍,185 dph (Ⅱ期)和210 dph (Ⅱ期)的表达量均较150 dph显著降低,而185、210和360 dph发育时间点的相对表达量无显著差异(图 5B)。
2.4 Rcdnd在性腺发育过程中的表达定位利用切片原位杂交技术检测Rcdnd mRNA在军曹鱼不同发育期性腺中的表达定位,结果显示,120 dph军曹鱼的精巢中,Rcdnd mRNA的杂交信号主要分布于精原细胞、初级精母细胞和次级精母细胞的周缘,其中,在精原细胞和初级精母细胞中检测到的杂交信号最强,次级精母细胞中的杂交信号明显减弱,而精细胞中几乎检测不到杂交信号(图 6A1)。Rcdnd mRNA在210 dph和360 dph军曹鱼精巢中的表达定位模式与120 dph相似,杂交信号主要分布于精子形成过程中的精原细胞和精母细胞,在精细胞和成熟精子中的表达极其微弱(图 6B1、C1)。
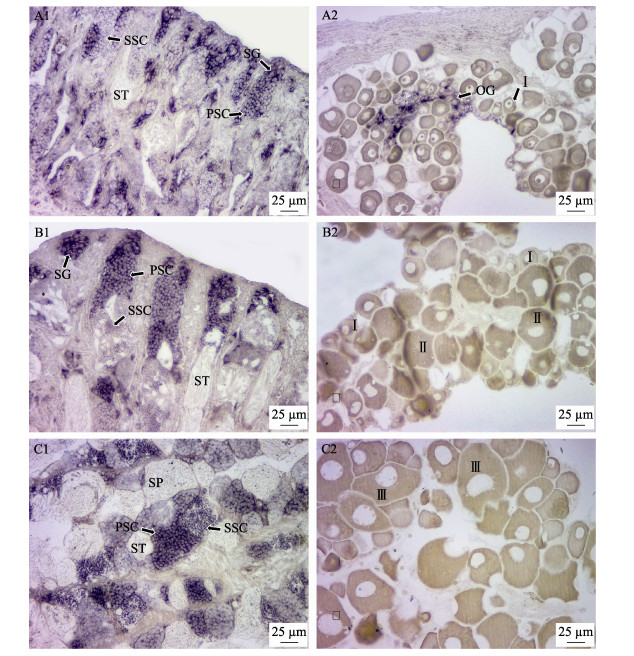
|
图 6 原位杂交法分析不同发育期性腺中Rcdnd mRNA的定位 Fig.6 Distribution of Rcdnd mRNA in different developmental stages of gonads by chemical in situ hybridization (CISH) A1、B1和C1:120 dph、210 dph和360 dph精巢切片;A2、B2和C2:120 dph、210 dph和360 dph卵巢切片SG:精原细胞;PSC:初级精母细胞;SSC:次级精母细胞;ST:精细胞;SP:精子;OG:卵原细胞;Ⅰ:第Ⅰ时相卵母细胞;Ⅱ:第Ⅱ时相卵母细胞;Ⅲ:第Ⅲ时相卵母细胞 A1, B1 and C1: Sections of testis at 120 dph, 210 dph and 360 dph; A2, B2 and C2: Sections of ovary at 120 dph, 210 dph and 360 dph SG: Spermatogonia; PSC: Primary spermatocyte; SSC: Secondary spermatocyte; ST: Spermatid; SP: Spermatozoa; OG: Oogonium; Ⅰ: Oocyte at Stage Ⅰ; Ⅱ: Oocyte at Stage Ⅱ; Ⅲ: Oocyte at Stage Ⅱ |
在卵巢发育过程中,Rcdnd mRNA主要在生殖细胞中表达,杂交信号均匀分布于细胞质及核仁内。Rcdnd mRNA在卵原细胞中的表达最强,而在初级卵母细胞中的表达相对较弱,且随着卵母细胞的生长发育,在第Ⅰ、Ⅱ和Ⅲ时相卵母细胞中检测到的杂交信号强度无明显变化(图 6A1、B1和C1)。
3 讨论本研究首次克隆了军曹鱼dnd基因的cDNA序列,其全长为1339 bp,编码368个氨基酸,推导的氨基酸序列与鲹科鱼类的一致性较高,和高体
基于半定量RT-PCR的组织表达分析结果显示,Rcdnd基因在精巢和卵巢中特异表达,这与大菱鲆(Lin et al, 2013)、牙鲆(Wang et al, 2015)、微鲤(Paedocypris progenetica) (陈克让等, 2018)、尼罗罗非鱼(Oreochromis niloticus) (Jin et al, 2019)、稀有
本研究采集的军曹鱼精巢样品包含了性腺发育分期的Ⅱ~Ⅴ期,其中,90 dph时,精巢处于精母细胞增长期(Ⅱ期);120 dph时,精巢处于Ⅱ~Ⅲ期;150 dph和185 dph时,精巢均处于精母细胞成熟期(Ⅲ期),而210和360 dph精巢分别处于精子细胞变态期(Ⅳ期)和精子成熟期(Ⅴ期)。这些样品中,Rcdnd的表达量随着精巢的发育呈逐渐上升趋势,在精子成熟期达到最大值,表明Rcdnd在军曹鱼精子发生过程中发挥一定的调控作用,这与大菱鲆(Lin et al, 2013)和蓝鳍金枪鱼(Yazawa et al, 2013)的研究结果相似。采集的卵巢样品包含3个发育分期,其中,90 dph卵巢属于卵原细胞增殖期(Ⅰ期),120 dph卵巢处于Ⅰ~Ⅱ期,150、185和210 dph卵巢均属于卵母细胞小生长期(Ⅱ期),360 dph卵巢发育至初级卵母细胞大生长期(Ⅲ期)。Rcdnd表达量在卵原细胞增殖期最低,随着卵巢的发育,Rcdnd表达量显著升高并趋于稳定,卵巢由Ⅰ期发育至Ⅲ期的过程中,卵母细胞通过积累糖类、蛋白质、核酸等原生质,细胞体积不断增大(Nishimura et al, 2014),因此,Rcdnd在卵巢发育过程中较高的表达水平可能与生殖细胞原生质的积累密切相关。此外,大菱鲆卵巢发育过程中,dnd基因的表达量在由Ⅰ期发育至Ⅱ期时出现显著的升高,但随着卵巢进一步发育至Ⅲ、Ⅳ期,其表达量呈下降趋势(Lin et al, 2013),这与dnd基因在军曹鱼卵巢发育过程中的表达模式存在一定差异,由此推测,dnd基因在卵巢发育过程中的表达模式存在物种差异性。
性腺切片原位杂交结果进一步证实,Rcdnd mRNA主要在生殖细胞中表达,而在性腺体细胞中的表达极微弱。Rcdnd mRNA在精巢发育过程中的表达定位与大菱鲆(Lin et al, 2013)、稀有
ARAMAKI S, KUBOTA K, SOH T, et al. Chicken dead end homologue protein is a nucleoprotein of germ cells including primordial germ cells. Journal of Reproduction and Development, 2009, 55(2): 214-218 DOI:10.1262/jrd.20154 |
BHATTACHARYA C, AGGARWAL S, ZHU R, et al. The mouse dead end gene isoform α is necessary for germ cell and embryonic viability. Biochemical and Biophysical Research Communications, 2007, 355(1): 194-199 DOI:10.1016/j.bbrc.2007.01.138 |
CASTELLANOS-GALINDO G A, BAOS R, ZAPATA L A. Mariculture-induced introduction of cobia Rachycentron canadum (Linnaeus, 1766), a large predatory fish, in the tropical eastern Pacific. BioInvasions Records, 2016, 5(1): 55-58 DOI:10.3391/bir.2016.5.1.10 |
CHEN G, ZHANG J D, YE N, et al. Introduction of culture technology of cobia, Rachycentron canadum(Ⅰ). Scientific Fish Farming, 2004(1): 10-11 [陈刚, 张健东, 叶宁, 等. 军曹鱼的养殖技术介绍(上). 科学养鱼, 2004(1): 10-11] |
CHEN K R, DING T Y, LI M Y. Cloning and expression analysis of dazl and dnd genes in minifish. Genomics and Applied Biology, 2018, 37(7): 2795-2803 [陈克让, 丁天宜, 李名友. Minifish dazl和dnd基因的克隆及表达分析. 基因组学与应用生物学, 2018, 37(7): 2795-2803] |
CHEN S H, CAI M Y, ZHANG Z P, et al. Preliminary studies on specification and development of the primordial germ cells from large yellowcroaker by dnd gene. Journal of Fisheries of China, 2015, 39(9): 1273-1282 [陈仕海, 蔡明夷, 张子平, 等. 基于dnd基因标记的大黄鱼原始生殖细胞发生发育的初步研究. 水产学报, 2015, 39(9): 1273-1282] |
CHENG L, HUANG T Q, LIU C B, et al. Research perspectives: Marker genes of primordial germ cells in fishes. Chinese Journal of Fisheries, 2020, 33(6): 80-88 [程琳, 黄天晴, 刘晨斌, 等. 鱼类原始生殖细胞标记基因研究进展. 水产学杂志, 2020, 33(6): 80-88 DOI:10.3969/j.issn.1005-3832.2020.06.015] |
DUAN J D, FENG G Q, CHANG P, et al. Germ cell-specific expression of dead end (dnd) in rare minnow (Gobiocypris rarus). Fish Physiology and Biochemistry, 2015, 41(2): 561-571 DOI:10.1007/s10695-015-0029-x |
FUJIMOTO T, NISHIMURA T, GOTO-KAZETO R, et al. Sexual dimorphism of gonadal structure and gene expression in germ cell-deficient loach, a teleost fish. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(40): 17211-17216 DOI:10.1073/pnas.1007032107 |
GROSS-THEBING T, YIGIT S, PFEIFFER J, et al. The vertebrate protein Dead End maintains primordial germ cell fate by inhibiting somatic differentiation. Development Cell, 2017, 43(6): 704-715 DOI:10.1016/j.devcel.2017.11.019 |
HAMILTON S, SEVERI W, CAVALLI R O. Biology and aquaculture of cobia: A review. Boletim do Instituto de Pesca, 2013, 39(4): 461-477 |
HONG N, LI M Y, YUAN Y M, et al. Dnd is a critical specifier of primordial germ cells in the medaka fish. Stem Cell Reports, 2016, 6(3): 411-421 DOI:10.1016/j.stemcr.2016.01.002 |
HORVAY K, CLAUBEN M, KATZER M, et al. Xenopus Dead end mRNA is a localized maternal determinant that serves a conserved function in germ cell development. Developmental Biology, 2006, 291(1): 1-11 DOI:10.1016/j.ydbio.2005.06.013 |
JIN Y H, DAVIE A, MIGAUD H. Expression pattern of nanos, piwil, dnd, vasa and pum genes during ontogenic development in Nile tilapia Oreochromis niloticus. Gene, 2019, 688: 62-70 DOI:10.1016/j.gene.2018.11.078 |
LI S Z, LIU W, LI Z, et al. Molecular characterization and expression pattern of a germ cell marker gene dnd in gibel carp (Carassius gibelio). Gene, 2016, 591(1): 183-190 DOI:10.1016/j.gene.2016.07.027 |
LI X N, LIU X Z, SHI B, et al. The expression patterns of membrane progestin receptor α (mPRα) during oocytes maturation in Cynoglossus semilaevis. Progress in Fishery Sciences, 2017, 38(1): 25-33 [李晓妮, 柳学周, 史宝, 等. 膜孕激素受体(mPRα)在半滑舌鳎(Cynoglossus semilaevis)卵母细胞成熟过程中的表达特征. 渔业科学进展, 2017, 38(1): 25-33] |
LIN F, ZHAO C Y, XU S H, et al. Germline-specific and sexually dimorphic expression of a dead end gene homologue in turbot (Scophthalmus maximus). Theriogenology, 2013, 80(6): 665-672 DOI:10.1016/j.theriogenology.2013.06.016 |
LIU C B, XU G F, HUANG T Q, et al. A review of research progress on gonadal development in fish. Chinese Journal of Fisheries, 2019, 32(1): 46-54 [刘晨斌, 徐革锋, 黄天晴, 等. 鱼类性腺发育研究进展. 水产学杂志, 2019, 32(1): 46-54 DOI:10.3969/j.issn.1005-3832.2019.01.009] |
LIU L X, HONG N, XU H Y, et al. Medaka dead end encodes a cytoplasmic protein and identifies embryonic and adult germ cells. Gene Expression Patterns, 2009, 9(7): 541-548 DOI:10.1016/j.gep.2009.06.008 |
LIU Y. Propagation physiology of main cultivated fish in China. Beijing: Agricultural Publishing Press, 1993: 20-42 [刘筠. 中国养殖鱼类繁育生理学. 北京: 农业出版社, 1993: 20-42]
|
NISHIMURA T, TANAKA M. Gonadal development in fish. Sexual Development, 2014, 8(5): 252-261 DOI:10.1159/000364924 |
SCHMITTGEN T D, LIVAK K J. Analyzing realtime PCR data by the comparative C(T) method. Nature Protocols, 2008, 3(6): 1101-1108 DOI:10.1038/nprot.2008.73 |
SHI B, LIU X Z, XU T, et al. Expression characterization of the novel membrane progestin receptor (mPR-Like) gene during the oocyte maturation of half-smooth tongue sole (Cynoglossus semilaevis). Progress in Fishery Sciences, 2017, 38(1): 10-17 [史宝, 柳学周, 徐涛, 等. 半滑舌鳎(Cynoglossus semilaevis)新型膜孕激素受体基因(mPRL)在卵母细胞成熟过程中的表达特征. 渔业科学进展, 2017, 38(1): 10-17] |
ŠKUGOR A, TVEITEN H, KRASNOV A, et al. Knockdown of the germ cell factor Dead end induces multiple transcriptional changes in Atlantic cod (Gadus morhua) hatchlings. Animal Reproduction Science, 2014, 144(3/4): 129-137 |
WANG X Y, LIU Q H, XIAO Y S, et al. The dnd RNA identifies germ cell origin and migration in olive flounder (Paralichthys olivaceus). BioMed Research International, 2015, 428591 |
WARGELIUS A, LEININGER S, SKAFTNESMO K O, et al. Dnd knockout ablates germ cells and demonstrates germ cell independent sex differentiation in Atlantic salmon. Scientific Reports, 2016, 6: 21284 DOI:10.1038/srep21284 |
WEIDINGER G, JÜRG S, SLANCHEV K, et al. Dead end, a novel vertebrate germ plasm component, is required for zebrafish primordial germ cell migration and survival. Current Biology, 2003, 13(16): 1429-1434 DOI:10.1016/S0960-9822(03)00537-2 |
YANG X G, YUE H M, YE H, et al. Identification of a germ cell marker gene, the dead end homologue, in Chinese sturgeon Acipenser sinensis. Gene, 2015, 558(1): 118-125 DOI:10.1016/j.gene.2014.12.059 |
YAZAWA R, TAKEUCHI Y, MORITA T, et al. The Pacific bluefin tuna (Thunnus orientalis) dead end gene is suitable as a specific molecular marker of type A spermatogonia. Molecular Reproduction and Development, 2013, 80(10): 871-880 DOI:10.1002/mrd.22224 |
YOUNGREN K, COVENEY D, PENG X, et al. The Ter mutation in the dead end gene causes germ cell loss and testicular germ cell tumours. Nature, 2005, 435(7040): 360-364 DOI:10.1038/nature03595 |
ZHANG H W, WANG Z R, ZHANG S C. Developmental Biology. Beijing: Higher Education Press, 2006: 399-407 [张红卫, 王子仁, 张士璀. 发育生物学. 北京: 高等教育出版社, 2006: 399-407]
|



