 全基因组微卫星分布特征分析
全基因组微卫星分布特征分析
 (Ictalurus punctatus)全基因组中完整型微卫星的分布特征,本研究使用生物信息学软件MISA对其全基因组微卫星进行了搜索并分析。结果显示,在斑点叉尾
(Ictalurus punctatus)全基因组中完整型微卫星的分布特征,本研究使用生物信息学软件MISA对其全基因组微卫星进行了搜索并分析。结果显示,在斑点叉尾 29条染色体中,筛选到完整型微卫星共510 256个,总长度达11 036 941 bp。其中,微卫星数量最多的是2号染色体(25 284个),其次分别是3号、1号和5号染色体,29号染色体的微卫星数量最少,只有11 591个。每条染色体长度与位于其上的微卫星的数量均显著相关(SPSS, r=0.98, P < 0.01)。27号染色体的微卫星相对丰度最大(785.03个/Mb),11号染色体的微卫星相对丰度最小(615.89个/Mb)。6种重复拷贝类型的微卫星中,单碱基的数量最多,占总数的45.31%,随后依次是二碱基(38.53%)、三碱基(8.73%)、四碱基(6.93%)、五碱基(0.46%)和六碱基(0.04%)。斑点叉尾
29条染色体中,筛选到完整型微卫星共510 256个,总长度达11 036 941 bp。其中,微卫星数量最多的是2号染色体(25 284个),其次分别是3号、1号和5号染色体,29号染色体的微卫星数量最少,只有11 591个。每条染色体长度与位于其上的微卫星的数量均显著相关(SPSS, r=0.98, P < 0.01)。27号染色体的微卫星相对丰度最大(785.03个/Mb),11号染色体的微卫星相对丰度最小(615.89个/Mb)。6种重复拷贝类型的微卫星中,单碱基的数量最多,占总数的45.31%,随后依次是二碱基(38.53%)、三碱基(8.73%)、四碱基(6.93%)、五碱基(0.46%)和六碱基(0.04%)。斑点叉尾 全基因组微卫星中,前10种优势重复拷贝类别为A、AC、AG、AT、AAT、AAAT、C、AAC、AAAC和AAG,表现出明显的A、T碱基优势。本研究结果可为进一步研究斑点叉尾
全基因组微卫星中,前10种优势重复拷贝类别为A、AC、AG、AT、AAT、AAAT、C、AAC、AAAC和AAG,表现出明显的A、T碱基优势。本研究结果可为进一步研究斑点叉尾 全基因组特征提供参考,并为今后进行斑点叉尾
全基因组特征提供参考,并为今后进行斑点叉尾 分子标记辅助育种及遗传信息评估等工作提供基础资料。
分子标记辅助育种及遗传信息评估等工作提供基础资料。 全基因组 微卫星 分布特征
全基因组 微卫星 分布特征 微卫星又称简单序列重复(simple sequence repeats, SSRs),广泛分布于真核、原核生物以及病毒中,是由1~6 bp碱基为单元重复串联而成的DNA序列。根据核心序列的排列差异,又分为完整型和不完整型(Morgante et al, 2001)。其中,完整型指重复序列中不存在其他碱基或重复序列的情况,不完整型指重复序列中存在错配情况。目前,对微卫星的研究主要集中于完整型(郑燕等, 2012),因其具有多态性高、共显性遗传等特点,目前被广泛应用于遗传图谱构建(Shen et al, 2007; Xia et al, 2010)、遗传多样性分析和种质资源保护(Narasimhamoorthy et al, 2008)等研究中。
斑点叉尾
近年来,由于远离原产地多年、亲本数量有限等原因,无可避免地对国内的斑点叉尾





斑点叉尾 全基因组数据从NCBI Genome数据库(https://www.ncbi.nlm.nih.gov/genome/?term=Ictalurus+punctatus)下载,登录号为GCA_004006655.3,以FASTA格式文件保存,共包含29条染色体,总大小为11 036 941 bp。
全基因组数据从NCBI Genome数据库(https://www.ncbi.nlm.nih.gov/genome/?term=Ictalurus+punctatus)下载,登录号为GCA_004006655.3,以FASTA格式文件保存,共包含29条染色体,总大小为11 036 941 bp。
利用微卫星搜索软件MISA (http://pgrc.ipk-gatersleben.de/misa/)对斑点叉尾 全基因组中完整型微卫星序列进行搜索和统计。本研究统计标准设置为MISA默认参数,经过前期的大量研究可知,以此标准搜索全基因组微卫星得出的结果最优(徐杰杰等, 2020),即单碱基重复序列最小拷贝数设置为10,二碱基重复序列最小拷贝数设置为6,三、四、五、六碱基重复序列最小拷贝数设置为5。考虑碱基互补配对原则和碱基排列顺序的差异,对可聚为同一类型的微卫星进行兼并统计。
全基因组中完整型微卫星序列进行搜索和统计。本研究统计标准设置为MISA默认参数,经过前期的大量研究可知,以此标准搜索全基因组微卫星得出的结果最优(徐杰杰等, 2020),即单碱基重复序列最小拷贝数设置为10,二碱基重复序列最小拷贝数设置为6,三、四、五、六碱基重复序列最小拷贝数设置为5。考虑碱基互补配对原则和碱基排列顺序的差异,对可聚为同一类型的微卫星进行兼并统计。

从斑点叉尾


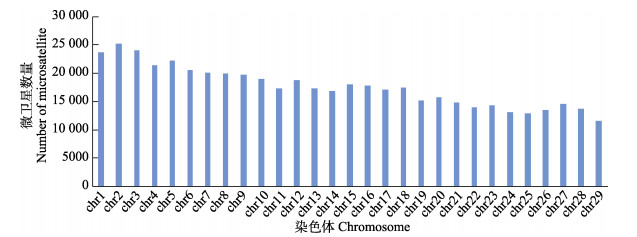
|
图 1 斑点叉尾 |
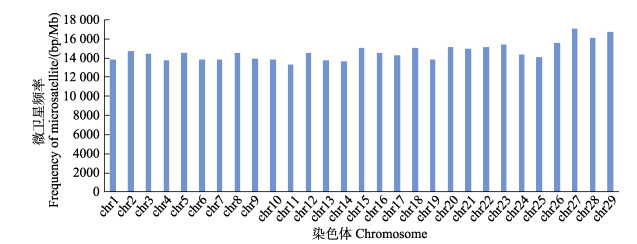
|
图 2 斑点叉尾 |

通过统计分析可知,斑点叉尾

|
|
表 1 斑点叉尾 |

|
图 3 斑点叉尾 |

在斑点叉尾

斑点叉尾
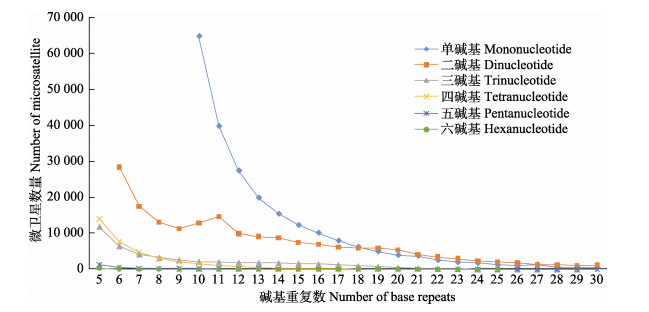
|
图 4 斑点叉尾 |

微卫星标记是一种应用广泛的分子标记技术,斑点叉尾




在本研究中,染色体DNA序列的长度与染色体上所含有的微卫星数量具有高度相关性(SPSS, r=0.98, P < 0.01),这与黄杰等(2012)和戚文华等(2013)的研究结果一致。同时也支持了Hancock (1996)的假说:染色体序列越长,微卫星含量越高的趋势。统计各染色体上的各种类型微卫星数量,发现各类型的比例除27号和29号染色体二碱基大于单碱基外,均符合29条染色体整体微卫星分布特征,即单碱基最多,其次是二碱基、三碱基、四碱基、五碱基和六碱基。这个规律与张琳琳等(2008)对赤拟谷盗(Tribolium castaneum)的研究结果相似,而与黄杰等(2012)对红原鸡(Gallus gallus)各染色体微卫星分布情况的研究结果相差甚远。高焕等(2005)认为,同一物种的不同染色体上,各种类型的重复序列分布有很大差异。而本研究的结果表明,各种类型的微卫星在各染色体上的分布符合整体规律,这可能和不同物种的不同类型微卫星的特定功能有关系。
|
|
表 2 斑点叉尾 |
斑点叉尾

不同物种的全基因组中重复序列不仅不同类型分布差异很大,同一类型不同类别也存在很大差异。斑点叉尾

单碱基类型微卫星以A类别为主,这与已公布的绝大多数生物的全基因组微卫星单碱基类型特征相符合,如食蟹猴(Macaca fascicularis)(涂飞云等, 2018)、红尾蚺(Boa constricto)(聂虎等, 2017)等。二碱基类型以AC所占比例最高,与大鼠(涂飞云等, 2015)、小鼠(Mus musculus)(童晓玲等, 2006)的二碱基类型的优势类别一致,而与德国小蠊(王晨等, 2015)、红原鸡(黄杰等, 2012)以AT碱基类别占优势的结果并不相同。Bachtrog等(2000)曾用6个黑腹果蝇(Drosophila melanogaster)群体作为研究对象,得出二碱基重复类型中不同重复类别的微卫星突变率不同的结论,表明在黑腹果蝇基因组微卫星中AC类别相对突变率比AT要大得多。而在斑点叉尾


三碱基类型中,数目最多的是AAT类别,其次是AAC和AAG,这与人的三碱基重复类型的前3种类别是一样的(Subramanian et al, 2003)。研究表明,某些三碱基重复类别可以形成特殊的结构,如发夹结构、三叶草结构和滑链结构等,这些结构非常的灵活,可能导致它们的遗传不稳定性(Pearson et al, 1996),此外,这些结构对基因的表达也起着十分重要的调控作用(戚文华等, 2016)。研究发现,人类的一些遗传疾病,如脆X综合征、肌强制性营养不良等与基因组中某些三碱基重复序列的增加有关(Schlötterer, 1998; Kovtun et al, 2001),而斑点叉尾

四碱基、五碱基的数量较多的前2种类别分别是AAAT、AAAC和ATAAT、AAAAC,六碱基的数量相对于前5种重复类型比较少,数量最多的2种类别是TGACTA和ATAGTC。与前3种碱基类型一样,四碱基、五碱基和六碱基也表现出明显的A/T碱基优势。有研究表明,DNA序列中G/C含量越高,微卫星分布越少。对于这个现象,倪守胜等(2018)通过分析虾夷扇贝(Patinopecten yessoensis)基因组的微卫星分布特征,得出DNA复制滑动机制和重组机制使得A/T重复类型的产生几率更高的结论。Schorderet等(1992)认为,由于基因组中CpG甲基化,胞嘧啶C容易在脱氨基作用下突变为胸腺嘧啶T。这可能是二碱基中CG含量较少、TG (AC类型)含量较多的原因。然而Stallings (1992)的研究则表明,无论是否有CpG甲基化过程,基因组中的CG重复类别都是偏小的,他提出CpG结构是一种有害结构。
3.3 斑点叉尾
斑点叉尾
综上所述,本研究使用软件MISA对斑点叉尾



BACHTROG D, AGIS M, IMHOF M, et al. Microsatellite variability differs between dinucleotide repeat motifs: Evidence from Drosophila melanogaster. Molecular Biology and Evolution, 2000, 17(9): 1277-1285 DOI:10.1093/oxfordjournals.molbev.a026411 |
BAI C C, LIU S F, ZHANG Z M. Characteristic analysis of microsatellite DNA in the genome of Gobiidae. Progress in Fishery Sciences, 2016, 37(5): 9-15 [白翠翠, 柳淑芳, 庄志猛. 虾虎鱼科(Gobiidae)基因组微卫星DNA的分布特征. 渔业科学进展, 2016, 37(5): 9-15] |
ELLEGREN H. Heterogeneous mutation processes in human microsatellite DNA sequences. Nature Genetics, 2000, 24(4): 400-402 DOI:10.1038/74249 |
GAO H, KONG J. Distribution characteristics and biological function of tandem repeat sequences in the genomes of different organisms. Zoological Research, 2005, 26(5): 555-564 [高焕, 孔杰. 串联重复序列的物种差异及其生物功能. 动物学研究, 2005, 26(5): 555-564 DOI:10.3321/j.issn:0254-5853.2005.05.017] |
HANCOCK J M. Simple sequences and the expanding genome. BioEssays, 1996, 18(5): 421-425 DOI:10.1002/bies.950180512 |
HE C B, CHEN S J, GAO X G, et al. Genetic diversity analysis of channel catfish (Ictalurus punctatus) germplasms using EST-SSR markers. Journal of Agricultural Biotechnology, 2008, 16(5): 815-823 [赫崇波, 陈姝君, 高祥刚, 等. EST-SSR标记对我国斑点叉尾  种质资源的研究. 农业生物技术学报, 2008, 16(5): 815-823 DOI:10.3969/j.issn.1674-7968.2008.05.016] 种质资源的研究. 农业生物技术学报, 2008, 16(5): 815-823 DOI:10.3969/j.issn.1674-7968.2008.05.016] |
HUANG J, DU L M, LI Y Z, et al. Distribution regularities of microsatellites in the Gallus gallus genome. Sichuan Journal of Zoology, 2012, 31(3): 358-363 [黄杰, 杜联明, 李玉芝, 等. 红原鸡全基因组中微卫星分布规律研究. 四川动物, 2012, 31(3): 358-363] |
HUANG J, ZHOU Y, LIU Y Z, et al. Characteristics of microsatellites in Arborophila rufipectus genome sequences using 454 GS FLX. Sichuan Journal of Zoology, 2015, 34(1): 8-14 [黄杰, 周瑜, 刘与之, 等. 基于454 GS FLX高通量测序的四川山鹧鸪基因组微卫星特征分析. 四川动物, 2015, 34(1): 8-14] |
KATTI M V, RANJEKAR P K, GUPTA V S. Differential distribution of simple sequence repeats in eukaryotic genome sequences. Molecular Biology and Evolution, 2001, 18(7): 1161-1167 DOI:10.1093/oxfordjournals.molbev.a003903 |
KOVTUN I V, GOELLNER G, MCMURRAY C T. Structural features of trinucleotide repeats associated with DNA expansion. Biochemistry and Cell Biology, 2001, 79(3): 325-336 DOI:10.1139/o01-101 |
LEOPOLDINO A M, PENSA S D. The mutational spectrum of human autosomal tetranucleotide microsatellites. Human Mutation, 2003, 21(1): 71-79 DOI:10.1002/humu.10153 |
LI W J, LI Y Z, DU L M, et al. Comparative analysis of microsatellite sequences distribution in the genome of giant panda and polar bear. Sichuan Journal of Zoology, 2014, 33(6): 874-878 [李午佼, 李玉芝, 杜联明, 等. 大熊猫和北极熊基因组微卫星分布特征比较分析. 四川动物, 2014, 33(6): 874-878] |
LIU Z J, LI P, ARGUE B J, et al. Inheritance of RAPD markers in channel catfish (Ictalurus punctatus) × blue catfish (I. furcatus) and their F1, F2 and backcross hybrids. Animal Genetics, 1998, 29: 58-62 DOI:10.1046/j.1365-2052.1998.00284.x |
LIU Z J, LI P, ARGUE B, et al. Random amplified polymorphic DNA markers: Usefulness for gene mapping and analysis of genetic variation of catfish. Aquaculture, 1999, 174(1): 59-68 |
LU T, WANG C, DU C, et al. Distribution regularity of microsatellites in Moschus berezovskii genome. Sichuan Journal of Zoology, 2017, 36(4): 420-424 [卢婷, 王晨, 杜超, 等. 林麝全基因组微卫星分布规律研究. 四川动物, 2017, 36(4): 420-424] |
MICKETT K, MORTON C, FENG J, et al. Assessing genetic diversity of domestic populations of channel catfish (Ictalurus punctatus) in Alabama using AFLP markers. Aquaculture, 2003, 228(1/2/3/4): 91-105 |
MORGANTE M, HANAFEY M, POWELL W. Microsatellites are preferentially associated with non-repetitive DNA in plant genomes. Nature Genetics, 2001, 30: 194-200 |
NARASIMHAMOORTHY B, SAHA M C, SWALLER T, et al. Genetic diversity in switch grass collections assessed by EST-SSR markers. Bioenergy Research, 2008, 1(2): 136-146 DOI:10.1007/s12155-008-9011-0 |
NI S S, YANG Y, LIU S F, et al. Microsatellite analysis of Patinopecten yessoensis using next-generation sequencing method. Progress in Fishery Sciences, 2018, 39(1): 107-113 [倪守胜, 杨钰, 柳淑芳, 等. 基于高通量测序的虾夷扇贝基因组微卫星特征分析. 渔业科学进展, 2018, 39(1): 107-113] |
NIE H, CAO S S, ZHAO M L, et al. Comparative analysis of microsatellite distributions in genomes of Boa constrictor and Protobothrops mucrosquamatus. Sichuan Journal of Zoology, 2017, 36(6): 639-648 [聂虎, 曹莎莎, 赵明朗, 等. 红尾蚺和原矛头蝮基因组微卫星分布特征比较分析. 四川动物, 2017, 36(6): 639-648] |
PEARSON C E, SINDEN R R. Alternative structures in duplex DNA formed within the trinucleotide repeats of the myotonic dystrophy and fragile X loci. Biochemistry, 1996, 35(15): 5041-5053 DOI:10.1021/bi9601013 |
QI W H, JIANG X M, XIAO G S, et al. Distribution regularities of microsatellites in the pig genome. Animal Husbandry and Veterinary Medicine, 2014, 46(8): 9-13 [戚文华, 蒋雪梅, 肖国生, 等. 猪全基因组中微卫星分布规律. 畜牧与兽医, 2014, 46(8): 9-13] |
QI W H, JIANG X M, XIAO G S, et al. Seeking and bioinformatics analysis of microsallite sequence in the genomes of cow and sheep. Acta Veterinaria et Zootechnica Sinica, 2013, 44(11): 1724-1733 [戚文华, 蒋雪梅, 肖国生, 等. 牛和绵羊全基因组微卫星序列的搜索及其生物信息学分析. 畜牧兽医学报, 2013, 44(11): 1724-1733] |
QI W H, YAN C C, XIAO G S, et al. Distribution regularities and bioinformatics analysis of microsatellite in the whole genomes of goat and Tibetan antelope. Journal of Sichuan University (Natural Science), 2016, 53(4): 937-944 [戚文华, 严超超, 肖国生, 等. 山羊和藏羚羊全基因组微卫星分布规律及其生物信息学分析. 四川大学学报(自然科学版), 2016, 53(4): 937-944] |
SCHLÖTTERER C. Genome evolution: Are microsatellites really simple sequences. Current Biology, 1998, 8(4): 132-134 DOI:10.1016/S0960-9822(98)70989-3 |
SCHORDERET D F, GARTER S M. Analysis of CpG suppression in methylated and nonmethylated species. Proceedings of the National Academy of Sciences of USA, 1992, 89(3): 957-961 DOI:10.1073/pnas.89.3.957 |
SHEN X, YANG G, LIU Y, et al. Construction of genetic linkage maps of guppy (Poecilia reticulata) based on AFLP and microsatellite DNA markers. Aquaculture, 2007, 271(1): 178-187 |
SIMMONS M, MICKETT K, KUCUKTAS H, et al. Comparison of domestic and wild channel catfish (Ictalurus punctatus) populations provides no evidence for genetic impact. Aquaculture, 2006, 252(2/3/4): 133-146 |
STALLINGS R L. CpG suppression in vertebrate genomes does not account for the rarity of (CpG)n microsatellite repeats. Genomics, 1992, 13(3): 890-891 DOI:10.1016/0888-7543(92)90178-U |
SUBRAMANIAN S, MISHRA R K, SINGH L. Genome-wide analysis of microsatellite repeats in humans: Their abundance and density in specific genomic regions. Genome Biology, 2003, 4(2): 1-10 DOI:10.1186/gb-2003-4-2-p1 |
TONG X L, DAI F Y, LI B, et al. Microsatellite repeats in mouse: Abundance, distribution and density. Current Zoology, 2006, 52(1): 138-152 [童晓玲, 代方银, 李斌, 等. 小鼠基因组中的微卫星重复序列的数量、分布和密度. 动物学报, 2006, 52(1): 138-152] |
TU F Y, LIU J, HAN W J, et al. Analysis of microsatellite distribution characteristics in the entire genome of Macaca fascicularis. Chinese Journal of Wildlife, 2018, 39(2): 400-404 [涂飞云, 刘俊, 韩卫杰, 等. 食蟹猴全基因组微卫星分布特征分析. 野生动物学报, 2018, 39(2): 400-404 DOI:10.3969/j.issn.1000-0127.2018.02.032] |
TU F Y, LIU X H, DU L M, et al. Distribution characteristics of microsatellites in the rat genome. Acta Agriculturae Universitatis Jiangxiensis, 2015, 37(4): 708-711 [涂飞云, 刘晓华, 杜联明, 等. 大鼠全基因组微卫星分布特征研究. 江西农业大学学报, 2015, 37(4): 708-711] |
WANG C, DU L M, LI P, et al. Distribution patterns of microsatellites in the genome of the German cockroach (Blattella germanica). Acta Entomologica Sinica, 2015, 58(10): 1037-1045 [王晨, 杜联明, 李鹏, 等. 德国小蠊全基因组中微卫星分布规律. 昆虫学报, 2015, 58(10): 1037-1045] |
WANG Y Y, LIU X X, DONG K Z, et al. Distribution difference of microsatellite in 7 domestic animals genomes. China Animal Husbandry and Veterinary Medicine, 2015, 42(9): 2418-2426 [王月月, 刘雪雪, 董坤哲, 等. 7种家养动物全基因组微卫星分布的差异研究. 中国畜牧兽医, 2015, 42(9): 2418-2426] |
XIA J, LIU F, ZHU Z, et al. A consensus linkage map of the grass carp (Ctenopharyngodon idella) based on microsatellites and SNPs. BMC Genomic, 2010, 11(1): 135-151 DOI:10.1186/1471-2164-11-135 |
XIANG J G, ZHOU J, JIN H. A study on the biological and biocheimica charaerisics of the Ictalurs punctatus. Hunan Journal of Hunan Agricultural University (Natural Science), 2004, 30(4): 355-358 [向建国, 周进, 金宏. 斑点叉尾  的生物学与生理生化特性研究. 湖南农业大学学报(自然科学版), 2004, 30(4): 355-358] 的生物学与生理生化特性研究. 湖南农业大学学报(自然科学版), 2004, 30(4): 355-358] |
XU J J, ZHENG X, LI J, et al. Distribution characteristics of whole genome microsatellite of Pelteobagrus fulvidraco. Genomics and Applied Biology, 2020, 39(12): 5488-5498 [徐杰杰, 郑翔, 李杰, 等. 黄颡鱼(Pelteobagrus fulvidraco)全基因组微卫星分布特征分析. 基因组学与应用生物学, 2020, 39(12): 5488-5498] |
XU J J, ZHENG X, ZHANG X Y, et al. Analysis of distribution characteristics of microsatellites in the four genomes of puffer fish. Genomics and Applied Biology, 2021, 40(4): 1441-1451 [徐杰杰, 郑翔, 张鑫宇, 等. 4种河鲀全基因组微卫星分布特征分析研究. 基因组学与应用生物学, 2021, 40(4): 1441-1451] |
ZHANG L L, WEI Z M, LIAN Z M, et al. Abundance of microsatellites in the entire genome and EST of Tribolium castaneum. Chinese Bulletin of Entomology, 2008, 45(1): 38-42 [张琳琳, 魏朝明, 廉振民, 等. 赤拟谷盗全基因组和EST中微卫星的丰度. 昆虫知识, 2008, 45(1): 38-42] |
ZHANG T, GAN J, CHEN J W, et al. Morphological characteristics and electrophoretic analysis of two kinds of isozymes from Ietalurus punetaus. Chinese Fishery Quality and Standards, 2019, 9(6): 57-64 [张涛, 甘金, 陈建武, 等. 斑点叉尾  形态观察及其两种同工酶的电泳分析. 中国渔业质量与标准, 2019, 9(6): 57-64] 形态观察及其两种同工酶的电泳分析. 中国渔业质量与标准, 2019, 9(6): 57-64] |
ZHENG Y, JIN G L, WU W R. Relationship between sequence completeness and polymorphism of microsatellites. Genomics and Applied Biology, 2012, 31(6): 587-591 [郑燕, 金谷雷, 吴为人. 微卫星序列完整性与多态性的关系. 基因组学与应用生物学, 2012, 31(6): 587-591] |



