2. 中国水产科学研究院 农业农村部水生动物基因组学重点实验室 北京 100141;
3. 河南省水产科学研究院 河南 郑州 450044
2. Key Laboratory of Aquatic Genomics, Ministry of Agriculture and Rural Affairs, Beijing Key Laboratory of Fishery Biotechnology, Chinese Academy of Fishery Sciences, Beijing 100141, China;
3. Henan Academy of Fishery Sciences, Zhengzhou, Henan 450044, China
天然免疫系统是宿主抵抗病原体感染的第一道防线,其对病原体的直接杀伤和清除作用是通过天然免疫分子和吞噬细胞实现的,其中,三重基序(tripartite motif, TRIM)家族蛋白作为具有抗病毒作用和天然免疫调节功能的代表性蛋白,成为近年来天然免疫领域的研究热点。TRIM蛋白是一个结构相对保守的蛋白家族,广泛存在于无脊椎动物和脊椎动物的细胞中,其结构包括1个RING锌指结构域、1~2个B-box结构域、卷曲螺旋结构域(Coiled-coil, CC)以及C端可变结构域,因此,TRIM蛋白又被称为RBCC蛋白(Micale et al, 2016)。TRIM蛋白家族作为一类重要的E3泛素连接酶,参与信号转导、细胞凋亡、蛋白质加工和运输以及机体对病毒的应答等重要的生命过程。在人类医学研究中发现,它们的改变与多种疾病的发生密切相关,如发育障碍、神经退行性疾病、病毒感染和肿瘤(Tocchini et al, 2015)。
在硬骨鱼类中,TRIM蛋白在天然免疫系统中同样发挥重要的作用。目前,已在斑马鱼(Danio rerio)基因组中鉴定出208个trim基因,在河豚(Tetraodontidae)基因组中发现了66个trim基因(Boudinot et al, 2011),而人类基因组中包含了75个trim基因(Versteeg et al, 2013)。与人类相比硬骨鱼类的trim基因家族进化速度更快,具有更高的多样性。在trim基因家族中,trim25是非常重要的成员之一。近几年,在鱼类的天然免疫系统研究中发现,TRIM25蛋白在多个重要养殖鱼类,如罗非鱼(Oreochromis mossambicus)、鲤、大黄鱼(Larimichthys crocea)、石斑鱼(Epinephelinae)等的抗病毒过程中发挥了重要作用(Manokaran et al, 2015)。周真真等(2019)通过实时荧光定量PCR发现,TRIM25在大黄鱼的心脏、脑、外周血、脾脏、皮肤、肌肉、头肾、肠和肝脏组织中均有表达,其中,在肝脏的表达水平最高;当对大黄鱼注射Poly (I: C)后,在外周血、头肾、肝脏和脾脏中均检测到trim25基因表达量显著上调,且均表现为先上升后下降的趋势,提示trim25基因在大黄鱼抗病毒免疫反应中起着重要作用。Jin等(2019)研究发现,斑马鱼TRIM25通过靶向调控RIG-I蛋白的2CARD和RD结构域的K63泛素化,促进斑马鱼对红点石斑鱼(Epinephelus guttatus)神经坏死病毒感染的先天免疫应答。Yang等(2016)研究发现,在石斑鱼脾脏细胞中过表达TRIM25可抑制病毒复制,提高干扰素信号通路相关基因的表达水平。这些研究表明,TRIM25在硬骨鱼对病毒感染的先天免疫应答中发挥了关键作用。在Ross团队开展的鲤抗疱疹病毒QTL研究中,定位了44号染色体上的关联SNP,而trim25基因位于该SNP下游6.5 kb,提示TRIM25在鲤抗病毒免疫应答中的作用(Palaiokostas et al, 2018)。鲤是研究基因组进化和开展性状解析的重要模式物种,由于鲤经历了第4轮全基因组复制,多倍化的基因组具有较高的环境适应性,基因组中多套拷贝的基因为性状的调控提供了丰富的变异来源和增加了调控的灵活性。以trim25为例,鲤基因组中trim25的拷贝数高达16个。如何精确定位在抗病和免疫应答过程中具体发挥调控作用的基因拷贝,阐述基因调控的遗传位点和作用机制,需要进一步的精细分析。考虑到SNP对基因表达的调控作用,将SNP信息与基因表达结果整合分析成为性状解析的重要手段。表达数量性状基因座(expression quantitative trait loci, eQTL)分析将每个基因的表达量作为数量性状,研究遗传突变与基因表达之间的相关性,来寻找调控基因表达的位点及协同作用的基因,以此来构建基因的调控网络(彭启迪, 2020)。eQTL分析方法广泛应用于人类医学研究和动植物的性状解析,通过分析与目的基因相关的基因组区域的表达水平(Guo et al, 2020; Kordas et al, 2019; Lowe et al, 2015; Nicolae et al, 2010; Walker et al, 2020),阐明由基因变异引起表型改变的分子机制。然而,在鱼类的性状解析中,eQTL仍处于起步阶段,仅在大西洋鲑(Salmo salar)(Aslam et al, 2020)、极地鱼(Jacobs et al, 2020)等几个物种中有少量报道。
本研究对鲤trim25基因的多个拷贝与人(Homo sapiens)、小鼠(Mus musculus)、爪蟾(Xenopus laevis)、原鸡(Gallus gallus)以及多种鱼类进行进化分析,分析鲤trim25与几个主要模式物种的共线性。同时,为比较鲤trim25基因各拷贝之间的差异,对鲤trim25的16个拷贝进行结构分析及在多个组织中的表达研究。通过trim25的eQTL网络分析,提取可能调控trim25基因表达的SNP位点,旨在为今后研究鲤trim25基因的相关调控和抗病研究提供参考。
1 材料与方法 1.1 TRIM25蛋白序列的提取和比对鲤TRIM25蛋白16个拷贝的氨基酸序列取自本课题组鲤全基因组序列和注释文件(Xu et al, 2019),人、小鼠、原鸡、爪蟾、象鼻鲨(Callorhinchus milii)、腔棘鱼(Latimeria chalumnae)、红鳍东方鲀(Takifugu rubripe)、大黄鱼、石斑鱼、斑马鱼、金线鲃(Sinocyclocheilus grahami)的TRIM25蛋白序列通过NCBI (https://www.ncbi.nlm.nih.gov/protein/)数据库查询获得。利用NCBI数据库中的BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi),对12个物种的TRIM25氨基酸序列进行比对,计算各物种与鲤TRIM25蛋白的一致性。
1.2 进化树的绘制、共线性分析和蛋白结构域预测利用ClustalW对鲤、人、小鼠、鸡、爪蟾、象鼻鲨、腔棘鱼、红鳍东方鲀、斑马鱼、大黄鱼、金线鲃、石斑鱼的TRIM25氨基酸序列进行比对,利用RAxML软件(V 7.2.8)构建进化树,将Bootstrap设为1000,其他参数设为默认值。各物种trim25基因的共线性数据,通过NCBI数据库(https://www.ncbi.nlm.nih.gov/)查询各物种trim25上下游基因获得。以鲤TRIM25的16个拷贝的蛋白序列为输入文件,用在线软件SMART(http://smart.emblheidelberg.de/)(高进等, 2016),预测鲤TRIM25的16个拷贝的蛋白结构域构成。
1.3 trim25在组织中的表达和eQTL调控网络分析采集40尾黄河鲤和40尾镜鲤的肝脏及脑组织,使用RNeasy试剂盒(Qiagen, 上海)提取RNA,并委托测序公司利用HiSeq2000测序平台进行150PE的转录组测序。使用Bowtie2 v2.3.4.2和Cufflinks 2.2.1软件对转录组数据进行基因表达(FPKM值)的计算,并提取每个样本中trim25的基因表达数据,采用R软件包ggplot2绘制箱式图来展示trim25的基因表达情况。从已有的鲤SNP变异数据集中提取对应样本的SNP分型数据和位置信息,结合基因表达数据和基因位置信息,利用MatrixEqtl软件进行顺式eQTL的分析,筛选标准为P < 10–5。
2 结果 2.1 鲤TRIM25蛋白的结构分析蛋白结构预测显示,完整的鲤TRIM25蛋白包括4个结构域:RING、B-box、Coiled-coil和PRY/SPRY结构域。其中,HHLG38G0859、HHLG11g0652、HHLG12g0509、HHLG29g0701、HHLG3g0722和HHLG4g0919这6个拷贝有完整的4个结构域;HHLG10g1061缺少PRY/SPRY结构域,HHLG37g0859缺少Coiled-coil结构域,HHLG13g0878、HHLG14g0181、HHLG6g0789和HHLG9g1073只包含RING和B-box结构域。HHLG16g0672只包含RING和Coiled-coil结构域,HHLG13g0902只包含B-box和Coiled-coil结构域,HHLG46g0068和HHLG5g0226只包含RING结构域。
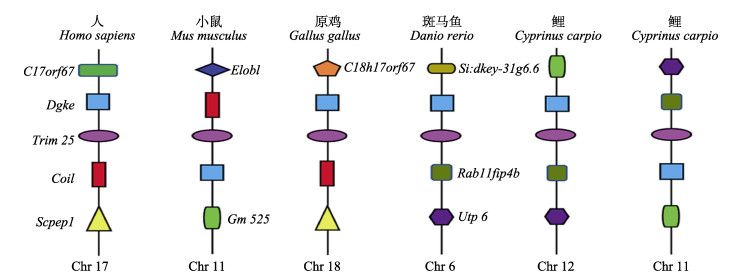
2.2 trim25共线性分析为了更好地理解trim25基因在进化上的保守性,对鲤、人、小鼠、鸡、斑马鱼5个物种的trim25基因及其上下游基因进行了共线性分析(图 2)。由于鲤的染色体与斑马鱼存在1∶2的映射关系,优先选择对应斑马鱼trim25基因所在染色体的鲤11 12号染色体上的HHLG11g0652和HHLG12g05092这2个拷贝来分析。从共线性图上可以看出,trim25基因在进化上高度保守。gdke是与trim25基因关联性最强的基因,存在于人、小鼠、鸡、斑马鱼及鲤5个物种。coil是与人、小鼠、鸡trim25基因关联性较强的另一个基因;而dgke、rab11fip4b和utp6这3个基因均存在于斑马鱼与鲤trim25基因上下游。其中,位于鲤12号染色体上的trim25基因上下游的基因分布与斑马鱼完全一致;位于鲤11和12号染色体上的trim25基因的上下游基因构成一致,只是上下游位置完全颠倒,这可能是鲤11号染色体的该区域在进化过程中发生了倒位。

|
图 1 鲤TRIM25蛋白结构域 Fig.1 Protein domain prediction of TRIM25 in Cyprinus carpio |
|
图 2 基因共线性分析 Fig.2 Synteny of genes neighboring trim25 |
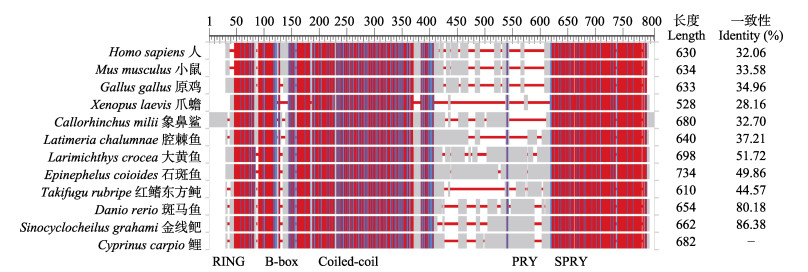
从NCBI数据库中收集代表性物种(哺乳动物、两栖动物、鸟类、软骨鱼类和硬骨鱼类)的TRIM25蛋白氨基酸序列。由于鲤的染色体与斑马鱼存在1 : 2的映射关系,优先选择对应斑马鱼TRIM25所在染色体的鲤的2个拷贝中结构完整、氨基酸序列最长的HHLG12g0509蛋白与其他11个物种TRIM25蛋白进行序列比对(图 3)。鲤16个TRIM25拷贝的位置信息见表 1。12个物种中,TRIM25蛋白长度从528个氨基酸(爪蟾)到734个氨基酸(石斑鱼),与鲤TRIM25蛋白一致性最高的是金线鲃,一致性最低的是爪蟾。
|
图 3 鲤TRIM25蛋白与其他物种的序列比对 Fig.3 Alignment of TRIM25 among C. carpio and other species |
|
|
表 1 鲤trim25基因位置信息 Tab.1 Location information of trim25 gene in C. carpio |
从序列比对的结果可以看出,哺乳动物中,人与小鼠TRIM25蛋白在RING、B-box、Coiled-coil和PRY/SPRY这4个结构域均高度相似,二者的主要差异在第420~470氨基酸之间。哺乳动物与鸡TRIM25蛋白在Coiled-coil和PRY/SPRY结构域高度相似,差异存在于RING和B-box结构域。腔棘鱼与象鼻鲨在B-box和Coiled-coil结构域高度相似,爪蟾TRIM25蛋白与其他物种相似性最低。海水鱼类中,大黄鱼与石斑鱼在RING结构域高度相近,红鳍东方鲀与前二者在RING结构域存在差异;三者的B-box和Coiled- coil结构域高度相似,但三者的PRY/SPRY结构域存在较大差异。鲤科鱼类中,斑马鱼、金线鲃和鲤在RING、B-box和Coiled-coil结构域高度相似;鲤与金线鲃的PRY/SPRY结构域高度相似,但斑马鱼的PRY/SPRY结构域与前二者存在差异。
2.4 TRIM25系统进化分析为了解析TRIM25在物种进化过程中的差异,构建了系统进化树(图 4)。可以看出,人与小鼠的TRIM25最为相近,其次是鸡。腔棘鱼作为一种原始的硬骨鱼类,其TRIM25也与上面3个物种相近。海水鱼类(大黄鱼、石斑鱼、红鳍东方鲀)单独聚为一支;鲤科鱼类中,位于鲤11和12号染色体上结构完整的HHLG11g0652、HHLG12g0509拷贝与金线鲃和斑马鱼聚为一支;鲤其余的TRIM25拷贝与象鼻鲨和爪蟾更为相近。鲤异源四倍化过程中产生了大量的多拷贝基因,基因的多个拷贝随着进化的压力出现了序列和结构的变化,最终导致亚功能化和去功能化。这些发生较大变异的基因拷贝,可能和其他物种聚到一起。

|
图 4 TRIM25蛋白的系统进化树 Fig.4 Phylogenetic tree of TRIM25 proteins |
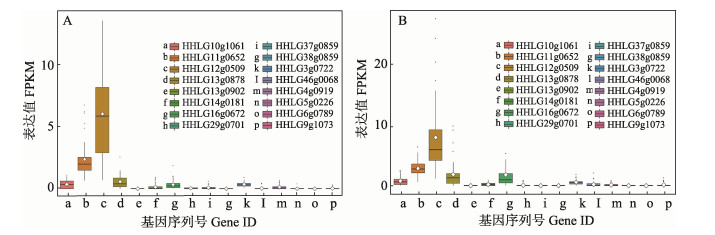
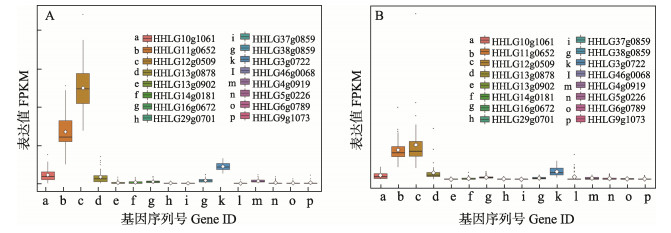
在黄河鲤和镜鲤的肝脏中,表达量较高的5个拷贝是一致的,包括HHLG10g1061、HHLG11g0652、HHLG12g0509、HHLG13g0878和HHLG16g0672。其他的11个拷贝在黄河鲤和镜鲤的肝脏组织中表达量极低或者几乎不表达。在黄河鲤和镜鲤的脑组织中,表达较高的5个拷贝相同,分别是HHLG10g1061、HHLG11g0652、HHLG12g0509、HHLG13g0878和HHLG3g0722,脑组织和肝脏组织的TRIM25表达情况非常相似(图 5和图 6)。
|
图 5 trim25基因在黄河鲤(A)和镜鲤(B)肝脏组织中的表达 Fig.5 Expression of trim25 genes of Yellow River carp (A) and Mirror carp (B) in liver tissue |
|
图 6 trim25基因在黄河鲤(A)和镜鲤(B)脑组织中的表达 Fig.6 Expression of trim25 genes of Yellow River carp (A) and Mirror carp (B) in brain tissue |
从课题组已构建的eQTL调控网络中提取出肝脏和脑组织中与trim25基因表达相关的顺式SNP位点。肝脏中筛选到的阳性SNP位点有5个,位于鲤第4、6、12、13和37号染色体上,分别调控HHLG4g0919、HHLG6g0789、HHLG12g0509、HHLG13g0878和HHLG37g0859这5个基因。脑组织中筛选到17个阳性位点,位于鲤第6、11和13号染色体上,其中,调控HHLG6g0789基因的有1个SNP位点,调控HHLG11g0652基因的有9个SNP位点,调控HHLG13g0878基因的有6个SNP位点,调控HHLG13g0902的有1个SNP位点(表 2和表 3)。
|
|
表 2 鲤肝脏组织顺式调控trim25基因表达的SNP Tab.2 SNP loci in cis-regulation of trim25 genes in liver of C. carpio |
|
|
表 3 鲤脑组织顺式调控trim25基因表达的SNP Tab.3 SNP loci in cis-regulation of trim25 genes in brain of Cyprinus carpio |
在进化过程中,脊椎动物出现了trim基因数目的显著增加,同时伴随着SPRY结构域的增加(Sardiello et al, 2008)。目前,已在人类基因组中鉴定出75个trim基因(Versteeg et al, 2013),在河豚基因组中发现了66个trim基因,在斑马鱼基因组中鉴定出208个trim基因(Boudinot et al, 2011),说明trim家族数量在不同物种中的差异很大,且与人类相比,硬骨鱼类的trim基因家族进化速度更快。硬骨鱼类基因组中trim基因存在的多样性和丰富变异,为trim基因家族的比较分析提供了理想的模型。鲤TRIM25蛋白属于TRIM家族第二类成员,通过氨基酸多序列比对分析结果发现,鲤与鲤科鱼类金线鲃、斑马鱼的TRIM25蛋白结构在3个保守结构域高度相似。鲤12号染色体上的trim25基因HHLG12g0509与斑马鱼上下游基因完全一致,trim25基因下游的2个基因分别是rab11fip4b和utp6;上游距离trim25最近的基因是dgke,dgke也是哺乳动物人、小鼠、鸡、斑马鱼、鲤共有的基因;coil基因是人、小鼠、鸡共有的基因。共线性的结果体现了硬骨鱼类与高等动物之间的进化差异,以及鲤在经历第4轮全基因组复制后,基因多样性有所增加。相对于鲤科其他二倍体鱼类,鲤经历了额外一轮全基因组复制,形成了四倍化的基因组,在基因组复制及之后的漫长进化过程中,基因组的许多区域产生大量的结构变异,如倒位、易位、插入、缺失等,类似鲤11号染色体的倒位现象大量存在。
本研究鉴定了鲤TRIM25蛋白有16个拷贝之多,这可能是进化过程中,由于环境压力导致的trim基因家族的扩张。此外,研究表明,trim基因家族的扩展与先天免疫及适应性免疫的进化惊人得平行,这表明TRIM蛋白可能在日益复杂的免疫功能中发挥核心调节因子的作用(Malfavon-Borja et al, 2013; Versteeg et al, 2014)。在鲤TRIM25的16个拷贝中,有6个拷贝具有完整的功能结构域,而另外10个拷贝虽然具有TRIM25的部分结构域,但并不完整,这可能由于进化过程中发生了基因的亚功能化或去功能化。
为了精细定位鱼类抗病毒性状相关的基因和通路,高通量分子标记的采集和大样本的表型分析是非常必要的。对于鲤这样经历了第4轮全基因组复制的异源四倍化物种,许多基因发生了扩增和结构变异,增加了性状精准定位的难度。在Ross团队发表的鲤抗疱疹病毒QTL研究中,初步定位到trim25基因(Palaiokostas et al, 2018),但由于其研究采用了旧版本参考基因组,装配和注释结果不够准确,因此,并未精确解析该基因的多拷贝的差异。本研究基于高质量的新版本基因组开展了鲤trim25基于多个拷贝的筛选和分析,为了解析trim25基因的调控机制,还开展了eQTL分析。eQTL是Jansen等(2001)提出的将全基因组中每个基因的表达量作为数量性状,对其进行QTL定位分析的方法。eQTL分析填补了基因型和表型之间的空白(Kim et al, 2012),与全基因组关联研究(GWAS)和差异表达分析的整合研究已经广泛应用于人类疾病研究和动植物育种研究(Joehanes et al, 2017; Nieuwenhuis et al, 2016; Pavlides et al, 2016),eQTL分析新统计方法和软件的开发加速了该领域的进展(Zhu et al, 2016)。本研究鉴定了肝脏组织中多个调控位点,表明了肝脏与免疫功能的密切相关(Huang et al, 2013; Lee et al, 2010)。根据鲤的TRIM25在脑和肝脏组织的表达分析及eQTL分析结果,初步筛选了候选的调控位点,对于目前处于起步阶段的鲤抗病毒QTL和GWAS研究有重要的提示作用。
本研究初步定位了鲤trim25基因的多个拷贝,比较了鲤与其他物种的trim25序列差异、进化关系和共线性相似度,揭示了鲤trim25各拷贝之间的结构多样性和在组织中表达的分化情况,筛选了可能调控trim25基因表达的顺时调控位点,为今后鲤trim25的基因功能精细研究奠定了基础,也为鱼类抗病研究和育种提供了重要参考。
ASLAM M L, ROBLEDO D, KRASNOV A, et al. Quantitative trait loci and genes associated with salmonid alphavirus load in Atlantic salmon: Implications for pancreas disease resistance and tolerance. Scientific Reports, 2020, 10(1): 10393 DOI:10.1038/s41598-020-67405-8 |
BOUDINOT P, VAN DER AA LM, JOUNEAU L, et al. Origin and evolution of TRIM proteins: New insights from the complete TRIM repertoire of zebrafish and pufferfish. PLoS One, 2011, 6(7): e22022 DOI:10.1371/journal.pone.0022022 |
GAO J, MA J L, LIU Y, et al. Bioinformatics analysis of sox gene family in Cynoglossus semilaevis. Progress in Fishery Sciences, 2016, 37(2): 41-48 [高进, 马佳璐, 刘洋, 等. 半滑舌鳎(Cynoglossus semilaevis) Sox基因家族生物信息学分析. 渔业科学进展, 2016, 37(2): 41-48] |
GUO J, CAO K, DENG C, et al. An integrated peach genome structural variation map uncovers genes associated with fruit traits. Genome Biology, 2020, 21(1): 258 DOI:10.1186/s13059-020-02169-y |
HUANG L R, WOHLLEBER D, REISINGER F, et al. Intrahepatic myeloid-cell aggregates enable local proliferation of CD8(+) T cells and successful immunotherapy against chronic viral liver infection. Nature Immunology, 2013, 14(6): 574-583 DOI:10.1038/ni.2573 |
JACOBS A, CARRUTHERS M, YURCHENKO A, et al. Parallelism in eco-morphology and gene expression despite variable evolutionary and genomic backgrounds in a Holarctic fish. PLoS Genetics, 2020, 16(4): e1008658 DOI:10.1371/journal.pgen.1008658 |
JANSEN R C, NAP J P. Genetical genomics: The added value from segregation. Trends in Genetics, 2001, 17(7): 388-391 DOI:10.1016/S0168-9525(01)02310-1 |
JIN Y, JIA K, ZHANG W, et al. Zebrafish TRIM25 promotes innate immune response to RGNNV infection by targeting 2CARD and RD regions of RIG-I for K63-linked ubiquitination. Frontiers in Immunology, 2019, 10: 2805 DOI:10.3389/fimmu.2019.02805 |
JOEHANES R, ZHANG X, HUAN T, et al. Integrated genome-wide analysis of expression quantitative trait loci aids interpretation of genomic association studies. Genome Biology, 2017, 18(1): 16 DOI:10.1186/s13059-016-1142-6 |
KIM Y A, PRZYTYCKA T M. Bridging the gap between genotype and phenotype via network approaches. Frontiers in Genetics, 2012, 3: 227 |
KORDAS G, RUDRA P, HENDRICKS A, et al. Insight into genetic regulation of miRNA in mouse brain. BMC Genomics, 2019, 20(1): 849 DOI:10.1186/s12864-019-6110-6 |
LEE W Y, MORIARTY T J, WONG C H, et al. An intravascular immune response to Borrelia burgdorferi involves Kupffer cells and iNKT cells. Nature Immunology, 2010, 11(4): 295-302 DOI:10.1038/ni.1855 |
LOWE W L Jr, REDDY T E. Genomic approaches for understanding the genetics of complex disease. Genome Research, 2015, 25(10): 1432-1441 DOI:10.1101/gr.190603.115 |
MALFAVON-BORJA R, SAWYER S L, WU L I, et al. An evolutionary screen highlights canonical and noncanonical candidate antiviral genes within the primate TRIM gene family. Genome Biology and Evolution, 2013, 5(11): 2141-2154 DOI:10.1093/gbe/evt163 |
MANOKARAN G, FINOL E, WANG C, et al. Dengue subgenomic RNA binds TRIM25 to inhibit interferon expression for epidemiological fitness. Science, 2015, 350(6257): 217-221 DOI:10.1126/science.aab3369 |
MICALE L, CHAIGNAT E, FUSCO C, et al. The tripartite motif: Structure and function. 2016, 770: 11-25
|
NICOLAE DL, GAMAZON E, ZHANG W, et al. Trait-associated SNPs are more likely to be eQTLs: Annotation to enhance discovery from GWAS. PLoS Genetics, 2010, 6(4): e1000888 DOI:10.1371/journal.pgen.1000888 |
NIEUWENHUIS M A, SIEDLINSKI M, VAN DEN BERGE M, et al. Combining genomewide association study and lung eQTL analysis provides evidence for novel genes associated with asthma. Allergy, 2016, 71(12): 1712-1720 DOI:10.1111/all.12990 |
PALAIOKOSTAS C, ROBLEDO D, VESELY T, et al. Mapping and sequencing of a significant quantitative trait locus affecting resistance to koi herpesvirus in common carp. G3 (Bethesda), 2018, 8(11): 3507-3513 DOI:10.1534/g3.118.200593 |
PAVLIDES J M, ZHU Z, GRATTEN J, et al. Predicting gene targets from integrative analyses of summary data from GWAS and eQTL studies for 28 human complex traits. Genome Medicine, 2016, 8(1): 84 DOI:10.1186/s13073-016-0338-4 |
PENG Q D. Method for constructing and analyzing biological network based on eQTL data. Masterxs Thesis of Harbin Institute of Technology, 2020 [彭启迪. 基于eQTL数据的生物网络构建与分析方法. 哈尔滨工业大学硕士研究生学位论文, 2020]
|
SARDIELLO M, CAIRO S, FONTANELLA B, et al. Genomic analysis of the TRIM family reveals two groups of genes with distinct evolutionary properties. BMC Evolutionary Biology, 2008, 8: 225 DOI:10.1186/1471-2148-8-225 |
TOCCHINI C, CIOSK R. TRIM-NHL proteins in development and disease. Seminars in Cell and Developmental Biology, 2015, 47-48: 52-59 DOI:10.1016/j.semcdb.2015.10.017 |
VERSTEEG G A, BENKE S, GARCÍA-SASTRE A, et al. InTRIMsic immunity: Positive and negative regulation of immune signaling by tripartite motif proteins. Cytokine and Growth Factor Reviews, 2014, 25(5): 563-576 DOI:10.1016/j.cytogfr.2014.08.001 |
VERSTEEG G A, RAJSBAUM R, SÁNCHEZ-APARICIO M T, et al. The E3-ligase TRIM family of proteins regulates signaling pathways triggered by innate immune pattern-recognition receptors. Immunity, 2013, 38(2): 384-398 DOI:10.1016/j.immuni.2012.11.013 |
WALKER R L, RAMASWAMI G, HARTL C, et al. Genetic control of expression and splicing in developing human brain informs disease mechanisms. Cell, 2020, 181(2): 484 DOI:10.1016/j.cell.2020.04.003 |
XU P, XU J, LIU G, et al. The allotetraploid origin and asymmetrical genome evolution of the common carp Cyprinus carpio. Nature communications, 2019, 10(1): 4625 DOI:10.1038/s41467-019-12644-1 |
YANG Y, HUANG Y, YU Y, et al. RING domain is essential for the antiviral activity of TRIM25 from orange spotted grouper. Fish and Shellfish Immunology, 2016, 55: 304-314 DOI:10.1016/j.fsi.2016.06.005 |
ZHOU Z Z, JING F, WEI K, et al. Molecular cloning and expression analysis of TRIM25 gene in Larimichthys crocea. Acta Hydrobiologica Sinica, 2019, 43(6): 1189-1196 [周真真, 景斐, 魏可, 等. 大黄鱼TRIM25基因克隆和表达分析. 水生生物学报, 2019, 43(6): 1189-1196] |
ZHU Z, ZHANG F, HU H, et al. Integration of summary data from GWAS and eQTL studies predicts complex trait gene targets. Nature Genetics, 2016, 48(5): 481-487 DOI:10.1038/ng.3538 |



