2. 山东省海洋生物研究院 山东 青岛 266104
2. Marine Biology Institute of Shandong Province, Qingdao, Shandong 266104, China
毛蚶(Scapharca kagoshimensis)俗称毛蛤、麻蛤、麻蚶等(王如才等, 2008),属广温广盐性经济贝类。毛蚶广泛分布于中国、日本、朝鲜沿岸,在中国以莱州湾、渤海湾、辽东湾、海州湾等浅海区资源尤为丰富(王庆志等, 2015)。对毛蚶的研究主要集中在形态学(陈蓉等, 2009; 宋菲菲等, 2012)、苗种繁育(翟林香等, 2010; 马云聪等, 2008)及养殖技术(王庆志等, 2015)等,关于群体遗传学的研究较少(赵文等, 2011; 田吉腾等, 2016)。进入20世纪80年代,受生境破坏及过度捕捞等因素的影响,毛蚶自然资源逐年减少,产量大幅度下降,已远远不能满足市场需求,毛蚶自然种群的遗传评估和资源恢复工作也亟待进行。
微卫星分子标记具有分布范围广、呈共显性遗传、多态性高和重复性好等特点,在群体遗传多样性分析、种质资源评价、遗传图谱构建等研究中有明显优势(Nikolic et al, 2009; Bao et al, 2016; Kewwuwan et al, 2016)。微卫星的开发方法主要有直接文库筛选法(战爱斌等, 2008)、磁珠富集法(宋丹丹等, 2019)、数据库检索法(魏大为等, 2015)、种间转移扩增法(Liu et al, 2007)等。目前,毛蚶已开发出部分微卫星标记,如Feng等(2009)利用磁珠富集法筛选了14对毛蚶微卫星引物,陈辰(2015)利用同样的方法获得41对多态性较好的微卫星引物。另外,Li等(2012)通过在魁蚶(Scapharca broughtonii)中构建cDNA文库,得到了25条在魁蚶和毛蚶中均通用的EST-SSR引物。Dong等(2012)利用泥蚶(Tegillarca granosa)的转录组数据,开发了62个EST-SSR引物,在毛蚶中的通用率为25.81%。但毛蚶微卫星标记的数量还远不能满足其遗传分析需求,需要开发更多可利用的微卫星标记。近年来,利用转录组测序平台开发SSR分子标记已经得到越来越多的应用,其相比于传统方法,具有通量大、周期短、成本低且直接与某些功能基因相关联的优点(于爱清等, 2019)。
本研究基于毛蚶鳃组织的转录组数据,对其中的SSR位点进行搜索,分析其分布特征及组成类型;并利用所筛选的引物在潍坊毛蚶野生群体中进行了遗传多样性分析,旨在为毛蚶微卫星分子标记的开发提供更为有效的方法,为毛蚶种群遗传学分析、遗传图谱构建及分子标记辅助育种提供有力工具。
1 材料与方法 1.1 实验材料及转录组测序实验所需毛蚶样品于2017年10月采自天津滨海新区大神堂海域,壳长为(26.19±1.46) mm,活体重为(20.01±2.89) g。选取活力好的毛蚶3只,取其鳃组织,构建高通量转录组测序文库,并利用Illumina HiSeq4000测序平台(杭州联川生物技术股份有限公司)进行测序,共获得93.57 Gb的数据量,原始测序数据经Trinity软件(http://trinityrnaseq.sourceforge.net/)组装并去除冗余后,获得了平均长度为621.23 bp的unigene 35 555条。
1.2 微卫星标记筛选和引物设计微卫星序列的识别和定位使用MISA软件进行,MISA搜索参数设置如下:单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸的最少重复次数分别为12、6、5、5、4和4,复合SSR两个位点间最大间隔碱基数设置为100,结合Primer 3软件批量设计引物。主要参数设置:引物长度为18~27 bp,PCR扩增产物在100~280 bp之间,57℃≤退火温度(Tm)≤63℃,20%≤GC含量≤80%。同时,利用NCBI数据库中的Primer-BLAST对结果进行进一步验证,主要参数设置:Self complementarity < 6,Self´3 complementarity < 3,且上下游引物Tm值差值不高于2℃。引物由生工生物工程(上海)股份有限公司合成。
1.3 微卫星多态性分析使用海洋动物组织基因组DNA提取试剂盒(天根生化科技(北京)有限公司)进行毛蚶肌肉组织DNA的提取,委托生工生物工程(上海)股份有限公司合成荧光引物,对毛蚶潍坊群体的30个个体进行STR检测(3730XL测序分析仪,美国ABI公司),利用GeneMapper 3.2软件对结果进行基因分型。
等位基因数(Na)、期望杂合度(He)和观测杂合度(Ho)用Microsatellite Analyser (MSA)软件计算(Dieringer et al, 2003)。多态信息含量(polymorphism information content, PIC)用PIC-CALC 0.6软件计算。利用Genepop 4.2软件进行哈迪–温伯格平衡(Hardy- Weinberg equilibrium, HWE)分析(http://genepop.curtin.edu.au/),并对P值进行Bonferroni校正。
2 结果与分析 2.1 毛蚶转录组中SSR的数量和分布特点对毛蚶转录组中序列总长度22 087 807 bp的35 555条unigene序列进行SSR检测,共得到3987个SSR (完美型SSR为3074个),分布在3162条unigene序列当中。SSR发生频率(含SSR的unigene数/unigene总数)为8.89%,SSR出现频率(检出SSR位点数/unigene总数)为11.21%,平均每5.54 kb含有1个SSR位点(总unigene长度/搜索到的SSR数量)。其中,612条unigene含有1个以上SSR位点,以复合微卫星形式出现的SSR数目为913个。
2.2 毛蚶转录组中SSR的重复基元类型和频率特征从毛蚶转录组中筛查到的3987个SSR位点中,二核苷酸重复SSR出现频率最高,为2315个,占总数的58.06%;其次是三核苷酸重复SSR (759个,19.04%)和单核苷酸重复SSR (696个,17.46%);四核苷酸、五核苷酸和六核苷酸重复SSR所占比例最少,分别占总数的4.01%、1.15%和0.28%。
3987个SSR共有182种重复基元类型,根据碱基互补配对原则和阅读起始碱基顺序的差异(栾生等, 2007),对每种长度类型的重复基元进行同类兼并后,单、二、三、四、五、六核苷酸重复基元类型分别为2、4、10、18、17和8种(表 1)。单核苷酸重复中出现最多的基元类型是A/T,占单核苷酸重复的比例为是83.62%。二核苷酸重复中出现较多的是AC/GT和AG/CT类型,占二核苷酸重复的比例分别为45.70%和33.09%,AAT/ATT基元在三核苷酸重复中出现的频率最高,占三核苷酸重复的34.78%。
|
|
表 1 毛蚶转录组SSR主要重复基元类型及分布比例 Tab.1 The type and percentage of main repeat motif of SSR in transcriptome of S. kagoshimensis |
本研究中,毛蚶转录组中SSR(完美型)长度在12~84 bp之间,其中,长度在12~19 bp之间的SSR占比最大,为SSR总数的61.06%。其次为长度20~ 29 bp的SSR,占总数的22.38%。长度≥50 bp的SSR仅占总数的2.64%。SSR的长度分布规律见图 1。
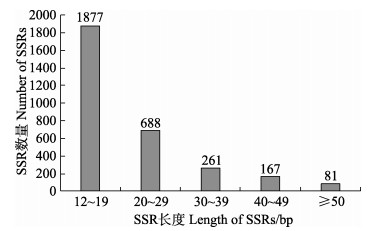
|
图 1 毛蚶转录组中的SSR长度分布 Fig.1 Length distribution of SSRs in transcriptome of S. kagoshimensis |
毛蚶转录组中不同类型SSR的重复次数分布见表 2。由表 2可知,SSR单核苷酸重复次数主要集中在12~14次,二核苷酸重复次数主要集中在6~8次,三核苷酸和四核苷酸重复次数主要集中在5~6次,五核苷酸和六核苷酸重复次数主要为4次。总的来看,重复6次的SSR占比最高,为778个(19.51%),其次为重复5次和7次,分别为488个(12.24%)和423个(10.61%)。重复次数≥15次的SSR位点共有803个,占总SSR个数的20.14%。
|
|
表 2 毛蚶转录组中不同类型SSR重复次数分布 Tab.2 Repeat number of SSR in transcriptome of S. kagoshimensis |
除部分微卫星序列侧翼序列过短或者本身结构不适合设计引物以外,使用Primer 3软件对其中含有SSR的3162条序列进行引物设计,最后从60对引物中筛选出扩增稳定、多态性较高的14对引物(表 3)。利用这14对引物对毛蚶潍坊野生群体的30个个体进行遗传多样性分析。等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)和哈迪–温伯格平衡检验(PHWE)等参数详见表 4。
|
|
表 3 毛蚶14对SSR引物信息 Tab.3 Information of SSR primers in S. kagoshimensis |
|
|
表 4 基于14个SSR位点的潍坊毛蚶群体遗传多样性分析 Tab.4 Genetic diversity of Weifang population in S. kagoshimensis at 14 SSR loci |
14个微卫星位点总共检测到216个等位基因,每个位点的等位基因个数从4个到26个不等,平均等位基因数为15.4个。Ho为0.379~0.967,平均值为0.682;He为0.623~0.962,平均值为0.852,PIC为0.817。经Bonferroni校正后发现,14个位点中有7个偏离哈迪–温伯格平衡。
3 讨论 3.1 毛蚶转录组SSR类型和分布规律近年来,越来越多的贝类中报道了利用转录组数据开发微卫星的方法,在墨西哥湾扇贝(Argopecten irradians concentricus)的转录组测序数据中,SSR的出现频率为10%(谭杰等, 2018),马氏珠母贝(Pinctada martensii)的SSR出现频率为13.34% (王忠良等, 2015),泥东风螺(Babylonia lutosa)的SSR出现频率为13.62% (熊钢等, 2020)。本研究中,毛蚶转录组中SSR的出现频率为11.21%,和上述报道相似,但低于扁玉螺(Neverita didyma)的86.53% (卢玮筱等, 2018)。在其他水生动物中,转录组中SSR出现频率差异较大,如牙鲆(Paralichthys olivaceus)为43.24% (李超等, 2015),大口黑鲈(Micropterus salmoides)为11.30% (黄勇等, 2019),金乌贼(Sepia esculenta)为90.66% (张金勇等, 2020),曼氏针乌贼(Sepiella japonica)为48.70%(管奥等, 2018),凡纳滨对虾(Litopenaeus vannamei)为22.1% (杨铭等, 2017)。在SSR检索标准一致的情况下,SSR出现频率主要与物种差异、转录组结构及测序数据大小有关。
除单核苷酸重复外,大多数贝类如墨西哥湾扇贝、马氏珠母贝、泥东风螺及菲律宾蛤仔(Ruditapes philippinarum)(谭杰等, 2018; 王忠良等, 2015; 熊钢等, 2020; 闫路路, 2015)等转录组中SSR均以二核苷酸重复为主,本研究中毛蚶转录组中SSR也以二核苷酸重复为主,和上述研究结论一致。多数鱼类,如牙鲆、刀鲚(Coilia ectenes)、密斑刺鲀(Diodon hystrix) (李超等, 2015; 于爱清等, 2019; 马军等, 2020)以及虾类,如凡纳滨对虾、红螯螯虾(Cherax quadricarinatus) (杨铭等, 2017; 李喜莲等, 2020)等,转录组中的SSR类型分布也符合这一规律。
本研究中二核苷酸重复基元类型以AC/GT最多,占二核苷酸重复的45.70%,这与合浦珠母贝(Pinctada fucata)、泥东风螺的研究结果一致(曲妮妮, 2008; 熊钢等, 2020)。目前,大多数鱼类的研究结果也验证了这一结论,如牙鲆、刀鲚、团头鲂(Megalobrama amblycephala)和
SSR标记多态性的高低是判断其物种可用性的重要依据。根据Temnykh等(2001)的研究,SSR的长度大小是影响其多态性高低的关键因素,当SSR长度≥20 bp时,呈高度多态性,12 bp≤长度≤20 bp时,呈中等多态性;前者更容易发生变异,原因是在较长的模板上滑动错配发生的几率更高(毛俐慧等, 2018)。本研究在设置筛选参数时,已剔除了长度在12 bp以下的SSR序列,12~19 bp和长度≥20 bp的序列分别占SSR总数量的66.23%和33.77%,在此基础上设计引物,保证了引物具有较高的多态性。
本研究筛选出的14对多态性较好的SSR引物中,二核苷酸重复主要为6次,三核苷酸重复主要为7次,随着重复次数的增加,各类型SSR的数量均逐渐降低。重复次数范围和SSR数量变化规律和大多数同类研究一致。相比之下,陈辰(2015)和Feng等(2009)用磁珠富集方法开发的毛蚶SSR中,高重复序列所占比例较大,其二核苷酸重复次数一般集中在13~27次。造成这种差异的原因,一方面是由于转录组中开发的SSR重复次数往往要低于基因组中的SSR (Xia et al, 2018),另一方面是磁珠富集的杂交洗脱等程序能够将大部分低重复序列除去(孙效文等, 2005)。Schlötterer等(2000)认为,重复次数与SSR位点突变率存在一定程度正相关。虽然本研究从转录组中开发的SSR重复次数相对较低,但从毛蚶群体遗传学分析的结果来看,依然显示了较高的多态性。
3.3 潍坊毛蚶群体遗传多样性分析本研究对基于转录组数据得到的微卫星引物进行筛选,并在潍坊毛蚶野生群体中进行了遗传多样性分析,其平均等位基因数(Na)、观测杂合度(Ho)和期望杂合度(He)分别为15.4、0.682和0.852,与陈辰(2015) (Na:15.2;Ho:0.719;He:0.856)的研究结果类似,相比Feng等(2009)(Na:21.64;Ho:0.798;He:0.931)多态性略低。14个微卫星位点的PIC值范围为0.549~0.943,平均值为0.817。根据Botstein等(1980)的标准,PIC≥0.5、0.25≤PIC < 0.5和PIC < 0.25分别代表高度多态性、中度多态性和低度多态性。从结果来看,本研究开发的14个微卫星标记均属高多态性,能够提供丰富的遗传信息,在毛蚶种群遗传学研究、遗传图谱构建及家系分析鉴定中具有较高的实用性。另外,本研究采集的样本为山东潍坊野生群体,也说明目前群体依然保持着较高的遗传多样性。
经Bonferroni校正后发现,14个微卫星位点中有7个显著偏离哈迪–温伯格平衡(P<0.05),微卫星位点偏离哈迪–温伯格平衡的现象在相关报道中并不少见,如在菲律宾蛤仔(闫路路, 2015)、青蛤(方军等, 2020)、里式拟石磺(Paraoncidium reevesii)(吴欣等, 2016)和熊本牡蛎(Crassostrea sikamea)(黄飘逸等, 2020)的群体遗传学研究中,微卫星位点偏离哈迪–温伯格的比例分别为75%、70.37%、35.71%和20%。一般来说,群体偏离平衡的原因主要是近交或存在无效等位基因而导致的纯合子过剩或取样样本量过小。其中,微卫星标记中存在无效等位基因是一个较为普遍的现象,尤其是在海洋贝类中(Reece et al, 2004)。本研究采集的群体为野生群体,从平均等位基因数、杂合度等遗传参数来看,均体现出较高的多态性,故推测微卫星中无效等位基因的存在或样本量较小是造成偏离哈迪–温伯格平衡的主要原因。
本研究结果表明,利用高通量转录组测序筛选毛蚶微卫星是一种高效可行的方法。研究结果增加了毛蚶的可用微卫星标记,对毛蚶的种群遗传学分析、遗传图谱构建及分子标记辅助育种都具有重要的意义。
BAO M M, ZHOU Q, XIE C X, et al. Ten polymorphic microsatellite markers characterized for Schizothorax pseudaksaiensis and applied for population genetic analysis. Journal of Genetics, 2016, 95: e5-e8 DOI:10.1007/s12041-016-0669-3 |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331 |
CHE R B. Development of molecule markers and gene differential expression analysis of Miichthys miiuy based on transcriptome sequencing data. Master´s Thesis of Zhejiang Ocean University, 2015 [车荣波. 基于转录组数据的  鱼分子标记筛选及基因差异表达分析. 浙江海洋学院硕士研究生学位论文, 2015] 鱼分子标记筛选及基因差异表达分析. 浙江海洋学院硕士研究生学位论文, 2015]
|
CHEN C. Population genetics of the ark shell. Doctoral Dissertation of Ocean University of China, 2015 [陈辰. 毛蚶群体遗传学研究. 中国海洋大学博士研究生学位论文, 2015]
|
CHEN R, LIU J Y, TANG L J, et al. Morphological variations analysis of five different populations of Scapharca subcrenata in China. Marine Sciences, 2009, 33(7): 64-69 [中国5个海区不同群体毛蚶形态差异分析. 海洋科学, 2009, 33(7): 64-69] |
DIERINGER D, SCHLÖTTERER C. Microsatellite analyser (MSA): A platform independent analysis tool for large microsatellite datasets. Molecular Ecology Notes, 2003, 3(1): 167-169 DOI:10.1046/j.1471-8286.2003.00351.x |
DONG Y H, YAO H H, LIN Z H, et al. Characterization of 62 polymorphic EST-SSR markers in the blood clam (Tegillarca ganosa) and their cross-amplification in Scapharca subcrenata. Conservation Genetics Resources, 2012, 4(4): 991-997 DOI:10.1007/s12686-012-9691-9 |
FANG J, SHEN Y P, ZHANG L L, et al. Development and verification of SSR markers in Cyclina sinensis. Journal of Applied Oceanography, 2020, 39(2): 214-220 [基于转录组数据的青蛤微卫星标记开发与验证. 应用海洋学学报, 2020, 39(2): 214-220 DOI:10.3969/J.ISSN.2095-4972.2020.02.008] |
FENG Y W, LI Q, KONG L F. Isolation and characterization of 14 polymorphic microsatellite loci in the ark shell Scapharca subcrenata (Bivalvia: Arcidae). Conservation Genetics, 2009, 10: 1125-1127 DOI:10.1007/s10592-008-9725-8 |
GUAN A, WU Y T, CHEN Y, et al. Deep sequence-based transcriptome analysis of microsatellites in the cuttlefish (Sepiella japonica). Progress in Fishery Sciences, 2018, 39(3): 144-151 [曼氏无针乌贼转录组微卫星特征分析. 渔业科学进展, 2018, 39(3): 144-151] |
HUANG P Y, MAO H T, YU Z N, et al. Evaluation of genetic diversity and versatility of wild and cultured populations based on high-throughput sequencing of Crassostrea sikamea microsatellite markers. Journal of Fisheries of China, 2020, 44(3): 368-377 [利用高通量测序开发的熊本牡蛎微卫星标记评价野生和养殖群体的遗传多样性及通用性. 水产学报, 2020, 44(3): 368-377] |
HUANG Y, GONG W B, CHEN H G, et al. Sequencing and bioinformatic analysis for transcriptome of Micropterus salmoides based on RNA-seq. South China Fisheries Science, 2019, 15(1): 106-112 [基于RNA-Seq高通量测序技术的大口黑鲈转录组分析. 南方水产科学, 2019, 15(1): 106-112] |
KEWWUWAN K, KUBOTA S, LIU Q, et al. Detection of growth-related quantitative trait loci and high-resolution genetic linkage maps using simple sequence repeat markers in the kelp grouper (Epinephelus bruneus). Marine Biotechnology, 2016, 18(1): 57-84 DOI:10.1007/s10126-015-9673-5 |
LI C, HOU J L, WANG G X, et al. Bioinformatic analysis of SSR markers in transcriptomic sequenceing Paralichthys olivaceus. Marine Fisheries, 2015, 37(2): 122-127 [基于牙鲆RNA-seq数据中SSR标记的信息分析. 海洋渔业, 2015, 37(2): 122-127 DOI:10.3969/j.issn.1004-2490.2015.02.004] |
LI H J, LIU X, DU X D, et al. Development of microsatellite markers in bay scallop and their inheritance patterns in an F1 hybrid family. Marine Sciences, 2009, 33(12): 4-8 [海湾扇贝微卫星标记开发及其分离方式分析. 海洋科学, 2009, 33(12): 4-8] |
LI M, ZHU L, ZHOU C Y, et al. Development and characterization of EST-SSR markers from Scapharca broughtonii and their transferability in Scapharca subcrenata and Tegillarca granosa. Molecules, 2012, 17(9): 10716-10723 DOI:10.3390/molecules170910716 |
LI X L, GUO J L, LI Q, et al. The information analysis of SSR loci in the red claw crayfish (Cherax quadricarinatus) transcriptome. Hubei Agricultural Sciences, 2020, 59(7): 207-211 [红螯螯虾转录组中的SSR位点信息分析. 湖北农业科学, 2020, 59(7): 207-211] |
LIU B Q, ZENG Q G, WANG Y J, et al. The cross-species amplification and validation of EST-SSR loci in Porphyra haitanensis. Acta Hydrobiologica Sinica, 2007, 31(2): 149-154 |
LU W X, CHEN Y X, QI P Z. Analysis of SSR Information in the transcriptional group of Neverita didyma. Journal of Zhejiang Ocean University (Natural Science), 2018, 37(3): 215-220 [扁玉螺转录组SSR信息分析. 浙江海洋大学学报(自然科学版), 2018, 37(3): 215-220 DOI:10.3969/j.issn.1008-830X.2018.03.005] |
LUAN S, KONG J, WANG Q Y, et al. Analysis of mirosatellites in the genome of Kuruma prawn Marsupenaeus japonicus. Progress in Natural Science, 2007, 17(6): 731-740 [日本囊对虾(Marsupenaeus japonicus)基因组微卫星特征分析. 自然科学进展, 2007, 17(6): 731-740 DOI:10.3321/j.issn:1002-008X.2007.06.005] |
MA J, LIU J X, JIANG Z J, et al. Development and identification of SSR markers based on RNA-seq data of Diodon hystrix. South China Fisheries Science, 2020, 16(1): 127-136 [基于RNA-seq数据的密斑刺鲀SSR分子标记开发及鉴定. 南方水产科学, 2020, 16(1): 127-136] |
MA Y C, YAN S H, SU W Q, et al. Practical techniques of breeding and culture of Scapharca subcrenata. Beijing: China Ocean Press, 2008 [毛蚶育苗和养殖实用技术. 北京: 海洋出版社, 2008]
|
MAO L H, LIU J X, DING H Q, et al. Microsatellite characterization analysis and primers design of the whole transcriptome of Curcuma alismatifolia. Molecular Plant Breeding, 2018, 16(22): 7407-7414 [姜荷花全长转录组微卫星特征分析和引物设计. 分子植物育种, 2018, 16(22): 7407-7414] |
NIKOLIC N, FEVE K, CHEVALET C, et al. A set of 37 microsatellite DNA markers for genetic diversity and structure analysis of Atlantic salmon Salmo salar populations. Journal of Fish Biology, 2009, 74(2): 458-466 DOI:10.1111/j.1095-8649.2008.02094.x |
QU N N. Microsatellite DNA isolation and genetic diversity of the pearl oyster Pinctada fucata. Master´s thesis of Huazhong Agricultural University, 2008 [曲妮妮. 合浦珠母贝微卫星DNA标记分离与遗传多样性研究. 华中农业大学硕士研究生学位论文, 2008]
|
REECE K S, RIBEIRO W L, GAFFNEY P M, et al. Microsatellite marker development and analysis in the eastern oyster (Crassostrea virginica): Confirmation of null alleles and non-mendelian segregation ratios. Journal of Heredity, 2004, 95(4): 346-352 DOI:10.1093/jhered/esh058 |
SCHLÖTTERER C. Evolutionary dynamics of microsatellite DNA. Chromosoma, 2000, 109(6): 365-371 DOI:10.1007/s004120000089 |
SONG D D, WANG L, WEI H J, et al. Construction and polymorphism analysis of a microsatellite library in red swamp crayfish Procambarus clarkii. Fisheries Science, 2019, 38(6): 819-826 [克氏原螯虾微卫星文库构建及多态性分析. 水产科学, 2019, 38(6): 819-826] |
SONG F F, WANG Q X, LIU M, et al. Morphological variation analysis of Scapharca subcrenata in China. Periodical of Ocean University of China (Natural Science), 2012, 42(11): 40-45 [不同地区毛蚶群体的形态差异的比较研究. 中国海洋大学学报(自然科学版), 2012, 42(11): 40-45] |
SUN X W, JIA Z Y, WEI D W, et al. Comparison between magnetic beads enriched and small inserted fragment library for microsatellite sequences of common carp. Journal of Fishery Sciences of China, 2005, 12(2): 126-132 [磁珠富集法与小片段克隆法筛选鲤微卫星的比较研究. 中国水产科学, 2005, 12(2): 126-132 DOI:10.3321/j.issn:1005-8737.2005.02.003] |
TAN J, YAO G Y, LIU Z G, et al. SSR information analysis and unigene functional annotation of transcriptome from Argopecten irradians "Bohaihong" and Argopecten irradians concentricus. Genomics and Applied Biology, 2018, 37(12): 5242-5250 [扇贝"渤海红"与墨西哥湾扇贝转录组SSR信息分析及unigene功能注释. 基因组学与应用生物学, 2018, 37(12): 5242-5250] |
TEMNYKH S, DECLERCK G, LUKASHOVA A, et al. Computational and experimental analysis of microsatellites in rice (Oryza sativa L. ): Frequency, length variation, transposon associations, and genetic marker potential. Genome Research, 2001, 11(8): 1441-1452 |
TIAN J T, HOU Y, LIU Z H, et al. Genetic diversity of different populations of Scapharca subcrenata based on mitochondrial COⅠ gene. Marine Sciences, 2016, 40(1): 1-9 [基于线粒体COI基因的毛蚶群体遗传多样性. 海洋科学, 2016, 40(1): 1-9 DOI:10.3969/j.issn.1671-6647.2016.01.001] |
TOTH G, GASPARI Z, JURKA J. Microsatellites in different eukaryotic genomes: Survey and analysis. Genome Research, 2000, 10(7): 967-981 DOI:10.1101/gr.10.7.967 |
WANG R C, WANG Z P. Science of marine shellfish culture. Qingdao: China Ocean University Press, 2008 [海水贝类养殖学. 青岛: 中国海洋大学出版社, 2008]
|
WANG Q Z, ZHANG M, TENG W M, et al. A review: Research progress of biology in ark shell Scapharca subcrenata insight from aquaculture. Journal of Dalian Ocean University, 2015, 30(4): 437-443 [毛蚶养殖生物学研究进展. 大连海洋大学学报, 2015, 30(4): 437-443] |
WANG Z L, DING Y, XU Y H, et al. Polymorphism of EST-SSRs from Pinctada martensii based on transcriptome datasets. Oceanologia et Limnologia Sinica, 2015, 46(3): 687-693 [基于转录组数据的马氏珠母贝EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 2015, 46(3): 687-693] |
WEI D W, LIAN Z Q, WU X D, et al. Preliminary screening of silurus lanzhouensis EST-SSR markers based on Clariidae and Siluridea ESTs. Freshwater Fisheries, 2015, 45(5): 19-23 [基于胡鲇科和鲇科ESTs序列初步筛选兰州鲇EST-SSR分子标记. 淡水渔业, 2015, 45(5): 19-23 DOI:10.3969/j.issn.1000-6907.2015.05.003] |
WU X. Development of SSR and SNP for Onchidiidae based on transcriptome sequencing. Master's Thesis of Shanghai Ocean University, 2016 [吴欣. 基于转录组测序的石磺科贝类SSR和SNP两种分子标记的开发. 上海海洋大学硕士研究生学位论文, 2016]
|
XIA Y, LUO W, YUAN S Q, et al. Microsatellite development from genome skimming and transcriptome sequencing: Comparison of strategies and lessons from frog species. BMC Genomics, 2018, 19: 886 DOI:10.1186/s12864-018-5329-y |
XIONG G, WANG X Q, WANG P, et al. Development and genetic diversity analysis of Babylonia lutosa with EST-SSR markers. Progress in Fishery Sciences, 2020, 41(4): 117-124 [泥东风螺EST-SSR开发及其群体遗传多样性分析. 渔业科学进展, 2020, 41(4): 117-124] |
YAN L L, QIN Y J, YAN X W, et al. Development of microsatellite markers in Ruditapes philippinarum using next-generation sequencing. Acta Ecologica Sinica, 2015, 35(5): 1573-1580 [基于转录组平台的蛤仔微卫星标记筛选. 生态学报, 2015, 35(5): 1573-1580] |
YANG M, YU Y, ZHANG X J, et al. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp (Litopenaus vannamei). Marine Sciences, 2017, 41(2): 96-102 [基于转录组数据的凡纳滨对虾微卫星标记开发. 海洋科学, 2017, 41(2): 96-102] |
YU A Q, SHI Y H, XU J B, et al. Characteristic analysis of microsatellites in selected Coilia ectenes using a transcriptome dataset. Progress in Fishery Sciences, 2019, 40(5): 101-109 [长江刀鲚选育群体转录组EST-SSR的分布特征分析. 渔业科学进展, 2019, 40(5): 101-109] |
ZENG C, GAO Z X, LUO W, et al. Characteristics of microsatellites in blunt snout bream (Megalobrama amblycephala) EST sequences using 454 FLX. Acta Hydrobiologica Sinica, 2013, 37(5): 982-988 [基于454 GS FLX高通量测序的团头鲂ESTs中微卫星特征分析. 水生生物学报, 2013, 37(5): 982-988] |
ZHAI L X, MAO H Q. Biological characteristics and artificial breeding techniques of Scapharca subcrenata. Modern Agricultural Science and Technology, 2010(4): 345-346 [毛蚶的生物学特性及人工育苗技术. 现代农业科技, 2010(4): 345-346 DOI:10.3969/j.issn.1007-5739.2010.04.225] |
ZHAN A B, HU J J, HU X L, et al. Isolation and characterization of microsatellite markers for Zhikong scallop by screening SSR-enriched library. Journal of Fisheries of China, 2008, 32(3): 353-361 [富集文库-菌落原位杂交法筛选栉孔扇贝的微卫星标记. 水产学报, 2008, 32(3): 353-361] |
ZHANG J Y, HE M C, XIANG Z L, et al. Microsatellite screening and characterization analysis of Sepia esculenta based on full-length transcriptome sequencing. Progress in Fishery Sciences, 2020, 41(6): 149-155 [基于全长转录组测序的金乌贼微卫星位点筛选与特征分析. 渔业科学进展, 2020, 41(6): 149-155] |
ZHAO W, ZHANG L, BI J H. ISSR analyses of genetic diversity within and among five stocks of Scapharca subcrenata alone Liaoning coast. Journal of Fisheries of China, 2011, 35(6): 854-862 [辽宁沿海5个毛蚶群体遗传多样性的ISSR分析. 水产学报, 2011, 35(6): 854-862] |



