2. 江苏省海洋生物产业技术协同创新中心 江苏 连云港 222005;
3. 江苏省农业种质资源保护与利用平台 江苏 南京 210014;
4. 连云港赣榆佳信水产开发有限公司 江苏 连云港 222100
2. Jiangsu Marine Biological Industry Technology Collaborative Innovation Center, Lianyungang, Jiangsu 222005, China;
3. Jiangsu Provincial Platform for Conservation and Utilization of Agricultural Germplasm, Nanjing, Jiangsu 210014, China;
4. Lianyungang Ganyu Jiaxin Aquatic Products Development Co., Ltd., Lianyungang, Jiangsu 222100, China
肠道微生物在对虾生长发育过程中发挥调控宿主营养代谢与抵抗病原菌侵染的作用,同时对其免疫功能也有重要影响(Rungrassamee et al, 2016)。而病原微生物的侵染则会破坏肠道菌群结构,使其功能失常,从而引发其他疾病。研究表明,肠道菌群结构及其代谢物的改变与甲壳类的生长和疾病有着密切联系,不同生长阶段和不同健康状态的虾体肠道菌群丰度与多样性存在一定差异,健康虾的肠道菌群多样性显著高于病虾(Xiong et al, 2015; Zheng et al, 2016; Gao et al, 2019)。
脊尾白虾(Exopalaemon carinicauda)是我国池塘单养和虾蟹混养模式常见的一种小型经济虾类,主要分布于黄渤海海域的浅海近岸水域,具有广温广盐、环境适应性强、繁殖能力强、生长快等优点(Zhang et al, 2015)。然而,随着高密度养殖模式与混合养殖模式的发展,病害的发生呈上升趋势,严重制约了虾蟹混养等养殖模式(Xu et al, 2010; 李新苍等, 2012; 王元等, 2013)。虾肝肠胞虫(Enterocytozoon hepatopenaei, EHP)是一种高传染性细胞内寄生的微孢子虫,主要寄生于虾的肝胰腺和肠道中,可通过摄取宿主细胞内的ATP进行繁殖(Santhoshkumar et al, 2017; Boakye et al, 2017; Aranguren et al, 2017; 程东远等, 2018)。EHP孢子肉眼不可见,感染EHP的个体前期无明显的感染症状,患病后期可发现群体内病虾体型大小不一,总体生长迟缓甚至停止生长(刘雅梅等, 2017; Singh et al, 2018)。近年来,在脊尾白虾养殖过程中也有发现EHP感染,对脊尾白虾养殖业造成了一定的损失。
研究健康虾和病虾肠道菌群结构之间的差异及功能有助于阐明疾病的致病机理,指示宿主的健康状态(郁维娜等, 2018)。利用高通量测序技术对病虾的肠道微生物和免疫基因分析,可以从生物学角度对肠道菌群结构、免疫应答以及虾体生长之间的相互作用机制进行阐释(Zhou et al, 2019)。蒋葛等(2019)研究表明,患有急性肝胰腺坏死病(acute hepatopancreatic necrosis disease, AHPND)的凡纳滨对虾(Litopenaeus vannamei)肠道菌群结构单一,弧菌属细菌在病虾肠道中占据优势地位。本研究拟采用Illumina MiSeq高通量测序技术,对感染EHP的脊尾白虾与健康虾肠道菌群进行16S rRNA基因测序,比较其肠道菌群结构多样性与群落差异,并利用Tax4Fun对肠道群落功能进行预测,进一步探究EHP感染对脊尾白虾肠道菌群的影响,以期为EHP的防治提供帮助。
1 材料与方法 1.1 样品采集与EHP人工感染健康虾取自江苏省南通市如东县养殖池塘,体长为(4.5±0.3) cm,体重为(1.3±0.3) g。运回实验室后,随机选取3尾,利用巢式PCR方法检测是否感染EHP,在水温24℃、盐度28、pH 7.9±0.2的条件下暂养1周。暂养期间连续曝气,每日按体重的5%投喂2次人工配合饲料(海大集团海龙牌),并及时清除残饵和排泄物,暂养结束后饥饿24 h。
随机抽取60尾健康个体用于人工感染,健康组与感染组各30尾分别置于50 L的塑料箱中,水温为24℃,盐度为28,pH为7.9±0.2,感染期间连续曝气。健康组正常投喂人工配合饲料,感染组投喂新鲜的EHP病虾肝胰腺及肌肉组织。病虾来源于实验室扩大培养的感染EHP脊尾白虾,体长为(4.8± 0.5) cm,体重为(1.7±0.6) g,投喂前将病虾肠道去除,剪碎肝胰腺和肌肉组织,并充分混匀,按体重的5%每日投喂2次,投喂2 h后及时清理残饵及粪便。每隔2 d换水1次,换水量为总量的1/3。连续感染10 d后,随机选取健康虾与感染虾各9尾,在无菌环境下用无菌刀片与镊子分别取其肝胰腺和肠道组织,3尾虾组织混为1个样品,共3个平行,肝胰腺样品用于PCR检测,肠道样品用于高通量测序,样品置于-80℃保存备用。
1.2 EHP巢式PCR检测取25 mg虾肝胰腺组织,按照Ezup柱式动物基因组DNA抽提试剂盒[生工生物工程(上海)股份有限公司]说明书进行DNA抽提,DNA洗脱溶解后于-20℃保存。
根据GenBank KF362130.1设计巢式PCR引物(外引物:EX-F: AAA AGC CAT TGA GTT TGT TG, EX-R: TTG CCT TCT CCC TCC TGT; 内引物:In-F: TAG CGG AAC GGA TAG GGA, In-R: CCA GCA TTG TCG GCA TAG) (已验证特异性); PCR反应体系(南京诺唯赞)为10 μL,包含3.8 μL ddH2O、5 μL 2×Taq Mix Ⅱ、EX-F/In-F和EX-R/In-R各0.4 μL、0.4 μL DNA。PCR反应程序:94℃预变性5 min,94℃ 45 s、55℃ 45 s、72℃ 1 min,共35个循环,最后72℃延伸7 min。第2轮PCR模板为第1轮产物,体系和程序与第1轮相同。PCR产物用1%琼脂糖凝胶电泳检测,送生工生物工程(上海)股份有限公司进行序列测定。
1.3 肠道总DNA提取与PCR扩增采用CTAB法提取健康虾(J组)和感染虾(G组)肠道总DNA,之后用1%琼脂糖凝胶电泳与分光光度计检测DNA纯度与浓度,将样品用无菌水稀释至1 ng/μL作DNA模板,用Phusion® High-Fidelity PCR Master Mix with GC Buffer (New England Biolabs)和高效高保真酶,PCR扩增引物为:515-F (5′-GTG CCA GCM CCG CGG TAA-3′)和806-R (5′-GGA CTA CHV GGG TWT CTA AT-3′)。所得PCR产物用2%琼脂糖凝胶电泳检测,采用QIAquick胶回收试剂盒(QIAGEN, 德国)回收目的条带。
1.4 上机测序与测序数据处理使用TruSeq DNA PCR-Free Sample Preparation Kit (Illumina)构建文库,文库质检合格后使用NovaSeq 6000进行上机测序(北京诺禾致源生物信息科技有限公司)。对下机数据使用FLASH (V1.2.7, http://ccb.jhu.edu/software/FLASH/)对样品的reads进行拼接、过滤,得到高质量Tags,参照QIIME (quantitative insights into microbial ecology, V1.7.0, http://qiime.org/)流程对Tags进行质控,去除嵌合体序列后得到有效数据(Caporaso et al, 2010)。
1.5 OTU聚类注释与多样性分析利用Uparse软件(V7.0.1001, http://www.drive5.com)对样品的全部Tags以97%的一致性将序列聚类成OTUs (operational taxonomic units)(Haas et al, 2011)。根据聚类结果,筛选出OTUs中出现频率最高的序列尾代表序列,参考Mothur方法与SILVA132 (http://www.arb-silva.de/)的SSUrRNA数据库进行物种注释分析(设定阈值为0.8~1) (Wang et al, 2007; Edgar, 2013)。
利用QIIME (V1.9.1)软件计算Observed-OTUs、Chao1、Shannon、Simpson和ACE等指数,使用R (V2.15.3)软件绘制稀释曲线和Rank abundance曲线,并进行Alpha多样性和Beta多样性指数组间差异分析。
1.6 肠道菌群功能预测采用Tax4Fun对肠道菌群功能进行预测,基于最小16S rRNA序列相似度的最近邻居法实现。经提取KEGG数据库原核生物全基因组16S rRNA基因序列与SILVA SSU Ref NR数据库进行比对并建立数据库,将KEGG数据库原核生物全基因组功能信息通过UProC和PAUDA两种方法进行注释并对应到SILVA数据库中,实现SILVA数据库功能注释。随后以SILVA数据库序列为参考序列聚类出OTU,进而获取功能注释信息(Aßhauer et al, 2015)。
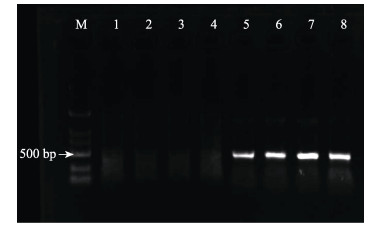
2 结果与分析 2.1 EHP感染检测结果对健康虾与感染虾进行巢式PCR检测,PCR产物经1%琼脂糖凝胶电泳验证(图 1),胶回收产物测序后通过GenBank BLAST同源检索分析,结果与GenBank KF362130.1序列一致。
|
图 1 样品EHP检测 Fig.1 EHP detection of samples M:DL 2000 DNA分子量标准; 1:阴性对照; 2~4:健康虾; 5:阳性对照; 6~8:感染虾 M: DL 2000 DNA marker; 1: Negative control; 2~4: Healthy shrimp; 5: Positive control; 6~8: Diseased shrimp |
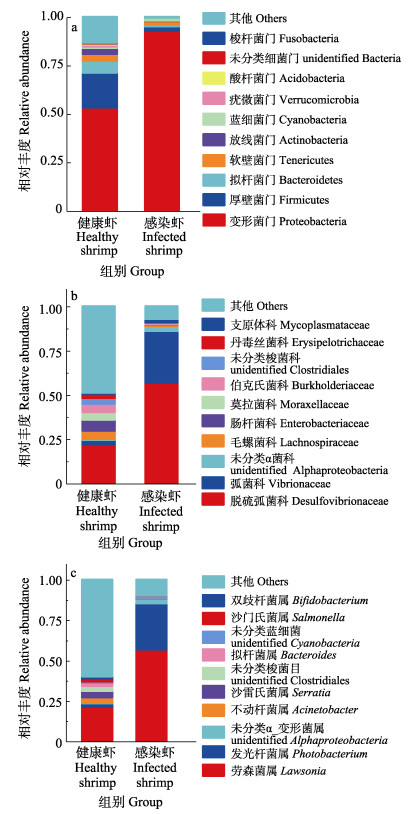
实验虾肠道微生物样品在97%的相似性水平上共获得2314个OTUs (472 451个reads)。归属于35个门、55个纲、122个目、233个科、542个属、318个种。如图 2a所示,感染虾中主要菌群为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、软壁菌门(Tenericutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),其相对丰度分别为92.64%、2.39%、1.88%、0.88%和0.24%,占细菌总量的98%。感染虾中变形菌门与拟杆菌门丰度显著高于健康虾(P < 0.05),而厚壁菌门、拟杆菌门和放线菌门丰度显著低于健康虾(P < 0.05)。
|
图 2 健康虾与感染虾肠道主要细菌物种相对丰度(n=3, x±SD) Fig.2 Relative abundance of intestinal bacteria in healthy and infected shrimp (n=3, x±SD) a:细菌门水平上物种相对丰度; b:细菌科水平上物种相对丰度; c:细菌属水平上物种相对丰度 a: Relative abundance of species at bacterial phylum level; b: Relative abundance of species at bacterial family level; c: Relative abundance of species at bacterial genus level |
如图 2b和图 2c所示,感染虾中优势菌为劳森菌属(Lawsonia)、发光杆菌属(Photobacterium)、未分类α变形菌属(unidentified Alphaproteobacteria)、不动杆菌属(Acinetobacter)和沙雷氏菌属(Serratia)等,主要归属于脱疏弧菌科(Desulfovibrionaceae)、弧菌科(Vibrionaceae)、莫拉式菌科(Moraxellaceae)和肠杆菌科(Enterobacteriaceae),分别归属于变形菌门和厚壁菌门。感染虾中变形菌门丰度显著高于健康虾(P < 0.05),而厚壁菌门丰度则显著低于健康虾(P < 0.05)。
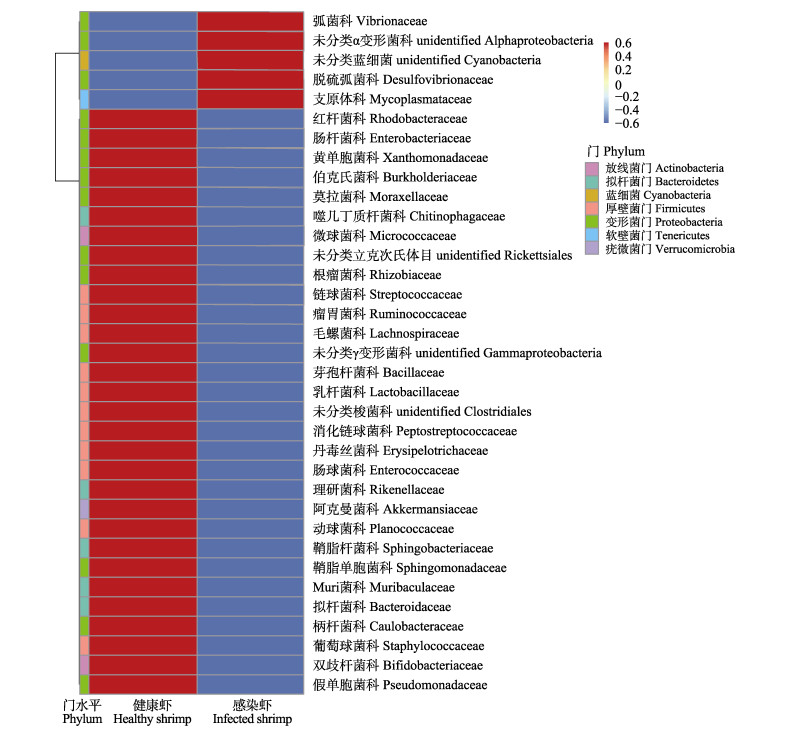
根据所有样本在科水平的物种注释及丰度信息,选取丰度排名前35的科,根据其在每个样本中的丰度信息,从物种和样本2个层面进行聚类,绘制成热图(图 3),感染虾共有5个优势菌科,健康虾共有30个优势菌科,分别归属于7个门。健康虾的30个优势菌科中共有4个潜在益生菌科,分别是乳杆菌科(Lactobacillaceae)、双歧杆菌科(Bifidobacteriaceae)、芽孢杆菌科和噬几丁质杆菌科(Chitinophagaceae)。感染虾中5个优势菌科分别是脱硫弧菌科、弧菌科、未分类蓝细菌科(unidentified Cyanobacteria)、支原体科(Mycoplasmataceae)和未分类α变形菌科(unidentified Alphaproteobacteria)。
|
图 3 健康虾与感染虾物种丰度聚类图(科水平)(n=3, x±SD) Fig.3 Cluster heatmap of healthy shrimp and infected shrimp (Family level)(n=3, x±SD) |
对不同样品在97%一致性阈值下的多样性分析指数进行统计分析(表 1),结果显示,样品覆盖率大于99%。对Shannon指数、Simpson指数、Chao1指数和ACE指数进行比较,感染组各指数均低于健康组,通过分析比较可以发现,感染组个体的肠道菌群多样性显著低于健康虾(P < 0.05)。
|
|
表 1 α多样性统计表(n=3;x±SD) Tab.1 Alpha diversity indices table (n=3;x±SD) |
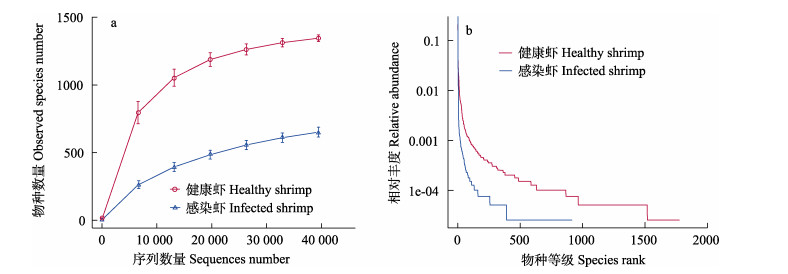
稀释曲线和等级聚类曲线可反映测序数据量的合理性,直观反映样品中物种的丰富度和均匀度(Lundberg et al, 2013)。图 4a为样品稀释曲线,由图 4a可见,稀释曲线趋于平缓,说明测序数据合理。图 4b为样品等级聚类曲线,曲线越宽说明样品丰富度越高,越平坦说明样品分布越均匀。由图 4b可见,与感染虾相比,健康虾的曲线更宽、更平缓。这与α多样性分析结果一致,可见感染虾肠道菌群多样性与丰富度显著低于健康虾(P < 0.05)。
|
图 4 健康虾与感染虾样品物种多样性曲线(n=3, x±SD) Fig.4 Species diversity curve of healthy and infected shrimp samples (n=3, x±SD) a:样品稀释曲线; b:等级聚类曲线 a: Rarefaction curve of sample; b: Rank abundance of sample |
功能注释聚类热图(Level 1)的结果显示(图 5),健康虾肠道菌群的主要功能包括细胞过程、未分类、环境信息处理、有机系统等,感染虾肠道菌群的主要功能包括新陈代谢、人类疾病和遗传信息处理。T-test检验结果显示,二组之间并无显著性差异(P > 0.05)。
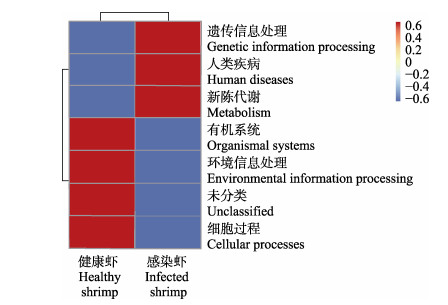
|
图 5 Tax4Fun功能注释聚类热图(Level 1)(n=3, x±SD) Fig.5 Tax4Fun functional annotation cluster heatmap (Level 1)(n=3, x±SD) |
肠道菌群在虾体健康生长发育与非特异性免疫调控过程中扮演重要角色,可促进虾体对营养的吸收及新陈代谢,为其抵抗病害侵袭提供屏障(Nie et al, 2017; Libertucci et al, 2018)。研究表明,凡纳滨对虾肠道菌群中丰度较高的菌群为变形菌门、拟杆菌门和厚壁菌门,主要包括弧菌属(Vibrio)、假单胞菌属(Pseudomonas)、拟杆菌属(Bacteroides)和蓝细菌(Cyanobacteria)等,同时与虾体不同健康状况有关(Zheng et al, 2016; Landsman et al, 2019; Zeng et al, 2020),这与本研究结果相似。本研究中,感染虾与健康虾肠道菌群丰度差异较大,感染虾肠道优势菌多归属于变形菌门与拟杆菌门,其丰度显著高于健康虾,而健康虾中厚壁菌门丰度则显著高于感染虾。研究发现,脊尾白虾肠道优势菌群为变形菌门和厚壁菌门,其中丰度较高的有假单胞菌属、肠杆菌属(Enterobater)、弧菌属、赤杆菌属(Erythrobacter)等(沈辉等, 2015)。本研究中,弧菌科(变形菌纲)丰度在感染虾中属于优势菌,弧菌病可对虾类产生较大的伤害,破坏其组织器官,造成非特异性免疫与代谢功能下降,导致大量死亡(Khimmakthong et al, 2017)。
Xiong等(2014)研究表明,从科水平上可简单有效对虾体健康进行预测。本研究对科水平OTUs分类热图进行分析,发现感染虾中优势潜在致病菌为脱硫弧菌科、弧菌科、未分类蓝细菌科、支原体科和未命名α_变形菌科,其弧菌科丰度显著高于健康虾(P < 0.05),可能是EHP的侵染破坏了虾肠道菌群结构,益生菌群的抑菌功能遭破坏,以至于致病菌群占据优势,增加了弧菌病的发病几率(Zokaeifar et al, 2014; Santhoshkumar et al, 2017; Aranguren et al, 2017)。健康虾中的乳杆菌科、芽孢杆菌科、双歧杆菌科的某些属可分泌多种胞外酶和抗菌肽,不仅可抑制致病菌繁殖,还可改善水质,提高水产动物的非特异性免疫(Zokaeifar et al, 2014; Lugli et al, 2017)。因此,推测在EHP感染初期向养殖水体或饲料中添加这些益生菌有望减缓或抑制EHP的传播(宁梓健等, 2020)。
3.2 肠道微生物多样性分析Zhou (2019)研究发现,“棉花状”的凡纳滨对虾肠道与健康虾肠道中优势菌群分属于变形菌门、软壁菌门和拟杆菌门,健康虾肠道菌群多样性显著高于病虾组,与本研究结果相似。吴金凤等(2016)通过高通量测序技术对凡纳滨对虾肠道及养殖水体进行检测,同样发现健康虾肠道微生物多样性显著高于病虾。在本研究中,受EHP感染的个体肠道微生物多样性显著低于健康个体,表明EHP的侵染对脊尾白虾肠道菌群多样性造成了一定的影响。EHP多和其他疾病共同发现,肠道菌群结构遭到破坏后,变形菌门丰度显著增加,厚壁菌门丰度显著降低,导致潜在致病菌暴发几率增加,感染虾更容易暴发弧菌病等疾病(Aranguren et al, 2017)。
虾肠道菌群多样性关系到虾体肠道群落生态功能稳定性,多样性越高说明肠道菌群越稳定,抗病能力越强(金若晨等, 2020)。本研究中,健康虾肠道内也存在多种潜在致病菌,但并没有出现患病症状,这与健康虾肠道菌群结构有一定关联。通过物种稀释曲线与等级聚类曲线可以看出,健康虾肠道菌群多样性更高,这与Shannon-Wiener指数和Simpson指数等多样性指数所表达的结果也一致。因此,健康虾肠道菌群结构更为完整,对致病菌的抑制能力更强。
3.3 肠道功能预测虾体感染EHP后群体间个体差异较大,出现生长缓慢甚至停止生长的现象。通过Tax4Fun功能预测,发现感染虾的肠道主要功能为新陈代谢和人类疾病,可能由于感染虾分配更多的能量用于抵抗EHP的侵染和其他疾病,减少了用于生长相关的能量; 而从健康虾的功能预测中可以看出,其将更多的能量用于生长和对环境的适应,从而保证安全存活与繁殖(Zhou et al, 2019)。
4 结论综上所述,健康虾与感染EHP虾的肠道菌群差异较大,其中感染虾肠道菌群结构多样性显著低于健康虾。通过16S rRNA基因测序分析,发现病虾中归属于变形菌门的脱硫弧菌科、弧菌科、未分类蓝细菌科、支原体科和未分类α变形菌科为优势菌,而归属于厚壁菌门的乳杆菌科、双歧杆菌科、芽孢杆菌科和噬几丁质菌科细菌在健康虾中占据优势地位。由此可见,EHP的侵蚀破坏了脊尾白虾肠道菌群结构,导致潜在致病菌丰度增加。
aARANGUREN L F, HAN J E, TANG K F J. Enterocytozoon hepatopenaei (EHP) is a risk factor for acute hepatopancreatic necrosis disease (AHPND) and septic hepatopancreatic necrosis (SHPN) in the Pacific white shrimp Penaeus vannamei. Aquaculture, 2017, 471: 37-42 DOI:10.1016/j.aquaculture.2016.12.038 |
AßHAUER K P, WEMHEUER B, DANIEL R, et al. Tax4Fun: Predicting functional profiles from metagenomic 16S rRNA data. Bioinformatics, 2015, 31(17): 2882-2884 DOI:10.1093/bioinformatics/btv287 |
BOAKYE D W, JAROENLAK P, PRACHUMWAT A, et al. Decay of the glycolytic pathway and adaptation to intranuclear parasitism within Enterocytozoonidae microsporidia. Environmental Microbiology, 2017, 19(5): 2077-2089 DOI:10.1111/1462-2920.13734 |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336 DOI:10.1038/nmeth.f.303 |
CHENG D Y, QIU L, SONG Z L, et al. Differences between populations and tissues of Litopenaeus vannamei infected with Enterocytozoon hepatopenaei. Progress in Fishery Sciences, 2018, 39(4): 83-92 [程东远, 邱亮, 宋增磊, 等. 凡纳滨对虾感染虾肝肠胞虫的群体及组织间差异性分析. 渔业科学进展, 2018, 39(4): 83-92] |
EDGAR R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998 DOI:10.1038/nmeth.2604 |
GAO S, PAN L Q, HUANG F, et al. Metagenomic insights into the structure and function of intestinal microbiota of the farmed Pacific white shrimp (Litopenaeus vannamei). Aquaculture, 2019, 499: 109-118 DOI:10.1016/j.aquaculture.2018.09.026 |
HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454- pyrosequenced PCR amplicons. Genome Research, 2011, 21(3): 494-504 DOI:10.1101/gr.112730.110 |
JIANG G, SHEN H, WAN X H, et al. Difference analysis of intestinal flora between healthy Litopenaeus vannamei and acute hepatopancreatic necrosis syndrome L. vannamei. Jiangsu Journal of Agricultural Sciences, 2019, 35(1): 142-148 [蒋葛, 沈辉, 万夕和, 等. 凡纳滨对虾急性肝胰腺坏死综合症病虾与健康虾肠道优势菌群比较分析. 江苏农业学报, 2019, 35(1): 142-148 DOI:10.3969/j.issn.1000-4440.2019.01.021] |
JIN R C, JIANG M, SUN S Y, et al. Microbial community in the Litopenaeus vannamei intestine and its aquaculture environment. Journal of Fisheries of China, 2020, 44(12): 2037-2054 [金若晨, 江敏, 孙世玉, 等. 凡纳滨对虾养殖环境及肠道微生物群落特征分析. 水产学报, 2020, 44(12): 2037-2054] |
KHIMMAKTHONG U, SUKKARUN P. The spread of Vibrio parahaemolyticus in tissues of the Pacific white shrimp Litopenaeus vannamei analyzed by PCR and histopathology. Microbial Pathogenesis, 2017, 113: 107-112 DOI:10.1016/j.micpath.2017.10.028 |
LANDSMAN A, ST-PIERRE B, ROSALES-LEIJA M, et al. Impact of aquaculture practices on intestinal bacterial profiles of Pacific whiteleg shrimp Litopenaeus vannamei. Microorganisms, 2019, 7(4): 93 DOI:10.3390/microorganisms7040093 |
LI X C, ZHOU J F, FANG W H, et al. Development and application of an economical real-time PCR for WSSV detection and quantification in ridgetail white prawns (Exopalaemon carinicauda). Journal of Fisheries of China, 2012, 36(10): 1554-1562 [李新苍, 周俊芳, 房文红, 等. 实用WSSV定量检测方法的建立及其应用于脊尾白虾病毒感染规律的研究. 水产学报, 2012, 36(10): 1554-1562] |
LIBERTUCCI J, YOUNG V B. The role of the microbiota in infectious diseases. Nature Microbiology, 2018, 4(1): 35-45 |
LIU Y M, QIU L, CHENG D Y, et al. The relationship of body length and weight in the Litopenaeus vannamei populations detected Enterocytozoon hepatopenaei. Progress in Fishery Sciences, 2017, 38(4): 96-103 [刘雅梅, 邱亮, 程东远, 等. 检出虾肝肠胞虫(Enterocytozoon hepatopenaei)的凡纳滨对虾(Litopenaeus vannamei)群体的体长和体重关系. 渔业科学进展, 2017, 38(4): 96-103] |
LUGLI G A, MILANI C, TURRONI F, et al. Comparative genomic and phylogenomic analyses of the Bifidobacteriaceae family. BMC Genomics, 2017, 18: 568 DOI:10.1186/s12864-017-3955-4 |
LUNDBERG D S, YOURSTONE S, MIECZKOWSKI P, et al. Practical innovations for high-throughput amplicon sequencing. Nature Methods, 2013, 10(10): 999-1002 DOI:10.1038/nmeth.2634 |
NIE L, ZHOU Q J, QIAO Y, et al. Interplay between the gut microbiota and immune responses of ayu (Plecoglossus altivelis) during Vibrio anguillarum infection. Fish and Shellfish Immunology, 2017, 68: 479-487 DOI:10.1016/j.fsi.2017.07.054 |
NING Z J, JIANG H B, LIU Q, et al. Genome-wide prediction and analysis of secreted proteins of Enterocytozoon hepatopenaei. Progress in Fishery Sciences, 2020, 41(6): 165-173 [宁梓健, 姜宏波, 刘琦, 等. 虾肝肠胞虫全基因组分泌蛋白的预测分析. 渔业科学进展, 2020, 41(6): 165-173] |
RUNGRASSAMEE W, KLANCHUI A, MAIBUNKAEW S, et al. Bacterial dynamics in intestines of the black tiger shrimp and the Pacific white shrimp during Vibrio harveyi exposure. Journal of Invertebrate Pathology, 2016, 133: 12-19 DOI:10.1016/j.jip.2015.11.004 |
SANTHOSHKUMAR S, SIVAKUMAR S, VIMAL S, et al. Biochemical changes and tissue distribution of Enterocytozoon hepatopenaei (EHP) in naturally and experimentally EHP-infected whiteleg shrimp, Litopenaeus vannamei (Boone, 1931), in India. Journal of Fish Diseases, 2017, 40(4): 529-539 DOI:10.1111/jfd.12530 |
SHEN H, WAN X H, HE P M, et al. Bacterial community structure in the intestine of Exopalaemon carinicauda Holehuis. Microbiology, 2015, 42(10): 1922-1928 [沈辉, 万夕和, 何培民, 等. 脊尾白虾肠道微生物菌群结构. 微生物学通报, 2015, 42(10): 1922-1928] |
SINGH M, SINGH P. Enterocytozoon hepatopenaei: A microsporidian in the midst of serious threat to shrimp aquaculture. Journal of Entomology and Zoology Studies, 2018, 6(6): 936-939 |
WANG Q, GARRITY G M, TIEDJE J M, et al. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267 DOI:10.1128/AEM.00062-07 |
WANG Y, FANG W H, ZHOU J F, et al. Pathogenic and pathological analysis of the muscular microsporidiasis of Exopalaemon carinicauda. Journal of Shanghai Ocean University, 2013, 22(5): 726-733 [王元, 房文红, 周俊芳, 等. 脊尾白虾肌肉微孢子虫病的病原和病理分析. 上海海洋大学学报, 2013, 22(5): 726-733] |
WU J F, XIONG J B, WANG X, et al. Intestinal bacterial community is indicative for the healthy status of Litopenaeus vannamei. Chinese Journal of Applied Ecology, 2016, 27(2): 611-621 [吴金凤, 熊金波, 王欣, 等. 肠道菌群对凡纳滨对虾健康的指示作用. 应用生态学报, 2016, 27(2): 611-621] |
XIONG J B, WANG K, WU J F, et al. Changes in intestinal bacterial communities are closely associated with shrimp disease severity. Applied Microbiology and Biotechnology, 2015, 99(16): 6911-6919 DOI:10.1007/s00253-015-6632-z |
XIONG J B, ZHU J L, ZHANG D M. The application of bacterial indicator phylotypes to predict shrimp health status. Applied Microbiology and Biotechnology, 2014, 98(19): 8291-8299 DOI:10.1007/s00253-014-5941-y |
XU W J, XIE J J, SHI H, et al. Hematodinium infections in cultured ridgetail white prawns, Exopalaemon carinicauda, in eastern China. Aquaculture, 2010, 300(1/2/3/4): 25-31 |
YU W N, DAI W F, TAO Z, et al. Characterizing the compositional and functional structures of intestinal micro-flora between healthy and diseased Litopenaeus vannamei. Journal of Fisheries of China, 2018, 42(3): 399-409 [郁维娜, 戴文芳, 陶震, 等. 健康与患病凡纳滨对虾肠道菌群结构及功能差异研究. 水产学报, 2018, 42(3): 399-409] |
ZENG S Z, KHORUAMKID S, KONGPAKDEE W, et al. Dissimilarity of microbial diversity of pond water, shrimp intestine and sediment in aquamimicry system. AMB Express, 2020, 10: 180 DOI:10.1186/s13568-020-01119-y |
ZHANG C S, LI Z G, LI F H, et al. Effects of starvation on survival, growth and development of Exopalaemon carinicauda larvae. Aquaculture Research, 2015, 46(9): 2289-2299 DOI:10.1111/are.12386 |
ZHENG Y F, YU M, LIU Y, et al. Comparison of cultivable bacterial communities associated with Pacific white shrimp (Litopenaeus vannamei) larvae at different health statuses and growth stages. Aquaculture, 2016, 451: 163-169 DOI:10.1016/j.aquaculture.2015.09.020 |
ZHOU L, CHEN C Z, XIE J, et al. Intestinal bacterial signatures of the"cotton shrimp-like"disease explain the change of growth performance and immune responses in Pacific white shrimp (Litopenaeus vannamei). Fish and Shellfish Immunology, 2019, 92: 629-636 DOI:10.1016/j.fsi.2019.06.054 |
ZOKAEIFAR H, BABAEI N, SAAD C R, et al. Administration of Bacillus subtilis strains in the rearing water enhances the water quality, growth performance, immune response, and resistance against Vibrio harveyi infection in juvenile white shrimp, Litopenaeus vannamei. Fish and Shellfish Immunology, 2014, 36(1): 68-74 DOI:10.1016/j.fsi.2013.10.007 |



