2. 上海海洋大学 水产科学国家级实验教学示范中心 上海 201306;
3. 中国海洋大学三亚海洋研究院 热带海洋生物种质资源开发与种业工程实验室 海南 三亚 572024
2. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China;
3. Laboratory of Tropical Marine Germplasm Resources and Breeding Engineering, Sanya Oceanographic Institution, Ocean University of China, Sanya, Hainan 572024, China
紫菜(Porphyra sensu lato,亦称laver)是一种深受世界人民喜爱的高营养价值海洋红藻,其广泛栽培主要集中在中国、韩国和日本等亚洲国家。由于部分海区的水质贫瘠、潮流不通、水体污染、温度不适、养殖管理不当等因素,近年来我国紫菜栽培业饱受频发病害的困扰(黄健等, 2000),部分栽培海区的病烂面积可达40%~50% (Yan et al, 2019; 何礼娟等, 2021)。拟油壶菌病(Oplidiopsis disease)是海上栽培紫菜的重要病害之一,可在短时间内蔓延并导致大面积病烂(Klochkova et al, 2016)。已确定该病病原属于专性细胞内寄生的卵菌纲拟油壶菌属(Olpidiopsis),已发现的Olpidiopsis pyropiae、O. porphyrae、O. porphyrae var. korenae、O. porphyrae var. scotiae、Olpidiopsis bostrychiae、O. heterosiphoniae以及O. muelleri等7种菌均具有感染紫菜的能力(West et al, 2006; Sekimoto et al, 2008、2009; Klochkova et al, 2016; Kwak et al, 2017; Badis et al, 2019)。我国最早于1992年在江苏省南部栽培的条斑紫菜(Neopyropia yezoensis,原名Pyropia yezoensis [Yang et al, 2020])中发现该病(马家海, 1992),并先后在栽培条斑紫菜和坛紫菜(Neoporphyra haitanensis, 原名Porphyra haitanensis [Yang et al, 2020])中发现感染该病(马家海等, 2007; 刘一萌等, 2012; 何礼娟等, 2021)。拟油壶菌病是对紫菜栽培威胁最大的病害之一,目前对病原尚没有体外分离培养的方法(Sekimoto et al, 2008),因此,对其致病机制还知之甚少,也缺乏对该病害长期有效的防控方法。已经施用的措施包括降低栽培密度、网帘干出以及酸洗等,但效果并不理想(Kim et al, 2014)。因此,探索病害发生的机理及其生物防控方法十分有必要。
近年来,附生细菌逐渐成为探究藻类病害发生机理和生物防控的热点和靶点。这是因为藻类附生细菌除了为藻类生长提供重要的营养物质和生长因子、帮助藻类完成生命周期等重要作用(Goecke et al, 2010; Singh et al, 2014),还与藻类病害密切相关。一方面,环境胁迫条件下藻类附生微生物的生态失调是导致病害的重要原因。某些致病微生物类群或其毒力相关的功能基因群在受胁迫个体上大量富集并最终致病(Egan et al, 2014、2016)。另一方面,藻类附生细菌还能通过分泌抗菌物质或群感效应(quorum sensing, QS)抑制剂等次级代谢产物干扰和抵抗致病菌的附着,保护海藻不受环境中病原的侵染等(Egan et al, 2014)。经统计,藻类附生细菌中约有12%~50%的菌株具有抗菌活力,是生防菌和抗病物质的重要来源(Singh et al, 2015)。
此外,对植物和动物卵菌病害的研究也充分体现了附生细菌在促病、防病方面的重要作用。一方面,植物表面附生细菌可产生卵菌毒力必需的细菌毒素或通过共感染以加强卵菌效应基因表达来促进卵菌的致病性(Larousse et al, 2017)。另一方面,附生细菌能抑制卵菌病原的附着或者利用其基因产物抑制卵菌孢子的萌发及菌丝的生长。例如,大西洋鲑(Salmo salar L.)的卵受水霉病侵染时,低发病率的鱼卵上附着的高丰度叶居菌属(Frondihabitans)细菌能有效抑制病原的附着(Liu et al, 2014);而阴沟肠杆菌(Enterobacter cloacae)则能利用fadB和fadL基因表达的氧化酶和蛋白与终极腐霉(Pythium ultimum)竞争植物产生的不饱和脂肪酸,从而抑制腐霉的萌发并达到抑病的效果(van Dijk et al, 2000)。
然而迄今为止,附生细菌与紫菜拟油壶菌病的关系尚未明确,一定程度上阻碍了附生细菌在促病、抗病方面的研究进展。因此,研究紫菜附生细菌与拟油壶菌病之间的关系,对病害的生物防控具有十分重要的意义。本研究分析了受拟油壶菌病感染的不同健康状态下的条斑紫菜附生菌群变化,拟为探究病害发生的微生态机制及寻找潜在生防细菌类群提供一定的基础数据。
1 材料与方法 1.1 样品采集样品采集地点为条斑紫菜的重要栽培区域,位于江苏省盐城市大丰区沿岸东沙海区(121°03′25.88″E, 33°20′11.28″N),海区面积约为1333 hm2。2018年10月—2019年1月,该海区条斑紫菜栽培面积共约333 hm2。2019年1月4日开始,约133 hm2区域条斑紫菜发生病烂,病烂面积达40%左右。从该区域选择3种不同状况的网帘进行条斑紫菜采样:网帘上紫菜未发生任何病烂(PyOlpH)、网帘上部分紫菜已发生病烂(PyOlpM)以及网帘上紫菜已经发生严重病烂(PyOlpS)(图 1)。每种类型网帘均采集3个生物重复,每个生物重复采集同一网帘上相对较为完整的多株(30~40株)条斑紫菜藻体,均匀混合后置于无菌采样袋中,同时用无菌采水袋采集网帘下部的原位海水以浸没采样袋中的紫菜样品。采集好的紫菜样品存放于低温保温箱中带回实验室进行后续研究。在每个生物重中随机挑选5~6株条斑紫菜,通过镜检确认感染情况。然后,在每个生物重复中再随机挑选5~6株紫菜藻体,混合后用于附生菌群DNA提取及微生物群落分析。

|
图 1 不同健康状态下的紫菜栽培网帘比较 Fig.1 Comparison of laver cultivation nets in different statuses |
病理观察及病原鉴定过程见何礼娟等(2021)。将紫菜样品用无菌海水冲洗3~5次后用灭菌的毛笔去除表面杂质,并用无菌刀片切取面积约为1 cm2的条斑紫菜典型病烂部位,在显微镜下观察后,记录病烂紫菜的组织病理变化。未发生病烂网帘的紫菜则随机切取相同面积的组织进行显微观察。
用真菌基因组DNA提取试剂盒(思科捷, 青岛)提取PyOlpM和PyOlpS组样品基因组DNA,并分别用真菌ITS通用引物ITS5和ITS4 (White et al, 1990)、腐霉(Pythium) cox1序列特异引物cox1-pyth-F和cox1-pyth-R (Lee et al, 2015)以及拟油壶菌cox1特异引物cox1-olpi-F和cox1-olpi-R (Kwak et al, 2017)进行PCR扩增。最后,PCR产物用1%琼脂糖凝胶电泳进行检测,并将目的片段胶回收、T载体连接及转化,将阳性克隆进行测序分析。所得序列在NCBI中进行BLAST同源性比对,并在MEGA-X软件中用邻接法(Neighbor-Joining, N-J)构建系统发育进化树,模型选择为Kimura2-paramete (Bootstrap设置为1000)。
1.3 附生菌群DNA提取、PCR扩增及高通量测序紫菜样品表面附生细菌群落DNA提取参照Burke等(2009)的方法并做适当修改:每个生物重复的5~6株紫菜藻体先用灭菌海水清洗3次以去除松散结合的浮游微生物;然后,将藻体置于15 mL CMFSW缓冲液(含0.45 mol/L NaCl、10 mmol/L KCl、7 mmol/L Na2SO4和0.5 mmol/L NaHCO3)中,并添加10 mmol/L EDTA和150 µL过滤除菌的快速多酶消化液(3M, 澳大利亚)于室温下孵育2 h;移除藻体后,清液用等体积的酚∶氯仿∶异戊醇(25∶24∶1)和氯仿∶异戊醇(24∶1)各抽提一次,取上清液并加入3倍体积的无水乙醇和0.1倍体积的醋酸钠溶液(3 mol/L, pH 5.2),混合均匀后于−20℃过夜沉淀;4℃20000×g离心30 min获得DNA沉淀后,用70%的乙醇溶液洗涤2次;最后,用200 µL的无酶灭菌水溶解DNA并用琼脂糖凝胶电泳确定DNA片段大小及质量。
取2 µL DNA,用Q5高保真DNA聚合酶及地球微生物组计划(Earth Microbiome Project)推荐的16S rRNA基因通用引物515F (5′-GTGYCAGCMGCCGCG GTAA-3′)和926R(5′-CCGYCAATTYMTTTRAGTTT -3′)扩增V4~V5可变区(Walters et al, 2016)。每个样品进行扩增时,515F引物5′端会附加6碱基的barcode以对不同样品加以区分。扩增条件:98℃预变性2 min;98℃变性15 s,55℃退火30 s,72℃延伸30 s,共25~27个循环;72℃后延伸5 min。每个样品的PCR产物用AxyPrepDNA凝胶回收试剂盒(Axygen, 美国)切胶回收,并用Quant-iT PicoGreen dsDNA Assay Kit (ThermoFisher Scientific, 美国)对PCR产物进行定量。PCR扩增子在上海派森诺公司用Illumina MiSeq平台进行2×300的双端测序。
1.4 数据分析每个样品的双端下机序列用USEARCH 10.0.240 (Edgar, 2010)进行拼接、质控、去除嵌合体,并按照97%的相似度聚类成可操作分类单元(operational taxonomic unit, OTU)。采用RDP classifier贝叶斯算法在QIIME 1.9.1 (Caporaso et al, 2010)中基于SILVA132数据库对OTU代表序列进行分类(cutoff值设为0.8)并生成OTU表,将注释为Unassignable、Unclassified、Chloroplast以及Mitochondria等的OTU信息从OTU表中去除,所得OTU表用以计算α多样性、β多样性和附生菌群组成等。用R (R core team, 2014)和Excel对微生物多样性及群落进行统计分析,包括各个类群相对丰度统计、α多样性指数的单因素方差分析(one-way ANOVA)、韦恩图分析、主坐标分析(PCoA)及置换多元方差分析(PERMANOVA)。
1.5 差异菌群分析用LEfSe [line discriminant analysis (LDA) effect size] (Segata et al, 2011)分析不同感染程度紫菜与未感染紫菜附生菌群中的差异类群。其中,本研究中LEfSe分析的参数分别设定为LDA score > 2.5,α=0.05。
1.6 共现网络分析用相对丰度前20的细菌属构建共现网络(co- occurrence network)。用pysch包中的corr.test ()函数在R中计算各细菌属间成对的Spearman相关系数矩阵,并用Gephi 0.9.2 (Bastian et al, 2009)对共现网络进行可视化。网络图中的每一个节点(node)均代表某个细菌属,节点之间的连线(也叫作边, edge)表示2个细菌属间具有显著的Spearman相关性(|r| > 0.6, 且FDR校正的P < 0.01)。
2 结果 2.1 病理观察及病原鉴定结果不同状况网帘上紫菜的显微观察结果见图 2。如图 2所示,PyOlpH组紫菜样品未观察到任何感染迹象。而PyOlpM和PyOlpS组样品在显微镜下观察到紫菜细胞受拟油壶菌感染后呈现的典型病理变化特征(红色箭头),且后者病变程度明显高于前者。详细的病理观察结果及分析见何礼娟等(2021)。

|
图 2 不同状态网帘上条斑紫菜的显微观察 Fig.2 Microscopic observation of N. yezoensis on cultivation nets in different statuses PyOlpS组条斑紫菜显微观察结果何礼娟等(2021) Microscopic observation of N. yezoensis in PyOlpS is a quotation from He et al (2021) |
利用ITS通用引物、腐霉cox1特异引物及拟油壶菌cox1特异引物对患病紫菜DNA进行检测,发现仅ITS通用引物和拟油壶菌cox1特异引物出现扩增条带(图 3A),经测序及构建N-J进化树发现该病原与O. pyropiae聚为一支(图 3B),说明引起大丰地区紫菜病烂的病原为拟油壶菌。
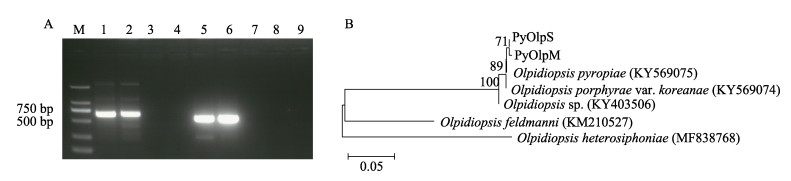
|
图 3 江苏省大丰病烂条斑紫菜病原鉴定
Fig.3 Identification of pathogen causing N. yezoensis disease in Dafeng, Jiangsu Province
A:ITS和cox1基因扩增的琼脂糖凝胶电泳图;B:基于cox1基因构建的N-J系统进化树 M:DL2000 DNA分子量标准;1、2:严重和中度病烂紫菜ITS扩增;3、4:严重和中度病烂紫菜的腐霉cox1扩增;5、6:严重和中度病烂紫菜的拟油壶菌cox1扩增;7、8和9分别为ITS、腐霉cox1和拟油壶菌cox1阴性对照(无菌水作模板) A: Argarose gel elecgtrophoresis for the amplification of ITS and cox1; B: N-J phylogenetic tree constructed based on cox1 gene sequences M: DL 2000 DNA marker; 1, 2: ITS amplification for PyOlpS and PyOlpM; 3, 4: Pythium cox1 amplification for PyOlpS and PyOlpM; 5, 6: Olpidiopsis cox1 amplification for PyOlpS and PyOlpM; 7, 8 and 9: Negative controls using sterilized water as template for ITS, Pythium cox1 and Olpidiopsis cox1 amplification, respectively |
各样品16S rRNA基因高通量测序结果见表 1。如表 1所示,下机序列经过拼接、质控等预处理,共获得有效序列283 911条。经过OTU聚类、代表序列注释以及剔除无效序列等,共获得247 657条高质量序列,分属于432个OTU。对测序覆盖度进行统计,结果显示,各样本Good′s coverage指数均大于0.99 (表 1),表明该测序深度能够在很大程度上反映各样品中的附生菌群多样性。
|
|
表 1 各样本测序结果统计 Tab.1 Statistics of the sequencing results of all samples |
用Observed species (Obs)、Chao1、PD whole tree (PD)和Shannon等指数来表征α多样性(图 4)。各组之间的多样性差异均不显著(P > 0.05, ANOVA, Tukey´s HSD test)。但是,PyOlpM组样品的4个α多样性指数(分别为133.00、143.00、11.01和5.30)均高于PyOlpH组(分别为122.00、134.00、10.40和5.26)和PyOlpS组(分别为125.00、132.00、9.91和4.41),而PyOlpS组除了Obs高于PyOlpH组外,其余指数也均低于PyOlpH组。
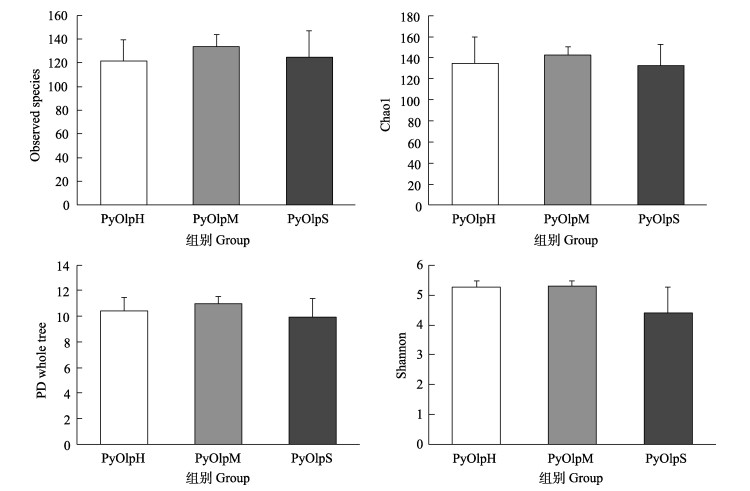
|
图 4 不同状态紫菜附生菌群的α多样性指数比较 Fig.4 Comparison of α diversity indices of epiphytic microbial communities of N. yezoensis in different statuses |
根据各组间附生菌群落结构多样性的相互关系在OTU水平上绘制了韦恩图(图 5A)。结果显示,3组样品特有的OTU分别为77、86和92个。有98个OTU在3组样品中均存在,占OTU总数的22.7%。3组样品两两之间也存在共有OTU,其中,PyOlpH和PyOlpM之间共有31个,PyOlpM和PyOlpS之间共有30个OTU,而PyOlpH和PyOlpS之间仅共有18个OTU。基于Bray-Curtis距离的PCoA显示,紫菜附生菌群可根据条斑紫菜被侵染的程度而分开(PCo1解释度为41.4%),表明不同状态紫菜附生菌群之间存在明显差异(图 5B)。基于Bray-Curtis距离的PERMANOVA表明,这种组间差异具有显著性(R2= 0.405, P=0.042 < 0.05)。

|
图 5 基于OTU的韦恩图分析(A)和主坐标分析(B) Fig.5 Venn diagram (A) and principal co-ordinates analysis (B) based on OTU |
经过序列注释及分类信息统计,所得高质量序列共注释出23门208属。其中,变形菌门(Proteobacteria)中的α-变形菌纲(α-Proteobacteria)和γ-变形菌纲(γ-Proteobacteria)以及厚壁菌门(Firmicutes)在所有样品中均占优势,在PyOlpH、PyOlpM和PyOlpS组中共分别占84.7%、83.4%和88.0% (图 6)。α-变形菌纲(PyOlpH: 26.91%; PyOlpM: 24.59%; PyOlpS: 20.44%)和厚壁菌门(PyOlpH: 17.29%; PyOlpM: 9.56%; PyOlpS: 5.95%)相对丰度随着感染程度增加呈递减趋势,而γ-变形菌纲相对丰度(PyOlpH: 40.5%; PyOlpM: 49.2%; PyOlpS: 61.6%)则随着感染程度增加呈递增趋势。
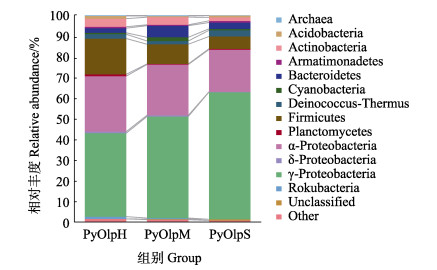
|
图 6 不同状态条斑紫菜附生菌群在门(或变形菌纲)水平上的相对丰度变化 Fig.6 Changes in relative abundance of epiphytic microbial communities of N. yezoensis in different statuses at the phylum or Proteobacterial level |
从细菌属水平来看,相对丰度前20的细菌属中共有16个属分布于α-变形菌纲、γ-变形菌纲和厚壁菌门(图 7)。例如,α-变形菌纲中的甲基杆菌属(Methylobacterium)、鞘氨醇单胞菌属(Sphingomonas)和柄杆菌属(Caulobacter),前二者相对丰度随着感染程度的增加而降低,而后者则相反;γ-变形菌纲中的贪铜菌属(Cupriavidus)、假单胞菌属(Pseudomonas)和Methyloversatilis属的相对丰度均随感染程度增加而增加;厚壁菌门中的Kroppenstedtia属的相对丰度随感染程度增加而减少。
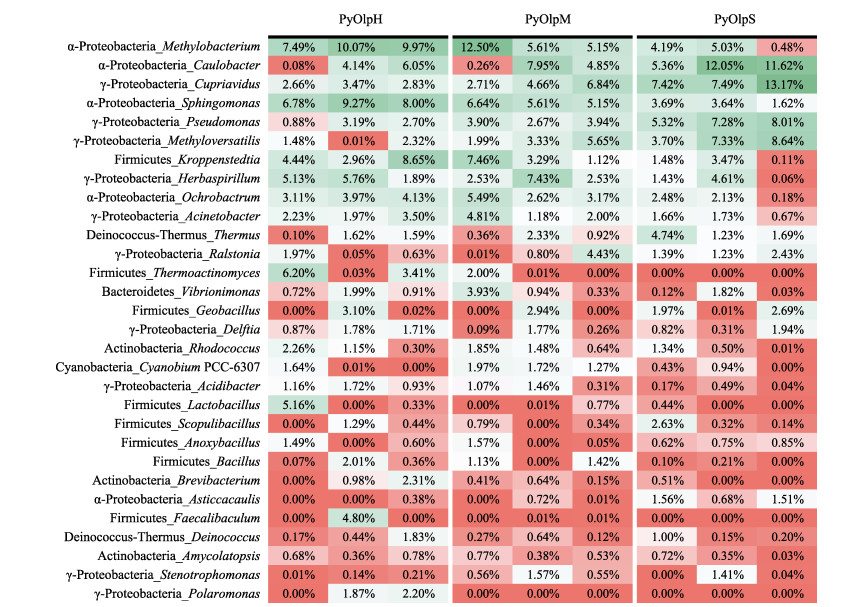
|
图 7 不同状态条斑紫菜附生菌群中前30个细菌属的相对丰度热图 Fig.7 Relative abundance heatmap of the top 30 bacterial genera in epibacterial communities of N. yezoensis in different health statuses 绿色表示相对丰度较高(> 1%)的属,红色表示相对丰度较低(< 1%)的属 Genus with high relative abundance (> 1%) are marked in green, and those with low relative abundance (< 1%) are marked in red |
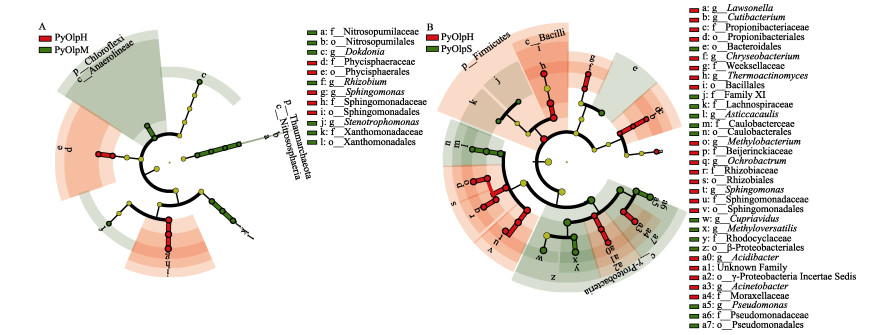
LefSe分析得到的差异菌群结果显示,PyOlpM与PyOlpH的附生菌群之间共有16个类群存在相对丰度的显著差异,其中鞘脂单胞菌目(Sphigomonadales)细菌在PyOlpH中显著富集,而黄单胞菌目(Xanthomonadales)则在PyOlpM中显著富集(图 8A)。PyOlpS与PyOlpH的附生菌群之间相对丰度存在显著差异的菌群数量则达到37个,如PyOlpS中的假单胞菌目(Pseudomonadales)和柄杆菌目(Caulobacterales),表明有更多的细菌类群参与到拟油壶菌的后期感染中(图 8B)。
|
图 8 健康紫菜附生菌群与不同感染程度条斑紫菜附生菌之间的差异菌群分析 Fig.8 Analysis of differential microbial taxa between healthy N. yezoensis and those in different infection states 红色表示相对丰度在PyOlpH组显著更高的微生物类群,绿色表示相对丰度在病烂紫菜中显著更高的类群 Microbial taxa with relative abundance significantly higher in PyOlpH are marked in red, and those with relative abundance significantly higher in diseased N. yezoensis are marked in green |
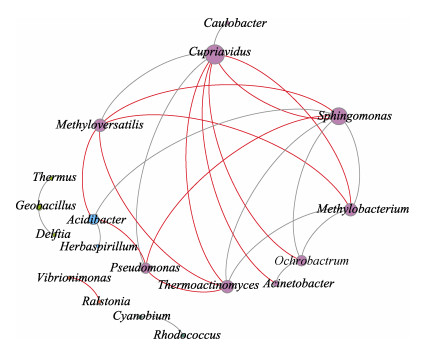
利用相对丰度前20的细菌属构建了一个节点数为18、边数为27的共现网络,每个节点的平均度为3,表示平均每个细菌属连接3个其他细菌属,正相互作用的边数为14 (51.85%),负相互作用的边数为13 (48.15%)。其中,贪铜菌属的度为最高(8),且与柄杆菌属、假单胞菌属和Methyloversatilis属之间存在正相互作用(图 9);鞘氨醇单胞菌属的度次之(7),该属细菌与甲基杆菌属、酸杆菌属(Acidibacter)、苍白杆菌属(Ochrobactrum)以及高温放线菌属(Thermoactinomyces)之间存在正相互作用,且这些细菌属均与贪铜菌属细菌或与贪铜菌属细菌存在正相互作用的细菌类群之间具有负相互作用(图 9)。
|
图 9 前20个细菌属之间的共现网络 Fig.9 Co-occurrence network of top 20 bacterial genera 节点的大小与某一属连接其他属数量的多少成正相关; 边的粗细与Spearman相关系数的大小成正比, 其中灰色的边表示细菌属之间的正显著相互作用, 而红色的边则表示负显著相互作用 The size of each node is proportional to the number of connections. The thickness of the edge is proportional to the Spearman's correlation coefficient. The gray edge indicates significantly positive interaction between bacteria genera, while the red edge indicates significantly negative interaction |
本研究以江苏省大丰区某栽培海区遭受拟油壶菌感染的条斑紫菜为研究对象,分析了不同感染状态下条斑紫菜附生菌群的多样性和组成差异以及主要主要细菌类群之间的互作关系。
研究结果显示,附生菌群的α多样性指数在不同状态样品之间不存在显著差异。这与赤腐病感染紫菜的菌群多样性变化较为一致(Yan et al, 2019)。但两项研究均发现,初期感染样品附生菌群的α多样性指数高于未发生感染样品,而且β多样性的结果显示了不同感染状态样品附生菌群之间存在显著差异,表明病原的感染与菌群的紊乱密切相关。另外,拟油壶菌病的发生与栽培环境的变化紧密相关,如水温的升高、海水盐度的降低、水流交换不佳以及干出时间不足等(马家海, 1992; Klochkova et al, 2012、2016)。综合两方面,与Egan等(2016)提出的观点较为符合,即环境胁迫条件下的微生物生态失调是造成藻类病害发生的主要原因,因此,藻类病害的发生发展是一个复杂的微生态过程。
研究表明,变形菌门和厚壁菌门通常是海藻表面占主导地位的2个细菌类群,在海藻表面广泛存在的细菌中它们可被看作是“微生物的杂草种类” (microbial weed species),并且具有多种多样的生物功能,例如帮助宿主抵抗多种胁迫环境因子、具有高效的产能系统以及诱导宿主的形态发生和后续的生长,表明这些类群的细菌对藻类宿主健康的重要性(Singh et al, 2014)。由于绝大多数细菌在藻类特别是紫菜叶状体上的功能还没有详尽的解析,因此,只能通过细菌在植物等宿主或者环境中的相应功能在一定程度上解析其在本研究中的生态功能。例如,α-变形菌纲中的主要类群(如甲基杆菌属和鞘氨醇单胞菌属)以及γ-变形菌纲中的假单胞菌属是陆生植物的叶际最常检出的微生物类群。其中,甲基杆菌属和假单胞菌属都能分泌AHLs (acyl homoserine lactones),是植物叶际QS的重要参与者,且后者往往对植物具有致病性,并通过QS分泌胞外聚合物增强其对附着生活的适应性(Vorholt, 2012)。此外,甲基杆菌属细菌还具有竞争利用植物叶际碳源的功能,因此,它可能通过QS或底物竞争等形式调节微生物的正常附着和聚集(Vorholt, 2012)。而善于从植物叶际吸收低浓度营养物质的鞘氨醇单胞菌属细菌则对病原假单胞菌的感染具有强烈的抑制作用,不仅能够抑制感染症状,还能够减弱病原的生长(Innerebner et al, 2011)。另外,土壤、水体以及植物等环境中常见的贪铜菌属细菌能利用多种碳源进行生长,并且具有抗生素和消毒剂的抗性,甚至可导致免疫力低下的人患上严重并发症(Balada-Llasat et al, 2010; Khan et al, 2016)。这些微生物的生态功能在一定程度上解释了这些细菌类群的相对丰度变化规律,亦即对宿主健康有益的细菌则相对丰度随着感染程度增加而减少,不利宿主健康的细菌则反之。
除此之外,细菌之间的共现网络图也能比较直观地反映细菌的功能和互作关系。比如,与鞘氨醇单胞菌属细菌具有正相互作用的细菌类群(如甲基杆菌等,图 9蓝色圆圈)往往与假单胞菌和贪铜菌等病害相关的细菌类群(图 9紫色圆圈)具有负相互作用,表明二者之间具有竞争、抑制的作用。这样的负相互作用为抑病生防菌的查找提供了重要的理论指导(Poudel et al, 2016)。研究还发现,PyOlpM比PyOlpH组的附生菌群显著富集了黄单胞菌目的细菌,这类细菌也是植物叶际表面QS的参与者之一。Berendsen等(2018)研究表明,叶片受霜霉病病原菌Hyaloperonospora arabidopsidis侵染的拟南芥在根际招募黄单胞菌目的黄单胞菌属、寡养单胞菌属(Stenotrophomonas)以及放线菌目(Actinomycetales)的微杆菌属(Microbacterium)等细菌协同帮助植物抵抗病害。因此可推测,拟油壶菌对紫菜的感染也可使宿主招募某些类群的益生菌来抵抗病害。但是,由于拟油壶菌和霜霉均为内寄生的病原,而由益生菌诱导出的宿主抗病力并不足以治愈已经感染的个体(Berendsen et al, 2018),因此,紫菜最终仍会被病原及后续生长起来的其他致病菌侵染。
基于本研究结果可提出以下设想:当环境条件变得适宜拟油壶菌侵染时,紫菜附生菌群发生紊乱并朝着利于病原及其他有害微生物附着生长的方向演变(如甲基杆菌、鞘氨醇单胞菌丰度的不断降低,贪铜菌和假单胞菌丰度的不断升高),这使得拟油壶菌病原不断附着到紫菜上并侵染、杀死宿主细胞;随着病原的持续感染,虽然紫菜会招募一些益生菌(如黄单胞菌目细菌)来帮助其抗病,但并不足以使其痊愈;贪铜菌和假单胞菌等有害细菌通过不断增殖甚至产生抵抗抗生素和消毒剂的能力,导致病害难以控制,紫菜组织持续病烂并凋亡。
综上所述,本研究探究了遭受拟油壶菌感染后不同健康状态下条斑紫菜的附生菌群多样性、群落结构和主要类群之间的相互作用,发现不同健康状态下的紫菜附生菌群存在明显差异,且主要细菌类群之间存在显著的互作关系,表明附生菌群的变化和互作在病害发生过程中起到了重要的作用。这些结果为阐明拟油壶菌致病的微生态机制及寻找生防细菌提供了一定的数据支持,今后仍需要大量实验验证发病的生态机制及附生细菌的生物功能。
BADIS Y, KLOCHKOVA T A, STRITTMATTER M, et al. Novel species of the oomycete Olpidiopsis potentially threaten European red algal cultivation. Journal of Applied Phycology, 2019, 31(2): 1239-1250 DOI:10.1007/s10811-018-1641-9 |
BALADA-LLASAT J M, ELKINS C, SWYERS L, et al. Pseudo-outbreak of Cupriavidus pauculus infection at an outpatient clinic related to rinsing culturette swabs in tap water. Journal of Clinical Microbiology, 2010, 48(7): 2645-2647 DOI:10.1128/JCM.01874-09 |
BASTIAN M, HEYMANN S, JACOMY M. Gephi: An open source software for exploring and manipulating networks. Dublin: Third International ICWSM Conference, 2009, 361-362
|
BERENDSEN R L, VISMANS G, YU K, et al. Disease-induced assemblage of a plant-beneficial bacterial consortium. ISME Journal, 2018, 12(6): 1496-1507 DOI:10.1038/s41396-018-0093-1 |
BURKE C, STAFFAN K, TORSTEN T. Selective extraction of bacterial DNA from the surfaces of macroalgae. Applied and Environmental Microbiology, 2009, 75(1): 252-256 DOI:10.1128/AEM.01630-08 |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336 DOI:10.1038/nmeth.f.303 |
EDGAR R C. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461 DOI:10.1093/bioinformatics/btq461 |
EGAN S, FERNANDES N D, KUMAR V, et al. Bacterial pathogens, virulence mechanism and host defence in marine macroalgae. Environmental Microbiology, 2014, 16(4): 925-938 DOI:10.1111/1462-2920.12288 |
EGAN S, GARDINER M. Microbial dysbiosis: Rethinking disease in marine ecosystems. Frontiers in Microbiology, 2016, 7: 991 |
GOECKE F, LABES A, WIESE J, et al. Chemical interactions between marine macroalgae and bacteria. Marine Ecology Progress Series, 2010, 409: 267-299 DOI:10.3354/meps08607 |
HE L J, YANG H C, LI J, et al. Diagnosis of Oplidiopsis disease in Pyropia yezoensis. Progress in Fishery Sciences, 2021, 42(4): 177-183 [何礼娟, 杨慧超, 李杰, 等. 一起条斑紫菜拟油壶菌病的诊断. 渔业科学进展, 2021, 42(4): 177-183] |
HUANG J, TANG X X, YAN X J, et al. Review and forecast of the study on the disease of kelp and laver in China. Marine Science, 2000, 24(9): 26-27 [黄健, 唐学玺, 严小军, 等. 我国海带和紫菜病害的研究展望. 海洋科学, 2000, 24(9): 26-27 DOI:10.3969/j.issn.1000-3096.2000.09.010] |
INNEREBNER G, KNIEF C, VORHOLT J A. Protection of Arabidopsis thaliana against leaf-pathogenic Pseudomonas syringae by Sphingomonas strains in a controlled model system. Applied and Environmental Microbiology, 2011, 77(10): 3202-3210 DOI:10.1128/AEM.00133-11 |
KHAN S, BEATTIE T K, KNAPP C W, et al. Relationship between antibiotic- and disinfectant- resistance profiles in bacteria harvested from tap water. Chemosphere, 2016, 152: 132-141 DOI:10.1016/j.chemosphere.2016.02.086 |
KIM G H, MOON K H, KIM J Y, et al. A revaluation of algal diseases in Korean Pyropia (Porphyra) sea farms and their economic impact. Algae, 2014, 29(4): 249-265 DOI:10.4490/algae.2014.29.4.249 |
KLOCHKOVA T A, SHIM J B, HWANG M S, et al. Host-parasite interactions and host species susceptibility of the marine oomycete parasite, Olpidiopsis sp., from Korea that infects red algae. Journal of Applied Phycology, 2012, 24(1): 135-144 DOI:10.1007/s10811-011-9661-8 |
KLOCHKOVA T A, SHIN Y J, MOON K H, et al. New species of unicellular obligate parasite, Olpidiopsis pyropiae sp. nov., that plagues Pyropia sea farms in Korea. Journal of Applied Phycology, 2016, 28(1): 73-83 DOI:10.1007/s10811-015-0595-4 |
KWAK M S, KLOCHKOVA T A, JEONG S, et al. Olpidiopsis porphyrae var. koreanae, an endemic endoparasite infecting cultivated Pyropia yezoensis in Korea. Journal of Applied Phycology, 2017, 29(7): 1-10 |
LAROUSSE M, GALIANA E. Microbial partnerships of pathogenic Oomycetes. PLoS Pathogens, 2017, 13(1): e1006028 DOI:10.1371/journal.ppat.1006028 |
LEE S J, HWANG M S, PARK M A, et al. Molecular identification of the algal pathogen Pythium chondricola (Oomycetes) from Pyropia yezoensis (Rhodophyta) using ITS and cox1 markers. Algae, 2015, 30(3): 217-222 DOI:10.4490/algae.2015.30.3.217 |
LIU Y M, MA J H, WEN Q. Simultaneous infection by red rot and olpidiops disease in laver Porphyra haitanensis. Journal of Dalian Ocean University, 2012, 27(6): 546-550 [刘一萌, 马家海, 文茜. 坛紫菜赤腐病与拟油壶菌病并发病的初步研究. 大连海洋大学学报, 2012, 27(6): 546-550 DOI:10.3969/j.issn.2095-1388.2012.06.011] |
LIU Y, DE BRUIJN I, JACK A L, et al. Deciphering microbial landscapes of fish eggs to mitigate emerging diseases. ISME Journal, 2014, 8(10): 2002-2014 DOI:10.1038/ismej.2014.44 |
MA J H, LIN Q S, MIN J, et al. Preliminary study on the olpidiops-disease of Porphyra yezoensis. Journal of Fisheries of China, 2007, 31(6): 860-864 [马家海, 林秋生, 闵建, 等. 条斑紫菜拟油壶菌病的初步研究. 水产学报, 2007, 31(6): 860-864] |
MA J H. An investigation of chytrid-disease in Porphyra yezoensis in the coastal water of south Jiangsu. Journal of Shanghai Fisheries University, 1992, 1(3/4): 185-188 [马家海. 江苏省南部沿海条斑紫菜壶状菌病的调查研究. 上海水产大学学报, 1992, 1(3/4): 185-188] |
POUDEL R, JUMPPONEN A, SCHLATTER D C, et al. Microbiome networks: A systems framework for identifying candidate microbial assemblages for disease management. Phytopathology, 2016, 106(10): 1083-1096 DOI:10.1094/PHYTO-02-16-0058-FI |
R CORE Team. R: A language and environment for statistical computing. Vienna: R Foundation for Statistical Computing, 2014
|
SEGATA N, IZARD J, WALDRON L, et al. Metagenomic biomarker discovery and explanation. Genome Biology, 2011, 12(6): R60 DOI:10.1186/gb-2011-12-6-r60 |
SEKIMOTO S, KLOCHKOVA T A, WEST J A, et al. Olpidiopsis bostrychiae sp. nov.: An endoparasitic oomycete that infects Bostrychia and other red algae (Rhodophyta). Phycologia, 2009, 48(6): 460-472 DOI:10.2216/08-11.1 |
SEKIMOTO S, YOKOO K, KAWAMURA Y, et al. Taxonomy, molecular phylogeny, and ultrastructural morphology of Olpidiopsis porphyrae sp. nov. (Oomycetes, straminipiles), a unicellular obligate endoparasite of Bangia and Porphyra spp. (Bangiales, Rhodophyta). Mycological Research, 2008, 112(3): 361-374 DOI:10.1016/j.mycres.2007.11.002 |
SINGH R P, KUMARI P, REDDY C R K. Antimicrobial compounds from seaweeds-associated bacteria and fungi. Applied Microbiology and Biotechnology, 2015, 99(4): 1571-1586 DOI:10.1007/s00253-014-6334-y |
SINGH R P, REDDY C R K. Seaweed-microbial interactions: Key functions of seaweed-associated bacteria. FEMS Microbiology Ecology, 2014, 88(2): 213-230 DOI:10.1111/1574-6941.12297 |
VAN DIJK K, NELSON E B. Fatty acid competition as a mechanism by which Enterobacter cloacae suppresses Pythium ultimum sporangium germination and damping-off. Applied and Environmental Microbiology, 2000, 66(12): 5340-5347 DOI:10.1128/AEM.66.12.5340-5347.2000 |
VORHOLT J A. Microbial life in the phyllosphere. Nature Reviews Microbiology, 2012, 10(12): 828-840 DOI:10.1038/nrmicro2910 |
WALTERS W, HYDE E R, BERG-LYONS D, et al. Improved bacterial 16S rRNA gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys. mSystems, 2016, 1(1): e00009-15 |
WEST J A, KLOCHKOVA T A, KIM G H, et al. Olpidiopsis sp., an oomycete from Madagascar that infects Bostrychia and other red algae: Host species susceptibility. Phycological Research, 2006, 54(1): 72-85 DOI:10.1111/j.1440-1835.2006.00410.x |
WHITE T J, BRUNS T, LEE S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR Protocols-A guide to methods and applications. San Diego: Academic Press, 1990, 315-322
|
YAN Y W, YANG H C, TANG L, et al. Compositional shifts of bacterial communities associated with Pyropia yezoensis and surrounding seawater co-occurring with red rot disease. Frontiers in Microbiology, 2019, 10: 1666 DOI:10.3389/fmicb.2019.01666 |
YANG L E, DENG Y Y, XU G P, et al. Redefining Pyropia (Bangiales, Rhodophyta): four new genera, resurrection of Porphyrella and description of Calidia pseudolobata sp. nov. from China. Journal of Phycology, 2020, 56(4): 862-879 DOI:10.1111/jpy.12992 |



