2. 中国水产科学研究院黄海水产研究所青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071;
3. 烟台开发区天源水产有限公司 山东 烟台 264006
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao, Shandong 266071, China;
3. Tianyuan Aquaculture Co., Ltd of Yantai Economic Development Zone, Yantai, Shandong 264006, China
vasa基因编码的蛋白是DEAD-box (Asp-Glu- Ala-Asp)蛋白家族成员,该蛋白参与多种细胞进程,如细胞RNA转录调节,RNA剪切、修饰和代谢,核内mRNA的运输及降解等(Dehghani et al, 2015)。Schüpbach等(1986)首次在黑腹果蝇(Drosophila melanogaster)中发现vasa的存在,证明其属母源基因,是生殖质的前体,在生殖细胞分化中发挥作用(Hay et al, 1988)。原始生殖细胞(PGCs)是由细胞分化而来,发育早期从体细胞中掉落,随后经过一系列复杂的趋化因子作用发生转移,至生殖脊时精卵结合促使原始性腺形成。随后通过一系列的分裂增殖与分化过程,在性成熟阶段由卵巢和精巢分泌成熟的配子(胡翔, 2015)。近年来,一些分子标记的发现帮助了PGCs的鉴定,硬骨鱼PGCs的第1个分子标记是vasa基因(Olsen et al, 1997)。vasa基因具有高度保守性,继在果蝇体内被克隆后,在无脊椎动物和脊椎动物相继展开研究,如家蚕(Bombyx mori) (Cao et al, 2012)、鸡(Gallus gallus) (Tsunekawa et al, 2000)和小鼠(Mus musculus)(Reunov et al, 2015)等。vasa基因作为母源性基因,在生殖细胞中特异性表达,因此,可能在生殖发育的调控过程中发挥作用。目前,已在多种鱼类成功克隆vasa序列并得到其同源基因,如斑马鱼(Danio rerio)(Krøvel et al, 2004)、青鳉(Oryzias latipes) (Herpin et al, 2007)、文昌鱼(Branchiostoma belcheri) (Wu et al, 2011)和七彩神仙鱼(Symphysodon haraldi) (林睿涓等, 2017)等,并深入研究了vasa在早期性腺分化及发育过程中发挥的作用。但未见有关中华蛸(Octopus sinensis) vasa基因的报道。
中华蛸属于八腕目(Octopoda)、蛸科(Octopodidae)、蛸属,在浙江、福建和广东等近海区域广泛分布。中华蛸喜穴居,肉质鲜美,蛋白质含量丰富且营养均衡(郑小东等, 2011),生鲜即食,加工后晒干也可,能食部分占比很高(92%以上),颇受人们喜爱。中华蛸作为我国重要的经济海产类,已开展了对其人工养殖及繁育的研究(蔡厚才等, 2009; 郑小东等, 2011),但目前对中华蛸繁育机制的研究仍处于探索阶段。中华蛸卵母细胞发育不同步,属分批产卵,给规模化苗种繁育增加了困难,工厂化养殖仍需努力。vasa是生殖细胞的分子标记物,开展vasa基因克隆和表达研究,可为中华蛸人工繁育和养殖提供理论依据。本研究以中华蛸为对象,运用cDNA末端快速扩增技术(RACE)克隆中华蛸vasa基因,并使用荧光定量PCR技术(qRT-PCR)对其早期发育阶段及各器官组织的表达状况进行检测,旨在完善中华蛸分子生物学和生殖调控方面的知识,为其生殖细胞的分化、早期性别鉴定及性别决定机制提供数据支撑,为实现工厂化繁育提供参考。
1 材料与方法 1.1 实验样品制备实验用中华蛸亲体在浙江南麂岛海域捕获,经长途运输至山东省烟台市牟平区天源水产有限公司进行人工培育,养殖水温为23.2℃~25.6℃,盐度为30~ 32,日换水量100%~200%。分别取活力旺盛、体质量为1.0~1.5 kg的雌雄成体中华蛸各3尾,观察其右侧第3条腕确认雌雄,经MgCl2 (浓度为20 g/L)麻醉后迅速解剖,分别取其卵巢、精巢、鳃、心脏、肾脏、肝胰腺、大脑、视腺和皮肤。所用受精卵、孵化出膜后幼体均由成体中华蛸自然产卵、人工培育而来。培育水温为23.8℃~26.0℃,溶氧 > 5.0 mg/L,受精卵孵化27 d得到初孵幼体。幼体孵出后,移入专用水泥池(6 m×2 m×1.5 m)。根据幼体密度和大小投喂合适密度的卤虫(Artemia),并连续充气确保氧气充足,前3 d使用静水培育,第4天使用流水,换水量为50%~ 70%,并进行吸底、排污。收集不同发育时间的受精卵(5、10、13、16、19、21、24和27 d)、孵化出膜后幼体(2、5、8、11、14、17、20、23和26 d)及未成熟、成熟期的卵巢若干,未成熟期体重为(182.31± 20.29) g,性腺指数(GSI)为(0.55±0.22)%,卵母细胞直径在80~100 μm之间;成熟期体重为(1326±100) g,GSI为(5.19±0.81)%,卵母细胞直径在400~500 μm之间。所有样品均快速装入含有RNA保存液的2.0 mL无酶管中,并在4℃冰箱放置12 h,保证保存液完全渗入组织。样品带回后贴上标签,转入–80℃冰箱,防止RNA降解,用于后续基因克隆和表达分析。
1.2 主要试剂SMARTTM RACE cDNA amplification试剂盒、PrimeScriptTM RT reagent with gDNA eraser反转录试剂盒、DNA Marker、Premix TaqTM (TaKaRa TaqTM V 2.0)大肠杆菌(Escherichia coli) DH5α菌株感受态细胞和pMDTM 18T vector cloning试剂盒均购自TaKaRa公司,SteadyPure DNA凝胶回收试剂盒购自艾克瑞生物,动物组织总RNA提取试剂盒(DP431)购自天根生化科技有限公司,ChamQTM SYBR Color qPCR master mix试剂盒购自诺唯赞生物科技股份有限公司。
1.3 总RNA提取及cDNA第一链合成本实验总RNA的提取使用动物RNA提取试剂盒(DP431),并参照其说明书提取中华蛸胚胎、孵化出膜后幼体各个时期和成体不同组织的总RNA。根据目的片段的长短采用不同浓度(1%~2%)的琼脂糖制成凝胶,点样后检测RNA的质量(条带是否清晰),使用NanoDrop 2000 (Thermo Scientific, 美国)微量分光光度计检测RNA的纯度及浓度(用量1 μL)。5′RACE、3′RACE模板链的制备按照SMARTTM RACE cDNA amplification kit说明进行。
1.4 vasa基因核心序列克隆从本实验室构建的中华蛸转录组数据的注释信息,筛选比对得到vasa基因的部分cDNA序列。通过Primer 5.0软件设计3对引物扩增核心序列(表 1),以中华蛸成熟卵巢组织cDNA为模板分别进行扩增。20 μL反应体系:10 μL Premix TaqTM (LA TaqTM V2.0),正向引物Os-vasa F (10 μmol/L) 0.8 μL,反向引物Os-vasa R (10 μmol/L) 0.8 μL,cDNA模板(1 μg/μL) 1 μL,ddH2O 6.4 µL补齐。反应程序:94℃ 5 min;94℃ 30 s,60℃ 40 s,72℃ 1 min,35个循环;72℃延伸10 min。产物扩增完成后,在电泳板中检测扩增条带,切胶仪(DUT-48超薄型)下观察合适的条带,并用SteadyPure DNA回收DNA。纯化后的DNA连接到pMD18-T vector中,连接体系:pMD18-T vector 1 μL, Solution Ⅰ 5 μL及DNA纯化产物4 μL),并置于PCR反应仪中反应3 h (16℃)。之后转入冰上融化的DH5α感受态细胞过夜培养,筛选阳性单克隆并进行菌落PCR鉴定,将含有目的基因的菌液测序(华大基因)。
|
|
表 1 本研究所用引物名称和序列 Tab.1 Names and sequences of primers used in this study |
测序后,经拼接比对确认为核心序列,设计RACE特异性引物Os-vasa 5′GSP-1、Os-vasa 5′GSP-2、Os-vasa 3′GSP-1和Os-vasa 3′GSP-2,使用巢式PCR进行3′和5′ RACE扩增,5′端扩增:5′RACE cDNA为模板,先用5′GSP-1与RACE通用引物UPM-long组合完成第1次反应;接着用第1次反应获取的目的液稀释后为模板,5′GSP-2与RACE通用引物UPM-short或NUP组合完成第2次反应。3′端扩增:3′RACE cDNA为模板,3′GSP-1与RACE通用引物UPM-long组合完成第1次反应;用第1次反应获取的目的液稀释后为模板,Os-vasa 3′GSP-2与RACE通用引物UPM-short或NUP组合进行第2轮扩增。
PCR反应体系(20 μL):10 μL Premix TaqTM (LA TaqTM V 2.0),3′或5′特异性引物(10 μmol/L) 0.8 μL,UPM (或NUP) 0.8 μL,RACE-cDNA模板(1 μg/μL) 1 μL,ddH2O 6.4 µL补齐。反应程序流程:94℃ 5 min;94℃ 30 s,3′和5′特异性引物退火温度30 s,72℃ 1 min,反应30次;72℃ 10 min。获取的产物检测后进行回收、纯化,之后连接转化培养,挑阳性菌株测序(华大基因公司)。
1.6 vasa基因序列分析测序完成的片段使用Contig Express 9.1软件进行拼接、验证,并用BLAST工具(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源性比对,确认实验得到的cDNA是DEAD-box家族的vasa基因。使用OFR Finder (http://www.ncbi.nlm.Nih.gov/projects/gorf/Orfig.cgi)在线软件推导vasa基因的开放阅读框,利用ExPASy (https://web.expasy.org/compute_pi/)网站分析分子量、理论等电点,利用Signal4.1 (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽,使用TMHMM 2.0 (http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜区分析,利用SMART (http://smart.embl-heidelberg.de/)和NCBI (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测结构和功能结构域。将Os-vasa与其他已知物种的vasa氨基酸序列使用NCBI进行同源蛋白比对,并使用DNAMAN对同源蛋白多重比较,利用MEGA 5.2软件使用邻近法(neighbor-joining)构建系统进化树(Koichiro et al, 2011)。
1.7 实时荧光定量PCR取不同发育时间的胚胎、孵化出膜后幼体、2个时期的卵巢和不同组织的总RNA,反转录为cDNA。将每个样品浓度加无菌无酶水补齐至50 ng/μL,根据Os-vasa基因的核心序列,利用Primer 5.0设计2对荧光定量特异性引物Os-vasa-RT-F/R,以β-actin为内参基因,使用StepOneTM Real-time PCR system (IBM, 美国),配制20 μL反应体系:ChamQTM SYBR color qPCR master mix (2×) 10 μL,ROX reference dyeⅠ(50×) 0.4 μL,Os-vasa-RT-F (10 μmol/L) 0.4 μL,Os-vasa-RT-R (10 μmol/L) 0.4 μL,cDNA模板(50 ng/μL) 2 μL,ddH2O 6.8 μL补齐。反应程序:95℃ 30 s (预变性);(95℃ 10 s;60℃ 30 s,然后95℃ 15 s;60℃ 60 s;95℃ 15 s) 40个循环。所有检测样本设3个生物学重复,并设置3个技术重复,反应结束后查看熔解曲线是否正常。根据所测数据,采用2–∆∆Ct法算出Os-vasa基因相对表达丰度,求其平均值±标准误(Mean±SE),使用SPSS 17.0软件对表达量进行方差检验,P < 0.05被认为差异显著。
2 结果与分析 2.1 Os-vasa基因序列分析中华蛸vasa基因全长为2438 bp,命名为Os-vasa其ORF长度为2067 bp,预测蛋白有688个氨基酸,5′-UTR长128 bp,3′-UTR长244 bp (包含A尾巴),理论等电点为5.89,氨基酸分子质量76 580.53 Da,无信号肽,跨膜区域没有明显的信号,推测其为胞内蛋白,不属于膜蛋白。具有vasa基因的DEXDc和HELICc 2个功能结构域,且包含DEAD-box家族蛋白的典型特点:9个保守区域,分别为AQTGSGKT(I)、PVLTLLLQ (Q)、PTRELA (Ⅰa)、TPGRI (Ⅰb)、DEAD (Ⅱ)、SAT (Ⅲ)、RGLD (Ⅴ)、LVFVE (Ⅳ)和HRIGRTGR(Ⅵ)。另外,预测的氨基酸序列N端有多个RG重复序列,C末端存在vasa基因常见的保守结构色氨酸(W)残基和酸性氨基酸残基(EEEE),序列中存在多个GG重复序列(图 1)。

|
图 1 Os-vasa基因cDNA序列全长和编码的氨基酸序列 Fig.1 Nucleotide sequence and deduced amino acids sequence of Os-vasa 起始密码子ATG和终止密码子TAA均用灰色阴影标出;PolyA用双下划线标出;GG重复序列、酸性氨基酸和色氨酸用方框标出;DExDc和HELICc 2个功能域用下划线标出;阴影加方框部分是Os-vasa保守基序 Start codon (ATG) and stop codon (TAA) are marked with gray shadow, and PolyA is marked with double underline; Nucleotide with a frame represents GG repeat sequence, acidic amino acid and tryptophan; Open reading fragment; DExDc and HELICc functional domains are underlined; Sequences in gray background and frame represents the Os-vasa conserved motifs |
通过在线软件NCBI blastx和DNAMAN软件,将中华蛸Os-vasa氨基酸序列与其他物种的vasa基因编码的氨基酸进行比对。结果发现,该氨基酸序列与加州双斑蛸(Octopus bimaculoides)同源性最高(98%),其次是虎斑乌贼(Sepia pharaonis)(68.74%)、太平洋牡蛎(Crassostrea gigas)(67.36%)、虾夷扇贝(Mizuhopecten yessoensis)(66.12%)、青螺(Lottia gigantea)(65.15%)海兔(Aplysia californica)(63%)和紫贻贝(Mytilus galloprovincialis)(62%),与人类(Homo sapiens)、小鼠(Mus musculus)同源性较低,分别为54%和53%。另外,DEXDc和HELICc 2个功能结构域的氨基酸序列保守性较高(图 2)。

|
图 2 中华蛸Os-vasa编码氨基酸序列与其他物种同源序列比对 Fig.2 Homologous amino acid sequences alignment of Os-vasa with other species 阴影部分表示氨基酸同源,左边为物种名称,右边为比对到的氨基酸位置。各物种蛋白NCBI登录号:加州双斑蛸(KOF70288.1)、虎斑乌贼(CAE1321294.1)、太平洋牡蛎(XP_034310873.1)、虾夷扇贝(XP_021370694.1)、青螺(XP_009057808.1)、海兔(XP_005113588.2)、紫贻贝(BAJ15435.1) The shaded area indicates amino acid homology species. Species NCBI Login No: Octopus bimaculoides (KOF70288.1), Sepia pharaonic (CAE1321294.1), Crassostrea gigas (XP_034310873.1), Mizuhopecten yessoensis (XP_021370694.1), Lottia gigantean (XP_009057808.1), Aplysia californica (XP_005113588.2), Mytilus galloprovincialis (BAJ15435.1) |
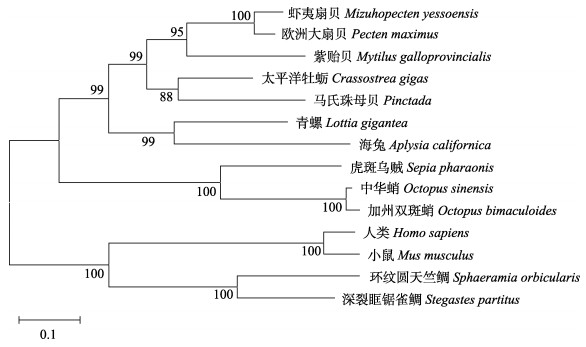
为了分析vasa基因在不同物种中的进化关系,利用MEGA 5.2软件对中华蛸及其他13个物种的DEAD-box蛋白序列构建系统进化树(图 3),结果显示,无脊椎动物和软体动物,脊椎动物各聚一支,其中,中华蛸先与加州双斑蛸、虎斑乌贼聚为一支,这表明它们的亲缘关系较近,vasa基因在头足类中可能较为保守,独立形成分支后再与软体动物的腹足纲(Gastropoda)和瓣鳃纲(Lamellibranchia)聚为一支。Os-vasa在系统进化树中呈现的结果与中华蛸在生物学分类中的地位基本一致。
|
图 3 不同物种vasa蛋白系统进化树 Fig.3 Phylogenetic analysis of vasa protein from different species 所用到的物种vasa基因序列登录号:虾夷扇贝(XP_021370694.1)、欧洲大扇贝(XP_033738811.1)、紫贻贝(BAJ15435.1)、太平洋牡蛎(XP_034310873.1)、马氏珠母贝(BAM75192.1)、青螺(XP_009057808.1)、海兔(XP_005113588.2)、虎斑乌贼(CAE1321294.1)、加州双斑蛸(KOF70288.1)、人类(CAB70750.1)、小鼠(NP_034159.1)、环纹圆天竺鲷(XP_030008903.1)、深裂眶锯雀鲷(XP_008278033.1) The Accession number of vasa gene sequence were used: Mizuhopecten yessoensis (XP_021370694.1), Pecten maximus (XP_033738811.1), Mytilus galloprovincialis (BAJ15435.1), Crassostrea gigas (XP_034310873.1), Pinctada fucata (BAM75192.1), Lottia gigantean (XP_009057808.1), Aplysia californica (XP_005113588.2), Sepia pharaonic (CAE1321294.1), Octopus bimaculoides (KOF70288.1), Homo sapiens (CAB70750.1), Mus musculus (NP_034159.1), Sphaeramia orbicularis (XP_030008903.1), Stegastes partitus (XP_008278033.1) |
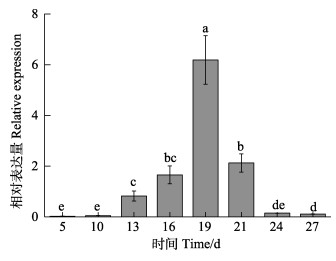
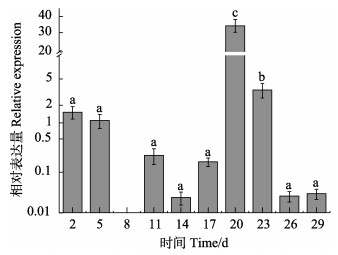
在胚胎时期的qRT-PCR检测结果如图 4所示,Os-vasa基因在发育前10 d微弱表达,从第13天开始,表达量逐渐上升,至第19天达到最高,之后表达量又开始下降,一直到孵化出膜。在仔蛸阶段,从出膜到17日龄,Os-vasa基因表达量都维持在较低水平,8日龄几乎检测不到表达信号,从20~23日龄期间的表达量较高,且在20日龄表达量最高,后期阶段(26和29日龄)又维持在较低水平(图 5)。
|
图 4 Os-vasa mRNA在胚胎发育时期的相对表达量 Fig.4 Expression distribution of Os-vasa mRNA at different development stages of the embryo 柱上不同字母代表具有显著差异(P<0.05),下同 Bar with different letters indicate significant differences (P < 0.05), the same as below |
|
图 5 Os-vasa mRNA在不同发育时期仔蛸中的相对表达量 Fig.5 Expression distribution of Os-vasa mRNA at the different stages of the larvae |
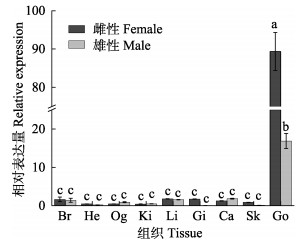
中华蛸雌性个体和雄性个体各组织的qRT-PCR检测结果如图 6所示,Os-vasa基因无论在雌性还是雄性个体性腺组织的表达量均较高,在其他组织中表达量均很低,并且在卵巢中的表达量高于精巢。
|
图 6 Os-vasa mRNA在雌雄中华蛸不同组织中的表达 Fig.6 Expression distribution of Os-vasa mRNA in different tissues of female and male O. sinensis Br:大脑;He:心脏;Og:视腺;Ki:肾;Li:肝胰腺;Gi:鳃;Ca:胴肌;Sk:皮肤;Go:性腺 Br: Brain; He: Heart; Og: Optic gland; Ki: Kidney; Li: Liver; Gi: Gill; Ca: Carcass; Sk: Skin; Go: Gonad |
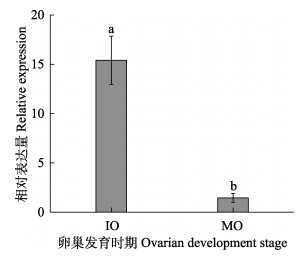
取未成熟的卵巢和成熟期的卵巢进行qRT-PCR检测,结果显示(图 7),未成熟卵巢表达量显著高于成熟卵巢(P < 0.05),未成熟期的表达量是成熟期的15.6倍左右。
|
图 7 Os-vasa mRNA在卵巢发育不同时期的表达 Fig.7 Expression distribution of Os-vasa mRNA at different stage of ovarian development of O. sinensis IO:未成熟卵巢;MO:成熟卵巢 IO: Immature ovary; MO: Mature ovary |
本研究从实验室构建的中华蛸性腺转录组注释中筛选得到vasa基因的部分片段,并通过NCBI进行比对确认,使用RACE技术首次克隆了vasa基因cDNA序列,并以Os-vasa命名。其全长为2438 bp,共编码688个氨基酸,理论等电点为5.89,氨基酸分子质量为76 580.53 Da。vasa蛋白属于DEAD-box蛋白家族,Ia-Ib和Ⅱ~Ⅵ这9个保守区域是DEAD-box家族蛋白共有的特点(Tanner et al, 2001),在大部分DEAD-box蛋白家族成员中保守性表达。本研究得到的Os-vasa基因同样发现了这9个保守基序的存在,每个保守基序都行使着特殊的生理功能,其中,Qmotif框在ATP的结合与水解过程中发挥重要作用,谷氨酰胺残基加上N可以参与氢键的形成,与功能性区域Ⅰ也有关联,例如,相互配合促使ATP、RNA两两结合(Rocak et al, 2004)。motifⅠ~Ⅲ又可以相互作用形成一个ATP水解的作用口袋(Caruthers et al, 2002)。motifⅠa和motifⅠb则主要参与RNA的结合过程,motif Ⅳ、Ⅴ和Ⅵ参与ATP酶和解旋酶的活性调节(Cordin et al, 2006)。经编码氨基酸的多序列比对,在保守基序区域,物种间呈现高度保守性,仅有少数氨基酸残基存在差异,表明vasa基因在进化过程中较为保守。另外,在Os-vasa基因中发现了一些vasa基因的普遍性特征,例如,存在多个GG重复序列,氨基酸C末端存在酸性氨基酸残基,终止密码子前端有1个色氨酸残基。GG基序参与蛋白因子elF4A的相互作用(Rogers et al, 2002),C末端的酸性氨基酸残基常与RNA结合过程有关(Fabioux et al, 2004)。RGG重复区也具有相似性,在昆虫和脊索动物中普遍存在且大多数位于vasa蛋白的N端,但并不普遍存在于所有vasa蛋白,例如,在马氏珠母贝(Pinctada fucata) (刘雅等, 2017)、刺参(Apostichopus japonicus) (隋娟等, 2008)的vasa蛋白的N端未发现RGG重复区的存在,本研究结果也是如此,说明这些序列并不是不可或缺的,其行使的功能具有可替代性。功能结构域结果显示,Os-vasa基因具有DEADc和HELICc 2个结构域,这些都是vasa蛋白家族特有的,表明所得cDNA属于vasa基因家族且高度保守。
中华蛸Os-vasa氨基酸序列与其他物种vasa基因编码的氨基酸比对结果显示,与加州双斑蛸相似度较高,达98%。系统进化树结果显示,中华蛸Os-vasa蛋白先与加州双斑蛸、虎斑乌贼聚为一支,预示中华蛸Os-vasa基因在生物体中发挥的功能与头足类中其他物种高度相似,然后再与软体动物中腹足纲和瓣鳃纲聚为一支,与脊椎动物则亲缘关系较远。Os-vasa在系统进化树中的分子学进化地位与中华蛸的生物学分类地位是相符的。
Os-vasa基因组织qRT-PCR结果显示,其在中华蛸卵巢和精巢中高表达,而在中华蛸其他组织中表达量较低,甚至检测不到表达信号,差异极显著(P < 0.01)。这一结果和多数报道的物种vasa mRNA表达模式一致,如栉孔扇贝(Chlamys farreri)(邵明瑜等, 2007)、马氏珠母贝、太平洋蓝鳍金枪鱼(Thunnus orientalis) (Nagasawa et al, 2009)。研究表明,通过RNAi技术使用vasa dsRNA处理太平洋牡蛎性腺会导致vasa mRNA沉默,性腺vasa mRNA表达水平明显降低,vasa蛋白表达量也随之降低或者不表达,且大部分太平洋牡蛎不育(Fabioux et al, 2009)。vasa基因在生殖细胞的特异性表达模式表明其参与调控生殖细胞发育过程并发挥重要作用。也有研究发现,vasa除在性腺组织中表达外,在其他组织也有表达,例如,在半滑舌鳎(Cynoglossus semilaevis)(Hay et al, 1988)的心脏组织,在虹鳟(Oncorhynchus mykiss)(Wu et al, 2014)的脑和心脏组织,在小鼠(Zamboni et al, 1983)的肾和肾上腺组织都检测到vasa基因的表达。vasa基因不仅参与生殖细胞发育,还可能通过调节有关mRNAs的转录参与体细胞的分化(Ikenishi et al, 2000)。vasa基因具体行使的功能随着生物体的进化演变而存在差异,仍需要大量的研究工作来阐明。
Özhan-Kizil等(2009)研究表明,vasa mRNA在夏威夷明钩虾(Parhyale hawaiensis)的1~16-细胞期均能检测到,且32-细胞期前均定位在生殖细胞中;中国对虾(Fenneropenaeus chinensis)最早从2-细胞期开始能检测到vasa mRNA的表达,且在后期表达没有消失,但有减弱趋势,出膜后表达信号消失(周倩如, 2007)。在模式生物斑马鱼中,vasa mRNA表达信号贯穿整个胚胎发育时期,在胚胎发育早期存在于各个细胞中,并随着胚胎的发育逐渐在生殖质区域聚集,vasa mRNA是斑马鱼生殖质的重要组成成分(周倩如等, 2007)。本研究中,vasa mRNA在发育10 d前微弱表达,从第13天开始表达量逐渐上升,至19 d达到最高,之后表达量又开始下降。在斑马鱼、栉孔扇贝中,vasa mRNA具有母源性特征遗传,在生殖过程中,随着配子的发生传递给下一代,分配到原始生殖质中,即早期vasa mRNA是由母系基因组传递给胚胎的(徐红艳等, 2010)。中华蛸可能在第13天细胞发生了分化,体细胞分化为原始生殖细胞(PGCs),原始生殖细胞逐渐积累并在第19天达到顶峰。随后从21 d到出膜阶段,各个组织器官逐渐发育形成,是体细胞分裂生殖的时期,体细胞数量大增,而生殖细胞的数量所占的比例减少,因此,vasa mRNA表达逐渐降低。这一表达结果和七彩神仙鱼相似(林睿涓等, 2017),推测中华蛸的原始生殖细胞在13 d时形成。Os-vasa基因在不同发育时期仔蛸中的表达结果显示,从出膜到17日龄,基因表达量都维持在较低水平,8日龄几乎检测不到表达信号,这可能因为随着受精过程的发生,母源性的vasa mRNA被激活以维持合子早期发育所需,随着发育的进行,母源性mRNA逐渐被消耗,仔蛸完成正常的生命活动则需依赖于自身基因组合成转录产物(许莉佳等, 2012);随着发育的推进,PGCs将穿过胚胎各组织(体细胞组织)在性原基聚集,并结合周围的体细胞,形成完整的初始生殖腺,进而分化为卵巢或精巢(朱新平等, 2017)。七彩神仙鱼出膜后30日龄和40日龄vasa的表达量较高,认为PGCs在分子水平可能已经开始分化(林睿涓等, 2017)。本研究结果与其相似,出膜后第20天和第23天vasa mRNA表达量较高,之后又维持稳定,推测第20天和第23天中华蛸的PGCs在分子水平可能已经开始分化,但这时期的高表达与性腺分化是否有关,仍需进一步在组织学水平进行鉴定。
未成熟的卵巢和成熟期的卵巢RT-qPCR检测结果显示,中华蛸未成熟和成熟卵巢均有vasa mRNA表达,并且未成熟期表达量较高。斑马鱼卵母细胞原位杂交结果显示,在卵母细胞的各阶段,vasa mRNA表达量存在差异,在Ⅱ期卵母细胞表达量最高,且定位到细胞质中,在第Ⅲ期开始下降并在后期表达量趋于稳定(项方, 2004)。对中华鳖(Trionyx sinensis)卵子发生的4个时期进行荧光原位杂交,在初级卵母细胞和最早期生长期卵母细胞检测出高表达信号,并认为与细胞进行RNA和蛋白质的储存有关,从生长期后表达信号逐渐减弱(朱新平等, 2017)。革胡子鲶(Clarias gariepinus)在性腺发育阶段qRT-PCR相关实验表明,与成熟卵母细胞(Ⅲ期和Ⅳ期卵母细胞)相比,未成熟卵母细胞(Ⅰ期和Ⅱ期)的vasa转录本较高(Raghuveer et al, 2010)。在罗非鱼(Oreochromis spp)(Kobayashi et al, 2000)和银鲫(Carassius auratus gibelio)(Xu et al, 2005)的研究中也有同样发现。中华蛸vasa mRNA在雌性生殖细胞的表达图谱与它们较为类似,未成熟期表达量较高,可能与此时期卵母细胞中RNA、蛋白质等物质大量积累有关;而在成熟期表达量减弱,这可能与转录产物的扩散使成熟卵细胞卵黄蛋白原的含量增加有关,而且随着卵母细胞体积的增大,Os-vasa mRNA相对密度减少。vasa mRNA在卵巢发育各时期的差异性表达表明,vasa mRNA与卵子发生密切相关。
4 结论本研究克隆了中华蛸vasa基因cDNA的全长序列,并以Os-vasa命名,对中华蛸胚胎发育、初孵幼体、卵巢的不同时期与中华蛸不同组织进行了表达分析。结果显示,中华蛸Os-vasa基因全长为2438 bp,其ORF长为2067 bp,编码688个氨基酸。氨基酸同源性序列分析表明,中华蛸Os-vasa与加州双斑蛸同源性最高。基因表达结果显示,Os-vasa基因主要在性腺中表达,且在卵巢的表达量高于精巢;vasa基因在中华蛸胚胎发育时期和出膜后29 d均有表达,胚胎期第19天表达量较高,出膜后第20天和第23天表达量较高;中华蛸未成熟和成熟卵巢均有vasa mRNA表达,且未成熟期表达量较高。本研究结果可为中华蛸的性分化、生殖细胞分子标记及发育研究提供参考。
CAI H C, ZHUANG D G, YE P, et al. Experiment on stock culturing, spawning and hatching of Octopus vulgaris. Marine Fisheries, 2009, 31(1): 58-65 [真蛸亲体培育、产卵及孵化试验. 海洋渔业, 2009, 31(1): 58-65 DOI:10.3969/j.issn.1004-2490.2009.01.009] |
CAO G L, ZHANG Y Y, XUE R Y, et al. Alternative splicing, expression patterns and promoter characters of vasa-like gene from the silkworm, Bombyx mori. Molecular Biology Reports, 2012, 39(5): 5967-5976 DOI:10.1007/s11033-011-1409-7 |
CARUTHERS J M, MCKAY D B. Helicase structure and mechanism. Current Opinion in Structural Biology, 2002, 12(1): 123-133 DOI:10.1016/S0959-440X(02)00298-1 |
CORDIN O, BANROQUES J, TANNER N K, et al. The DEAD-box protein family of RNA helicases. Gene, 2006, 367: 17-37 DOI:10.1016/j.gene.2005.10.019 |
DEHGHANI M, LASKO P. In vivo mapping of the functional regions of the DEAD-box helicase Vasa. Biology Open, 2015, 4: 450-462 DOI:10.1242/bio.201410579 |
FABIOUX C, CORPOREAU C, QUILLIEN V, et al. In vivo RNA interference in oyster-vasa silencing inhibits germ cell development. FEBS Journal, 2009, 276(9): 2566-2573 DOI:10.1111/j.1742-4658.2009.06982.x |
FABIOUX C, POUVREAU S, ROUX F L. The oyster vasa-like gene: A specific marker of the germline in Crassostrea gigas. Biochemical and Biophysical Research Communications, 2004, 315(4): 897-904 DOI:10.1016/j.bbrc.2004.01.145 |
HAY B, JAN L Y, JAN Y N. A protein component of Drosophila polar granules is encoded by vasa and has extensive sequence similarity to ATP-dependent helicases. Cell, 1988, 55(4): 577-587 DOI:10.1016/0092-8674(88)90216-4 |
HERPIN A, ROHR S, RIEDEL D, et al. Specification of primordial germ cells in medaka (Oryzias latipes). BMC Developmental Biology, 2007, 7(1): 3 DOI:10.1186/1471-213X-7-3 |
HU X. The effect of atrazine on ICR mice´s oocyte development ability. Master´s Thesis of Shanghai Ocean University of Shandong Normal University, 2015 [胡翔. 阿特拉津对ICR小鼠卵母细胞发育潜能的影响. 山东师范大学硕士研究生学位论文, 2015]
|
IKENISHI K, TANAKA T S. Spatio-temporal expression of Xenopus vasa homolog, XVLG1, in oocytes and embryos: The presence of XVLG1 RNA in somatic cells as well as germline cells. Development, Growth and Differentiation, 2000, 42(2): 95-103 DOI:10.1046/j.1440-169x.2000.00493.x |
KOBAYASHI T, KAJIURA-KOBAYASHI H, NAGAHAMA Y. Differential expression of vasa homologue gene in the germ cells during oogenesis and spermatogenesis in a teleost fish, tilapia, Oreochromis niloticus. Mechanisms of Development, 2000, 99(1): 139-142 |
KOICHIRO T, DANIEL P, NICHOLAS P, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Narnia, 2011, 28(10): 2731-2739 |
KRØVEL A V, OLSEN L C. Sexual dimorphic expression pattern of a splice variant of zebrafish vasa during gonadal development. Developmental Biology, 2004, 271(1): 190-197 DOI:10.1016/j.ydbio.2004.04.004 |
LIN R J, GAO J Z, JIN S R, et al. Cloning and expression analysis of Vasa gene in Symphysodon haraldi. Journal of Shanghai Ocean University, 2017, 26(3): 330-338 [七彩神仙鱼Vasa基因cDNA的克隆及表达分析. 上海海洋大学学报, 2017, 26(3): 330-338] |
LIU Y, WANG Q H, HUANG R L, et al. Cloning and expression analysis of vasa gene from Pinctada fucata martensii. Genomics and Applied Biology, 2017, 36(7): 2832-2839 [马氏珠母贝Pm-vasa基因的克隆与表达分析. 基因组学与应用生物学, 2017, 36(7): 2832-2839] |
NAGASAWA K, TAKEUCHI Y, MIWA M, et al. cDNA cloning and expression analysis of a vasa-like gene in Pacific bluefin tuna Thunnus orientalis. Fisheries Science, 2009, 75(1): 71-79 DOI:10.1007/s12562-008-0021-9 |
OLSEN L, AASLAND, RFJOSE A. A vasa-like gene in zebrafish identifies putative primordial germ cells. Mechanisms of Development, 1997, 66(1/2): 95-105 |
ÖZHAN-KIZIL G, HAVEMANN J, GERBERDING M. Germ cells in the crustacean Parhyale hawaiensis depend on Vasa protein for their maintenance but not for their formation. Developmental Biology, 2009, 327(1): 230-239 DOI:10.1016/j.ydbio.2008.10.028 |
RAGHUVEER K, SENTHILKUMARAN B. Cloning and differential expression pattern of vasa in the developing and recrudescing gonads of catfish, Clarias gariepinus. Comparative Biochemistry and Physiology Part A, Molecular and Integrative Physiology, 2010, 157(1): 79-85 DOI:10.1016/j.cbpa.2010.04.017 |
REUNOV A A, REUNOVA Y A. In mouse oocytes the mitochondrion-originated germinal body-like structures accumulate mouse Vasa homologue (MVH) protein. Zygote, 2015, 23(4): 501-506 DOI:10.1017/S0967199414000124 |
ROCAK S, LINDER P. DEAD-box proteins: The driving forces behind RNA metabolism. Nature Reviews Molecular Cell Biology, 2004, 5(3): 232-241 DOI:10.1038/nrm1335 |
ROGERS G JR, KOMAR A A, MERRICK WC. eIF4A: The godfather of the DEAD box helicases. Progress in Nucleic Acid Research and Molecular Biology, 2002, 72: 307-331 |
SHAO M Y. CDNA cloning and developmental expression patterns of reproduction-related DEAD-box family and boule genes from Chlamys farreri. Doctoral Dissertation of Qingdao Ocean University of China, 2007 [邵明瑜. 栉孔扇贝生殖相关因DEAD-box家族和boule的cDNA克隆及其发育表达图. 中国海洋大学博士研究生学位论文, 2007]
|
SUI J, ZHANG Z F, SHAO M Y, et al. Cloning and characterization of a vasa-like gene in Apostichopus japonicus and its expression in tissues. Journal of Fishery Sciences of China, 2008, 15(3): 407-413 [刺参vasa-like基因克隆及其在组织中的表达分析. 中国水产科学, 2008, 15(3): 407-413 DOI:10.3321/j.issn:1005-8737.2008.03.005] |
TANNER N K, LINDER P. DExD/H box RNA helicases from generic motors to specific dissociation functions. Molecular Cell, 2001, 8(2): 251-262 DOI:10.1016/S1097-2765(01)00329-X |
TSUNEKAWA N, NAITO M, SAKAI Y, et al. Isolation of chicken vasa homolog gene and tracing the origin of primordial germ cells. Development, 2000, 127: 2741-2750 DOI:10.1242/dev.127.12.2741 |
WU H R, CHEN Y T, SU Y H, et al. Asymmetric localization of germline markers Vasa and Nanos during early development in the amphioxus Branchiostoma floridae. Developmental Biology, 2011, 353(1): 147-159 DOI:10.1016/j.ydbio.2011.02.014 |
WU X, WANG Z, JIANG J, et al. Cloning, expression promoter analysis of vasa gene in Japanese flounder (Paralichthys olivaceus). Comparative Biochemistry and Physiology Part B, Biochemistry and Molecular Biology, 2014, 167: 41-50 DOI:10.1016/j.cbpb.2013.06.004 |
XIANG F. The expression of zebrafish vasa gene and gcl gene during oogenesis. Master′s Thesis of Wuhan University, 2004 [项方. 斑马鱼vasa基因和gcl基因在卵母细胞发生过程中的表达. 武汉大学硕士研究生学位论文, 2004]
|
XU H Y, LI M Y, GUI J F, et al. Fish germ-cell. Scientia Sinica Vitae, 2010, 40(2): 124-138 [鱼类生殖细胞. 中国科学: 生命科学, 2010, 40(2): 124-138] |
XU H, GUI J, HONG Y. Differential expression of vasa RNA and protein during spermatogenesis and oogenesis in the gibel carp (Carassius auratus gibelio), a bisexually and gynogenetically reproducing vertebrate. Developmental Dynamics, 2005, 233(3): 82-872 |
XU L J, ZHANG X, WU S, et al. Progress on research of germ-cell transplantation in fish. Chinese Bulletin of Life Sciences, 2012, 24(3): 280-286 [鱼类生殖细胞移植的研究进展. 生命科学, 2012, 24(3): 280-286] |
ZAMBONI L, UPADHYAY S. Germ cell differentiation in mouse adrenal glands. Journal of Experimental Zoology, 1983, 228(2): 173-193 DOI:10.1002/jez.1402280204 |
ZHENG X D, LIU Z S, ZHAO N, et al. Embryonic development and paralarvae growth of Octopus vulgaris. Oceanologia et Limnologia Sinica, 2011, 42(2): 317-323 [真蛸(Octopus vulgaris)胚胎发育及浮游期幼体生长研究. 海洋与湖沼, 2011, 42(2): 317-323] |
ZHOU Q R, SHAO M Y, ZHANG Z F. Structure characterization and application prospects of vasa protein. Transactions of Oceanology and Limnology, 2007(4): 129-134 [Vasa基因编码蛋白的结构特征和应用展望. 海洋湖沼通报, 2007(4): 129-134 DOI:10.3969/j.issn.1003-6482.2007.04.020] |
ZHOU Q R. cDNA cloning and expression analysis of two DEAD-box family genes, Fc-vasa and Fc-PL10a, from Fenneropenaeus chinensis. Master′s Thesis of Ocean University of China, 2007 [周倩如. 中国明对虾(Fenneropenaeus chinensis)两个DEAD-box家族基因Fc-vasa和Fc-PL10的克隆和表达分析. 中国海洋大学硕士研究生学位论文, 2007]
|
ZHU X P, XU H Y, ZHANG P Y, et al. Cloning and expression analysis of vasa in Chinese soft-shell turtle oocytes. Acta Hydrobiologica Sinica, 2017, 3(2): 306-313 [中华鳖vasa基因克隆及在卵母细胞中的表达分析. 水生生物学报, 2017, 3(2): 306-313] |



