2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业 可持续发展重点实验室 山东 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程 功能实验室 山东 青岛 266071;
4. 青岛瑞滋集团有限公司 山东 青岛 266408
2. Key Laboratory of Sustainable and Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao, Shandong 266071, China;
3. Pilot National Laboratory for Marine Science and Technology (Qingdao), Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao, Shandong 266071, China;
4. Qingdao Ruizi Company, Qingdao, Shandong 266408, China
仿刺参又称刺参(Apostichopus japonicus),具有重要的营养价值和药用价值,是我国第五次海水养殖浪潮的代表性物种之一,其养殖业为我国沿海经济结构调整和渔民增收开辟了一条新途径(王印庚等, 2014)。在人工养殖刺参过程中,刺参的病害问题日益凸显,给刺参养殖业造成了严重的经济损失,严重制约了该产业的稳定发展。开展刺参抗病机制解析对刺参良种选育和病害防控具有重要意义。DNA甲基化是表观遗传修饰的主要方式,在不改变DNA序列的前提下,改变遗传表现在调控基因表达和染色质构象等方面发挥着重要作用(Moore et al, 2013)。研究表明,刺参在肠道组织再生、夏眠、化皮及倍性等过程或现象中,相关组织均有显著的基因组甲基化水平差异变化,表明甲基化在刺参相关生理过程中发挥重要的调控作用(Yang et al, 2020; Zhao et al, 2015; Sun et al, 2020; Han et al, 2021; 高杉等, 2017; 李晓妮, 2018; 赵业, 2015)。本研究以刺参腐皮综合征重要致病原——灿烂弧菌(Vibrio splendidus)为病原对刺参苗种进行人工攻毒侵染实验,以攻毒后发病个体和未攻毒的健康个体的体壁组织为样本,采用全基因组甲基化测序技术(WGBS)和转录组高通量测序技术,分析灿烂弧菌胁迫下刺参体壁DNA甲基化水平变化及基因表达差异变化,并进一步对甲基化位点和转录组差异进行关联分析,筛选出关键位点及关联基因,为解析刺参响应灿烂弧菌侵染的分子机制提供基础数据,也有助于为刺参抗病性状选育提供参考。
1 材料与方法 1.1 实验材料本研究所用健康苗种取自山东青岛瑞滋集团有限公司,选取活力良好、健康的个体,苗种规格为(50.0±2.0) g/只。苗种运回实验室后暂养3 d,暂养水温为(13.0±0.5)℃,待苗种状态稳定后用于后续实验。
攻毒用菌株为本团队病原库中保存的分离自患腐皮综合征的刺参病样的灿烂弧菌菌株(AJ-Vb 1801)。对该菌株用胰蛋白胨大豆肉汤培养基(TSB)固体培养基复苏,然后用TSB液体培养基扩大培养。
1.2 实验样品的采集灿烂弧菌人工攻毒侵染与取样:根据实验目的将实验分为2组,每组设3个平行,每个平行实验苗种数量为30头,实验水槽容积为50 L。对照组(PT10H)为在水质良好的自然海水中养殖的健康苗种,侵染组(PT16S)按照水体体积在养殖水槽中添加培养的灿烂弧菌菌液至终浓度1×106 CFU/mL (该浓度为灿烂弧菌对刺参苗种的半致死浓度)。养殖条件:温度(13.0±0.5)℃,盐度(28.0±0.5),每天换1/3水,换水后及时添加灿烂弧菌菌液,使其维持在1×106 CFU/mL。每天换水后定时投喂刺参配合饲料,投喂量为刺参苗种体重的2%,养殖期间持续充气。实验期间,每天观察刺参苗种的生理状态,及时收集侵染组发生化皮的濒死个体,分别标记为PT16S1、PT16S2和PT16S3,在相同时间节点采集对照组健康个体,分别标记为PT10H1、PT10H2和PT10H3。剖取所采集样品的体壁组织,置于2 mL冻存管后迅速放入液氮罐中,送回实验室,–80℃保存,用于后期DNA和RNA的提取。
1.3 DNA和RNA的提取以对照组和侵染组的体壁组织为材料,分别采用Omega公司的Mollusc DNA kit和QIAGEN RNeasy mini kit提取各样品体壁基因组DNA和RNA,1.5%琼脂糖凝胶电泳检测DNA和RNA完整性,NanoDrop核酸测定仪测定所提取核酸样品的纯度,检测合格的样品于–80℃保存备用。
1.4 全基因组DNA甲基化测序将所提取的6个DNA样品送至杭州联川生物技术股份有限公司分别构建甲基化文库,并利用HiSeq4000测序平台150PE上机策略进行测序。
1.5 数据处理与比对测序完成后对原始数据进行预处理,去除可能含有测序接头序列和低质量的测序数据(含有N的比例大于5%,以及质量值Q≤10的碱基数占整个read的20%以上的reads),得到有效数据,用于后续的生物信息学分析。
以本团队组装的刺参全基因组序列(数据待发表)为基础,对预处理后的有效测序数据使用Bismark (Krueger et al, 2011)进行参考基因组序列比对,统计基因组比对结果。
1.6 刺参体壁基因组DNA甲基化分布特征分析根据基因组比对分析结果,统计所测样品各类型甲基化C位点(mCpG、mCHG和mCHH)的数目,及其在全部mC的位点中所占的比例,解析物种的全基因组DNA甲基化修饰特征。
1.7 差异甲基化区域分析使用R语言程序包中的methylKit筛选差异甲基化区域(differentially methylated regions, DMRs),默认选择1000 bp windows、500 bp overlap、FDR < 0.05为差异筛选阈值,进行DMR分析。统计差异表达分析中侵染组的甲基化水平显著性升高或下降的基因数目。对筛选到的具有DMR的基因进行注释,按照基因结构绘制柱状统计图。
1.8 转录组中差异表达基因的筛选将所采集的6个样品的RNA送至杭州联川生物技术股份有限公司分别构建cDNA文库,采用Illumina NovaseqTM 6000进行测序,测序读长为双端2×150 bp (PE150)。使用String Tie软件计算不同样品间各基因表达定量的结果,以FPKM (fragments per kilobase of exon model per million mapped reads)为单位对已知基因统计其在不同样本中的表达丰度,筛选差异表达基因(different expression genes, DEGs)。
1.9 DNA甲基化与转录组联合分析对转录组测序和基因甲基化测序数据进行联合分析,统计DMRs和DEGs注释的共有基因,检测这些共有基因的DMR甲基化水平与DEG表达水平之间的相关性,统计DMR甲基化水平与DEG表达水平存在负相关的基因,分别采用GOseq和KOBAS软件对筛选到的差异甲基化基因进行GO和KEGG pathway富集分析,采用ggplot2软件绘制差异甲基化基因的KEGG散点图。
2 结果 2.1 甲基化测序结果统计对实验获得的对照组(PT10H)和侵染组(PT16S)的6个样品的WGBS测序和数据统计分析结果见表 1。高通量测序平均每个样品产出165 510 349条的原始数据,对原始数据进行预处理后,截去测序数据的测序接头和去除低质量的数据,得到均值为23.20 Gb的有效数据,有效序列占比在99.18%以上,Q30以上的数据占比大于91.96%,有37.31%~39.81%的mapped reads可比对到刺参的参考基因组,说明测序数据的质量较好。
|
|
表 1 WGBS测序数据统计 Tab.1 Statistics of the WGBS sequencing data |
根据基因组比对分析结果,表 2统计了对照组和侵染组刺参体壁组织中C位点以及不同类型(CG、CHG和CHH)的平均甲基化水平。侵染组样品基因组甲基化C位点占全部C位点数的(3.87±0.27)%,对照组为(3.59±0.04)%,说明在病原菌侵染胁迫下,基因组甲基化C位点占比显著升高(P < 0.05)。不同类型(CpG、CHG和CHH)的平均甲基化水平统计结果显示,对照组mCpG、mCHG和mCHH占CpG、CHG和CHH的比例分别为24.14%、0.56%和0.47%,而侵染组的mCpG、mCHG和mCHH占CpG、CHG和CHH的比例分别为24.22%、0.98%和0.76%。由此可见,对于刺参基因组来说,CpG位点发生甲基化的比例显著高于CHG和CHH。从对照组和侵染组不同类型位点甲基化对比结果可以看出,侵染组的mCHG和mCHH比例显著高于对照组。
|
|
表 2 不同类型C位点甲基化水平 Tab.2 Methylation level of different cytosine contexts |
对照组和侵染组基因组DNA不同形式甲基化C位点占总甲基化C位点的比例见图 1。如图 1所示,在总的mC位点中,侵染组和对照组mCpG占比分别为83.06%和81.91%,mCHH占比分别为13.41%和14.28%,mCHG占比分别为3.53%和3.81%,说明甲基化位点主要集中在CpG二核苷酸序列的胞嘧啶上,CpG甲基化是主要的甲基化形式。
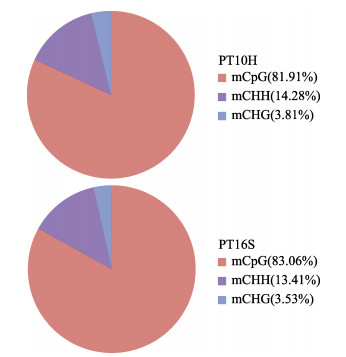
|
图 1 对照组(PT10H)和侵染组(PT16S) mCpG、mCHG和mCHH占总mC的百分比 Fig.1 Methylation percentage of mCpG, mCHG, and mCHH to total mC in PT10H and PT16S groups |
使用R语言程序包中的methylKit分析自对照组和侵染组样品测序结果中筛选到的626 677个DMRs。根据分组的差异比较结果,对发生显著甲基化的基因数量的统计结果见图 2。从对照组和侵染组共筛选到的启动子区域DMRs共有40 329个(6.44%),其中,甲基化水平升高的有22 613个,下降的有17 716个;外显子区域存在58 522个DMRs (9.34%),其中,升高的有29 731个,下降的有28 791个;内含子区域DMRs共有109 635个(17.49%),其中,升高的有56 603个,下降的有53 032个;基因间区DMRs共有401 174个(64.02%),其中,升高的有216 152个,下降的有185 022个;CGI.CG岛DMRs共有17 017个(2.72%),其中,升高的有8877个,下降的有8140个。刺参基因间区的差异甲基化基因比例最高,CGI.CG岛区域的差异甲基化比例最低。
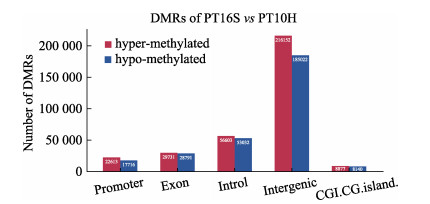
|
图 2 不同基因功能区域的DMR数量 Fig.2 The number of DMRs in different gene functional regions |
由对照组和侵染组转录组测序分析共检测出29 290个基因,对所检测的基因表达进行差异显著性分析,共筛选到496个显著性差异表达基因(DEGs),其中,上调性基因214个,下调性基因有282个(图 3)。从整体上来看,下调基因数目多于上调基因数目。
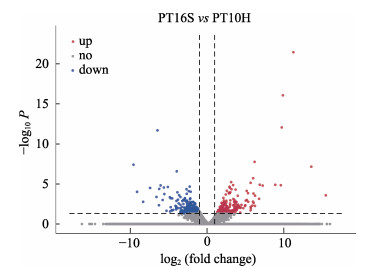
|
图 3 PT16S与PT10H差异表达基因火山图 Fig.3 Valcano plot of DEGs between PT16S and PT10H |
对DMRs功能基因注释筛选到的23 706个功能基因和DEGs筛选到的496个功能基因进行比对,筛选到DMRs和DEGs注释的共有基因有261个(图 4)。对这261个基因的DMRs差异和基因表达差异进行关联分析,结果见表 3。共筛选到180个负相关关联基因,其中,甲基化水平高而基因表达下调的负相关关联基因有58个,甲基化水平低而基因表达上调的负相关关联基因有122个。对所筛选的负相关关联基因差异甲基化区域进行统计,差异甲基化区域位于启动子区域的负相关基因有60个。

|
图 4 差异表达基因和差异甲基化区域共注释基因的韦恩图 Fig.4 Venn graph between the differentially expressed genes and the differentially methylated regions |
|
|
表 3 基因组甲基化DMRs与转录组DEGs关联分析筛选基因统计 Tab.3 Statistics of gene number detected by association analysis between DMRs and DEGs |
对所筛选的甲基化和转录组负相关的180个基因进行GO富集分析,结果显示(图 5),甲基化水平与表达水平呈负相关的180个基因中,有124个基因注释到896个GO terms,其中,50个GO terms显著富集(P < 0.05)。富集到生物过程的GO terms主要包括细胞粘附、DNA转录模板、先天性免疫应答、氧化还原过程、RNA聚合酶Ⅱ对转录的负调控等;富集到细胞成分的GO terms主要包括膜的组成部分、细胞质、细胞膜、细胞核等;富集到分子功能的GO terms主要包括蛋白结合、金属离子结合、钙离子结合、ATP结合、锌离子结合、DNA结合、转运蛋白活性等。筛选到的功能基因包括免疫球蛋白、富含亮氨酸的重复序列(LRR)、有机阴离子转运蛋白多肽(OATP)、半胱天冬酶募集结构域(CARD)、黄素结合单加氧酶(almA)、CD36、血红素结合蛋白等。
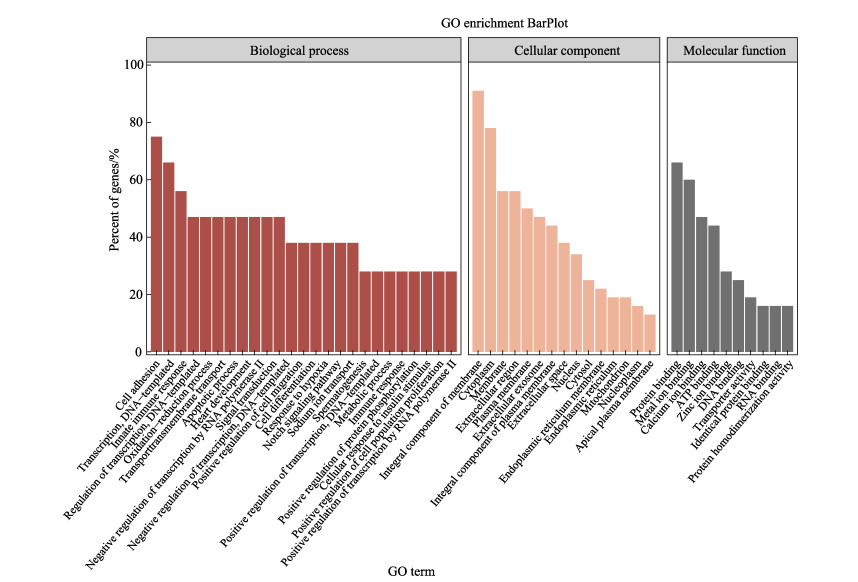
|
图 5 甲基化与基因表达负相关基因GO富集 Fig.5 GO enrichment of negative correlated genes detected by association analysis between DMRs and DEGs |
利用KEGG数据库对差异表达基因进行pathway分析,结果见图 6,甲基化水平与基因表达水平呈负相关的有180个基因,其中,39个富集基因注释到112个KEGG信号通路中,有20个通路显著富集(P < 0.05),内质网中的蛋白质加工、破骨细胞分化、乙型肝炎、维生素消化吸收这4个通路注释基因最多,均有3个负相关关联基因富集在相应通路上,筛选到的功能基因包括hsp20和CD36等。

|
图 6 甲基化与基因表达负相关基因KEGG富集 Fig.6 KEGG enrichment of negative correlated genes detected by association analysis between DMRs and DEGs |
DNA甲基化是表观遗传学研究的重要内容,是基因组DNA的一种重要修饰方式,参与生物体或细胞的生物调控过程(Moore et al, 2013)。水生生物在响应温度、病原、饲料、倍性及杂种优势过程中DNA甲基化均发生了改变(吴彪等, 2016; 高杉等, 2017; 卓梅琴等, 2019; Han et al, 2021; 李炎璐等, 2019)。
目前,水产动物研究中常用的DNA甲基化检测方法有基于限制性酶切预处理的甲基化敏感扩增多态性技术(MASP)和基于高通量测序的亚硫酸氢盐处理的全基因组DNA甲基化测序技术(WGBS)。MASP技术具有简便、高效和可靠的特点。研究人员利用该技术探究了刺参不同群体(左之良等, 2016; 左闪等, 2021)、不同组织(郭婷婷, 2013)、高温胁迫(李尚俊等, 2017)、夏眠(赵业, 2015)和化皮(高杉等, 2017)的甲基化水平差异。但MASP无法完全准确地显示基因组中甲基化情况的全貌及具体的甲基化位点的序列等信息。
随着高通量测序技术的发展,WGBS成为检测DNA甲基化的最可靠技术之一,该方法可以直观地反映全基因组DNA甲基化的详细信息,可以直接提供筛选出的位点及其对应的信息序列。Yang等(2020)运用该技术筛选到夏眠期刺参的DMR区域和关键功能基因;温争争等(2021)利用WGBS技术完成了刺参高温胁迫下基因组DNA甲基化水平及模式的变化研究;Sun等(2020)利用该技术筛选到了高浓度灿烂弧菌侵染下刺参化皮组织DMR区域。全基因组序列信息是开展WBGS分析的关键,参考基因组拼接的完整性对DMR区域的筛选和注释效果产生直接影响。Zhang等(2017)和Li等(2018)获得NCBI数据库中的刺参基因组序列信息,共包括3821个Scaffolds (N50为786 kb)。Sun等(2020)利用高浓度灿烂弧菌(5×109 CFU/mL)攻毒制备化皮实验材料,利用NCBI中的全基因组数据比对筛选得到116 522个DMRs。本团队于2020年采用纳米孔测序技术和染色体构象捕获技术(Hi-C)开展了刺参基因组精细图谱绘制,拼接获得911 Mb的染色体水平的刺参基因组,共定位到23条染色体,N50为39.61 Mb (相关数据未发表),基因组拼接质量得到较大提升。
本研究利用灿烂弧菌半致死浓度(1×106 CFU/mL)制备化皮实验材料,利用染色体水平基因组序列筛选得到的DMRs数目为626 677个,显著高于Sun等(2020)筛选到的116 522个DMRs,所筛选的DMRs的差异数除了与实验条件差异有关外,对比时所选用的基因组拼接质量是产生此差异的主要原因之一。
3.2 刺参基因组甲基化特征及其响应病原胁迫的变化高通量测序获得的所有样品的基因组甲基化水平检测结果表明,本研究所测定的刺参体壁组织甲基化水平为3.52~4.17%。Yang等(2020)测定的夏眠期刺参基因组甲基化水平在3.5%左右;Sun等(2020)检测刺参甲基化水平在4%左右。可以看出,刺参基因组整体甲基化水平较低,这与Tweedie等(1997)发现的无脊椎动物基因组甲基化水平通常低于脊椎动物的结果相一致。
对刺参的不同类型C位点甲基化水平的统计结果可以看出,CG类型位点的甲基化比例较高,而CHG和CHH位点甲基化比例很低,与目前已报道的其他海洋无脊椎动物CG类型甲基化水平范围(20%~ 50%)相一致(曹哲明等, 2009; 于涛等, 2010; Sun et al, 2014)。进一步对发生甲基化C位点的分型比例统计结果可以看出,mCpG占甲基化位点的比例达到80%以上,表明mCpG是最主要的甲基化形式。与李玉强等(2018)利用methylRAD-seq技术对仿刺参的甲基化图谱研究结果相一致。这些结果表明海洋动物的DNA甲基化模式都有一定的相似性和保守性。
对病原侵染刺激下基因组甲基化水平的变化可以看出,侵染组样本的甲基化水平显著高于对照组。虽然CG位点甲基化比例未发生显著变化,但CHH和CHG的甲基化比例显著升高,证明病原胁迫会引起基因组甲基化水平的升高。高杉等(2017)、Yang等(2020)、Sun等(2020)和温争争等(2021)研究结果也表明,温度和高浓度病原刺激等外界胁迫会引起基因组甲基化水平的升高,尤其是CHH和CHG的甲基化比例的升高。虽然mCpG是刺参最主要的甲基化形式,但在不同刺激影响下的占比仍存在差异。本研究在病原半致死浓度刺激下,mCpG的占比在82%左右。Sun等(2020)测定的在致死浓度病原刺激下,mCpG的占比为87%~89%,但在温度刺激或夏眠状态下,mCpG的占比达到94%以上。相关对比分析证明,不同的刺激形式造成的基因组甲基化存在差异。
3.3 刺参响应灿烂弧菌侵染差异甲基化位点和差异表达基因分析本研究利用染色体水平基因组为基础,筛选得到626 677个DMRs,且在侵染组中筛选到的高甲基化DMR数量大于低甲基化DMR数量,这与侵染组全基因组甲基化水平增高相一致。根据这些DMR所在的功能区域的位置统计结果可以看出,基因间区的差异甲基化位点数量最高,这与李玉强(2018)利用methylRAD-seq技术分析结果解释的甲基化标签较大比例分布于基因间区的结果相一致。对筛选的DMR区域进行功能基因注释得到23 706个功能基因,所注释的功能基因数目也远高于Sun等(2020),这可能也与实验条件的差异和比对所选用的基因组序列不同有关。转录组测序结果表明,侵染组筛选到的下调基因数目多于上调基因数目,这个趋势与侵染组筛选到高甲基化位点数目较多正好相反,也从侧面验证了Choy等(2010)提出的甲基化水平的升高会抑制基因表达,二者存在负调控关系。在所筛选到的496个差异表达基因中发现DNA甲基化转移酶的表达显著上调,相关结果与Yang等(2020)和李尚俊等(2017)检测到的相关基因上调表达相一致,也解释了该基因上调表达与基因组甲基化水平的升高直接相关。
3.4 刺参响应病原胁迫的基因组甲基化与基因表达的关联分析基因组DNA甲基化作为基因的表达调控的重要方式,在通常情况下,DNA低甲基化会激活基因的转录,而高甲基化则抑制基因的转录(Bird, 2002)。如早期的海胆(Sphaerechinus granularis)甲基化研究报道了DNA甲基化对于海胆发育具有重要意义(Tosi et al, 1995)。本研究自甲基化和转录组关联分析中筛选到261个共同注释的基因,其中180个为负相关基因,占比为68.97%,说明所筛选到的基因其DNA甲基化对基因表达发挥了重要作用。这些负相关基因是后期解析刺参表观调控需要重点关注的对象。
本研究对所筛选的180个负相关基因进行GO和KEGG分析。在甲基化和转录组负相关的180个基因的GO富集分析中,筛选到生物进程、细胞成分和分子功能多个GO terms中的富含亮氨酸的重复序列LRR基因,该基因包含许多抗病基因,能参与固有免疫(Gou et al, 2010)。富集到的CARD蛋白参与先天性免疫应答、细胞质、ATP结合、乙型肝炎、RIG-I样受体信号通路、麻疹、甲型流感等通路,进而参与包括炎症反应、免疫、分化、细胞生长、肿瘤发生和凋亡等许多生物过程(Beg et al, 1994)。此外,筛选到的免疫球蛋白能够避免免疫系统的过分激活,降低炎症反应。虽然在KEGG通路中富集的负相关基因数量仅为39个,但筛选到的Hsp20和CD36基因参与免疫调控、细胞粘附等多种生理和病理过程(Li et al, 2015; 魏书磊等, 2013)。这些重要的功能基因将成为解析刺参响应病原侵染分子机理的重要关注点。
综上所述,本研究以染色体水平的基因组序列为基础,通过对病原侵染后化皮和健康样品体壁组织的WGBS和转录组测序分析,筛选到相应的差异甲基化位点和差异表达基因,进一步通过关联分析筛选到刺参响应病原侵染的关键基因及其甲基化特征,相关结果将为解析刺参抗病调控机理奠定基础。
BEG A A, BALDWIN A S. Activation of multiple NF-kappa B/Rel DNA-binding complexes by tumor necrosis factor. Oncogene, 1994, 9(5): 1487-1492 |
BIRD A. DNA methylation patterns and epigenetic memory. Genes and Development, 2002, 16(1): 6-21 DOI:10.1101/gad.947102 |
CAO Z M, YANG J. Analysis of the methylation in genome DNA from different tissues of Anodonta woodiana. Ecology and Environmental Sciences, 2009, 18(6): 2011-2016 [曹哲明, 杨健. 背角无齿蚌不同组织的基因组DNA甲基化分析. 生态环境学报, 2009, 18(6): 2011-2016 DOI:10.3969/j.issn.1674-5906.2009.06.001] |
CHOY M K, MOVASSAGH M, GOH H, et al. Genome-wide conserved consensus transcription factor binding motifs are hyper-methylated. BMC Genomics, 2010, 11(1): 519 DOI:10.1186/1471-2164-11-519 |
GAO S, YANG A F, DONG Y, et al. MSAP analysis of DNA methylation in the body wall of Apostichopus japonicus. Acta Hydrobiologica Sinica, 2017, 41(3): 637-642 [高杉, 杨爱馥, 董颖, 等. 仿刺参"化皮病"体壁组织DNA甲基化的MSAP分析. 水生生物学报, 2017, 41(3): 637-642] |
GOU X P, HE K, YANG H, et al. Genome-wide cloning and sequence analysis of leucine-rich repeat receptor-like protein kinase genes in Arabidopsis thaliana. BMC Genomics, 2010, 11: 19 DOI:10.1186/1471-2164-11-19 |
GUO T T. The method to assess genome DNA methylation of Apostichopus japonicus by HPLC and MSAP analysis in different tissues. Masterxs Thesis of Shanghai Ocean University, 2013 [郭婷婷. 刺参不同组织基因组DNA甲基化状态MSAP分析及HPLC方法的建立. 上海海洋大学硕士研究生学位论文, 2013]
|
HAN L S, SUN Y, CAO Y, et al. Analysis of the gene transcription patterns and DNA methylation characteristics of triploid sea cucumbers (Apostichopus japonicus). Scientific Reports, 2021, 11(1): 7564 DOI:10.1038/s41598-021-87278-9 |
KRUEGER F, ANDREWS S R. Bismark: A flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics, 2011, 27(11): 1571-1572 DOI:10.1093/bioinformatics/btr167 |
LI J K, WU X W, TAN J, et al. Molecular cloning of the heat shock protein 20 gene from Paphia textile and its expression in response to heat shock. Chinese Journal of Oceanology and Limnology, 2015, 33(4): 919-927 DOI:10.1007/s00343-015-4223-6 |
LI S J, SUN G H, LI X Y, et al. Characteristics of epigenetic regulation of related genes under high temperature stress in sea cucumber Apostichopus japonicus. Journal of Fishery Sciences of China, 2017, 24(3): 470-476 [李尚俊, 孙国华, 李雪燕, 等. 高温胁迫下仿刺参表观遗传调控相关基因的表达特征. 中国水产科学, 2017, 24(3): 470-476] |
LI X N. Analysis of DNA methylation regulation and function of candidated genes during intestinal regeneration in the sea cucumber Apostichopus japonicus. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, University of Chinese Academy of Sciences, 2018 [李晓妮. 刺参肠道再生的DNA甲基化调控解析及再生候选基因的功能分析. 中国科学院大学(中国科学院海洋研究所)博士研究生学位论文, 2018]
|
LI Y L, CHEN C, CHEN J G, et al. DNA methylation analysis of Epinephelus moara, Epinephelus lanceolatus and their F1 hybrid. Progress in Fishery Sciences, 2019, 40(6): 98-104 [李炎璐, 陈超, 陈建国, 等. 云纹石斑鱼、鞍带石斑鱼及其杂交F1的DNA甲基化分析. 渔业科学进展, 2019, 40(6): 98-104] |
LI Y Q, WANG R J, LI Y L, et al. Genome-wide profiling of DNA methylation in Apostichopus japonicus based on methylRAD-Seq. Periodical of Ocean University of China (Natural Science), 2018, 48(9): 41-50 [李玉强, 王睿甲, 李语丽, 等. 基于MethylRAD-Seq技术对仿刺参DNA甲基化图谱的研究. 中国海洋大学学报(自然科学版), 2018, 48(9): 41-50] |
LI Y, WANG R, XUN X, et al. Sea cucumber genome provides insights into saponin biosynthesis and aestivation regulation. Cell Discovery, 2018, 4(1): 29 DOI:10.1038/s41421-018-0030-5 |
MOORE L D, LE T, FAN G P. DNA methylation and its basic function. Neuropsychopharmacology, 2013, 38(1): 23-38 DOI:10.1038/npp.2012.112 |
SUN H J, ZHOU Z C, DONG Y, et al. Insights into the DNA methylation of sea cucumber Apostichopus japonicus in response to skin ulceration syndrome infection. Fish and Shellfish Immunology, 2020, 104: 155-164 DOI:10.1016/j.fsi.2020.05.005 |
SUN Y, HOU R, FU X, et al. Genome-wide analysis of DNA methylation in five tissues of Zhikong scallop, Chlamys farreri. PloS One, 2014, 9(1): e86232 DOI:10.1371/journal.pone.0086232 |
TOSI L, ANIELLO F, GERACI G, et al. DNA methyltransferase activity in the early stages of a sea urchin embryo-evidence of differential control. FEBS Letters, 1995, 361(1): 115-117 DOI:10.1016/0014-5793(95)00160-B |
TWEEDIE S, CHARLTON J, CLARK V, et al. Methylation of genomes and genes at the invertebrate-vertebrate boundary. Molecular and Cellular Biology, 1997, 17(3): 1469-1475 DOI:10.1128/MCB.17.3.1469 |
WANG Y G, RONG X J, LIAO M J, et al. Sea cucumber culture and disease control technology. Beijing: China Agriculture Press, 2014 [王印庚, 荣小军, 廖梅杰, 等. 刺参健康养殖与病害防控技术丛解. 北京: 中国农业出版社, 2014]
|
WEI S L. Expression and functional analysis of CD36 gene in zebrafish. Master′s Thesis of China Ocean University, 2013 [魏书磊. 斑马鱼(Danio rerio)重组CD36基因的原核表达及功能研究. 中国海洋大学硕士研究生学位论文, 2013]
|
WEN Z Z, ZUO S, CHEN M, et al. DNA methylation level of genomic DNA of Apostichopus japonicus at different temperatures. Progress in Fishery Sciences, 2021, 42(3): 46-54 [温争争, 左闪, 陈梦, 等. 刺参基因组DNA甲基化水平及模式对温度变化的响应. 渔业科学进展, 2021, 42(3): 46-54] |
WU B, YANG A G, SUN X J, et al. Effects of acute temperature stress on genome-wide DNA methylation profiles in Patinopecten yessoensis. Progress in Fishery Sciences, 2016, 37(5): 140-146 [吴彪, 杨爱国, 孙秀俊, 等. 急性温度胁迫对虾夷扇贝(Patinopecten yessoensis)基因组DNA甲基化的影响. 渔业科学进展, 2016, 37(5): 140-146] |
YANG Y J, ZHENG Y Q, SUN L N, et al. Genome-wide DNA methylation signatures of sea cucumber Apostichopus japonicus during environmental induced aestivation. Genes, 2020, 11(9): 1020 DOI:10.3390/genes11091020 |
YU T, YANG A G, WU B, et al. Analysis of Chlamys farreri, Patinopecten yessoensis and their offspring using methylation-sensitive amplification polymorphism (MSAP). Journal of Fisheries of China, 2010, 34(9): 1335-1342 [于涛, 杨爱国, 吴彪, 等. 栉孔扇贝、虾夷扇贝及其杂交子代的MSAP分析. 水产学报, 2010, 34(9): 1335-1342] |
ZHANG X J, SUN L, YUAN J B, et al. The sea cucumber genome provides insights into morphological evolution and visceral regeneration. PLoS Biology, 2017, 15(10): e2003790 DOI:10.1371/journal.pbio.2003790 |
ZHAO Y, CHEN M Y, STOREY K B, et al. DNA methylation levels analysis in four tissues of sea cucumber Apostichopus japonicus based on fluorescence-labeled methylation- sensitive amplified polymorphism (F-MSAP) during aestivation. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2015, 181: 26-32 DOI:10.1016/j.cbpb.2014.11.001 |
ZHAO Y. Study on gene expression patterns and basic characteristics of DNA methylation in Apostichopus japonicus (Selenka) during aestivation. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2015 [赵业. 刺参Apostichopus japonicus (Selenka)夏眠期间基因表达模式及DNA甲基化基础特征研究. 中国科学院研究生院(海洋研究所)博士研究生学位论文, 2015]
|
ZHUO M Q, YANG S B, LING S C, et al. Effects of dietary lipid on lipid metabolism, methylation and expression of PI3KCa in the ovary of yellow catfish (Pelteobagrus fulvidraco). Journal of Fisheries of China, 2019, 43(10): 2186-2196 [卓梅琴, 杨水波, 凌仕诚, 等. 饲料脂肪对黄颡鱼卵巢脂类代谢以及PI3KCa甲基化和转录水平的影响. 水产学报, 2019, 43(10): 2186-2196] |
ZUO S, WEN Z Z, ZHOU H X, et al. Evaluation of epigenetic and genome sequence diversity in sea cucumber Apostichopus japonicus selected population based on MSAP technology. Progress in Fishery Sciences, 2021, 42(3): 38-45 [左闪, 温争争, 周红学, 等. 基于MSAP技术的刺参选育群体基因组表观与序列遗传多样性分析. 渔业科学进展, 2021, 42(3): 38-45] |
ZUO Z L, TAN J, WU B, et al. MSAP analysis of genomic DNA in the tissues of Apostichopus japonicus and white A. japonicus. japonicus. Progress in Fishery Sciences, 2016, 37(3): 93-100 [左之良, 谭杰, 吴彪, 等. 普通刺参(Apostichopus japonicus)和白刺参不同组织基因组DNA的MSAP研究. 渔业科学进展, 2016, 37(3): 93-100] |



