2. 山东省资源与环境研究院山东省海洋生态修复重点实验室 山东 烟台 264006;
3. 中国科学院烟台海岸带研究所 山东 烟台 264003
2. Shandong Marine Resource and Environment Research Institute, Shandong Key Laboratory of Marine Ecological Restoration, Yantai, Shandong 264006, China;
3. Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai, Shangdong 264003, China
免疫识别是先天免疫的第一道防线,通过模式识别蛋白(pattern recognition proteins, PRPs)结合微生物表面高度保守的病原相关分子结构,如细菌中的脂多糖、肽聚糖,真菌中的β-1, 3-葡聚糖以及病毒双链RNA等(Huang et al, 2019; Li et al, 2020; Rout et al, 2020)。目前,已在无脊椎动物中鉴定出肽聚糖识别蛋白(peptidoglycan recognition proteins, PGRPs)、C1q结构域蛋白、NOD2、革兰氏阴性菌结合蛋白、含硫酯键蛋白、Toll样受体、脂多糖和(或)β-1, 3葡聚糖结合蛋白和清道夫受体等多种PRPs,它们对入侵病原微生物的免疫识别及应答起重要作用(曹丹丹等, 2018; Wang et al, 2013; Gerdol et al, 2015)。
PGRPs首次在家蚕(Bombyx mori)的血淋巴和角质层中被鉴定,它能特异性结合肽聚糖,并触发酚氧化酶级联反应(Li et al, 2020)。随后,在果蝇(Drosophila melanogaster)中成功克隆出PGRP后,其他无脊椎动物的PGRPs也相继被鉴定克隆(Hu et al, 2020)。在结构上,PGRP包含一个保守的肽聚糖结合2型酰胺酶结构域,也被称为PGRP结构域,它能够水解N-乙酰胞壁酸(N-acetylmuramic)和L-丙氨酸(L-alanine)之间的酰胺键,以达到抗菌目的(Zaidman-Rémy et al, 2006; Mellroth et al, 2006)。根据PGRPs分子量及结构的不同,可将其分为短型(PGRP-S)、中间型(PGRP-I)和长型(PGRP-L) 3类(Dziarski, 2004)。PGRPs在抵御病原菌的先天免疫中具有多种功能,其中,识别并结合细菌肽聚糖是其最基本的功能(Yang et al, 2010)。另外,PGRPs可作为免疫识别蛋白激活免疫通路(Wei et al, 2018),通过诱导细菌的氧化应激、硫醇应激和金属应激等作为直接杀菌剂(Kashyap et al, 2014; Hu et al, 2020),此外,PGRPs可作为调理素来诱导凝集或吞噬作用(Coteur et al, 2007; Yang et al, 2019)。
皱纹盘鲍(Haliotis discus hannai)是我国重要的海水养殖经济贝类,主要分布在辽东半岛和山东半岛(姜向阳等, 2021)。自20世纪80年代以来,由于病原菌入侵,尤其是弧菌的感染造成人工养殖和野生鲍鱼出现大规模死亡,其中鳗弧菌(Vibrio anguillarum)通过产生热稳定和热不稳定的毒素使宿主发病,导致全球鲍鱼产量大幅下降(王瑞旋等, 2006; 何婷婷, 2018; 吴富村等, 2020)。因此,研究免疫相关分子的特性对鲍鱼养殖业的疾病控制和健康管理具有重要意义。迄今为止,已在软体动物中鉴定出100多种PGRPs,如大竹蛏(Solen grandis)中的SgPGRP-S1 (Wei et al, 2018),仿刺参(Apostichopus japonicus)中的AjPGRP-S (Hu et al, 2020)。然而,目前对鲍鱼中PGRP功能的认识还很有限。本研究从皱纹盘鲍中鉴定了一种PGRP (命名为HdPGRP),并对其组织分布、入侵病原菌时的转录表达模式、抗菌活性等进行研究,以更好地了解鲍鱼对病原体入侵的免疫应答反应。
1 材料与方法 1.1 鲍鱼选取及细菌刺激选取平均壳长为60 mm的皱纹盘鲍,在海水中驯养1周(20℃~22℃),将300只鲍鱼平均分为6组。其中3组分别用鳗弧菌(1×107 CFU/mL)进行浸泡刺激,每组分别在0、3、6、12、24、48和72 h时随机选6个个体抽取血淋巴,2600 r/min离心10 min,获得血细胞。另外3组为对照组,每组随机选取6个个体,取血细胞、外套膜、鳃、肝胰腺和肌肉进行hdpgrp的组织特异性表达分析。
1.2 总RNA的提取及cDNA的合成使用TRIzol (Invitrogen, 美国)从鲍鱼血细胞、外套膜、鳃、肝胰腺和肌肉中提取总RNA,1.0%琼脂糖凝胶电泳检测RNA质量后,用2 μg总RNA和0.5 mmol/L Oligo (dT) 70℃热变性5 min后逆转录合成cDNA。反应体系(25 μL):已完成变性的RNA,5 μL dNTPs,1 μL RNase抑制剂(Promega, 美国),1 μL M-MLV逆转录酶(Promega, 美国)以及5 μL 5×M- MLV反应缓冲液,加无RNA酶水至25 μL。
1.3 hdpgrp基因的克隆通过对本实验室构建的皱纹盘鲍cDNA文库中EST序列进行BLAST分析,得到皱纹盘鲍肽聚糖识别蛋白的EST序列。以P1、P2为正向引物(表 1),Oligo (dT)为反向引物,进行巢式PCR扩增hdpgrp的3′端。步骤如下:加入聚合酶延长变性(94℃ 5 min);94℃ 50 s,58℃ 45 s,72℃ 30 s,35次循环扩增;PCR产物经凝胶纯化后,连接到pMD18-T载体(TaKaRa, 大连)进行测序。将原始EST序列与扩增片段重叠得到hdpgrp的全长cDNA。
|
|
表 1 实验所用引物 Tab.1 Primers used in this study |
用BLAST算法对hdpgrp的cDNA序列进行分析,并通过ExPASy (expert protein analysis system)推导出蛋白序列进行分析。使用SMART (simple modular architecture research tool)预测蛋白结构域。采用
ClustalW软件进行多序列比对。利用MEGA 4.0软件的邻位连接(neighbor-joining, NJ)算法构建系统发育树(重复1000次)。
1.5 hdpgrp mRNA的转录表达采用实时荧光定量PCR (qRT-PCR)技术分析hdpgrp mRNA的转录表达。用基因特异性引物(P3、P4, 表 1)扩增hdpgrp片段,β-actin作为内参基因,以1.2所合成的cDNA为模板,使用7500 Fast Real-time PCR系统(Applied Biosystems, 美国)进行qRT-PCR。反应程序:94℃,5 min变性;94℃ 50 s,60℃ 60 s,72℃ 50 s循环40次,72℃延伸10 min。用2–ΔΔCt法分析hdpgrp mRNA的相对表达水平(Livak et al, 2001)。所有数据以mRNA相对表达量的平均值±标准差(Mean±SD, n=6)表示,采用SPSS 16.0软件对所得数据进行统计学分析,P < 0.05为具有显著性差异。
1.6 HdPGRP的重组表达用一对特异性引物(P7和P8, 表 1)扩增hdpgrp,然后将PCR产物克隆到pEASY-blunt E1表达载体(Transgen Biotech, 中国)。在大肠杆菌(Escherichia coli) BL21 (DE3)中表达重组质粒,经IPTG诱导后,用15% SDS-PAGE检测蛋白表达结果。重组蛋白HdPGRP (rHdPGRP)用Ni2+螯合Sepharose柱纯化,然后用梯度尿素-TBS甘油缓冲液(10%甘油, 2 mmol/L还原型谷胱甘肽,0.2 mmol/L氧化型谷胱甘肽,50 mmol/L NaCl, 50 mmol/L Tris-HCl, pH 8.0)透析复性。用BCA法测定重组蛋白的浓度(Smith et al, 1985)。
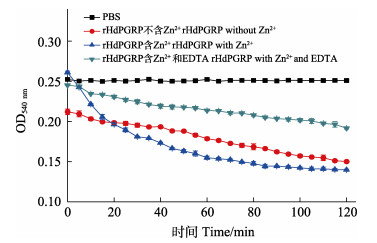
1.7 酰胺酶活性参照Mellroth等(2003)的方法测定rHdPGRP对肽聚糖的相对酶活性。将不溶于水的肽聚糖(来自金黄色葡萄球菌Staphylococcus aureus, 1 mg/mL)分别溶解于HEPES缓冲液(20 mmol/L, pH 7.2, 150 mmol/L NaCl),HEPES-ZnCl2缓冲液(20 mmol/L, pH 7.2, 150 mmol/L NaCl, 10 mmol/L ZnCl2)和HEPES-ZnCl2 EDTA缓冲液(20 mmol/L, pH 7.2, 150 mmol/L NaCl, 10 mmol/L ZnCl2, 10 mmol/L EDTA)并加入rHdPGRP (50 µg/mL),其中以PBS缓冲液作为阴性对照。在120 min内每隔15 min检测OD540 nm值。
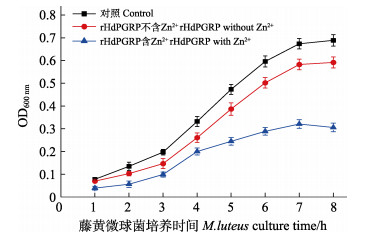
1.8 rHdPGRP的抑菌活性将藤黄微球菌(Micrococcus luteus)培养至对数期,用培养基稀释至1.0 × 107 CFU/mL。在100 μL的细菌悬液中加入rHdPGRP (终浓度为100 μg/mL),设含10 mmol/L ZnCl2和不含ZnCl2 2组。无rHdPGRP的细胞悬液作为阴性对照。220 r/min,37℃振荡培养,每隔1 h测定OD600 nm值。
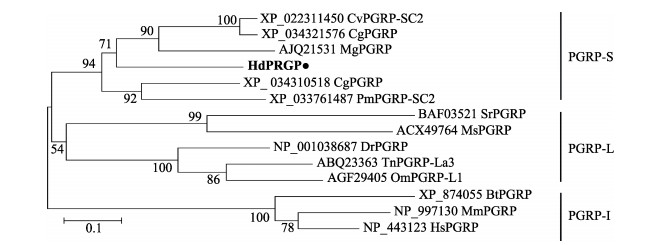
2 结果 2.1 hdpgrp的cDNA克隆及序列分析hdpgrp的cDNA全长为1467 bp,共编码354个氨基酸,在GenBank数据库中编号为MZ150581。通过ExPASy分析,HdPGRP含有1个糖基化位点(N133),用SMART软件预测,HdPGRP含有1个信号肽(1~18氨基酸)、1个SH3b结构域(93~160氨基酸)、1个PGRP结构域(179~322氨基酸)和1个Ami_2结构域(191~332氨基酸) (图 1),说明HdPGRP可能分泌到细胞外识别并结合肽聚糖。多序列比对分析结果表明,HdPGRP与太平洋牡蛎(Crassostrea gigas) (58%, XP_034310518)和光滑双脐螺(Biomphalaria glabrata) (55%, NP_001298227)的PGRPs之间具有高度相似性,且HdPGRP有4个保守的Zn2+结合位点(H209、Y255、H318和C330)以及5个保守的酰胺酶催化位点(H209、Y255、H318、T328和C330) (图 2)。采用MEGA 4.0软件的邻接法构建的系统发育树显示,PGRPs分为3个分支:短型PGRPs (PGRP-S)、中长型PGRPs (PGRP-I)和长型PGRPs (PGRP-L),HdPGRP与太平洋牡蛎等其他海洋无脊椎动物的PGRPs聚于短型PGRPs (图 3)。

|
图 1 HdPGRP核酸及推导的氨基酸序列 Fig.1 The complete nucleotide and deduced amino acid sequence of HdPGRP 加粗:起始密码子和终止密码子;方框:糖基化位点;下划线:信号肽;深灰色阴影:SH3b结构域;波浪线:PRGP结构域;浅灰色阴影:Ami_2结构域。 Bold: Initial codon and termination codon; Box: Glycosylation sites; Underline: Signal peptide; Dark grey shadow: SH3b domain; Wave line: PRGP domain; Light gray shadow: Ami_2 domain. |

|
图 2 HdPGRP与其他物种中PGRPs的多序列比对 Fig.2 Multiple sequence alignments of HdPGRP with PGRPs in other animals PGRPs来源及GenBank登录号如下:夏威夷短尾鱿(EsPGRP-2, AAY27974)、三角帆蚌(HcPGRP-S2, AHK22786)、太平洋牡蛎(CgPGRP-S1S, BAG3 1896; CgPGRP, XP_034310518)、大竹蛏(SgPGRP, AEW43446)、豹蛙(LcPGRP, PIO34231)、光滑双脐螺(BgPGRP, NP_001298227)。圆形:Zn2+结合位点;三角形:酰胺酶催化位点。 The PGRPs source and GenBank number are as follows: Euprymna scolopes (EsPGRP-2, AAY27974), Hyriopsis cumingii (HcPGRP-S2, AHK22786), Crassostrea gigas (CgPGRP-S1S, BAG3 1896; CgPGRP, XP_034310518), Solen grandis (SgPGRP, AEW43446), Lithobates catesbeianus (LcPGRP, PIO34231), Biomphalaria glabrata (BgPGRP, NP_001298227). Round: Zn2+ binding site; Triangle: Amidase catalytic site. |
|
图 3 HdPGRP的N-J进化树 Fig.3 Neighbor-joining phylogenetic tree analysis of HdPGRP 利用MEGA 4.0软件,采用邻接法构建系统发育树,重复1000次。PGRPs来源及GenBank登录号如下:美洲牡蛎(CvPGRP-SC2, XP_022311450)、太平洋牡蛎(CgPGRP-SC2, XP_034321576; CgPGRP, XP_034310518)、紫贻贝(MgPGRP-C, AJQ2153)、欧洲扇贝(PmPGRP-SC2, XP_033761487)、印度蚕(SrPGRP, BAF03521)、烟草天蛾(MsPGRP, ACX49764)、斑马鱼(DrPGRP, NP_001038687)、黑斑鲀(TnPGRP-La3, ABQ23363)、虹鳟(OmPGRP-L1, AGF29405)、牛(BtPGRP-4, XP_874055)、鼠(MmPGRP-3, NP_997130)、人(HsPGRP-3, NP_443123)。 Using MEGA 4.0 software, the phylogenetic tree was constructed by Neighbor-Joining method and repeated 1000 times. The PGRPs source and GenBank number are as follows: Crassostrea virginica (CvPGRP-SC2, XP_022311450), Crassostrea gigas (CgPGRP-SC2, XP_034321576; CgPGRP, XP_034310518), Mytilus galloprovincialis (MgPGRP-C, AJQ2153), Pecten maximus (PmPGRP-SC2, XP_033761487), Samia ricini (SrPGRP, BAF03521), Manduca sexta (MsPGRP, ACX49764), Danio rerio (DrPGRP, NP_001038687), Tetraodon nigroviridis (TnPGRP-La3, ABQ23363), Oncorhynchus mykiss (OmPGRP-L1, AGF29405), Bos taurus (BtPGRP-4, XP_874055), Mus muscu (MmPGRP-3, NP_997130), Homo sapiens (HsPGRP-3, NP_443123). |
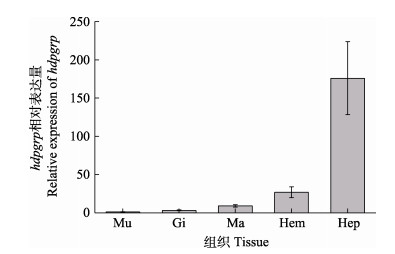
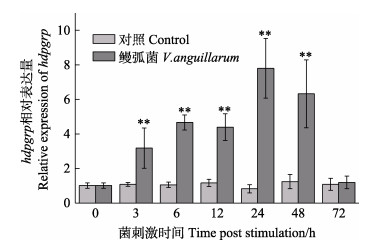
利用qRT-PCR技术对鲍鱼各组织中hdpgrp转录表达进行分析。hdpgrp在所有被检测的组织中均有表达,其中,hdpgrp主要分布于肝胰腺,其次为血细胞、外套膜和鳃,在肌肉中表达量最低(图 4)。在鳗弧菌刺激后,血细胞中hdpgrp表达量在3—48 h内显著升高(P < 0.01),并在24 h时达到最大值,是对照组的9.4倍(P < 0.01),随后,hdpgrp表达量下降,在72 h时,hdpgrp水平与对照组相比无显著差异,说明hdpgrp表达恢复至正常水平(图 5)。
|
图 4 不同组织中hdpgrp的相对表达量(平均值±标准差, n=6) Fig.4 Relative expression levels of hdpgrp in different tissues (Mean±SD, n=6) Mu:肌肉;Gi:鳃;Ma:外套膜;Hem:血细胞;Hep:肝胰腺。 Mu: Muscle; Gi: Gills; Ma: Mantle; Hem: Hemocytes; Hep: Hepatopancreas. |
|
图 5 鳗弧菌刺激后血细胞中hdpgrp的相对表达量(平均值±标准差, n=6) Fig.5 Relative expression levels of hdpgrp in hemocytes after V. anguillarum stimulation (Mean±SD, n=6) *: P < 0.05, **: P < 0.01. |
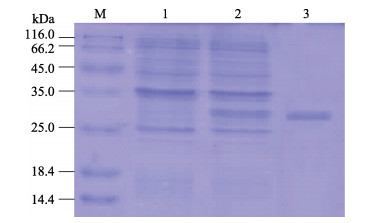
用IPTG对含HdPGRP重组质粒的表达菌株BL21(DE3)进行诱导表达,SDS-PAGE显示,位于30 kDa处出现1条与预期重组蛋白大小一致的条带。经Ni2+螯合琼脂糖柱纯化并复性后的rHdPGRP蛋白条带单一,约30 kDa (图 6)。
|
图 6 rHdPGRP的SDS-PAGE分析 Fig.6 SDS-PAGE analysis of rHdPGRP 泳道M:标准蛋白分子量标记物;泳道1:阴性对照(未诱导);泳道2:诱导大肠杆菌表达rHdPGRP;泳道3:纯化的rHdPGRP。 Lane M: Marker; Lane 1: rHdPGRP in E. coli without induction; Lane 2: Induced rHdPGRP in E. coli; Lane 3: Purified rHdPGRP. |
酰胺酶活性是PGRP水解不溶性肽聚糖的N-乙酰胞壁酸和L-丙氨酸之间肽键,从而降解肽聚糖的一种活性。结果显示,在Zn2+存在的情况下,rHdPGRP对肽聚糖显示出较高的降解活性,在120 min内OD540 nm从0.268降到0.142。与含Zn2+组相比,不含Zn2+组和Zn2+-EDTA组的rHdPGRP对肽聚糖的降解活性明显降低(图 7)。表明rHdPGRP的酰胺酶活性是Zn2+依赖型。rHdPGRP对革兰氏阳性菌藤黄微球菌的抑制实验显示,在Zn2+存在情况下,经rHdPGRP孵育后,藤黄微球菌的生长被强烈抑制(图 8),然而,在无Zn2+的情况下,rHdPGRP对其生长的抑制作用减弱(图 8)。
|
图 7 rHdPGRP的酰胺酶活性 Fig.7 Amidase activity of rHdPGRP |
|
图 8 rHdPGRP的抗菌活性(平均值±标准差, n=3) Fig.8 Antibacterial activity of rHdPGRP (Mean±SD, n=3) |
皱纹盘鲍作为一种海洋无脊椎动物,被认为缺乏复杂的适应性免疫,它们主要由先天免疫发挥机体防御,需要通过一些模式识别蛋白来识别异己,进而抵御外界病原菌的入侵。PGRP作为特异性识别肽聚糖的模式识别蛋白,在抵御以肽聚糖为细胞壁主要成分的微生物的免疫反应中发挥重要作用(Wei et al, 2018; Yang et al, 2019; 杨嘉龙, 2011)。本研究从皱纹盘鲍中鉴定了一种PGRP,并对其在组织中的分布情况、病原菌入侵时转录表达模式以及抗菌活性进行了研究。
多序列比对结果显示,HdPGRP与其他软体动物的PGRPs具有高度的相似性,PGRP结构域与噬菌体的T3溶菌酶同源,表明HdPGRP具有抗菌和凝集作用。此外,HdPGRP中还发现了SH3b结构域,在细菌蛋白中发现的SH3结构域同源物能介导不同的生理过程,如增加局部蛋白浓度,改变其亚细胞位置,并介导大型多蛋白复合物的组装(Huang et al, 2019; Morton et al, 1994)。这些结构特征表明,HdPGRP可能具有细菌识别和清除的效应功能。值得注意的是,在软体动物中发现的PGRPs均为短型,说明软体动物PGRPs介导的免疫防御机制可能与非软体动物不同。
鲍鱼属于半开放循环系统,易受到多种微生物的侵入。在本研究中,尽管hdpgrp在所测组织中均有表达,但在不同组织其表达量完全不同,表现出一定的组织特异性。与三角帆蚌的pgrps类似(Huang et al, 2019; Tao et al, 2014),hdpgrp主要在皱纹盘鲍肝胰腺中表达,而在日本盘鲍(Haliotis discus discus)中发现的另一种短型pgrp主要在血淋巴中表达(Premachandra et al, 2014)。类似地,在太平洋牡蛎中,cgpgrp-s1l和cgpgrp-s3分别在外套膜和消化盲囊中的表达量最高(Itoh et al, 2008)。在大竹蛏中,sgpgrp-s1主要在肌肉和肝胰腺中表达,而sgpgrp-s2主要在鳃和外套膜中表达(杨顶珑, 2014)。不同病原菌引发的病害不同,pgrps在不同的组织中选择性表达,表明PGRPs有可能针对不同病原菌进行特异性识别。鳗弧菌作为皱纹盘鲍的一种重要致病菌,通过产生热稳定和热不稳定的毒素使宿主发病(王瑞旋等, 2006),在鳗弧菌刺激后,血细胞中hdpgrp的表达显著升高,并在24 h时达到最高,可能是鳗弧菌细胞壁中的肽聚糖被PGRP识别,进行信号传递并触发免疫反应(Hu et al, 2020)。
PGRPs具有酰胺酶活性,通过水解连接L-丙氨酸和N-乙酰胞壁酸之间的酰胺键降解肽聚糖,防止细菌过度激活免疫系统(Zaidman-Rémy et al, 2006; Premachandra et al, 2014; 杨嘉龙, 2011)。前期研究表明,存在4个保守的Zn2+结合残基是PGRP酰胺酶活性能够被激活的前提(Hu et al, 2020),与此相一致的是,HdPGRP也鉴定出4个保守的Zn2+结合位点(H210、Y255、H318和C330)。在Zn2+存在的情况下,肽聚糖被rHdPGRP显著降解,这与栉孔扇贝(Chlamys farreri) (Yang et al, 2010)和菲律宾蛤仔(Ruditapes philippinarum)的酰胺酶活性一致(Yang et al, 2019)。研究表明,PGRPs通过酰胺酶活性降解细胞壁上的肽聚糖,细胞壁的损伤抑制细菌的生长,从而发挥一定的抗菌作用(Hu et al, 2020; Wang et al, 2003)。藤黄微球菌作为革兰氏阳性菌,其细胞壁成分主要为肽聚糖,本研究发现,rHdPGRP在Zn2+的作用下显著抑制藤黄微球菌的生长,这表明HdPGRP对藤黄微球菌的抑菌活性是通过酰胺酶活性实现的。
4 结论本研究在皱纹盘鲍中鉴定了一种新的模式识别蛋白(HdPGRP),对其在免疫防御中的作用进行了初步研究。HdPGRP在进化及结构上较为保守,具有保守的Zn2+结合位点和酰胺酶催化位点。hdpgrp在血细胞及各组织中均有表达,血细胞中的hdpgrp能够响应鳗弧菌刺激显著上调,表明其可能参与皱纹盘鲍抗细菌免疫防御。此外,证实了HdPGRP具有酰胺酶活性,对微生物病原菌具有一定的抗菌活性。这些结果为了解HdPGRP的功能特性及其在皱纹盘鲍免疫防御机制中的作用提供了基础依据。
CAO D D, LIU J X, WANG Z G, et al. Expression analysis and functional characterization of NOD2 in the resistance of Japanese flounder (Paralichthys olivaceus) to Edwardsiella tarda infection. Progress in Fishery Sciences, 2018, 39(3): 53-64 [曹丹丹, 刘金相, 王志刚, 等. 牙鲆NOD2基因的表达分析及在抗迟缓爱德华氏菌感染过程中的功能. 渔业科学进展, 2018, 39(3): 53-64] |
COTEUR G, MELLROTH P, LEFORTERY C D, et al. Peptidoglycan recognition proteins with amidase activity in early deuterostomes (Echinodermata). Developmental and Comparative Immunology, 2007, 31(8): 790-804 DOI:10.1016/j.dci.2006.11.006 |
DZIARSKI R. Peptidoglycan recognition proteins (PGRPs). Molecular Immunology, 2004, 40(12): 877-886 DOI:10.1016/j.molimm.2003.10.011 |
GERDOL M, VENIER P. An updated molecular basis for mussel immunity. Fish and Shellfish Immunology, 2015, 46(1): 17-38 DOI:10.1016/j.fsi.2015.02.013 |
HE T T. Comparative study on immunological responses of Pacific abalone and its hybrids with green abalone under Vibrio harveyi stress. Masterxs Thesis of Xiamen University, 2018 [何婷婷. 哈维氏弧菌胁迫下不同规格皱纹盘鲍和绿盘鲍免疫应答研究. 厦门大学硕士研究生学位论文, 2018]
|
HU Z G, CAO X B, GUO M, et al. Identification and characterization of a novel short-type peptidoglycan recognition protein in Apostichopus japonicus. Fish and Shellfish Immunology, 2020, 99: 257-266 DOI:10.1016/j.fsi.2020.02.013 |
HUANG Y, PAN J L, LI X G, et al. Molecular cloning and functional characterization of a short peptidoglycan recognition protein from triangle-shell pearl mussel (Hyriopsis cumingii). Fish and Shellfish Immunology, 2019, 86: 571-580 DOI:10.1016/j.fsi.2018.12.007 |
ITOH N, TAKAHASHI K G. Distribution of multiple peptidoglycan recognition proteins in the tissues of Pacific oyster, Crassostrea gigas. Comparative Biochemistry and Physiology, Part B: Biochemistry and Molecular Biology, 2008, 150(4): 409-417 DOI:10.1016/j.cbpb.2008.04.011 |
JIANG X Y, YU G L, SONG X K, et al. Survey and evaluation of the Qianliyan national aquatic germplasam reserve. Progress in Fishery Sciences, 2021, 42(5): 1-7 [姜向阳, 于广磊, 宋秀凯, 等. 千里岩海域国家级水产种质资源保护区现状调查与评价. 渔业科学进展, 2021, 42(5): 1-7] |
KASHYAP D R, ROMPCA A, GABALLA A, et al. Peptidoglycan recognition proteins kill bacteria by inducing oxidative, thiol, and metal stress. PLOS Pathogens, 2014, 10(7): e1004280 DOI:10.1371/journal.ppat.1004280 |
LI X, YUAN S, SUN Z S, et al. Gene identification and functional analysis of peptidoglycan recognition protein from the spotted sea bass (Lateolabrax maculatus). Fish and Shellfish Immunology, 2020, 106: 1014-1024 DOI:10.1016/j.fsi.2020.08.041 |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method. Methods, 2001, 25(4): 402-408 DOI:10.1006/meth.2001.1262 |
MELLROTH P, KARLSSON J, STEINER H. A scavenger function for a Drosophila peptidoglycan recognition protein. Journal of Biological Chemistry, 2003, 278(9): 7059-7064 DOI:10.1074/jbc.M208900200 |
MELLROTH P, STEINER H. PGRP-SB1: An N-acetylmuramoyl L-alanine amidase with antibacterial activity. Biochemical and Biophysical Research Communications, 2006, 350(4): 994-999 DOI:10.1016/j.bbrc.2006.09.139 |
MORTON C J, CAMPBELL I D. SH3 Domains: Molecular 'Velcro'. Current Biology, 1994, 4(7): 615-617 DOI:10.1016/S0960-9822(00)00134-2 |
PREMACHANDRA H K A, ELVITIGALA D A S, WHANG I, et al. Identification of a novel molluscan short-type peptidoglycan recognition protein in disk abalone (Haliotis discus discus) involved in host antibacterial defense. Fish and Shellfish Immunology, 2014, 39(1): 99-107 DOI:10.1016/j.fsi.2014.04.018 |
ROUT A K, PARAMANIK S, DEHURY B, et al. Elucidating the molecular interaction of zebrafish (Danio rerio) peptidoglycan recognition protein 2 with diaminopimelic acid and lysine type peptidoglycans using in silico approaches. Journal of Biomolecular Structure and Dynamics, 2020, 38(12): 3687-3699 DOI:10.1080/07391102.2019.1666742 |
SMITH P K, KROHN R I, HERMANSON G T, et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry, 1985, 150(1): 76-85 DOI:10.1016/0003-2697(85)90442-7 |
TAO Y, YANG Z Y, ZHANG X, et al. Molecular cloning and mRNA expression of the peptidoglycan recognition protein gene HcPGRP1 and its isoform HcPGRP1a from the freshwater mussel Hyriopsis cumingi. Genetics and Molecular Biology, 2014, 37(3): 508-517 DOI:10.1590/S1415-47572014000400006 |
WANG R X, XU L W, FENG J, et al. Advances in studies on diseases caused by microorganisms of abalone. Transactions of Oceanology and Limnology, 2006, 90(2): 117-124 [王瑞旋, 徐力文, 冯娟, 等. 鲍类微生物性疾病研究进展. 海洋湖沼通报, 2006, 90(2): 117-124 DOI:10.3969/j.issn.1003-6482.2006.02.019] |
WANG X W, WANG J X. Pattern recognition receptors acting in innate immune system of shrimp against pathogen infections. Fish and Shellfish Immunology, 2013, 34(4): 981-989 DOI:10.1016/j.fsi.2012.08.008 |
WANG Z M, LI X N, COCKLIN R R, et al. Human peptidoglycan recognition protein-L is an N-acetylmuramoyl- L-alanine amidase. Journal of Biological Chemistry, 2003, 278(49): 49044-49052 DOI:10.1074/jbc.M307758200 |
WEI X M, YANG D L, LI H Y, et al. Peptidoglycan recognition protein of Solen grandis (SgPGRP-S1) mediates immune recognition and bacteria clearance. Fish and Shellfish Immunology, 2018, 73: 30-36 DOI:10.1016/j.fsi.2017.12.001 |
WU F C, QUE H Y, ZHANG G F. History, current status, and future development of the Pacific abalone seed release and sea ranching in China. Marine Sciences, 2020, 44(8): 56-68 [吴富村, 阙华勇, 张国范. 我国皱纹盘鲍底播增养殖历史、现状及未来发展趋势. 海洋科学, 2020, 44(8): 56-68] |
YANG D L, HAN Y J, LIU Y L, et al. A peptidoglycan recognition protein involved in immune recognition and immune defenses in Ruditapes philippinarum. Fish and Shellfish Immunology, 2019, 88: 441-448 |
YANG D L. Study on the immune defense functions of peptidoglycan recognition protein in razor clam (Solen grandis). Masterxs Thesis of Shanghai Ocean University, 2014 [杨顶珑. 大竹蛏肽聚糖识别蛋白的免疫防御功能研究. 上海海洋大学硕士研究生学位论文, 2014]
|
YANG J L, WANG W, WEI X M, et al. Peptidoglycan recognition protein of Chlamys farreri (CfPGRP-S1) mediates immune defenses against bacterial infection. Developmental and Comparative Immunology, 2010, 34(12): 1300-1307 |
YANG J L. Study on the mechanism of immune response mediated by key pattern recognition receptors in Zhikong scallop Chlamys farreri. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2011 [杨嘉龙. 栉孔扇贝关键模式识别受体介导的免疫应答机制研究. 中国科学院研究生院(海洋研究所)博士研究生学位论文, 2011]
|
ZAIDMAN-RÉMY A, HERVÉ M, POIDEVIN M, et al. The Drosophila amidase PGRP-LB modulates the immune response to bacterial infection. Immunity, 2006, 24(4): 463-473 |



