2. 中国水产科学研究院黄海水产研究所 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Pilot National Laboratory for Marine Science and Technology (Qingdao), Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao, Shandong 266071, China
细胞毒性淋巴细胞(CLs)可以识别感染病毒或转化的细胞,并随后诱导其凋亡,是免疫系统的重要组成部分(Naneh et al, 2014)。CLs包括细胞毒性T淋巴细胞(CTL)和自然杀伤(NK)细胞,CTL和NK细胞在其自身分泌的细胞毒性颗粒的帮助下,介导病原体或病毒感染的细胞以及肿瘤细胞的死亡(Bolitho et al, 2007)。细胞毒性颗粒的主要内容物包括穿孔素以及颗粒酶等可溶性的细胞毒蛋白(Sutton et al, 2016)。颗粒酶(granzyme, Gzm)是以His-Asp-Ser三联体催化位点为特征的丝氨酸蛋白酶,具有底物特异性(Wernersson et al, 2006)。到目前为止,在人类(Homo sapiens)中发现有5种Gzms (Gzm A、B、H、K和M),在小鼠(Mus musculus)中发现有11种Gzms (Gzm A~G、K~N) (Bots et al, 2006)。鱼类Gzms与哺乳动物相应Gzms的相似性较低,其Gzms的鉴定与命名存在争议,例如,首次在斑点叉尾
GzmB是目前研究最深入、含量最丰富的丝氨酸蛋白酶之一(Turner et al, 2019a、b),它是免疫炎症反应必不可少的介质,可诱导靶细胞的凋亡(Trapani, 2001; Matsuura et al, 2014)。活化的CTL识别靶细胞后,从细胞毒颗粒中释放成熟的GzmB,然后,与穿孔素通过内吞作用进入细胞质基质中(Darmon et al, 1996),可激活caspase-3,进而诱导靶细胞凋亡。除了caspase依赖途径,GzmB也参与caspase非依赖途径的靶细胞凋亡(Boivin et al, 2009)。人类GzmB长度约3500 bp,由5个外显子和4个内含子组成,其ORF编码247个氨基酸,包含2个6链β折叠和3个反式结构域片段(Klein et al, 1989; Estébanez-Perpiñá et al, 2000)。目前,关于GzmB功能的研究在人类和小鼠中已有不少报道(Turner et al, 2019b; Smyth et al, 1995; Larimer et al, 2017)。然而,在鱼类中,关于GzmB的研究仅在鲤(Cyprinus carpio)、银鲫(Carassius auratus langsdorfii)、罗非鱼(Oreochromis niloticus)、大西洋鲑(Atlantic salmon)、斑点叉尾
半滑舌鳎(Cynoglossus semilaevis)是我国重要的海水养殖经济鱼类之一,主要分布在我国沿海地区,其肉质鲜美、营养丰富,深受广大消费者喜爱,具有良好的市场需求和养殖开发潜力。然而,高密度集约化养殖以及环境污染等导致半滑舌鳎多种疾病频繁暴发。其中,最严重的是由哈维氏弧菌(Vibrio harveyi)引起的细菌性疾病,严重制约了半滑舌鳎养殖业的健康发展(陈政强等, 2012; 于孟君等, 2017; 王双艳等, 2019)。近年来,以提高鱼体抗病力为目标的良种选育,被认为是当下解决半滑舌鳎病害的一种行之有效的方法(Zhou et al, 2019)。长期以来,鱼类免疫机制及抗病分子机制的基础研究比较薄弱,严重阻碍了鱼类抗病育种的发展(陈松林, 2004; 周欣等, 2021)。因此,对半滑舌鳎抗病分子机制的研究迫在眉睫,这将为其病害防治提供重要的理论依据。目前,关于GzmB基因在半滑舌鳎中的研究尚未见报道。因此,本研究对半滑舌鳎GzmB基因(CsGzmBl)的全长cDNA序列进行克隆,采用实时荧光定量PCR技术(qRT-PCR)检测了该基因在健康半滑舌鳎不同组织中的表达模式,以及哈维氏弧菌感染后不同组织的表达水平,旨在为进一步研究该基因在半滑舌鳎免疫应答中的作用机制奠定基础。
1 材料与方法 1.1 样品采集 1.1.1 正常组织样品采集本实验所用半滑舌鳎均购自山东省海阳市黄海水产公司,在22℃循环海水中养殖。选取5尾健康、体重为(104.9±4.6) g的半滑舌鳎个体,分别收集鳃、头肾、肝脏、肠、皮肤、脑、中肾、肌肉和脾脏共9个组织,立即置于液氮中冷冻,然后转入–80℃保存。
1.1.2 哈维氏弧菌感染与样品采集采用Wei等(2018)的方法,对10月龄健康半滑舌鳎进行哈维氏弧菌感染实验。首先定量培养哈维氏弧菌(Wang et al, 2017),然后用无菌PBS将哈维氏弧菌稀释至终浓度为1.0×104 CFU/mL,用于半滑舌鳎腹腔注射实验,对照组注射等体积的无菌PBS。分别在感染前0 h与感染后12、24、48、72及96 h共6个时间点取样,每个时间点分别取3尾半滑舌鳎,取其肝脏、脾脏、肾脏,肠、鳃和皮肤共6个组织,放入液氮中速冻,随后转入–80℃保存。
1.2 RNA提取与cDNA合成使用RNA提取试剂盒(Invitrogen, 美国)提取各组织的总RNA,用1%的琼脂糖凝胶电泳检测RNA的完整性,用分光光度计检测RNA的浓度和纯度。RNA质量鉴定合格后,使用Prime Script RT reagent kit with gDNA eraser试剂盒(TaKaRa, 日本)合成cDNA。使用SmartTM RACE cDNA amplification kit (TaKaRa, 日本)合成RACE-Ready cDNA。
1.3 CsGzmBl基因全长cDNA的克隆根据半滑舌鳎全基因组测序结果(Chen et al, 2014),获得CsGzmBl基因的预测序列(登录号:XM_008328902.3),以该序列片段设计引物(表 1),先进行普通PCR扩增,验证其ORF区。普通PCR扩增体系:2×ExTaq Mix(TaKaRa, 日本) 25 µL,CsPRF1l-ORF-F/R引物各1 µL,cDNA模板2 µL,ddH2O补至50 µL。PCR扩增条件:94℃ 4 min;35个循环(94℃ 30 s,58℃ 30 s,72℃ 1 min 45 s);72℃ 10 min。将PCR产物进行琼脂糖凝胶电泳检测,并使用DNA胶回收试剂盒(天根,中国)进行纯化回收。将纯化的片段连接到pEasy-T1载体(全式金,中国),通过42℃热激法转入到Trans-T1感受态细胞中(全式金,中国),最后挑取单克隆,送华大基因进行测序,成功获得CsGzmBl的ORF区序列。
|
|
表 1 本研究所用的引物 Tab.1 Primers used in this study |
以得到的ORF区序列设计RACE引物(表 1),以合成的RACE-Ready-cDNA为模板,通过降落PCR进行5′-RACE和3′-RACE PCR扩增。第1轮反应体系为10 μL,按照TaqTM hot start (TaKaRa, 日本)说明书加样。PCR程序:94℃ 30 s;72℃ 3 min,5个循环;94℃ 30 s,70℃ 30 s,72℃ 5 min,5个循环;94℃ 30 s,70℃ 30 s,72℃ 3 min,25个循环;72℃延伸10 min。将第1轮的PCR反应产物稀释50倍后作为第2轮普通PCR的模板,普通PCR体系和程序均同上所述。扩增完成后,进行分子克隆实验,并挑取单克隆送测序。成功获得5′和3′端序列,然后,通过DNAstar软件进行拼接,最终获得CsGzmBl的cDNA全长序列。
1.4 序列分析利用在线程序ORFinder (https://www.ncbi.nlm.nih.gov/orffinder/)推导CsGzmBl基因的氨基酸序列。使用BLAST程序(http://www.ncbi.nlm.nih.gov/BLAST/)对不同物种GzmB的氨基酸序列进行同源性搜索。使用ExPASy在线软件的ProtParam程序(http://au.expasy.org/tools/protparam.html)计算CsGzmB的相对分子质量和理论等电点等理化参数。基于SMART 4.0程序(http://smart.embl-heidelberg.de/)预测该蛋白的信号肽序列及功能结构域。使用在线程序SoftBerry-Psite (http://linux1.softberry.com/berry.phtml?topic=psite&group=programs&subgroup=proloc)预测该蛋白的功能位点。使用Predict Protein (https://www.predictprotein.org/)预测该蛋白的二级结构。使用在线软件GSDS 2.0 (http://gsds.gao-lab.org/index.php)比较不同物种GzmB的基因组结构。使用BioEdit软件进行脊椎动物GzmB的氨基酸序列比对。使用MEGA 7.0软件通过NJ法–邻位相连法构建GzmB的系统进化树。
1.5 检测CsGzmBl基因的表达模式应用实时荧光定量PCR(qRT-PCR)检测CsGzmBl基因在半滑舌鳎健康成鱼不同组织以及哈维氏弧菌刺激后免疫相关组织中的表达水平。选用β-actin作为内参基因,用于qRT-PCR的引物均列在表 1中。使用SYBR® Premix Ex TaqTM (TaKaRa, 日本)试剂盒,按照说明书在ABI 7500 Fast Real-time (Applied Biosystems, 美国)仪器上进行CsGzmBl基因的定量分析。每个样品设置3个平行,使用2–ΔΔCt方法(Livak et al, 2001)计算目的基因的相对表达量。所有实验数据以平均值±标准误(Mean±SE)表示,使用SPSS 16.0软件进行单因素方差分析(one-way ANOVA)和Duncan多重比较。当P < 0.05时为差异显著(P < 0.05);当P < 0.01时为差异极显著(P < 0.01)。
2 结果 2.1 CsGzmBl基因的cDNA序列分析CsGzmBl基因的cDNA全长为923 bp,其开放阅读框(ORF)为780 bp,5′非编码区(UTR)为49 bp,3′UTR为94 bp (在polyA尾部上游5 bp处存在一个多聚腺苷酸加尾信号(AATAAA))。ORF编码一个由259个氨基酸残基组成的蛋白,分子量为29.06 kDa,理论等电点为9.23,总平均疏水系数为–0.353,脂肪系数为77.53,不稳定指数(Ⅱ)为42.36,表明该蛋白不稳定。信号肽预测显示,该蛋白的N端存在一个由19个氨基酸残基组成的信号肽。功能位点预测发现,该蛋白包含2个N端糖基化位点(分别为NSSH和NNSK)以及5个二硫键。亚细胞定位预测发现,CsGzmBl属于胞外分泌蛋白(图 1)。

|
图 1 CsGzmBl基因的核苷酸与氨基酸序列 Fig.1 Nucleotide and deduced amino acid sequence of CsGzmBl 非编码区用小写字母表示,编码区用大写字母表示。2个预测的N端糖基化位点(NSSH和NNSK)用灰色阴影标注。起始密码子和终止密码子用红色加粗字母表示。聚腺苷酸化信号和多聚腺昔氨酸加尾信号分别用方框和下划线标出。 UTR is in lowercase, and ORF is in uppercase. Two predicted N-glycosylation sites (NSSH and NNSK) are shaded in greys. The start codon and stop codon are red in bold. The polyadenylation signal and polyadenylation tail are boxed and underlined, respectively. |
对不同物种的GzmB氨基酸序列进行比对发现,在脊椎动物GzmB中比较保守的一些功能域及位点也同样存在于CsGzmBl中。CsGzmBl具有一个典型的丝氨酸蛋白酶家族所特有的“催化三联体His63Asp112Ser207”,一个保守的蛋白活性中心,一个保守的前导肽(ED),一个保守的“PHSRPYMA”序列以及6个保守的半胱氨酸(Cys)(图 2)。保守的Cys分别为Cys 48、Cys 64、Cys 146、Cys 177、Cys 192和Cys 213,其中,Cys 48与Cys 64,Cys 146与Cys 213,Cys 177与Cys 192分别各自形成了二硫键。此外,CsGzmBl的Cys 5与Cys 13,Cys 203与Cys 229也分别各自形成了二硫键。CsGzmBl 5个二硫键位置如图 2所示。

|
图 2 CsGzmBl基因与其他脊椎动物GzmB基因的多氨基酸序列比对 Fig.2 Multiple alignment of the GzmB amino acid sequences among vertebrates 相同氨基酸和相似氨基酸分别用黑色和浅灰色阴影表示。星号表示6个保守的半胱氨酸的位置,三角形表示保守的催化三联体,黑色长方形表示保守的蛋白活性中心。不同物种信号肽的位置用白色框标注。CsGzmBl蛋白的二硫键由双箭头表示,其前导肽由双线表示。 Identical and similar amino acids are shaded in black and light gray, respectively. Asterisks indicate 6 conserved cysteines, black triangles indicate conserved catalytic triads, and black rectangles indicate conserved protein active centers. The positions of signal peptides of different species are marked with white boxes. The disulfide bond of the CsGzmBl protein is represented by a double arrow, and its activation di-peptide peptide is represented by a double line. |
BLAST同源性搜索发现,CsGzmBl与其他硬骨鱼类的GzmB氨基酸序列的一致性在41.00%~63.35%之间,如大菱鲆(Scophthalmus maximus)(63.35%)、黄鳝(Monopterus albus)(61.66%)、尖吻鲈(Lates calcarifer)(61.39%)、孔雀鱼(Poecilia reticulata) (60.41%)、小丑鱼(Amphiprion ocellaris) (59.92%)、金头鲷(Sparus aurata)(59.85%)、剑尾鱼(Xiphophorus maculatus)(59.06%)、弹涂鱼(Periophthalmus magnuspinnatus)(57.54%)、荫平

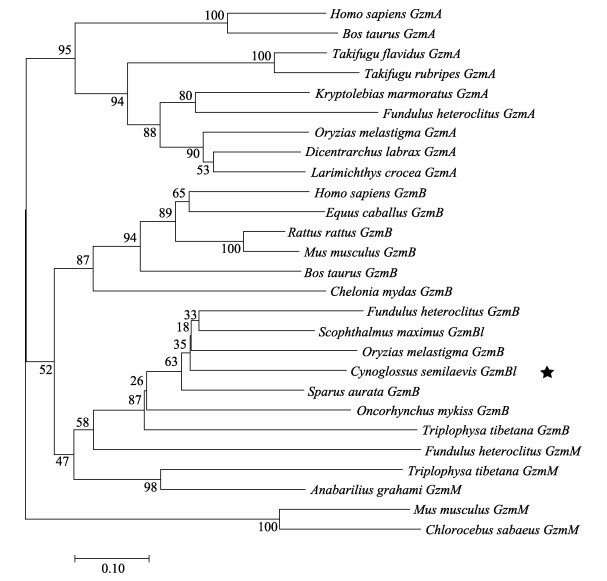
为更明确CsGzmBl的进化地位,选用CsGzmBl与其他物种的GzmB、GzmA及GzmM的氨基酸序列,构建系统发育树。如图 3所示,所有物种的GzmA聚为一大支;CsGzmBl与其他物种的GzmB聚为另一大支,其中,部分物种的GzmM与GzmB聚在一起。在GzmB这一支中,发现有2个主要的不同分支,其中,所有硬骨鱼类的GzmB聚为一支,其他脊椎动物的GzmB聚为一支,这2大支最终汇聚在一起。因此,脊椎动物Gzm氨基酸的同源性符合进化规则。
|
图 3 GzmB、GzmA以及GzmM氨基酸序列的系统发育树 Fig.3 Phylogenetic tree based on the amino acid sequences of GzmB, GzmA and GzmM from various species CsGzmBl用黑色五角星表示。 CsGzmBl was marked with a black five-pointed star. |
本研究使用的所有氨基酸序列登录号为:人GzmA (NP_006135.2),牛GzmA (Bos taurus, XP_019838691.1),鲈鱼GzmA (Dicentrarchus labrax, AIK66548.1),红树林鱂鱼GzmA (Kryptolebias marmoratus, XP_017267744.1),青鳉GzmA (XP_024131649.1),底鳉GzmA(Fundulus heteroclitus, XP_036000696.1),大黄鱼GzmA (TMS12193.1),菊黄东方鲀GzmA (Takifugu flavidus, TWW60312.1),红鳍东方鲀GzmA (Takifugu rubripes, XP_029685142.1),小鼠GzmM (XP_031205327.1),绿猴GzmM (Chlorocebus sabaeus, XP_037852248.1),西藏高原鳅GzmM (Triplophysa tibetana, KAA0712223.1),底鳉GzmM (Fundulus heteroclitus, JAR24158.1),

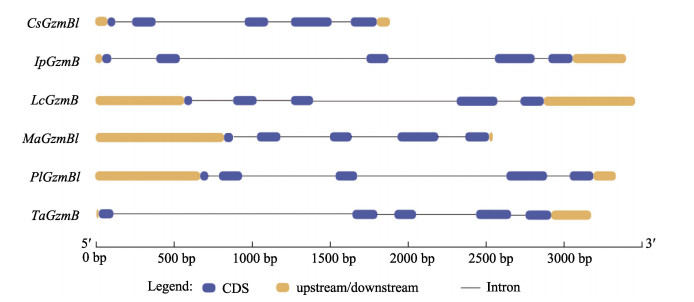
以CsGzmB基因的全长cDNA序列与半滑舌鳎全基因组序列进行比对,获得CsGzmBl的基因组DNA序列,将之与CsGzmBl的ORF序列进行比较分析,得到CsGzmBl的基因结构。CsGzmBl基因由1890个碱基组成,包括5个外显子与4个内含子。外显子–内含子边界均符合AG/GT拼接规则,所有的3′末端受体位点具有共同的AG序列,所有的5′末端供体位点部位都有共同的GT序列。将半滑舌鳎与其他物种的GzmB基因组结构进行比较,发现GzmB的基因组结构比较保守,均是由5个外显子和4个内含子组成(图 4)。
|
图 4 CsGzmBl和其他物种GzmB基因的外显子–内含子结构比较
Fig.4 Comparison of the exon/intron structure of between the CsGzmBl and GzmB genes among vertebrates
外显子用蓝色的圆角矩形标注,5′和3′非编码区均用橙色的圆角矩形表示,黑色的线代表内含子。不同物种GzmB基因的mRNA序列和基因组序列登录号:CsGzmBl:XM_008328902.3,NC_024322.1;斑点叉尾 :XM_017478999.1,NC_030425.1;大黄鱼:XM_019268918.2,NC_040028.1;黄鳝:XM_020594827.1,NW_018127904.1;玛丽鱼:XM_015052332.1,NW_015113718.1;南美石头鱼:XM_034180280.1,NC_047112.1
Exons are in blue rounded rectangles; 5′UTR and 3′UTR are in orange rounded rectangles. Lines indicate the introns. The mRNA and gene accession numbers of different species GzmBs are as follows: CsGzmBl: XM_008328902.3, NC_024322.1; IpGzmB (Ictalurus punctatus GzmB): XM_017478999.1, NC_030425.1; LcGzmB (Larimichthys crocea GzmB): XM_019268918.2, NC_040028.1; MaGzmBl (Monopterus albus GzmBl): XM_020594827.1, NW_018127904.1; PlGzmBl (Poecilia latipinna GzmBl): XM_015052332.1, NW_015113718.1; TaGzmB (Thalassophryne amazonica GzmB): XM_034180280.1, NC_047112.1 :XM_017478999.1,NC_030425.1;大黄鱼:XM_019268918.2,NC_040028.1;黄鳝:XM_020594827.1,NW_018127904.1;玛丽鱼:XM_015052332.1,NW_015113718.1;南美石头鱼:XM_034180280.1,NC_047112.1
Exons are in blue rounded rectangles; 5′UTR and 3′UTR are in orange rounded rectangles. Lines indicate the introns. The mRNA and gene accession numbers of different species GzmBs are as follows: CsGzmBl: XM_008328902.3, NC_024322.1; IpGzmB (Ictalurus punctatus GzmB): XM_017478999.1, NC_030425.1; LcGzmB (Larimichthys crocea GzmB): XM_019268918.2, NC_040028.1; MaGzmBl (Monopterus albus GzmBl): XM_020594827.1, NW_018127904.1; PlGzmBl (Poecilia latipinna GzmBl): XM_015052332.1, NW_015113718.1; TaGzmB (Thalassophryne amazonica GzmB): XM_034180280.1, NC_047112.1
|
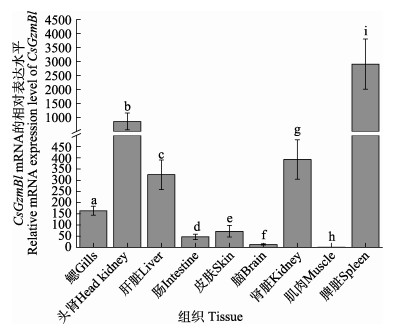
半滑舌鳎健康成鱼鳃、头肾、肝脏、肠、皮肤、脑、中肾、肌肉、脾脏共9个组织中CsGzmBl的表达情况如图 5所示。从图 5可以看出,CsGzmBl在这些组织中呈现出不同的表达情况,其中,在脾脏中表达量最高,在头肾、中肾、肝脏和鳃中表达量次之,在其他组织中表达量较低,在肌肉中表达量最低。
|
图 5 CsGzmBl mRNA在健康半滑舌鳎成鱼不同组织中的表达水平 Fig.5 Expression of CsGzmBl mRNA in different tissues of healthy adult C. semilaevis 数据均以平均值±标准误表示(n=3)。柱状图上方的不同字母表示各个组织之间存在显著性差异(P < 0.05)。 All data are shown as Mean±SE (n=3). Different letters above the bars indicate significant difference at the level of P < 0.05. |
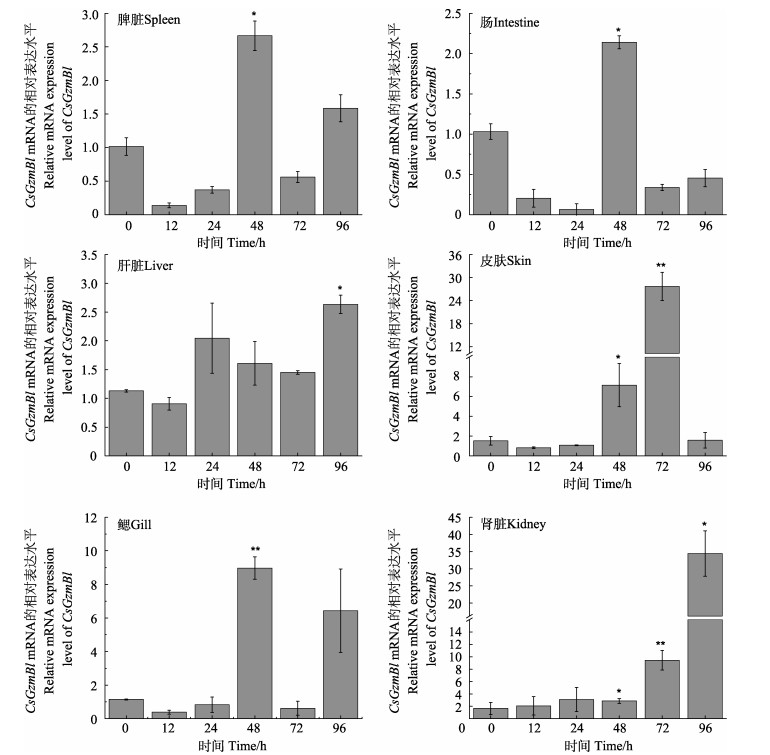
哈维氏弧菌刺激后,与对照组0 h相比,CsGzmBl mRNA的表达水平在脾脏、肠、肝脏、皮肤、鳃和肾脏中均有不同程度的上调。如图 6所示,在脾脏、肠和鳃中,CsGzmBl的表达水平仅在细菌感染48 h后有明显的上调趋势(P < 0.05或P < 0.01),在其他时间点无明显变化。在皮肤中,CsGzmBl mRNA的表达量在细菌感染72 h后显著增加(P < 0.05),在其余时间点与对照组相比无显著差异(P > 0.05)。在肝脏和肾脏中,细菌感染后96 h,CsGzmBl的表达水平显著上升(P < 0.05),其余时间点与对照组相比也无显著差异(P > 0.05)。
|
图 6 哈氏弧菌感染后脾脏、肠、肝脏、皮肤、鳃和肾脏中CsGzmBl mRNA的表达情况 Fig.6 Expression pattern of CsGzmBl mRNA in the liver, spleen, kidney, intestine, gills and skin after V. harveyi infection 样品取自感染后0、12、24、48、72及96 h。数值以平均值±标准误表示(n=3)。星号表示显著性差异(** P < 0.01,* P < 0.05)。 The samples are tested at 0, 12, 24, 48, 72 and 96 h after infection. All the data are shown as Mean±SE (n=3). The asterisk indicates a significant difference (** P < 0.01, * P < 0.05). |
本研究通过RACE方法获得了CsGzmBl基因的cDNA全长,并对其序列进行分析,发现CsGzmBl基因具有一些保守的功能域或功能位点。CsGzmBl具有一个序列为“His63Asp112Ser207”的催化三联体,催化三联体是肤凝乳蛋白酶的典型特征,是形成可实现精确三维定向的支架所必需的部分(Branden et al, 1991),在Gzm蛋白家族成员中高度保守(Smyth et al, 1996),如大西洋鳕GzmA/K、尼罗罗非鱼Gzm及银鲫Gzm中均具有催化三联体“His57Asp102Ser195”(Matsuura et al, 2014; Praveen et al, 2006; Wernersson et al, 2006)。此外,CsGzmBl蛋白还具有一个保守的前导肽ED(GluAsp),在携带翻译信息的酶转移到粗面内质网和胞外颗粒过程中起不可或缺的作用(Praveen et al, 2004)。大多数哺乳动物,如马、人、大鼠及小鼠的GzmB蛋白的N末端都具有一个高度保守的序列“GlyGlu(EG)或Glu-Glu(GG)”的前导肽(Caputo et al, 1993)。研究表明,Gzm氨基酸激活的前肽后面1~4位(IIGG)与9~16位(PHSRPYMA)高度保守(Trapani, 2001; Piuko et al, 2007; Pham et al, 1999)。哺乳动物,如人、大鼠,小鼠及马Gzm蛋白中均具有这2个保守结构域,其中,IIGG结构域是前肽裂解激活蛋白酶所必需的(Smyth et al, 1995)。本研究中,CsGzmBl前导肽后1~4位为“IVNG”,类似于哺乳动物Gzm蛋白的“IIGG”,第10~16位是保守的“PHSRPYMA”结构域。
本研究发现,从哺乳类到爬行动物,各个物种GzmB均具有6个保守的Cys,参与形成3个二硫键。Gzm蛋白家族成员具有多个高度保守的Cys,这对Gzm蛋白加工过程中的正确折叠和激活至关重要(Praveen et al, 2006)。据报道,斑点叉尾
Huang等(2010)报道称,鱼类Gzms与哺乳动物的GzmA/B/K关系更近,可能是因为GzmA/B是更古老的祖先基因。因此,推测GzmB在进化上处于更高地位,而GzmM可能由GzmB进化而来。本研究选择A和M作为进化分析的组成部分,而最终的进化分析发现,鱼类的GzmM与GzmB聚在一起,证实了此推测。本研究还发现,所有物种的GzmB始终聚在一起,符合GzmB的同源进化规则。基因结构比较发现,CsGzmBl与其他鱼类GzmB基因均由5个外显子与4个内含子组成。据报道,Gzm家族所有成员的基因结构极其保守,如鲤GzmA/K、斑马鱼(Danio rerio)Gzm、尼罗罗非鱼Gzm及人GzmA与GzmK基因均由5个外显子和4个内含子组成(Huang et al, 2010; Praveen et al, 2006)。本研究中,CsGzmBl的基因结构符合外显子和内含子法则(Mount, 1982),但与其他鱼类GzmB基因相比,CsGzmBl基因的长度明显较短。以上结果表明,CsGzmBl既具备Gzm家族的典型特征,也具有GzmB的特点,进化上与Gzms家族其他成员有明确区别,因此,该基因属于硬骨鱼类GzmB同源基因。
关于鱼类Gzms基因组织表达模式的研究十分有限,仅在几种鱼中有报道。鲤中GzmA/K基因在脾脏、鳃及头肾中高表达(肾脏中最高),其次是肾脏和肠,在肝脏和皮肤及肌肉中表达量最低(Huang et al, 2010)。尼罗罗非鱼的Gzm仅在肾脏和血液中被检测到,在肝脏、脾脏、鳃及肌肉中均未发现表达(Praveen et al, 2006)。在银鲫中,Gzm mRNA在鳃和脾脏中表达量最高,在肝脏中表达量最低(Matsuura et al, 2014)。虽然鱼类Gzm基因呈现出不同的组织表达模式,但它总能在免疫相关组织中被检测到。本研究发现,CsGzmBl mRNA在半滑舌鳎健康成鱼9个组织中呈现不同的表达水平,其中,在脾脏中表达量最高,头肾、中肾、肝脏和鳃中表达量次之,肌肉中表达量最低。综合表明,CsGzmBl基因可能在半滑舌鳎先天性免疫中具有重要作用。
许多研究表明,Gzms基因在病毒和细菌感染过程中发挥重要作用,相关研究在哺乳动物中有大量报道。然而,关于鱼类Gzms基因在细菌感染后的表达研究十分有限。在鲤中发现GzmA/K可以被SVCV病毒所诱导(Huang et al, 2010)。鲫鱼的Gzm基因可以被迟缓爱德华氏菌(Edwardsiella tarda)所调节。本研究发现,哈维氏弧菌感染后,CsGzmBl基因在肝脏、肾脏、脾脏、肠、鳃及皮肤6个组织中均显著上调,说明CsGzmBl参与半滑舌鳎的免疫防御作用,但其具体的功能和作用机制尚不清楚,还需进一步研究。
综上所述,本研究通过对CsGzmBl基因的克隆、序列特征描述以及表达模型分析,初步表明GzmBl参与半滑舌鳎免疫应答过程,为进一步研究GzmBl在半滑舌鳎抗病免疫中的作用机制奠定了基础。
BOIVIN W A, COOPER D M, HIEBERT P R, et al. Intracellular versus extracellular granzyme B in immunity and disease: Challenging the dogma. Laboratory Investigation, 2009, 89(11): 1195-1220 DOI:10.1038/labinvest.2009.91 |
BOLITHO P, VOSKOBOINIK I, TRAPANI J A, et al. Apoptosis induced by the lymphocyte effector molecule perforin. Current Opinion in Immunology, 2007, 19(3): 339-347 DOI:10.1016/j.coi.2007.04.007 |
BOTS M, MEDEMA J P. Granzymes at a glance. Journal of Cell Science, 2006, 119(24): 5011-5014 DOI:10.1242/jcs.03239 |
BRANDEN C, TOOZE J. Introduction to protein structure. New York: Garland Publishing, 1991
|
CAPUTO A, GARNER R S, WINKLER U, et al. Activation of recombinant murine cytotoxic cell proteinase-1 requires deletion of an amino-terminal dipeptide. Journal of Biological Chemistry, 1993, 268(24): 17672-17675 DOI:10.1016/S0021-9258(17)46755-X |
CHAVES-POZO E, VALERO Y, LOZANO M T, et al. Fish granzyme A shows a greater role than granzyme B in fish innate cell-mediated cytotoxicity. Frontiers in Immunology, 2019, 10: 2579 DOI:10.3389/fimmu.2019.02579 |
CHEN S L, ZHANG G J, SHAO C W, et al. Whole-genome sequence of a flatfish provides insights into ZW sex chromosome evolution and adaptation to a benthic lifestyle. Nature Genetics, 2014, 46(46): 253-260 |
CHEN S L. Progress in molecular breeding for disease-resistant marine fishes and its prospects. Science and Technology Review, 2004(9): 10-13 [陈松林. 海水养殖鱼类抗病分子育种研究进展及前景展望. 科技导报, 2004(9): 10-13 DOI:10.3321/j.issn:1000-7857.2004.09.004] |
CHEN Z Q, YAO Z X, LIN M. Study on pathogen of skin ulcer disease of half-smooth tongue sole (Cynoglossus semilaevis). Journal of Fisheries of China, 2012, 36(5): 764-771 [陈政强, 姚志贤, 林茂, 等. 半滑舌鳎皮肤溃疡病病原研究. 水产学报, 2012, 36(5): 764-771] |
DARMON A J, LEY T J, NICHOLSON D W, et al. Cleavage of CPP32 by granzyme B represents a critical role for granzyme B in the induction of target cell DNA fragmentation. Journal of Biological Chemistry, 1996, 271(36): 21709-21712 DOI:10.1074/jbc.271.36.21709 |
ESTÉBANEZ-PERPIÑÁ E, FUENTES-PRIOR P, BELORGEY D, et al. Crystal structure of the caspase activator human granzyme B, a proteinase highly specific for an Asp-P1 residue. Biological Chemistry, 2000, 381(12): 1203-1214 |
HUANG R, ZHONG S, LIU H, et al. Identification and characterization of common carp (Cyprinus carpio L.) granzyme A/K, a cytotoxic cell granule-associated serine protease. Fish and Shellfish Immunology, 2010, 29(3): 388-398 DOI:10.1016/j.fsi.2010.04.002 |
KLEIN J L, SHOWS T B, DUPONT B, et al. Genomic organization and chromosomal assignment for a serine protease gene (CSPB) expressed by human cytotoxic lymphocytes. Genomics, 1989, 5(1): 110-117 DOI:10.1016/0888-7543(89)90093-1 |
LARIMER B M, WEHRENBERG-KLEE E, DUBOIS F, et al. Granzyme B PET imaging as a predictive biomarker of immunotherapy response. Cancer Research, 2017, 77(9): 2318-2327 DOI:10.1158/0008-5472.CAN-16-3346 |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods, 2001, 25: 402-408 DOI:10.1006/meth.2001.1262 |
MATSUURA Y, YABU T, SHIBA H, et al. Identification of a novel fish granzyme involved in cell-mediated immunity. Developmental and Comparative Immunology, 2014, 46(2): 499-507 DOI:10.1016/j.dci.2014.06.006 |
MOUNT S M. A catalogue of splice junction sequences. Nucleic Acids Research, 1982, 10(2): 459-472 DOI:10.1093/nar/10.2.459 |
NANEH O, AVČIN T, ZAVEC A B. Perforin and human diseases. Sub-Cellular Biochemistry, 2014, 80: 221-239 |
PHAM C T N, LEY T J. Dipeptidyl peptidase I is required for the processing and activation of granzymes A and B in vivo. Proceedings of the National Academy of Sciences, 1999, 96(15): 8627-8632 DOI:10.1073/pnas.96.15.8627 |
PIUKO K, BRAVO I G, MÜLLER M. Identification and characterization of equine granzyme B. Veterinary Immunology and Immunopathology, 2007, 118(3/4): 239-251 |
PRAVEEN K, EVANS D L, JASO-FRIEDMANN L. Evidence for the existence of granzyme-like serine proteases in teleost cytotoxic cells. Journal of Molecular Evolution, 2004, 58(4): 449-459 DOI:10.1007/s00239-003-2566-7 |
PRAVEEN K, LEARY J H, EVANS D L, et al. Molecular characterization and expression of a granzyme of an ectothermic vertebrate with chymase-like activity expressed in the cytotoxic cells of Nile tilapia (Oreochromis niloticus). Immunogenetics, 2006, 58(1): 41-55 DOI:10.1007/s00251-005-0063-4 |
SMYTH M J, MCGUIRE M J, THIA K Y T. Expression of recombinant human granzyme B. A processing and activation role for dipeptidyl peptidase I, 1995, 154(12): 6299-6305 |
SMYTH M J, O'CONNOR M D, TRAPANI J A. Granzymes: A variety of serine protease specificities encoded by genetically distinct subfamilies. Journal of Leukocyte Biology, 1996, 60(5): 555-562 DOI:10.1002/jlb.60.5.555 |
SUTTON V R, BRENNAN A J, ELLIS S, et al. Serglycin determines secretory granule repertoire and regulates natural killer cell and cytotoxic T lymphocyte cytotoxicity. FEBS Journal, 2016, 283(5): 947-961 DOI:10.1111/febs.13649 |
TRAPANI J A. Granzymes: A family of lymphocyte granule serine proteases. Genome Biology, 2001, 2(12): 1-7 |
TURNER C T, HIROYASU S, GRANVILLE D J. Granzyme B as a therapeutic target for wound healing. Expert Opinion on Therapeutic Targets, 2019b, 23(9): 745-754 DOI:10.1080/14728222.2019.1661380 |
TURNER C T, LIM D, GRANVILLE D J. Granzyme B in skin inflammation and disease. Matrix Biology, 2019a, 75/76: 126-140 DOI:10.1016/j.matbio.2017.12.005 |
WANG L, FAN C, XU W, et al. Characterization and functional analysis of a novel C1q-domain-containing protein in Japanese flounder (Paralichthys olivaceus). Developmental and Comparative Immunology, 2017, 67: 322-332 DOI:10.1016/j.dci.2016.09.001 |
WANG S Y, WANG L, CHEN Z F, et al. Cloning and expression analysis of the polymeric immunoglobulin receptor (pIgR) in half smooth tongue sole (Cynoglossus semilaevis). Progress in Fishery Sciences, 2019, 40(2): 51-57 [王双艳, 王磊, 陈张帆, 等. 半滑舌鳎多聚免疫球蛋白受体(pIgR)基因的克隆和表达分析. 渔业科学进展, 2019, 40(2): 51-57] |
WEI M, XU W T, LI K M, et al. Cloning, characterization and functional analysis of dctn5 in immune response of Chinese tongue sole (Cynoglossus semilaevis). Fish and Shellfish Immunology, 2018, 77: 392-401 DOI:10.1016/j.fsi.2018.04.007 |
WERNERSSON S, REIMER J, POORAFSHAR M, et al. Granzyme-like sequences in bony fish shed light on the emergence of hematopoietic serine proteases during vertebrate evolution. Developmental and Comparative Immunology, 2006, 30(10): 901-918 DOI:10.1016/j.dci.2005.10.014 |
YU M J, SUN L M, CHEN Y D, et al. Cloning and expression of the c-Jun gene in immune response to pathogen challenge in half-smooth tongue sole (Cynoglossus semilaevis). Progress in Fishery Sciences, 2017, 38(2): 31-39 [于孟君, 孙璐明, 陈亚东, 等. 半滑舌鳎(Cynoglossus semilaevis) c-Jun基因的克隆及免疫应答分析. 渔业科学进展, 2017, 38(2): 31-39] |
ZHOU Q, SU Z, LI Y, et al. Genome-wide association mapping and gene expression analyses reveal genetic mechanisms of disease resistance variations in Cynoglossus semilaevis. Frontiers in Genetics, 2019, 10: 1167 DOI:10.3389/fgene.2019.01167 |
ZHOU X, GAO F Y, LU M X. The progress of fish disease resistance breeding. Journal of Dalian Ocean University, 2021, 36(3): 510-523 [周欣, 高风英, 卢迈新. 鱼类抗病性育种研究进展. 大连海洋大学学报, 2021, 36(3): 510-523] |



