2019年我国的鲍养殖产量高达18.03万t,占世界鲍养殖量的90%之上(农业农村部渔业渔政管理局等, 2020)。虽然我国的鲍养殖产量不断提高,养殖规模不断扩大,但转口期稚鲍的高死亡率是困扰鲍养殖业的瓶颈之一,制约了鲍产业的健康可持续发展。
在水产动物中,转口期指动物早期幼体阶段由摄食活体生物饵料逐渐转变为完全摄食人工配合饲料的过程,是水产动物早期发育的重要阶段(郭忠娣, 2016; Zhao et al, 2020)。鲍的转口期特指稚鲍生长到一定规格后(4~5 mm左右),其消化器官逐渐发育成熟,由摄食底栖硅藻转变为摄食人工配合饲料的时期(Fleming et al, 1996; Johnston et al, 2005; Nel et al, 2018)。饵料的转变往往影响水产动物的生理和生化功能(黄仲园等, 2020)。Zhao等(2020)研究发现,大口黑鲈(Micropterus salmoides)的仔稚鱼在转口期消化能力降低,肝脂代谢能力在转口后显著提高。Hamza等(2007)研究发现,梭鲈(Sander lucioperca)在转口之后碱性磷酸酶活性显著升高,影响了仔鱼的消化能力。饵料的转变除影响水产动物的生理状态外,直接引起肠道微生物的演替。肠道微生物作为动物肠道的重要组成部分,对宿主的食物消化吸收、代谢及免疫等有着重要影响(陈贞年等, 2021; Wu et al, 2012; Nicholson et al, 2012; Sampson et al, 2016)。水产动物的转口影响了肠道微生物的群落结构和多样性,进而影响水产动物的生长发育,如大口黑鲈(Zhao et al, 2020)、牙鲆(Paralichthys olivaceus) (刘增新等, 2017)和草鱼(Ctenopharyngodon idellus) (祝东梅, 2008)。对此,深入研究水产动物转口期的肠道微生物演替变化,对于更好地理解水产动物的生理变化及改善转口饲料配方均具有重要指导意义。
目前,有关鲍转口期的肠道微生物研究相对较少。Tanaka等(2003)研究发现,转口后皱纹盘鲍(Haliotis discus hannai)的肠道优势菌群被具有降解海藻酸盐能力的菌群演替。Nel等(2018)研究发现,使用不同的转口饵料投喂南非鲍(H. midae),稚鲍的肠道菌群结构和多样性具有显著差异。为从微生态层面了解皱纹盘鲍饵料转换后的适应机制,并尝试初步探索肠道微生物与稚鲍转口期高死亡率的关系,本研究通过16S rDNA高通量测序和生物信息学方法,分析皱纹盘鲍在转口期不同阶段肠道菌群结构演替规律和多样性,为皱纹盘鲍健康养殖提供理论基础。
1 材料与方法 1.1 实验材料本研究所用皱纹盘鲍鲍苗由山东省威海长青海洋科技股份有限公司(122.57°N, 37.16°E)提供。育苗生产于2020年3月20日进行,所有稚鲍均由本次育苗所得,来自同一批苗种。育苗40 d后,育苗板上的稚鲍生长到一定规格(约4 mm)后进行剥离。稚鲍的剥离标志着转口期的开始,剥离后的稚鲍放入流水池中进行养殖,养殖水温为(18.0±0.5)℃,养殖密度控制在5000 ind./m2左右。本研究选用金牌鲍鱼配合饲料502BY [主要成分为鱼粉、豆粕、海带(Laminaria japonica)粉和裙带菜(Undaria pinnatifida)粉等,粗蛋白含量≥30%]作为稚鲍的转口饲料,投喂量为稚鲍总体重的5%,每日傍晚投喂,次日早晨清理残饵、粪便和死亡的鲍鱼。
1.2 样品采集所有实验稚鲍均取自同一养殖池,且健康无病害。转口前,稚鲍以摄食底栖硅藻为主,食物来源稳定且死亡率低,在稚鲍剥离前2 d (T0)采集8个样本。经人工剥离后,稚鲍由摄食底栖硅藻转变为摄食人工配合饲料,死亡率上升至60%左右。根据稚鲍的剥离时间,分别在剥离后第4天(T4)、第10天(T10)、第35天(T35)和第40天(T40)进行样本采集,每次采集8个样本。用无菌海水冲洗鲍贝壳和腹足上的黏液,解剖取鲍肠道组织,放入1.5 mL无菌离心管内,–80℃超低温冰箱内保存待用。
1.3 DNA的提取、PCR扩增和高通量测序采用CTAB法提取鲍肠道微生物的总DNA,1%琼脂糖凝胶电泳检测DNA的纯度和浓度。通过PCR扩增16S rDNA的V3~V4可变区域,扩增引物分别为341F (5′-CCTAYGGGRBGCASCAG-3′)和806R (5′-GGACTACNNGGGTATCTAAT-3′)。PCR扩增体系为30 µL,包括Phusion® High-Fidelity PCR Master Mix (New England Biolabs) 15 µL,正、反向引物各2 µmol/L,模板DNA 10 ng。PCR扩增条件:98℃预变性1 min;98℃变性10 s,50℃退火30 s,72℃延伸30 s,30个循环;72℃延伸5 min。使用2%琼脂糖凝胶电泳检测PCR产物后,使用胶回收试剂盒(Qiagen, 德国)回收产物。
使用TruSeq® DNA PCR-Free建库试剂盒(Illumina, 美国)进行文库构建,使用Qubit® 2.0 Fluorometer (Thermo Scientific)对构建好的文库进行质量评估,并使用Illumina NovaSeq6000进行上机测序。去除原始数据的Barcode和引物序列,并使用FLASH (V1.2.7) (Magoč et al, 2011)进行拼接;之后,进一步使用Qiime (V1.9.1) (Caporaso et al, 2010)对拼接后的原始数据精准除杂;最后,通过UCHIME软件与SILVA132数据库进行嵌合体序列的对比检验并去除,得到优质数据用于后续分析(Edgar et al, 2011; Haas et al, 2011)。
1.4 数据处理和分析利用Uparse软件(V7.0.1001)对各个样本的有效数据进行聚类,以97%的一致性将序列聚类为操作分类单元(operational taxonomic units, OTUs),筛选出OTUs中出现频数最高的序列作为代表序列(Edgar et al, 2013)。使用Mothur算法与SILVA132数据库对OTUs序列进行物种注释分析(Quast et al, 2013)。使用MUSCLE软件(V 3.8.31)进行多序列比对分析,获得OTUs代表序列的系统发生关系(Edgar et al, 2004)。最后,对数据进行均一化处理。
利用QIIME软件计算Alpha多样性指数(Chao1指数、ACE指数、Shannon-Wiener指数、Simpson指数和测序深度)。使用R软件(V 2.15.3)进行数据统计分析及Beta多样性分析,使用ade4和ggplot2软件包绘制主成分分析图(PCA)。通过Tax4Fun软件包进行样本微生物的功能预测。使用LEfSe软件进行LEfSe分析,LDA Score的筛选值设置为4.0。根据物种丰度,计算相对丰度前100的各菌属之间的皮尔森相关系数,筛选相关系数 > 0.6或 < –0.6,且显著(P < 0.05)的菌属,以菌属为节点,相关性系数为边,使用Gephi软件(V 0.9.2)绘制共发生网络图。
2 结果 2.1 测序结果初步分析皱纹盘鲍转口期不同阶段的肠道菌群测序数据经过拼接、除杂和质控后,通过剔除较少数据量的样本,T0、T4、T10、T35和T40的最终有效样本数量分别为8、7、5、8和8。测序共获得2 220 598条高质量有效序列,经97%的一致性聚类分析得到3609个OTU。将转口期稚鲍的各组样品之间的共有和特有OTU数量通过Venn图展示(图 1)。5组样品的共有菌群为792种,其中,T4组的特有菌群种类最多,共419种,T0组次之,共359种,而T10、T35和T40的特有菌群种类依次减少。表明随着转口期的推进,稚鲍肠道微生物的特有菌群数量变小,且远小于转口前及转口后第4天。

|
图 1 皱纹盘鲍肠道菌群OTU Venn图 Fig.1 OTU Venn diagram of the intestinal microflora of H. discus hannai |
皱纹盘鲍转口期各样品组的Alpha多样性指数见表 1。由表 1可知,各组的覆盖率均不低于99.50%,表明测序深度合理且已经基本覆盖样品中所有的物种。Chao1和ACE指数自转口开始后即呈下降趋势,其中,T0和T4组的Chao1和ACE指数都显著高于T35和T40组(P < 0.05),说明稚鲍肠道微生物群落的丰富度在转口前和转口后第4天高于转口后第35天和第40天。Shannon-Wiener和Simpson指数在稚鲍转口期过程呈先上升后下降的趋势,其中,T10组的2个指数数值最高,T35的数值最低,且显著低于前3组样品(P < 0.05),说明T35组的物种丰富度和均匀度最低,即T35组的物种多样性最低。T40组的数据较T35组略有回升但不显著(P > 0.05),Shannon- Wiener指数仍显著低于T10组(P < 0.05),Simpson指数仍显著低于T4和T10组(P < 0.05)。综合比较4种Alpha多样性指数,皱纹盘鲍转口后第35天和第40天的肠道菌群多样性低于转口前和转口后第4天。
|
|
表 1 Alpha多样性指数 Tab.1 Statistics of alpha diversity index |
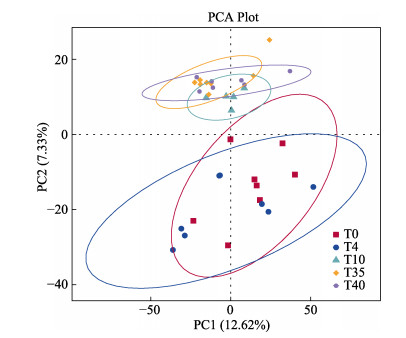
通过Beta多样性分析,对转口期各组间的稚鲍肠道微生物群落结构多样性进行差异比较。本研究采用基于OTU水平的主成分分析(PCA),将原本测序得到的所有微生物物种丰度数据重新进行线性组合,得到主成分PC1和PC2,它们的贡献率分别是12.62%和7.33%。由图 2可见,T0与T4的样品组间距离较近,T35与T40的样品组间距离较近,而T10处于T0和T4集群与T35和T40集群相对中间的位置,即T10属于转口期菌群演替变化的过渡阶段。本研究在每一时期采集8个样本,但由于高通量测序所获得的部分样本的有效数据较少,影响数据分析效果,将数据量较少的样本剔除,导致有效样本数量并不都是8个。
|
图 2 皱纹盘鲍肠道菌群的主成分分析(PCA) Fig.2 Principal component analysis (PCA) of the intestinal microflora of H. discus hannai |
通过与数据库进行比对汇总,共有3562条(98.70%) OTU能够被注释。在所有的有效样本中共检测出34个门类,分类至47个纲、106个目、212个科和516个属。
在门水平上,各组样品中群落结构组成相似、相对丰度均 > 1%的菌门包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes),相对丰度占比达到88.25%。从皱纹盘鲍转口期的各个采样时间点来看,尽管变形菌门的相对丰度随着转口的进行而有所降低,但始终是绝对优势菌门。拟杆菌门在转口前及转口初期的占比较小,但在转口后第35天和第40天出现剧增。厚壁菌门的相对丰度变化较小,在转口后的第35天为最小值。
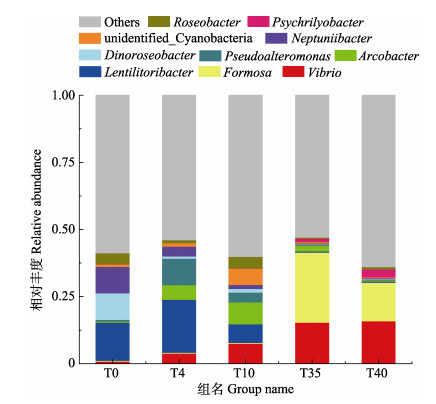
在属水平上(图 3),各组样品的菌群组成和相对丰度差异显著。在转口前,稚鲍肠道中相对丰度 > 1%的菌属共有9个,其中相对丰度前三的菌属分别为Lentilitoribacter (14.18%)、Dinoroseobacter (9.90%)和Neptuniibacter (9.86%)。在转口后第4天,相对丰度 > 1%的菌属共有11个,Lentilitoribacter (19.70%)依旧为绝对优势菌属且相对丰度升高,而第2和第3优势菌属则被假交替单胞菌属(Pseudoalteromonas) (9.86%)和弓形杆菌属(Arcobacter) (5.52%)取代。转口后第10天,相对丰度 > 1%的菌属在5组中最多,共有15个,Lentilitoribacter的相对丰度下降至6.95%,弓形杆菌属(8.15%)和弧菌属(Vibrio) (7.50%)则上升为第1和第2优势菌属。然而,转口后第35天和第40天,相对丰度 > 1%的菌属相较于前3组有明显减少,分别为5个和6个,Formosa和弧菌属均为绝对优势菌属。
|
图 3 基于属水平的皱纹盘鲍肠道菌群相对丰度 Fig.3 Relative abundance of microflora communities of H. discus hannai at genus level |
除以上各组的优势菌属,物种相对丰度分析结果还发现,未鉴别蓝细菌(Unidentified Cyanobacteria)主要集中在T0、T4和T10,其中T10组的相对丰度达到最高值。海洋玫瑰菌属(Roseobacter)的相对丰度呈先减少后增加再减少的趋势,在T10相对丰度最高(4.27%),在T35和T40均≤1%。Psychrilyobacter仅在T35和T40的相对丰度 > 1%。综上所述,皱纹盘鲍的肠道优势菌群在整个转口期过程中发生了明显的演替变化。
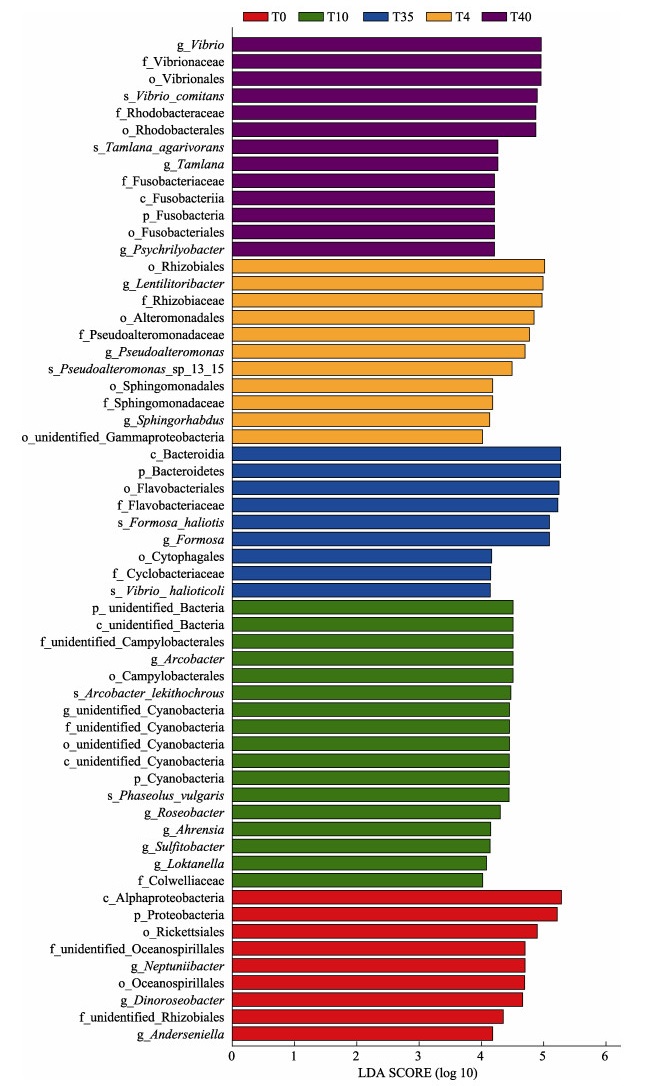
2.4 LEfSe分析利用LEfSe分析皱纹盘鲍转口期不同阶段存在统计学差异的生物标志物,本研究共获得59个生物标志物,T0到T40的生物标志物数目分别为9、11、17、9和13 (图 4)。从门水平上看,T0和T4组的生物标志物主要为变形菌门,T10组主要为蓝细菌门(Cyanobacteria)和unidentified bacteria,T35主要为拟杆菌门,T40主要为梭杆菌门(Fusobacteria)和变形菌门。从属水平上看,T0组主要为Dinoroseobacter、Neptuniibacter和Anderseniella;T4组主要为Lentilitoribacter、假交替单胞菌属和Sphingorhabdus;T10主要为弓形菌属、海洋玫瑰菌属、阿赫兰氏菌属(Ahrensia)、Sulfitobacter和洛克氏菌(Loktanella);T35主要富集在弧菌属和Formosa;T40主要为弧菌属、Tamlana和Psychrilyobacter。LEfSe结果显示,皱纹盘鲍的肠道优势菌群随着转口期的进行而发生显著改变,弧菌属是转口后第35天和第40天的主要生物标志物。
|
图 4 LEfSe分析的LDA分布直方图 Fig.4 LDA histogram of LEfSe analysis |
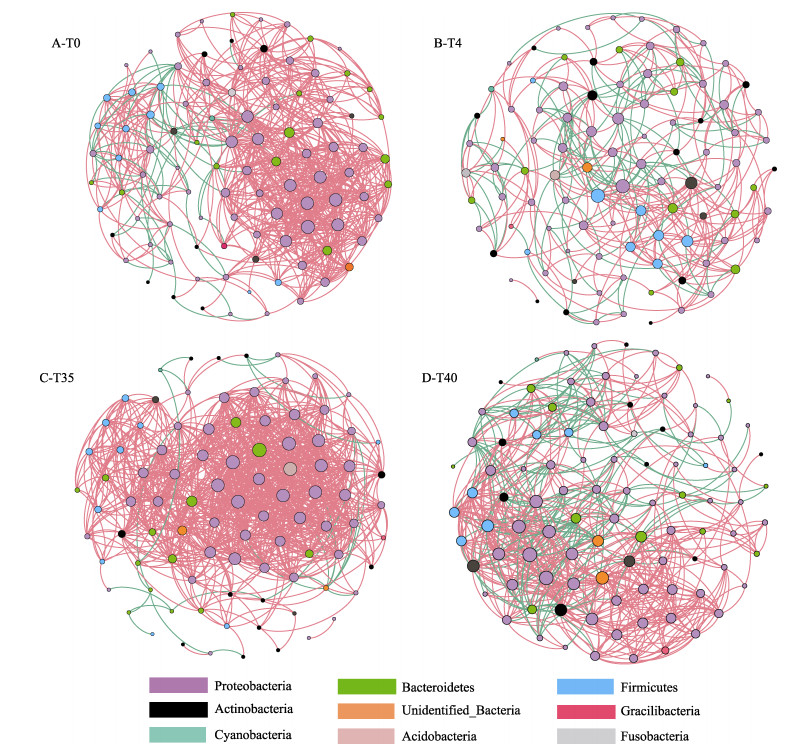
本研究共构建了T0、T4、T35和T40的4个共发生网络(图 5),以探索转口期不同阶段皱纹盘鲍肠道微生物之间的潜在相互作用,并计算网络拓扑特征以描述微生物属间复杂的关联模式(表 2)。各种网络拓扑特征差异表明,4组样本的微生物共现模式存在差异。T35的边、平均度和聚类系数最大,表明该转口期阶段的肠道微生物网络节点之间的交互最多,网络关系最复杂。T0、T4和T40的模块性均 > 0.4,说明这3个网络形成模块化结构,相较于T35的网络更加稳定。
|
图 5 转口期皱纹盘鲍肠道微生物的共发生网络关系图 Fig.5 Co-network of the intestinal microflora of H. discus hannai in weaning period |
|
|
表 2 网络拓扑特征值 Tab.2 Key topological features of microbial networks |
在网络图中,节点代表各个菌属,节点的大小表示菌属的相对丰度大小;边的颜色(红色为正相关,绿色为负相关)则用来表明菌属之间的相关关系。其中,T4网络的负相关关系占比最高(33.80%),T35网络的负相关关系占比最低(2.40%)。皱纹盘鲍的肠道菌群在整个转口期过程中以变形菌门、拟杆菌门和厚壁菌门的数量最多,为主要优势菌门,它们之间的关系变化规律为:在T0和T4阶段,大多数变形菌门与拟杆菌门存在正相关关系,与大多数厚壁菌门为负相关关系,而到了T35和T40阶段,三者之间则多为正相关关系。皱纹盘鲍的肠道菌群之间的关系随着转口的进行发生明显的改变,这些肠道菌群之间存在着复杂的合作与竞争关系,这将对稚鲍的生长发育、消化代谢及免疫等功能产生影响。
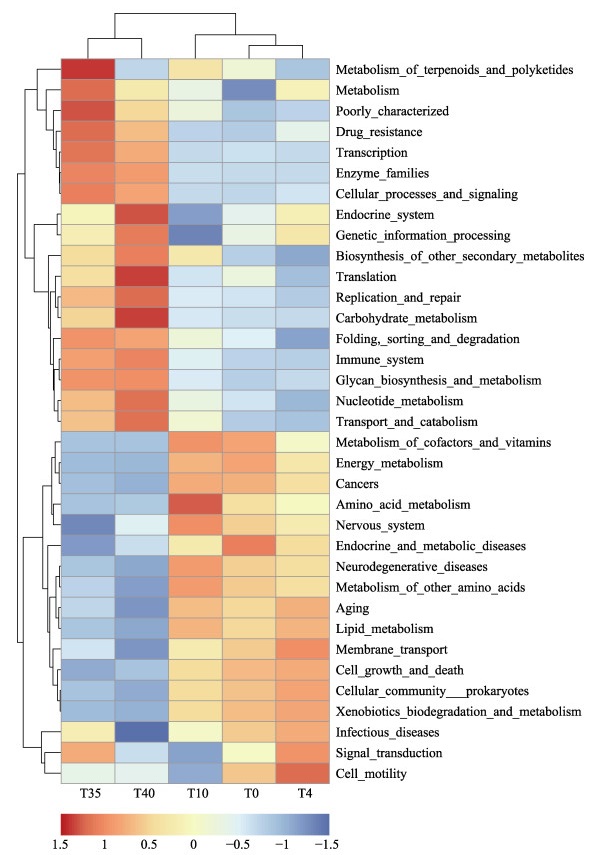
2.6 Tax4Fun功能预测本研究利用Tax4Fun对所有样本的稚鲍肠道微生物进行功能预测分析。在一级功能层面上,各组皱纹盘鲍稚鲍肠道微生物的主要功能包括代谢(metabolism)、遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、细胞加工(cellular processes)、unclassified、人类疾病(human diseases)和生物系统(organismal systems)等。在二级功能层面上,对丰度排名前35的功能基因信息进行注释和聚类,结果见图 6。T0组以内分泌代谢疾病(endocrine and metabolic diseases)、辅助因子和维生素代谢(metabolism of cofactors and vitamins)和肿瘤(cancers)为主要功能;T4组中,细胞运动性(cell motility)占绝对优势,信号传导(signal transduction)和膜运输(membrane transport)等次之;T10组中,氨基酸代谢(amino acid metabolism)占绝对优势,其次为神经系统(nervous system)、辅助因子和维生素代谢、其他氨基酸代谢(metabolism of other amino acids)等;T35组中,萜类和多酮类的代谢(metabolism of terpenoids and polyketides)占绝对优势,代谢(metabolism)、缺乏特征(poorly characterized)和酶家族(enzyme families)等次之;T40组以翻译(translation)、内分泌系统(endocrine system)和碳水化合物代谢(carbohydrate metabolism)等为主要功能。另外,通过观察聚类分支可发现,T0、T4和T10的功能较为接近,聚为一支,而T35和T40聚为另一支,与前3组的功能差异较大,说明随着转口期的进行,皱纹盘鲍肠道菌群数量和结构发生显著改变,影响了功能基因的表达。
|
图 6 第2水平上的Tax4Fun功能注释聚类热图 Fig.6 Cluster heatmap of annotated function by Tax4Fun in second level |
肠道微生物被称为动物的“第二基因组”,在宿主的生长发育过程中具有重要作用(Gao et al, 2019)。能够影响肠道微生物结构组成的因素很多,例如,饵料种类、环境条件、遗传基因和宿主的生理状态等(Nayak, 2010; Wang et al, 2020)。在这些因素中,饵料种类对肠道微生物的影响极为显著(Wang et al, 2018)。鲍在转口期发生了明显的饵料转变,即由摄食底栖硅藻转变为摄食人工配合饲料,对其肠道微生物产生巨大影响。本研究利用高通量测序技术,分析了转口期过程中,皱纹盘鲍肠道微生物结构组成和多样性的变化情况。PCA结果显示,转口前和转口后第4天的菌群结构与转口后第35天和第40天明显不同,表明转口期过程改变了鲍苗肠道菌群的组成。本研究所用的鲍人工配合饲料主要成分为鱼粉、豆粕以及海带粉等,与底栖硅藻的主要成分差别很大,促成了鲍肠道微生物群落的演替,表现为细菌的类别和相对丰度的改变。
通过生物信息学分析,发现变形菌门、拟杆菌门和厚壁菌门在各组样品中的相对丰度都 > 1%,说明它们是鲍转口期的核心菌门。变形菌门的含量在各组样品中均为最高,但在转口后第35天和第40天的占比有所下降。拟杆菌门能降解植物多糖,促进核糖、脂质和蛋白质之间的相互转换(Tran et al, 2018; Michaud et al, 2009)。配合饲料营养丰富,且添加了一定的海藻成分,促使拟杆菌门大量增加,从一定程度上反映出鲍在转口后第35天起对配合饲料具有较强的消化能力。厚壁菌门与降解复杂的多糖分子有关(Gao et al, 2019),在鲍的转口期过程中未发生明显的变化。
从属水平上看,转口前后的优势菌属差异较大,且各组的优势菌属多为该阶段的生物标志物。在转口前,Lentilitoribacter、Dinoroseobacter和Neptuniibacter为主要的鲍肠道优势菌属。随着配合饲料的摄入,Dinoroseobacter和Neptuniibacter的相对丰度迅速下降。Lentilitoribacter在转口前和转口后第4天的相对丰度较高,但在转口后的第10天开始下降,到了转口后第35天和第40天,其相对丰度仅不足1%。Lentilitoribacter和Neptuniibacter都是好氧型海洋细菌(Park et al, 2013; Arahal et al, 2007)。其中,Lentilitoribacter表现出过氧化氢酶和氧化酶活性,推测该菌属功能与叶杆菌属功能相近,可分解糖和有机酸盐,这能够促进转口期前皱纹盘鲍对微藻类食物的消化。Dinoroseobacter为好氧自养型细菌,常与藻类共生(Biebl et al, 2005; Wagner-Döbler et al, 2010)。Dinoroseobacter可能随着底栖硅藻而进入稚鲍肠道,所以在转口前的含量高,但在转口开始后,由于稚鲍不再摄食硅藻,该菌的丰度也逐渐下降。在转口后的第4天和第10天,发现相对丰度较高的假交替单胞菌属和弓形杆菌属。假交替单胞菌属能生产多种消化酶,如淀粉酶、脂肪酶和蛋白酶,能够协助宿主消化吸收(Tzuc et al, 2014)。此外,Zhao等(2012)认为,假交替单胞菌属还参与了宿主的免疫调节,协助免疫系统尚未成熟的宿主抵抗外来病原体。弓形杆菌属是一种常见的水体病原体,能够引起动物的腹泻和菌血症(许小红, 2008)。本研究中,弓形杆菌出现在稚鲍死亡率较为集中的转口期,可能对稚鲍的生长具有一定的负面作用。转口后第35天起,弧菌属和Formosa的相对丰度大幅增加,并远超其他菌属。弧菌属在许多海洋动物的肠道内均有发现,既有益生菌也有致病菌。其中,有益弧菌具有降解海藻酸盐的功能,如鲍肠道弧菌(V. halioticoli)和牡蛎弧菌(V.crassostreae)等(Gobet et al, 2018),而致病弧菌,如副溶血弧菌(V. parahaemolyticus)、河流弧菌(V. fluvialis)、哈维氏弧菌(V. harveyi)和溶藻弧菌(V. alginolyticus)等,会导致鲍和其他水产动物的相关疾病(Shi et al, 2017; 吴欢欢等, 2019)。因此,弧菌属的大量增加,需要及时关注。通过与数据库匹配,发现本研究中的弧菌属主要为鲍肠道弧菌、巨型弧菌(V. gigantis)和V. comitans,均为非致病性弧菌,可推测弧菌属的大量增加与配合饲料中的褐藻成分有关,并起着协助稚鲍消化的作用。Formosa具有降解褐藻成分的功能,曾经在大鲍(H. gigantean)肠道内发现(Tanaka et al, 2015)。另外,本研究还在鲍苗转口后的第40天发现了能分泌多种降解酶的Tamlana (刘邦涛, 2020)。通过以上关于转口期稚鲍的肠道微生物结构和组成分析结果,说明肠道优势菌群是随着稚鲍的摄食习惯改变而转变的,这些优势菌群多为益生菌。但是,实验结果发现,相对丰度 > 1%的优势菌属数量在转口后第35天和第40天显著减少,表明转口期过程可能降低了稚鲍的肠道微生物多样性。另外,本研究仅初步探讨了饵料种类的转变对转口期皱纹盘鲍的肠道菌群结构的影响,但在其他水产动物[中华绒螯蟹(Eriocheir sinensis) (王晨赫, 2019)、凡纳滨对虾(Litopenaeus vannamei) (金若晨等, 2020)和尼罗罗非鱼(Oreochromis niloticus) (刘志刚等, 2018)等]的研究中均提出,环境菌群对于肠道菌群也具有一定的影响。因此,在今后的研究中,还应加强关注皱纹盘鲍转口期间养殖水体、饵料表面菌群和肠道菌群的关联分析。
肠道微生物多样性代表宿主的健康状态和代谢能力,与菌群功能稳定性有关,多样性低说明菌群的功能稳定性差,导致宿主被病菌侵害的可能性增加(Jena et al, 2018; 金若晨等, 2020)。Shi等(2017)发现,在同一无大规模病害暴发的养殖池内,健康皱纹盘鲍的肠道微生物多样性高于患病鲍。Xiong等(2015)发现,病池内对虾的肠道微生物多样性低于健康对虾。根据Alpha多样性分析的结果发现,稚鲍的肠道微生物多样性在转口后第35天和第40天明显低于转口前和转口后第4天,与前人的研究结果一致。Zhao等(2012)研究指出,杂色鲍(H. diversicolor)在摄食底栖硅藻阶段稚鲍的肠道细菌消化酶多样性高于摄食人工饲料和大型藻类阶段。Nel等(2018)研究发现,南非鲍摄食底栖硅藻组的Shannon-Wiener指数也高于转口后摄食海带组和人工饲料组。通过OTU分析,发现T4组的特有菌群种类最多,T0组次之,T10、T35和T40的特有菌群种类依次减少。由于宿主的饵料转变,肠道菌群处于不断演替的动态变化,可体现为OTU数量的变化(Zhang et al, 2021)。T4组处于饵料转变的初期,稚鲍表现出对饵料突然转变的不适应,且摄食状态不佳,处于一种半饥饿状态。研究表明,饥饿状态下,皱纹盘鲍的肠道菌群多样性升高(Choi et al, 2021)。这与本研究中T4组特有OTU数量突然增多的结果相符合。
Beta多样性分析结果显示,T0与T4的样本组间距离接近,在PCA图中的聚类圈重叠;而T35与T40的样本组间距离接近,但与T0和T4的距离较远。在共发生网络关系分析中,通过比较变形菌门、厚壁菌门和拟杆菌门的相关关系,发现T0与T4组菌群间的相关关系更为接近,而T35与40的菌群相关关系更接近。另外,T0与T4、T35与T40的物种组成也更为接近。因此,将Beta多样性分析、共发生网络分析和物种组成分析相结合来剖析转口期各组样本的组间关系,可见转口前及转口后第4天的菌群结构、组成及各个优势菌门间的相关关系都更为相似,但与转口后第35天和第40天的菌群存在一定的差异。
Tax4Fun功能预测结果表明,T0和T4的肠道菌群所编码的基因多与疾病和细胞加工有关,而T10、T35和T40则多与代谢功能有关。随着皱纹盘鲍对人工配合饲料的适应,其肠道菌群所编码的基因与代谢功能的相关性增加,包括氨基酸代谢、碳水化合物代谢和酶家族等,说明肠道微生物参与了多种分子代谢,协助稚鲍适应与底栖硅藻相比营养成分更加丰富的人工配合饲料,这与相关优势菌群,如拟杆菌门(Tran et al, 2018)、假交替单胞菌属(Tzuc et al, 2014)和Tamlana (刘邦涛, 2020)等随着转口期过程的演替和增加相符合。
4 结论本研究通过对皱纹盘鲍转口前、转口后第4天、第10天、第35天和第40天的肠道菌群变化进行研究分析,发现皱纹盘鲍的肠道微生物群落结构在转口期的变化规律:在门水平上,厚壁菌门丰度保持稳定,变形菌门丰度总体下降,拟杆菌门丰度总体升高;在属水平上,弧菌属与Formosa丰度升高,Lentilitoribacter丰度降低。转口后的优势菌群多为能利用配合饲料成分为能量源的细菌,代替了以底栖硅藻成分为能量源或与底栖硅藻共生的细菌,对协助稚鲍顺利完成转口具有重要意义。另外,转口后第35天和第40天稚鲍肠道微生物多样性的降低,说明其肠道内环境的不稳定性可能增加了稚鲍的患病风险。本研究分析了转口前后皱纹盘鲍的肠道菌群变化情况,为改善鲍育苗工作和健康养殖提供了新思路。
ARAHAL D R, LEKUNBERRI I, GONZÁLEZ J M, et al. Neptuniibacter caesariensis gen. nov., sp. nov., a novel marine genome-sequenced gammaproteobacterium. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(5): 1000-1006 DOI:10.1099/ijs.0.64524-0 |
BIEBL H, ALLGAIER M, TINDALL B J, et al. Dinoroseobacter shibae gen. nov., sp. nov., a new aerobic phototrophic bacterium isolated from dinoflagellates. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(3): 1089-1096 DOI:10.1099/ijs.0.63511-0 |
Bureau of Fisheries, Ministry of Agriculture and Rural Affairs, National Fisheries Technology Extension Center, China Society of Fisheries. China fishery statistical yearbook 2020. Beijing: China Agriculture Press, 2010 [农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 2020中国渔业统计年鉴. 北京: 中国农业出版社, 2010]
|
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336 DOI:10.1038/nmeth.f.303 |
CHEN Z N, WANG X Q, LUO L T, et al. High-throughput sequencing analysis of the effects of sanguinarine on Trionyx sinensis intestinal microbiota. Progress in Fishery Sciences, 2021, 42(1): 177-185 [陈贞年, 王晓清, 罗来婷, 等. 高通量测序分析血根碱对中华鳖肠道菌群结构的影响. 渔业科学进展, 2021, 42(1): 177-185] |
CHOI M J, OH Y D, KIM Y R, et al. Intestinal microbial diversity is higher in Pacific abalone (Haliotis discus hannai) with slower growth rates. Aquaculture, 2021, 537: 736500 DOI:10.1016/j.aquaculture.2021.736500 |
EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME improves sensitivity and speed of chimera detection. Bioinformatics, 2011, 27(16): 2194-2200 DOI:10.1093/bioinformatics/btr381 |
EDGAR R C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research, 2004, 32(5): 1792-1797 DOI:10.1093/nar/gkh340 |
EDGAR R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998 DOI:10.1038/nmeth.2604 |
FLEMING A E, HONE P W. Abalone aquaculture. Aquaculture, 1996, 140(1/2): 1-4 |
GAO S, PAN L Q, SONG M S, et al. Metagenomic insights into the structure and function of intestinal microbiota of the farmed Pacific white shrimp (Litopenaeus vannamei). Aquaculture, 2019, 499: 109-118 DOI:10.1016/j.aquaculture.2018.09.026 |
GOBET A, MEST L, PERENNOU M, et al. Seasonal and algal diet-driven patterns of the digestive microbiota of the European abalone Haliotis tuberculata, a generalist marine herbivore. Microbiome, 2018, 6(1): 60 DOI:10.1186/s40168-018-0430-7 |
GUO Z D. Effects of different weaning strategies on the survival rate and digestive system structure and function of larvae and juvenile Chinese sucker, Myxocyprinus asiaticus. Master´s Thesis of Southwest University, 2016 [郭忠娣. 不同转食策略对胭脂鱼仔稚鱼成活率和消化系统结构功能的影响. 西南大学硕士研究生学位论文, 2016]
|
HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454- pyrosequenced PCR amplicons. Genome Research, 2011, 21(3): 494-504 DOI:10.1101/gr.112730.110 |
HAMZA N, MHETLI M, KESTEMONT P. Effects of weaning age and diets on ontogeny of digestive activities and structures of pikeperch (Sander lucioperca) larvae. Fish Physiology and Biochemistry, 2007, 33(2): 121-133 DOI:10.1007/s10695-006-9123-4 |
HUANG Z Y, HE W H, PENG Z R, et al. Effects of adding Vallisneria natans meal to diet on growth performance, physiological and biochemical properties of juvenile grass carps (Ctenopharyngodon idellus). Progress in Fishery Sciences, 2020, 41(1): 169-177 [黄仲园, 何文辉, 彭自然, 等. 苦草粉对草鱼幼鱼生长性能与生理生化性能的影响. 渔业科学进展, 2020, 41(1): 169-177] |
JENA P K, SHENG L, NAGAR N, et al. The effect of synbiotics Bifidobacterium infantis and milk oligosaccharides on shaping gut microbiota community structure and NASH treatment. Data in Brief, 2018, 19: 1025-1029 DOI:10.1016/j.dib.2018.05.127 |
JIN R C, JIANG M, SUN S Y, et al. Microbial community in Litopenaeus vannamei intestine and its aquaculture environment. Journal of Fisheries of China, 2020, 44(12): 2037-2054 [金若晨, 江敏, 孙世玉, 等. 凡纳滨对虾养殖环境及肠道微生物群落特征分析. 水产学报, 2020, 44(12): 2037-2054 DOI:10.11964/jfc.20200312188] |
JOHNSTON D, MOLTSCHANIWSKYJ N, WELLS J. Development of the radula and digestive system of juvenile blacklip abalone (Haliotis rubra): Potential factors responsible for variable weaning success on artificial diets. Aquaculture, 2005, 250(1): 341-355 |
LIU B T. Analysis of microbial diversity in the abdominal intestine and studies on the alginate lyase from strain Agarivorans sp. B2Z047. Master´s Thesis of Shandong University, 2020 [刘邦涛. 鲍鱼肠道微生物多样性分析及菌株Agarivorans sp. B2Z047产褐藻胶裂解酶研究. 山东大学硕士研究生学位论文, 2020]
|
LIU Z G, LU M X, KE X L, et al. Correlation between microflora structure in intestinal tract and aquaculture environment of tiapia (Oreochromis niloticus) and streptococcicosis. Journal of Fisheries of China, 2018, 42(10): 1635-1647 [刘志刚, 卢迈新, 可小丽, 等. 尼罗罗非鱼肠道及养殖环境中菌群结构与链球菌病的相关性. 水产学报, 2018, 42(10): 1635-1647 DOI:10.11964/jfc.20170910951] |
LIU Z X, LIU X Z, SHI B, et al. Composition of intestinal bacterial community of Japanese flounder (Paralichthys olivaceus) during early life stages. Progress in Fishery Sciences, 2017, 38(1): 111-119 [刘增新, 柳学周, 史宝, 等. 牙鲆(Paralichthys olivaceus)仔稚幼鱼肠道菌群结构比较分析. 渔业科学进展, 2017, 38(1): 111-119] |
MAGOČ T, SALZBERG S L. FLASH: Fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963 DOI:10.1093/bioinformatics/btr507 |
MICHAUD L, GIUDICE A L, TROUSSELLIER M, et al. Phylogenetic characterization of the heterotrophic bacterial communities inhabiting a marine recirculating aquaculture system. Journal of Applied Microbiology, 2009, 107(6): 1935-1946 DOI:10.1111/j.1365-2672.2009.04378.x |
NAYAK S K. Role of gastrointestinal microbiota in fish. Aquaculture Research, 2010, 41(11): 1553-1573 DOI:10.1111/j.1365-2109.2010.02546.x |
NEL A, JONE C L W, BRITZ P J, et al. The effect of juvenile abalone Haliotis midae (Linnaeus, 1758) weaning diet on gut-bacterial formation. Journal of Shellfish Research, 2018, 37(1): 191-197 DOI:10.2983/035.037.0117 |
NICHOLSON J K, HOLMES E, KINROSS J, et al. Host-gut microbiota metabolic interactions. Science, 2012, 336(6086): 1262-1267 DOI:10.1126/science.1223813 |
PARK S, LEE J S, LEE K C, et al. Lentilitoribacter donghaensis gen. nov., sp. nov., a slowly-growing alphaproteobacterium isolated from coastal seawater. Antonie van Leeuwenhoek, 2013, 103(3): 457-464 DOI:10.1007/s10482-012-9825-9 |
QUAST C, PRUESSE E, YILMAZ P, et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research, 2013, 41: 590-596 |
SAMPSON T R, DEBELIUS J W, THRON T, et al. Gut microbiota regulate motor deficits and neuroinflammation in a model of Parkinson's disease. Cell, 2016, 167(6): 1469-1480 DOI:10.1016/j.cell.2016.11.018 |
SHI L Y, LIANG S, LUO X, et al. Microbial community of Pacific abalone (Haliotis discus hannai) juveniles during a disease outbreak in South China. Aquaculture Research, 2017, 48(3): 1080-1088 DOI:10.1111/are.12950 |
TANAKA R, CLEENWERCK I, MIZUTANI Y, et al. Formosa haliotis sp. nov., a brown-alga-degrading bacterium isolated from the gut of the abalone Haliotis gigantea. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(12): 4388-4393 |
TANAKA R, SUGIMURA I, SAWABE T, et al. Gut microflora of abalone Haliotis discus hannai in culture changes coincident with a change in diet. Fisheries Science, 2003, 69(5): 951-958 DOI:10.1046/j.1444-2906.2003.00712.x |
TRAN N T, XIONG F, HAO Y T, et al. Starvation influences the microbiota assembly and expression of immunity-related genes in the intestine of grass carp (Ctenopharyngodon idellus). Aquaculture, 2018, 489: 121-129 |
TZUC J T, ESCALANTE D R, HERRERA R R, et al. Microbiota from Litopenaeus vannamei: Digestive tract microbial community of Pacific white shrimp (Litopenaeus vannamei). SpringerPlus, 2014, 3: 280 |
WAGNER-DÖBLER I, BALLHAUSEN B, BERGER M, et al. The complete genome sequence of the algal symbiont Dinoroseobacter shibae: A hitchhiker´s guide to life in the sea. The ISME Journal, 2010, 4(1): 61-77 |
WANG A R, RAN C, RINGØ E, et al. Progress in fish gastrointestinal microbiota research. Reviews in Aquaculture, 2018, 10(3): 626-640 |
WANG C H. Chinese mitten crab (Eriocheir sinensis) gut microbes structure characteristics and correlation research and breeding environment microorganisms. Master´s Thesis of Nanjing Agricultural University, 2019 [王晨赫. 中华绒螯蟹(Eriocheir sinensis)肠道微生物结构特征及与养殖环境微生物相关性研究. 南京农业大学硕士研究生学位论文, 2019]
|
WANG X Z, TANG B, Luo X, et al. Effects of temperature, diet and genotype-induced variations on the gut microbiota of abalone. Aquaculture, 2020, 524: 735269 |
WU H H, WANG W J, LÜ D, et al. Turbot (Scophthalmus maximus) biodiversity assessment using high-throughput Illumina sequencing to analyze juvenile turbot intestines and their bacterial cultures. Progress in Fishery Sciences, 2019, 40(4): 84-94 [吴欢欢, 王伟继, 吕丁, 等. 应用高通量测序技术分析大菱鲆幼鱼肠道及其养殖环境的微生物群落结构. 渔业科学进展, 2019, 40(4): 84-94] |
WU S G, WANG G T, ANGERT E R, et al. Composition, diversity, and origin of the bacterial community in grass carp intestine. PLoS One, 2012, 7(2): e30440 |
XIONG J B, WANG K, WW J F, et al. Changes in intestinal bacterial communities are closely associated with shrimp disease severity. Applied Microbiology and Biotechnology, 2015, 99(16): 6911-6919 |
XU X H. Research on ecological characteristics and the retrieve of emerging pathogenic bacteria in urban water body. Doctoral Dissertation of Jiangsu University, 2008 [许小红. 城市水体中新型病原细菌生态学特征及其溯源研究. 江苏大学博士研究生学位论文, 2008]
|
ZHANG Y, WEN B, MENG L J, et al. Dynamic changes of gut microbiota of discus fish (Symphysodon haraldi) at different feeding stages. Aquaculture, 2021, 531(5): 735912 |
ZHAO J, SHI B, JIANG Q R, et al. Changes in gut-associated flora and bacterial digestive enzymes during the development stages of abalone (Haliotis diversicolor). Aquaculture, 2012, 338/339/340/341: 147-153 |
ZHAO L L, HE K, LUO J, et al. Co-modulation of liver genes and intestinal microbiome of largemouth bass larvae (Micropterus salmoides) during weaning. Frontiers in Microbiology, 2020, 11: 1332 |
ZHU D M. Study on bacterial flora with early life stages of the grass carp (Ctenopharyngodon idellus). Master´s Thesis of Huazhong Agricultural University, 2008 [祝东梅. 草鱼早期发育阶段细菌群落的研究. 华中农业大学硕士研究生学位论文, 2008]
|



