2. 江苏省淡水水产研究所 农业农村部淡水虾蟹遗传育种与养殖重点实验室 江苏 南京 210017;
3. 中国科学院南京地理与湖泊研究所 江苏 南京 210008
2. Freshwater Fisheries Research Institute of Jiangsu Province, Key Laboratory of Genetic Breeding and Cultivation for Freshwater Crustacean, Ministry of Agriculture and Rural Affairs, Nanjing, Jiangsu 210017, China;
3. Nanjing Institute of Geography and Lakes, Chinese Academy of Sciences, Nanjing, Jiangsu 210008, China
纤维素是由β-(1, 4)-D-葡萄糖单元聚合形成的线性多聚糖,是自然界中分布最广、含量最丰富的可再生性资源,其合成与降解是自然界碳循环的中心环节。纤维素酶属于糖苷水解酶(glycoside hydrolase, GH)。早期的研究认为,动物自身不能合成内源性纤维素酶,而主要依靠消化器官内共生菌分泌的外源性纤维素酶来消化分解植物性饵料。直到Smant等(1998)从马铃薯金线虫(Globodera rostochiensis)和黄胸散白蚁(Reticulitermes speratus)体内分别成功克隆了糖苷水解酶GH9基因序列,才证实动物体内内源性纤维素酶的存在。随后,纤维素酶基因在植物寄生线虫类、软体类、昆虫类以及甲壳类等动物中被广泛发现(Watanabe et al, 2001; Tanimura et al, 2013)。
中华绒螯蟹(Eriocheir sinensis)又名河蟹,广泛分布于我国沿海的咸淡水与内陆淡水江河湖库水域,是我国主要养殖的经济甲壳动物之一(邵洋等, 2021)。中华绒螯蟹为典型的杂食性甲壳动物,自然水域的中华绒螯蟹胃含物中食物组分多样,既有植物性饵料,又有动物性饵料,但常以植物性饵料为主;人工养殖条件下的中华绒螯蟹偏好动物性饵料,但仍然会摄入一定量的水生植物(陈炳良等, 1989)。中华绒螯蟹自身能否合成内源性纤维素酶类来消化分解水生植物纤维素尚不清楚。本研究通过转录组测序结合分子克隆获得中华绒螯蟹内源性纤维素酶基因(EsGH9-1)的DNA和cDNA序列,分析其组织分布与发育阶段表达特征,比较不同饵料条件下纤维素酶基因及酶活性的差异,以期为中华绒螯蟹的养殖生产提供借鉴参考。
1 材料与方法 1.1 实验材料 1.1.1 实验对象中华绒螯蟹1龄幼蟹取自江苏省淡水水产研究所养殖基地,运至江苏省淡水水产研究所室内循环养殖实验室暂养3~5 d后,选取附肢健全、健康有活力的个体作为实验样本用于后续实验。
1.1.2 实验试剂Total RNA提取试剂盒、反转录试剂盒、DNA提取试剂盒、RT-PCR试剂(SYBR Premix Ex TaqTM Ⅱ、荧光染料ROX)、DNA marker购自宝日医生物技术有限公司,PCR试剂、引物合成服务以及序列测序服务均由生工生物工程(上海)股份有限公司提供,其他试剂均为国产分析纯。
1.2 样品采集选取1龄幼蟹进行不同组织部位的解剖,包括肝胰腺、胃、肠道、心脏、血淋巴、肌肉、表皮和再生附肢。采集不同发育时期的中华绒螯蟹,包括溞状幼体时期(Ⅰ、Ⅲ和Ⅴ期)、大眼幼体、仔蟹(Ⅰ、Ⅲ和Ⅴ期)和幼蟹。每个组织部位或发育阶段时期取3个生物学重复。在解剖样品前,先对样品进行清洗并置于冰上降低中华绒螯蟹的活跃度。所有收集样本经液氮速冻后置于–80℃冰箱保存备用。
1.3 总DNA、总RNA提取与cDNA的合成根据Universal DNA extraction试剂盒操作步骤,提取中华绒螯蟹肌肉组织总DNA,于–20℃保存备用。根据MiniBEST Universal RNA Extraction (TaKaRa)试剂盒操作步骤,提取中华绒螯蟹各组织和不同发育阶段个体总RNA,于–80℃保存备用。根据Prime Script™ RT reagent kit with gDNA eraser (Perfect Real Time)试剂说明书进行逆转录反应,获得cDNA后于–20℃保存备用。
1.4 EsGH9-1基因组DNA与cDNA序列的克隆利用已知近缘红螯螯虾(Cherax quadricarinatus) β-1, 4-内切葡聚糖酶cDNA序列(AF148497)比对中华绒螯蟹肝胰腺转录组文库(PRJNA310144),筛选到中华绒螯蟹β-1, 4-内切葡聚糖酶cDNA序列。以中华绒螯蟹肝胰腺cDNA为模板,采用Primer 5.0软件设计引物,对EsGH9-1基因的cDNA序列进行扩增和确认。在获得EsGH9-1基因外显子序列的基础上,设计引物对所预测的内含子进行PCR扩增和测序,引物序列见表 1。将PCR扩增产物送至生工生物工程(上海)股份有限公司测序,后拼接得到纤维素酶基因组DNA序列。
|
|
表 1 本研究所用引物序列 Tab.1 Primers used in this study |
应用EditSeq程序进行开放阅读框(ORF)分析并将其推导为相应的氨基酸序列;使用NCBI (http://www.ncbi.nlm.nih.gov) BLAST进行蛋白质序列同源性检索分析;使用ExPASy (https://web.expasy.org/protparam/)在线工具分析蛋白质的分子量、等电点及基本性质;使用SMART (http://smart.embl-heidelberg.de/)预测蛋白功能域;使用Prosite (https://prosite.expasy.org/)预测蛋白氨基酸结构功能位点;使用ClustalW2软件将克隆序列与同源氨基酸序列对比分析;应用MEGA 6软件,采用Neighbor-Joining法构建系统进化树。
1.6 EsGH9-1基因mRNA实时荧光PCR定量检测采用RT-PCR技术检测EsGH9-1在不同组织、不同发育时期及不同饵料条件下的相对表达量(张宏叶等, 2017)。将第一链cDNA模板按1∶20稀释,用CFX96实时PCR检测系统(Eppendorf, 德国)进行RT-PCR。以β-actin作为内参基因,无菌超纯水为阴性对照,β-actin和纤维素酶基因的引物如表 1所示。RT-PCR在3个独立的实验中每个样品设置3个生物学重复,用2−ΔΔCt法计算相对表达量。
1.7 不同饵料组实验随机选取规格为(25±5) g的1龄幼蟹分为3个实验组,包括配合饲料饲喂组(江苏海普瑞饲料有限公司,主要原料为鱼粉、鱼油、大豆磷脂油、豆粕、花生粕和面粉,粗蛋白≥40.0%,粗灰分≤18.0%,粗脂肪≥5.0%,粗纤维≤6.5%)、动物性饵料饲喂组(冰带鱼)和植物性饵料饲喂组(伊乐藻)。每池放置24只,每个实验组3个平行。实验在9个圆柱形塑料缸中进行,实验持续30 d,每天17:30进行投喂,投喂量约为河蟹体重的3%~5%,每日1次,每天08:00清除池内粪便、少量残饵和死蟹,实验期间,保持水温为28.0~29.5℃,pH 7.5~7.9,溶解氧≥5.0 mg/L,氨氮 < 0.05 mg/L。实验结束后,在最后一次投喂后2 h进行取样,每缸各取3只中华绒螯蟹的肝胰腺组织,经液氮冷却后于–80℃保存备用,用于测定EsGH9-1在不同饵料条件下的相对表达量以及β-1, 4-内切葡聚糖酶活性。
1.8 β-1, 4-内切葡聚糖酶活性测定采用DNS法测定β-1, 4-内切葡聚糖酶活性,测量方法参照谢骜李畅等(2020)。
1.9 数据分析所得数据均以平均值±标准误(Mean±SE)表示,采用SPSS统计软件中的邓肯法分析各组数据的差异情况,P < 0.05表示差异显著(以不同字母标记),采用Origin 2018软件绘图。
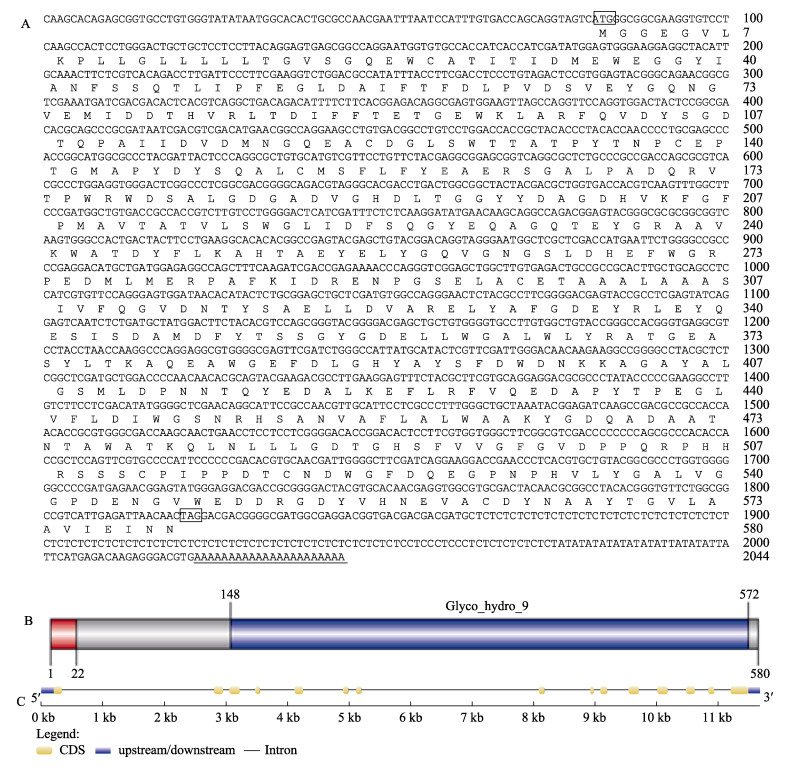
2 结果 2.1 EsGH9-1基因组DNA和cDNA序列的克隆EsGH9-1 cDNA序列长为2044 bp (GenBank登录号:MZ447859),其中,包括80 bp的5′端非翻译区、221 bp的3′端非翻译区和编码580个氨基酸残基的1740 bp开放阅读框(图 1A)。利用ExPASy预测了编码蛋白的理化性质,推测该蛋白的分子式为C2876H4209N727O901S19,相对分子量为63, 993.53,理论等电点为4.14,为亲水性蛋白。EsGH9-1包含信号肽和糖苷水解酶家族9功能催化区(glycoside hydrolase family 9) (图 1B)。EsGH9-1基因组DNA为11 679 bp,包含15个外显子和14个内含子,外显子和内含子接头区符合受体拼接点和供体拼接点的Chambon法则,详见基因结构示意图(图 1C)。
|
图 1 中华绒螯蟹纤维素酶EsGH9-1序列分析 Fig.1 Analysis of EsGH9-1 gene from E. sinensis A:cDNA序列分析:起始密码子(ATG)和终止密码子(TAG)用方框标出,下划黑线表示poly(A)尾巴;B:功能结构域示意图;C:DNA序列结构示意图 A: cDNA sequence analysis: The start codon (ATG) and termination codon (TAG) are marked with boxes, the poly (A) was underlined; B: Functional domain schematic; C: Schematic diagram of DNA sequence structure |
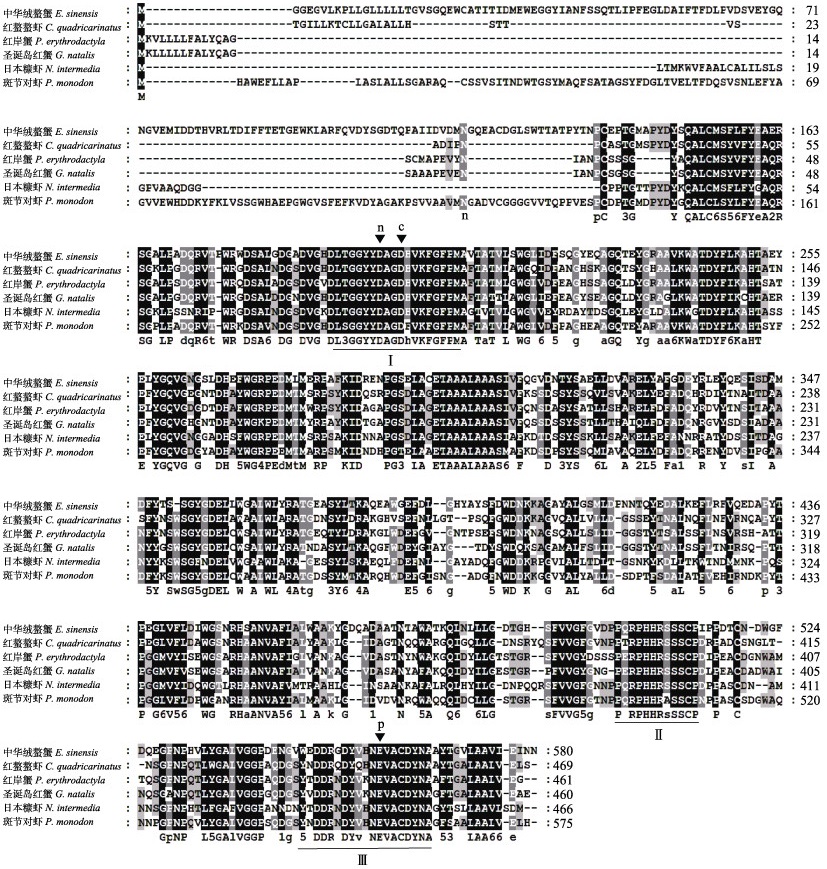
SWISS-MODEL三维预测结果显示,EsGH9-1蛋白的三级结构由α螺旋、β折叠以及连接的无规则卷曲构成,催化活性中心谷氨酸Glu559是质子供体,天冬氨酸Asp198是亲核基团,天冬氨酸Asp201是共亲核基团(图 2)。
|
图 2 中华绒螯蟹纤维素酶EsGH9-1与其他甲壳类序列比对分析 Fig.2 Amino acid sequence alignment of EsGH9-1 and other cellulase sequence in crustaceans 深黑色区域:相似性100%;深灰色区域:相似性≥75%;浅灰色区域:相似性≥50%。催化残基用黑色倒三角标出,n表示亲核基团,c表示共亲核基团,p表示质子供体。3个保守区以罗马数字Ⅰ、Ⅱ和Ⅲ表示。物种的GenBank ID:中华绒螯蟹MZ447859;红螯螯虾AAD38027.1;红岸蟹AIT11911.1;圣诞岛红蟹APF29556.1;日本糠虾BAL60587.1;斑节对虾XP_037800235.1。 Black area: Similarity 100%; Grey area: Similarity ≥75%; Lighter grey area: Similarity ≥50%. The catalytic residues are marked with a black inverted triangle. n=Nucleophile, c=Co-nucleophile, p=Proton donor. Three conserved regions are marked with Ⅰ, Ⅱ, and Ⅲ respectively. The GenBank IDs of reference sequences are shown as follows: E. sinensis (MZ447859), C. quadricarinatus (AAD38027.1), P. erythrodactyla (AIT11911.1), G. natalis (APF29556.1), N. intermedia (BAL60587.1), P. monodon (XP_037800235.1). |
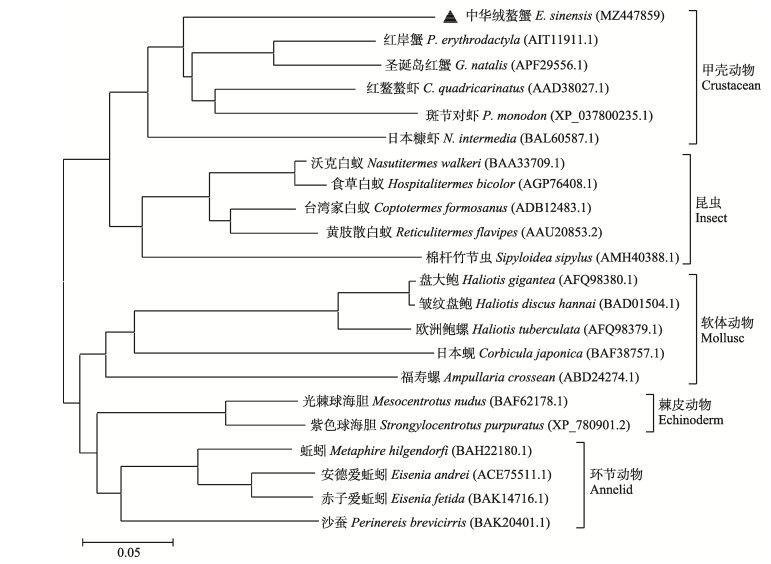
中华绒螯蟹EsGH9-1预测氨基酸序列与其他近缘物种的纤维素酶蛋白进行多重序列比对,结果显示,EsGH9-1与红螯螯虾、红岸蟹(Parasesarma erythrodactyla)、圣诞岛红蟹(Gecarcoidea natalis)、日本糠虾(Neomysis intermedia)、斑节对虾(Penaeus monodon)的同源性在57.81%~64.69%之间(图 2)。系统进化分析结果显示,EsGH9-1首先与红螯螯虾、红岸蟹、日本糠虾等甲壳类聚为一支,之后与昆虫类聚类,其余GH9以软体动物、棘皮动物和环节动物为单位形成独立分支,与传统的分类方法结果相一致(图 3)。
|
图 3 基于氨基酸序列构建的中华绒螯蟹及其他物种GH9的系统进化树 Fig.3 Phylogenetic relationship of GH9 from E. sinensis and different species based on amino acid sequences |
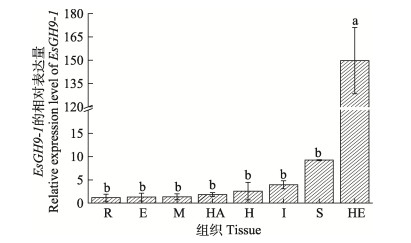
RT-PCR检测结果显示,EsGH9-1主要分布在消化器官,在肝胰腺组织中表达量最高,其次在胃和肠道组织,而在其他组织中表达量低(图 4)。
|
图 4 中华绒螯蟹纤维素酶EsGH9-1在不同组织中的表达量
Fig.4 Expression of the EsGH9-1 in different tissues of E. sinensis
R:再生附肢;E:表皮;M:肌肉;HA:血淋巴;H:心脏;I:肠道;S:胃;HE:肝胰腺 柱形图上的不同字母表示mRNA表达水平存在显著差异(P < 0.05)。下同。 R: Regenerated appendage; E: Epidermis; M: Muscle; HA: Haemolymph; H: Heart; I: Intestines; S: Stomach; HE: Hepatopancreas Different letters on the column chart indicate significant differences in mRNA expression levels (P < 0.05). The same as below. |
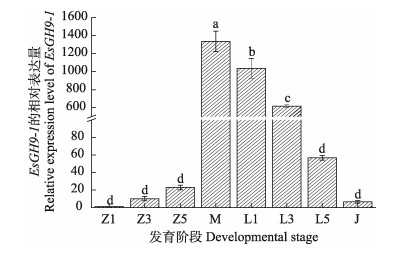
RT-PCR检测结果显示,中华绒螯蟹EsGH9-1在溞状幼体时期表达量较低,进入到大眼幼体期和仔蟹前期表达量最高,仔蟹Ⅴ期后EsGH9-1的表达显著下降(图 5)。
|
图 5 中华绒螯蟹纤维素酶EsGH9-1在不同发育时期的表达量 Fig.5 Expression of the EsGH9-1 in different developmental stages of E. sinensis Z1:溞状幼体Ⅰ期;Z3:溞状幼体Ⅲ期;Z5:溞状幼体Ⅴ期;M:大眼幼体;L1:Ⅰ期仔蟹;L3:Ⅲ期仔蟹;L5:Ⅴ期仔蟹;J:幼蟹 Z1: Zoea Ⅰ; Z3: Zoea Ⅲ; Z5: ZoeaⅤ; M: Megalopa; L1: Larvae Ⅰ; L3: Larvae Ⅲ; L5: Larvae Ⅴ; J: Juvenile |
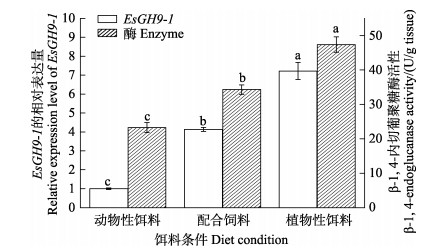
RT-PCR检测结果显示,EsGH9-1在植物性饵料组表达量最高,配合饲料组次之,动物性饵料组表达量最低,不同饵料条件下肝胰腺β-1, 4-内切葡聚糖酶活性与EsGH9-1表达趋势一致(图 6)。
|
图 6 不同饵料条件下中华绒螯蟹纤维素酶EsGH9-1的表达量及β-1, 4-内切葡聚糖酶活性的变化 Fig.6 The expression of EsGH9-1 and β-1, 4-endoglucanase activity from E. sinensis under different diet conditions |
纤维素酶广泛存在于植物、细菌、真菌等微生物体内。随着陆生昆虫、水生贝类、鱼类等动物内源性纤维素酶基因的发现,有关水生甲壳类内源性纤维素酶基因的报道也越来越多(Watanabe et al, 2001; Tanimura et al, 2013)。Byrne等(1999)首次在红螯螯虾肝胰腺组织中克隆获得了GH9家族的纤维素酶基因cDNA序列。随后Crawford等(2004)在红螯螯虾肌肉组织克隆获得了GH9基因组DNA全长序列。Linton等(2006)在多个海洋蟹类中肠组织中扩增获得GH9基因序列片段,并在肌肉组织中扩增获得GH9基因的DNA序列。Gray等(2017)研究发现,GH9基因序列在陆生草食性与非草食性蟹类体内也广泛存在。本研究通过转录组测序结合分子克隆从中华绒螯蟹肝胰腺组织中获得EsGH9-1 cDNA序列,从肌肉组织中获得EsGH9-1基因组DNA序列,证实了中华绒螯蟹自身含有内源性纤维素酶类GH9基因。
作为甲壳类动物重要的消化器官,肝胰腺是合成消化酶的主要场所。本研究中,内源性EsGH9-1主要在肝胰腺组织中表达,这与一些虾蟹类,如红岸蟹、红螯螯虾、罗氏沼虾(Macrobrachium rosenbergii)等结果相一致(Bui et al, 2015; Tan et al, 2016; Ong et al, 2016)。此外,在一些草食性虾蟹肝胰腺中检测到纤维素酶活性也印证了肝胰腺是纤维素酶的合成部位(Linton et al, 2004、2011)。中华绒螯蟹肝胰腺是一个大型致密腺体,包被在中肠前端及幽门胃处,与中肠的起始段相连,内源性纤维素酶在肝胰腺合成后,并分泌到胃液和肠道中进行纤维素的消化与分解。
EsGH9-1在大眼幼体期与仔蟹前期高表达,可能与其不同生长发育阶段的摄食能力与食性转变有关。在溞状幼体时期,中华绒螯蟹的摄食以被动滤食为主,进入到大眼幼体阶段,中华绒螯蟹已具备大螯和口器,主动摄食能力增强,对浮游动、植物的摄入量都增多(付龙龙等, 2015);到仔蟹阶段,蟹体由浮游型转为底栖型,摄食种类也由浮游性食物向底栖性食物如沉积有机碎屑、水生植物根茎转变(崔莹等, 2019)。此外,何林岗等(1988)研究发现,中华绒螯蟹各期幼体的摄食量变化不呈线性相关的递增性,其摄食量在大眼幼体阶段暴发式增长,仔蟹Ⅰ期稍有下降,这与本研究中EsGH9-1在大眼幼体期与仔蟹前期表达量的飞跃式增长具有一致性。而仔蟹Ⅴ期后EsGH9-1表达量下降的原因还有待于进一步研究。
饵料种类与构成和动物消化酶类的表达活性密切相关,草食性动物通常表现出较高的糖苷水解酶活性,而肉食性动物则表现出较高的脂质和蛋白酶活性(Guo et al, 2019; Wang et al, 2020)。本研究中,中华绒螯蟹肝胰腺组织中EsGH9-1相对表达量及β-1, 4-内切葡聚糖酶活性在植物性饵料组中最高,这与罗氏沼虾、锯缘青蟹(Scylla serrata)肝胰腺组织纤维素酶活性随纤维素摄入量的增加而升高的结果相一致,表明EsGH9-1可能参与植物性饵料的消化与吸收(Gonzalez-Pena et al, 2002; Pavasovic et al, 2004)。微型藻类与水生植物等植物性饵料是中华绒螯蟹的重要饵料,对中华绒螯蟹的食物贡献率为40% (Duffy et al, 2011; Mao et al, 2016)。纤维素酶活性水平代表对植物性饵料纤维素等的同化效率,影响甚至决定着甲壳类的生态营养位与空间分布(Kawaida et al, 2019)。内源性纤维素酶基因表达受到抑制会导致酶活性降低、体重减轻甚至死亡(Shelomi et al, 2019; Wu et al, 2019)。因此,纤维素酶在植物性饵料的摄食与消化过程中起着重要作用。中华绒螯蟹内源纤维素酶活性对植物饲料的响应促进了富含纤维素的植物细胞壁的溶解,有利于中华绒螯蟹对纤维素和其他植物性食物的消化和分解。
本研究从中华绒螯蟹肌肉与肝胰腺组织中克隆获得内源性纤维素酶EsGH9-1基因组DNA和cDNA序列,证实了中华绒螯蟹自身具有内源性纤维素酶基因。EsGH9-1具有糖苷水解酶第9家族的典型结构特征,主要分布在肝胰腺组织,在大眼幼体期以及仔蟹前期高表达。不同饵料条件下,植物性饵料组EsGH9-1相对表达量以及β-1, 4-内切葡聚糖酶活性显著上调,推测EsGH9-1参与中华绒螯蟹对纤维素的消化与分解。但由于缺少EsGH9-1体外表达、活性检测等功能验证,EsGH9-1对植物性饵料消化分解的贡献作用、与肠道菌群消化分解是否存在协作互补等还有待于进一步研究。
BUI T, LEE S Y. Endogenous cellulase production in the leaf litter foraging mangrove crab Parasesarma erythodactyla. Comparative Biochemistry and Physiology Part B Biochemistry and Molecular Biology, 2015, 179: 27-36 DOI:10.1016/j.cbpb.2014.09.004 |
BYRNE K A, LEHNERT S A, JOHNSON S E, et al. Isolation of a cDNA encoding a putative cellulase in the red claw crayfish Cherax quadricarinatus. Gene, 1999, 239(2): 317-324 DOI:10.1016/S0378-1119(99)00396-0 |
CHEN B L, DU N S, YE H F. Analysis on the feeding habit of the mitten crab Eriocheir sinensis. Fisheries Science and Technology Information, 1989(1): 1-5 [陈炳良, 堵南山, 叶鸿发. 中华绒螯蟹的食性分析. 水产科技情报, 1989(1): 1-5 DOI:10.16446/j.cnki.1001-1994.1989.01.001] |
CRAWFORD A C, KRICKER J A, ANDERSON A J, et al. Structure and function of a cellulase gene in redclaw crayfish, Cherax quadricarinatus. Gene, 2004, 340(2): 267-274 DOI:10.1016/j.gene.2004.06.060 |
CUI Y, WU X G, ZHAO F, et al. Diet sources of juvenile Chinese mitten crab (Eriocheir sinensis) during upstream migration indicated by the carbon and nitrogen stable isotopes. Oceanologia et Limnologia Sinica, 2019, 50(4): 822-829 [崔莹, 吴旭干, 赵峰, 等. 基于碳、氮稳定同位素分析中华绒螯蟹(Eriocheir sinensis)幼蟹溯河洄游期食物来源变化. 海洋与湖沼, 2019, 50(4): 822-829] |
DUFFY R E, GODWIN I, NOLAN J, et al. The contribution of naturally occurring food items to the diet of Cherax destructor when fed formulated diets of differing protein levels. Aquaculture, 2011, 313(1/2/3/4): 107-114 |
FU L L, LI Y H, LU Q P, et al. Analysis of key points in the initial stage of crab seedling transportation and stocking. Journal of Aquaculture, 2015, 36(3): 40-40 [付龙龙, 李跃华, 陆全平, 等. 蟹苗运输及放养初期关键点剖析. 水产养殖, 2015, 36(3): 40-40 DOI:10.3969/j.issn.1004-2091.2015.03.014] |
GONZALEZ-PENA M C, ANDERSON A J, SMITH D M, et al. Effect of dietary cellulose on digestion in the prawn Macrobrachium rosenbergii. Aquaculture, 2002, 211(1/2/3/4): 291-303 |
GRAY M, LINTON S M, ALLARDYCE B J. cDNA sequences of GHF9 endo-β-1, 4-glucanases in terrestrial Crustacea. Gene, 2017, 642: 408-422 |
GUO H, TANG D, SHI X, et al. Comparative transcriptome analysis reveals the expression and characterization of digestive enzyme genes in the hepatopancreas of the Chinese mitten crab. Fisheries Science, 2019, 85(6): 979-989 DOI:10.1007/s12562-019-01358-4 |
HE L G, GU Z M. Food requirement of the crab Eriocheir sinensis in larval stages. Oceanologia et Limnologia Sinica, 1988, 19(4): 391-395 [何林岗, 顾志敏. 中华绒螯蟹幼体摄饵量的研究. 海洋与湖沼, 1988, 19(4): 391-395] |
KAWAIDA S, NANJO K, OHTSUCHI N, et al. Cellulose digestion abilities determine the food utilization of mangrove estuarine crabs. Estuarine Coastal and Shelf Science, 2019, 222(30): 43-52 |
LINTON S M, GREENAWAY P, TOWLE D W. Endogenous production of endo-β-1, 4-glucanase by decapod crustaceans. Journal of Comparative Physiology B Biochemical Systems and Environmental Physiology, 2006, 176(4): 339-348 DOI:10.1007/s00360-005-0056-5 |
LINTON S M, GREENAWAY P. Presence and properties of cellulase and hemicellulase enzymes of the gecarcinid land crabs Gecarcoidea natalis and Discoplax hirtipes. Journal of Experimental Biology, 2004, 207(23): 4095-4104 DOI:10.1242/jeb.01252 |
LINTON S M, SHIRLEY A J. Isozymes from the herbivorous gecarcinid land crab, Gecarcoidea natalis that possess both lichenase and endo-β-1, 4-glucanase activity. Comparative Biochemistry and Physiology Part B Biochemistry and Molecular Biology, 2011, 160(1): 44-53 DOI:10.1016/j.cbpb.2011.05.007 |
MAO Z G, GU X H, ZENG Q F. Food sources and trophic relationships of three decapod crustaceans: Insights from gut contents and stable isotope analyses. Aquaculture Research, 2016, 47(9): 2888-2898 DOI:10.1111/are.12739 |
ONG S S, BHASSU S, KWONG Q B, et al. Identification of a putative cellulase gene in the giant freshwater prawn, Macrobrachium rosenbergii (De Man, 1879). Aquaculture Research, 2016, 47(11): 3653-3661 DOI:10.1111/are.12818 |
PAVASOVIC M, RICHARDSON N A, ANDERSON A J, et al. Effect of pH, temperature and diet on digestive enzyme profiles in the mud crab, Scylla serrata. Aquaculture, 2004, 242(1/2/3/4): 641-654 |
SHAO Y, YU D W, YANG F, et al. Comparison of the taste compounds in different grades of Chinese mitten crab (Eriocheir sinensis). Progress in Fishery Sciences, 2021, 42(4): 192-198 [邵洋, 余达威, 杨方, 等. 不同等级中华绒螯蟹滋味物质的比较分析. 渔业科学进展, 2021, 42(4): 192-198] |
SHELOMI M, WIPFLER B, ZHOU X, et al. Multifunctional cellulase enzymes are ancestral in Polyneoptera. Insect Molecular Biology, 2019, 29(1): 124-135 |
SMANT G, STOKKERMANS J, YAN Y, et al. Endogenous cellulases in animals: Isolation of β-1, 4-endoglucanase genes from two species of plant-parasitic cyst nematodes. Proceedings of the National Academy of Sciences, 1998, 95(9): 4906-4911 DOI:10.1073/pnas.95.9.4906 |
TAN M H, GAN H M, GAN H Y, et al. First comprehensive multi-tissue transcriptome of Cherax quadricarinatus (Decapoda: Parastacidae) reveals unexpected diversity of endogenous cellulase. Organisms Diversity and Evolution, 2016, 16(1): 185-200 DOI:10.1007/s13127-015-0237-3 |
TANIMURA A, LIU W, YAMADA K, et al. Animal cellulases with a focus on aquatic invertebrates. Fisheries Science, 2013(79): 1-13 |
WANG Z F, TANG D, GUO H Y, et al. Evolution of digestive enzyme genes associated with dietary diversity of crabs. Genetica, 2020, 148(8): 87-99 |
WATANABE H, NODA H, TOKUDA G, et al. A cellulase gene of termite origin. Nature, 1998, 394(6691): 330-331 |
WATANABE H, TOKUDA G. Animal cellulases. Cellular and Molecular Life Sciences, 2001, 58(9): 1167-1178 |
WU W G, GU D F, YAN S C, et al. RNA interference of endoglucanases in the formosan subterranean termite Coptotermes formosanus shiraki (Blattodea: Rhinotermitidae) by dsRNA injection or ingestion. Journal of Insect Physiology, 2019, 112: 15-22 |
XIE A L C, YANG Y X, WANG X L, et al. Cloning endo- glucanase gene of cellulolytic microorganisms from Apriona germari and its expression in Lactobacillus. Chinese Journal of Animal Nutrition, 2020, 32(3): 1344-1352 [谢骜李畅, 杨雨鑫, 王小龙, 等. 桑天牛肠道微生物内切葡聚糖酶基因的克隆及在乳酸杆菌中的表达. 动物营养学报, 2020, 32(3): 1344-1352] |
ZHANG H Y, YU X D, WANG P P, et al. Cloning and expression analysis of Mn-SOD gene of Odontobutis potamophila. Progress in Fishery Sciences, 2017, 38(4): 34-43 [张宏叶, 于兴达, 王佩佩, 等. 河川沙塘鳢(Odontobutis potamophila) Mn-SOD基因的克隆及表达分析. 渔业科学进展, 2017, 38(4): 34-43] |



