2. 中国水产科学研究院黄海水产研究所农业农村部海洋渔业可持续发展重点实验室 山东 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071;
4. 青岛瑞滋集团有限公司 山东 青岛 266409
2. Key Laboratory of Sustainable and Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao, Shandong 266071, China;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao, Shandong 266071, China;
4. Qingdao Ruizi Company, Qingdao, Shandong 266409, China
仿刺参(Apostichopus japonicus)又称刺参,是我国第五次海水养殖浪潮的代表性经济物种之一,年产量近20万t。然而,随着养殖产业的迅猛发展,养殖过程中出现的病害频发、养殖成活率降低等一系列问题已经成为限制刺参产业可持续发展的主要瓶颈。解析刺参的抗病机制对于指导良种选育和健康养殖具有重要意义。课题组前期完成的刺参重要疾病的流行病学和病原学研究结果表明,刺参腐皮综合征是刺参养殖中最主要的疾病种类,灿烂弧菌(Vibrio splendidus)是该疾病的主要致病原之一(张春云等, 2006)。因此,解析刺参响应灿烂弧菌侵染的分子机制可为揭示刺参抗病机制提供重要参考。
microRNA (miRNA)是一类内源性的、进化上高度保守的非编码小RNA单链分子,它作为转录后水平的关键调控因子,通过与靶基因3´端非翻译区(UTR)结合而抑制靶基因的翻译或降解靶基因,在调控真核生物的生长发育、细胞分化和免疫防御等生命活动中发挥了重要作用(Krol et al, 2010)。近年来,水生生物miRNA的鉴定及其对靶基因的调控作用机制逐渐成为水产养殖研究的热点之一。研究表明,miRNA可以参与鱼、虾、蟹、贝、参等水生生物的免疫调控过程(Zhang et al, 2017; Chen et al, 2017; Boonchuen et al, 2020; Li et al, 2018)。Li等(2012)利用miRNA-seq技术筛选出刺参体腔细胞响应灿烂弧菌的差异表达miRNA,但目前缺乏关于腐皮综合征发病的靶器官——体壁组织miRNA及其调控网络等相关机制的研究报道。因此,本研究以刺参体壁组织为研究对象,采用miRNA-seq技术对健康刺参和灿烂弧菌侵染后患病刺参的体壁组织进行测序,通过生物信息学软件筛选DEmiRNAs并进行验证,构建关键免疫相关通路上miRNA-mRNA的调控网络,以期为解析刺参抗病网络调控机制提供基础数据。
1 材料与方法 1.1 实验材料本研究所用苗种取自山东青岛瑞滋集团有限公司培育的1龄刺参苗种。本研究选取活力良好、健康无异的刺参苗种,苗种规格为(50.0±2.0) g/只。实验用刺参苗种运回实验室后暂养3 d,暂养水温为(13.0± 0.5)℃,待苗种状态稳定后用于后续实验。
侵染菌株为本团队水生动物病原库中保存的分离自患腐皮综合征刺参体壁组织的灿烂弧菌菌株(AJ-Vb1801)。对该菌株用胰蛋白胨大豆肉汤固体培养基(TSB固体培养基)复苏,然后用TSB液体培养基扩大培养。
1.2 实验样品的采集灿烂弧菌人工侵染与取样:根据实验目的将实验分为2个组,每组设置3个平行,每个平行组中刺参苗种数量为30头,实验水槽容积为30 L。对照组(PT10H)刺参苗种使用取自水质良好的自然海域的海水进行健康养殖,侵染组(PT16S)按照水体体积在水槽中投放制备好的灿烂弧菌菌悬液至浓度为1×106 CFU/mL (该浓度为灿烂弧菌对刺参苗种的LD50)。2组实验在温度为(13.0±0.5)℃、盐度为(28.0±0.5)的条件下进行养殖,每天换1/3水,换水后及时添加制备好的灿烂弧菌菌悬液,使其维持在1×106 CFU/mL,每天投喂刺参配合饲料,投喂量为刺参苗种体重的2%,养殖期间保持溶氧充足。实验期间,每天观察刺参苗种的生理状态,及时收集并记录侵染组发生化皮的患病个体。实验开始7 d后,分别从侵染组的患病个体和对照组的健康个体中随机挑取3头刺参,剖取刺参样品的体壁组织,置于2 mL冻存管后迅速置于液氮中保藏并送回实验室,在–80℃超低温冰箱中保存,用于每个个体组织样品RNA的提取。对照组样品的编号分别为PT10H1、PT10H2和PT10H3,侵染组的编号分别为PT16S1、PT16S2和PT16S3。
1.3 总RNA提取及质量检测以PT10H组和PT16S组的刺参体壁组织为材料,采用QIAGEN RNeasy mini kit (74106)提取所采集样品的总RNA,以1.5%琼脂糖凝胶电泳检测总RNA的完整性,用NanoDrop核酸测定仪测定所提取核酸样品的纯度,检测合格的样品(浓度≥200 ng/μL; RIN≥8.0; 28S/18S≥1.5)于–80℃保存备用。
1.4 miRNA文库的建立对于所提取的总RNA样本,采用15% PAGE进行miRNA (18~30 nt)分离,分离后的miRNA经乙醇沉淀离心富集,然后利用TruSeq Small RNA Sample Preparation kit (Illumina, RS-200-0048)的方法及流程进行文库制备。文库构建完成后,先使用Qubit 2.0进行初步定量,稀释至1 ng/μL,随后使用Agilent 2100对文库的insert size进行检测,使用Bio-Rad CFX 96荧光定量PCR仪和Bio-Rad iQ SYBR Green进行实时荧光定量PCR (qRT-PCR),对文库的有效浓度进行准确定量(文库有效浓度 > 2 nmol/L)。检测合格后,将所构建的文库利用HiSeq2500测序平台进行单端测序(SE50),得到50 bp的序列reads。HiSeq深度测序由杭州联川生物技术股份有限公司完成。
1.5 miRNA测序数据处理原始数据经过质控处理后得到clean reads,去除3´接头,并进行长度筛选,保留碱基长度在18~26 nt的序列。再将剩余序列分别与mRNA数据库、Rfam数据库(包含rRNA、tRNA、snRNA、snoRNA等)和Repbase数据库进行比对和过滤,最后获得的数据即有效数据,用于后续miRNA数据分析。
1.6 miRNA鉴定和靶基因预测分析使用Bowtie 2 (2.2.3)将高质量的valid reads映射到本团队前期测序所得刺参基因组(组装完成,待发表),鉴定出相关miRNA。miRNA的差异表达分析使用edgeR软件,筛选标准是基因表达差异倍数 > 2且FDR≤0.05为差异显著的基因(DEmiRNA)。
使用TargetScan (Nam et al, 2014)、miRanda (Betel et al, 2008)软件对DEmiRNA分别进行靶基因预测。对2款软件预测出的靶基因分别按照每款软件的评分标准进行筛选。TargetScan算法中去除context score percentile < 50的靶基因,miRanda算法中去除最大自由能(max energy) > –10的靶基因。最后取2款软件的交集作为DEmiRNA的靶基因。
1.7 DEmiRNAs靶基因GO富集与KEGG通路分析利用GOseq R软件包进行DEmiRNAs靶基因的GO (gene ontology)富集分析(P < 0.05)。利用KOBAS软件对差异基因进行KEGG (Kyoto encyclopedia of genes and genomes; http://www.genome.jp/kegg/)信号通路富集分析。
1.8 qRT-PCR验证差异表达DEmiRNA表达量根据上述实验结果,随机选择9个DEmiRNA序列,并用加尾法设计引物(表 1)。参照Mir-XmiRNA first-strand synthesis试剂盒(TR201)说明书,将测序所用样本的总RNA反转录为cDNA。得到的cDNA保存于–20℃,用于后续荧光定量实验。qRT-PCR扩增体系为(20 μL):10 μL 2×miRNA qRT-PCR master mix (TaKaRa),上下游引物(10 μmol/L)各0.5 μL,cDNA 2 μL,RNase-free water定容至20 μL。以U6作为qRT-PCR的内参基因,每个样本3个重复,同时设置不加模板的阴性对照。采取2–ΔΔCt法计算所选DEmiRNAs的相对表达量。用3个重复实验的平均值±标准差(Mean±SD)来表示DEmiRNA的相对表达值。最后通过GraphPad 6软件对DEmiRNA的qRT-PCR结果和miRNA-seq结果进行绘图。
|
|
表 1 用于DEmiRNA表达验证的qRT-PCR引物 Tab.1 qRT-PCR primers used for validation of selected DEmiRNA |
根据DEmiRNAs与mRNAs的靶向结合关系,利用联川生物在线分析软件(https://www.omicstudio.cn/tool/56)构建基于KEGG分析得到的关键免疫途径的miRNA-mRNA调控网络。
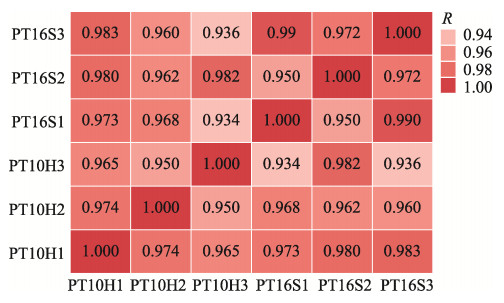
2 结果 2.1 刺参体壁样品miRNA测序数据结果PT10H和PT16S组刺参体壁样品的miRNA-seq分别平均产生11 929 100和10 107 557条原始序列,过滤后得到的平均有效序列分别为5 902 588和5 053 529条,说明本研究的测序数据质量良好。与miRbase数据库比对结果显示,PT10H比对到194个已知miRNA和19个新的miRNA,PT16S组比对到182个已知miRNA和42个新的miRNA (表 2)。对PT10H和PT16S组内各生物学重复之间的Pearson相关性系数分析结果显示,组内样品的相关性系数均在0.95以上,说明各样品的重复性良好(图 1)。
|
|
表 2 刺参体壁样品miRNA-seq数据概况 Tab.2 Overview of miRNA-seq data of body wall samples from A. japonicus |
|
图 1 miRNA测序结果的各样品间不同生物学重复的Pearson相关性分析 Fig.1 Pearson correlations between all the different biological repeats based on the miRNA-seq |
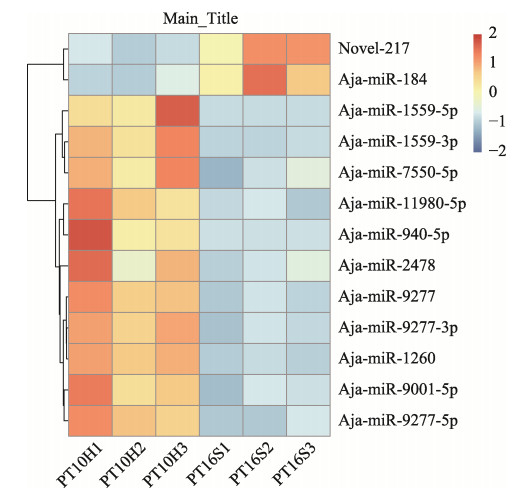
用Bowtie 2.2.3软件将长度筛选后的RNA定位到刺参基因组参考序列上,将定位成功的序列与Rfam database数据库进行比对,除去tRNAs、rRNAs、snRNA、snoRNAs和重复序列。利用miRDeep2软件共鉴定出PT10H与PT16S之间的差异表达miRNAs (P≤1) 299个,其中包括Aja-miR-2012、Aja-miR-92a和Aja-miR-286在内的127个miRNAs上调表达,Aja-miR-2007和Aja-miR-9277在内的172个miRNAs下调表达。进一步筛选出PT10H与PT16S之间显著性差异表达的miRNAs (DEmiRNAs) 13个,其中上调和下调DEmiRNAs的数量分别为2个和11个(表 3)。筛选出的13个DEmiRNAs中包含1个新的miRNA (Novel-217)和12个已知miRNAs。根据13个显著差异的DEmiRNAs在6个样品间的表达量构建聚类分析图(图 2),结果显示,Novel-217和Aja-miR-184上调表达幅度较大,Aja-miR-1559-5p、Aja-miR-940-5p和Aja-miR-1559-3p下调幅度较大。
|
|
表 3 PT10H与PT16S间差异表达miRNAs信息统计 Tab.3 Information of differentially expressed miRNAs between PT10H and PT16S |
|
图 2 PT10H与PT16S间差异表达miRNAs聚类分析热图 Fig.2 Cluster analysis heatmap of differentially expressed miRNAs between PT10H and PT16S |
采用TargetScan和miRanda软件对上述13个DEmiRNAs进行靶基因预测,共预测出22 082个靶基因。其中,上调的DEmiRNA共3010个靶基因,下调的DEmiRNA共19 072个靶基因。
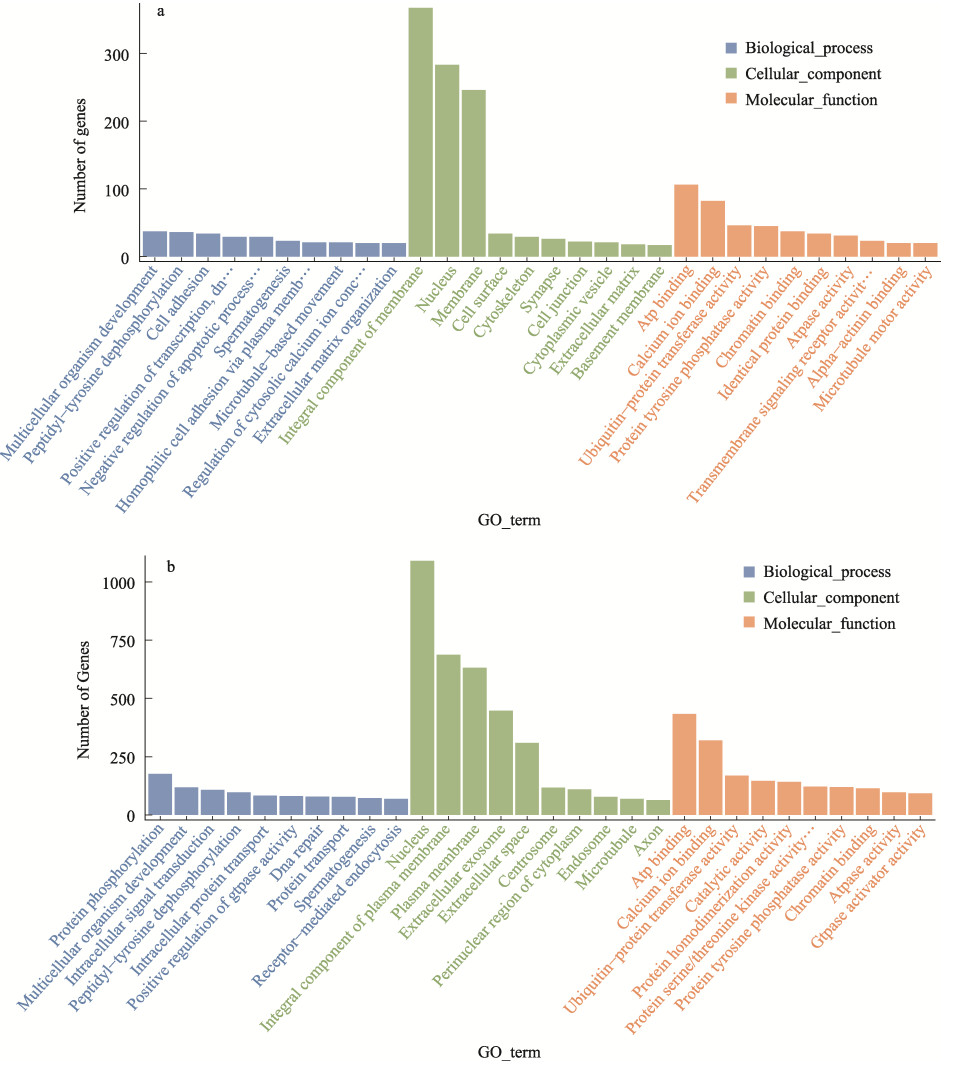
GO数据库注释结果显示,上调DEmiRNAs的2119个靶基因共注释到了585个GO terms (P≤0.05),包括生物进程368个terms,细胞成分79个terms,分子功能138个terms。分别选取3个本体显著性排名前10的GO terms构建GO富集柱状图(图 3a),结果显示,富集到生物进程的GO terms主要包括多细胞生物发育(GO: 0007275)、肽酰酪氨酸去磷酸化(GO: 0035335)和细胞粘附(GO: 0007155);富集到细胞成分的GO terms主要包括膜的组成部分(GO: 0016021)、核(GO: 0005634)和细胞膜(GO: 0016020);富集到分子功能的GO terms主要包括ATP结合(GO: 0005524)、钙离子结合(GO: 0005509)和泛素蛋白转移酶活性(GO: 0004842)。下调DEmiRNAs的8652个靶基因共注释到514个GO terms (P≤0.05),包括生物进程285个terms,细胞成分83个terms,分子功能146个terms。分别选取3个本体显著性排名前10的GO terms构建GO富集柱状图(图 3b)。结果显示,富集到生物进程的GO terms主要包括多蛋白质磷酸化(GO: 0006468)、细胞内信号转导(GO: 0035556)和多细胞生物发育(GO: 0007275);富集到细胞成分的GO terms主要包括膜的组成部分(GO: 0016021)、核(GO: 0005634)和质膜的组成部分(GO: 0005887);富集到分子功能的GO terms主要包括ATP结合(GO: 0005524)、钙离子结合(GO: 0005509)和泛素蛋白转移酶活性(GO: 0004842)。
|
图 3 PT10H与PT16S间DEmiRNAs靶基因GO富集 Fig.3 GO categorization of the target genes of DEmiRNAs between PT10H and PT16S a:上调DEmiRNAs靶基因;b:下调DEmiRNAs靶基因 a: Target genes of up-regulated DEmiRNAs; b: Target genes of down-regulated DEmiRNAs |
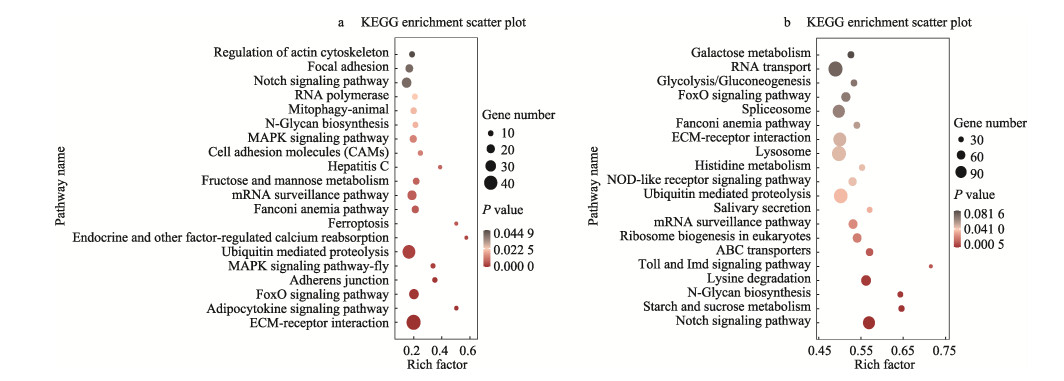
进一步对DEmiRNAs的靶基因进行KEGG通路富集分析,结果显示,上调DEmiRNAs的829个靶基因注释到24条代谢通路(P≤0.05),其中富集基因数较多的是ECM受体相互作用(43个)、泛素介导的蛋白水解(38个)、FoxO信号通路(26个)、Notch信号通路(26个)、mRNA监测通路(23个)、粘着力(18个)和MAPK信号通路(16个)(图 4a)。下调DEmiRNAs的3499个靶基因注释到22条代谢通路(P≤0.05),其中,富集基因数较多的是神经活性配体–受体相互作用(254个)、嘌呤代谢(141个)、泛素介导的蛋白水解(115个)、溶酶体(115个)、Notch信号通路(99个)和FoxO信号通路(66个)(图 4b)。
|
图 4 PT10H与PT16S间DEmiRNAs靶基因KEGG富集图 Fig.4 KEGG pathway of the target genes of DEmiRNAs between PT10H and PT16S a:上调DEmiRNAs靶基因;b:下调DEmiRNA靶基因 a: Target genes of up-regulated DEmiRNAs; b: Target genes of down-regulated DEmiRNAs |
为验证高通量测序数据的准确性,从筛选到的13个DEmiRNAs中随机选取10个进行qRT-PCR验证。结果显示,2个上调DEmiRNAs (Novel-217和Aja-miR-184)和5个下调DEmiRNAs (Aja-miR-2478、Aja-miR-9277、Aja-miR-1559-3p、Aja-miR-7550-5p和Aja-miR-11980-5p)与测序数据中相应的DEmiRNAs的表达量变化趋势一致(图 5),验证一致率达到70%,上述结果说明本研究的测序结果真实可靠。
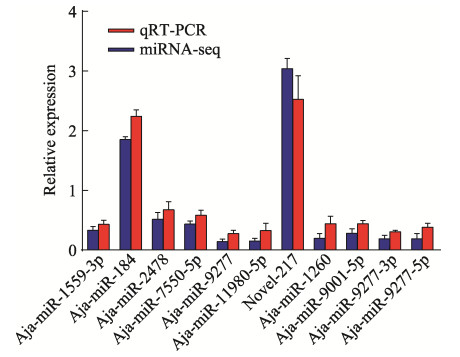
|
图 5 PT10H与PT16S组间刺参体壁组织DEmiRNAs的qRT-PCR验证 Fig.5 qRT-PCR validation of DEmiRNAs in body wall of A. japonicus between PT10H and PT16S |
根据KEGG富集分析结果,使用联川生物在线分析软件对泛素介导的蛋白水解和Notch信号通路上的DEmiRNAs及其靶基因构建miRNA-mRNA基因共表达网络。结果显示,13个DEmiRNAs共靶向结合115个与细胞免疫中泛素介导的蛋白水解相关的mRNAs (图 6a)。结合mRNAs较多的DEmiRNA为Aja-miR-9277-5p、Aja-miR-9277-3p和Aja-miR-2478,分别结合了31、24和23个mRNAs。其中,UBR5、UB2G1、UBE3C和XIAP作为关键基因靶向结合多个DEmiRNAs,可作为后续泛素介导的蛋白水解通路中的重点基因进行研究。共有108个与体液免疫中Notch信号通路相关的mRNAs被13个DEmiRNAs靶向结合(图 5b)。Aja-miR-9277、Aja-miR-9277-5p和Novel-217靶向结合的mRNAs最多,分别达到36、34和32个。其中,NOTC1、SVEP1和FBP1作为关键基因靶向结合多个DEmiRNAs,可作为后续Notch信号通路中的重点基因进行研究。

|
图 6 富集在刺参体壁免疫相关通路上的差异表达miRNA-mRNA的调控网络 Fig.6 Regulation networks of miRNA-mRNA enriched in immune-related pathways in the body wall of A. japonicus a:泛素介导蛋白水解;b:Notch信号通路 a: Ubiquitin-mediated proteolysis; b: Notch singling pathway |
目前,刺参腐皮综合征是对刺参养殖影响最大的疾病种类之一,本团队前期研究结果显示,灿烂弧菌是刺参腐皮综合征的重要致病原(张春云等, 2006)。虽然刺参腐皮综合症的致病原菌已被分离鉴定,但刺参响应该病原侵染的相关分子机制尚不清晰。Li等(2012)运用Illumina Hiseq2000平台对健康和患腐皮综合征的刺参体腔细胞进行深度测序后,在2个文库中找到了40个共同的保守miRNA;该研究还发现,miR-31和miR-2008在2个刺参文库中呈显著差异表达。Li等(2016)通过转染miR-210模拟物,证明miR-210能够抑制刺参AjToll基因,并且其ROS变化与AjToll基因的表达趋势一致,这意味着miR-210在一种新的免疫途径中起到了关键作用。Lv等(2017)研究发现,miR-137在刺参发病机制中具有调节免疫功能的作用。Lu等(2015a)发现,miR-31通过靶向作用于Ajp105来调节呼吸爆发。Shao等(2017)研究证明,在灿烂弧菌和刺参相互作用期间,miR-31负向调节肿瘤坏死因子的表达,使得半胱氨酸蛋白酶-3和半胱氨酸蛋白酶-8依赖性细胞凋亡。Lv等(2015)通过转染miR-200的模拟物验证其靶基因AjTollip下游分子的表达情况,表明miR-200能增强刺参体腔细胞的抗菌活性。Lu等(2015b)通过转染miR-133模拟物和si-AjIRAK-1,表明miR-133参与刺参TLR联级调节,并通过双荧光素酶报告基因实验确定了miR-133和AjIRAK-1的靶向关系。以上研究成果都是基于刺参体腔细胞测序,进而对差异miRNAs的表达及功能进行分析,本研究以刺参患病的病灶组织作为研究对象,通过高通量测序从转录组学角度筛选差异表达miRNAs,对其靶基因进行富集分析,可为刺参与病原菌互作关系和发病机制解析提供新思路。
miRNAs具有高度保守性、组织特异性和表达时序性。本研究通过生物信息学分析筛选到与免疫相关的3个DEmiRNAs,包括Aja-miR-184、Aja-miR-2478和Aja-miR-9277。在其他水产动物中,梁金荣(2017)研究发现,在经过副溶血弧菌(Vibrio parahaemolyticus)感染后,凡纳滨对虾(Litopenaeus vannamei)的肠道、心脏、肌肉等组织中miR-184的表达量显著升高,并进一步证实miR-184可以通过增强对虾体内抗菌肽的合成参与宿主的免疫调控,从而达到提高机体免疫活力的效果;Ou等(2013)研究发现,克氏原螯虾(Procambarus clarkia)的miR-184在受到蟹螺原体(Spiroplasma eriocheiris)刺激后表达量显著上调;Xu等(2019)研究发现,河南华溪蟹(Sinopotamon henanense)在应对镉(Cd)诱导的氧化应激反应中miR-184显著上调。同源miRNAs在不同水产动物中可能发挥类似的调控作用。本研究发现,刺参体壁在灿烂弧菌胁迫后Aja-miR-184表达量显著上调,推测刺参通过上调Aja-miR-184的表达来激活相关免疫通路,以应对灿烂弧菌的侵染。林锋(2018)从罗氏沼虾(Macrobrachium rosenbergii)野田村病毒(Macrobrachium rosenbergii nodavirus, MrNV)的致病性出发,确定了bta-miR-2478参与罗氏沼虾体液免疫,并可作为免疫增强剂在对虾野田村病毒的诊断和治疗上应用。林德麟(2017)在对牛乳乳清进行高通量测序后发现,miR-2478在乳清中显著表达,并靶向多种免疫球蛋白的合成。本研究发现,刺参体壁的Aja-miR-2478在灿烂弧菌胁迫后显著下调,表明Aja-miR-2478参与了刺参对灿烂弧菌的胁迫应答,推测Aja-miR-2478通过下调其表达量以解除对部分免疫相关基因的限制,从而增强刺参抵抗灿烂弧菌侵染的能力。相关结果表明,在水产动物中,Aja-miR-184和Aja-miR-2478在响应细菌侵染过程中有着相似的表达趋势。郭睿等(2019)研究发现,意大利蜜蜂(Apis mellifera ligustica)幼虫经蜜蜂球囊菌(Ascosphaera apis)胁迫后,肠道中miR-9277显著上调表达,其靶基因富集分析结果显示,miR-9277主要参与了丝氨酸蛋白酶和泛素介导的蛋白水解的调控。本研究发现,刺参体壁在灿烂弧菌胁迫后,Aja-miR-9277显著下调表达,并参与了促丝裂原活化蛋白激酶和泛素介导的蛋白水解的调控。然而,Aja-miR-9277与意大利蜜蜂中的同源miRNA表达趋势相反,推测同源miRNAs在不同物种中可能发挥不同的作用,但这种差异产生的分子机制仍需要进一步研究。
本研究中,筛选到的上调miRNA数目及其靶基因的数目都显著低于下调miRNA及其靶基因的数目,同时,靶基因的GO富集显示,靶基因主要富集在蛋白质磷酸化、膜的组成部分、泛素蛋白转移酶活性等GO terms上,表明刺参体壁的新陈代谢和细胞生命活动在受到灿烂弧菌胁迫后受到了一定程度的抑制。FOXO信号通路可以直接参与黄颡鱼(Pelteobagrus fulvidraco)胰岛素调控脂肪代谢的合成(卓梅琴, 2018),其下游的靶基因FOXO1对缢蛏(Sinonovacula constricta)的生长发育起着重要的调控作用(谢淑媚, 2018)。Toll和IMD信号通路在调控抗菌肽(antimicrobial peptides, AMPs)的表达中占主导地位(Vallet-Gely et al, 2008; Aggarwal et al, 2008)。真菌和革兰氏阳性菌的感染会激活Toll信号通路,从而调控一系列抗菌肽的表达;革兰氏阴性菌和病毒的感染会激活IMD信号通路,从而参与调控多种抗菌肽的表达(Lan et al, 2013)。促丝裂原活化蛋白激酶(mitogen-activated protein kinases, MAPKs)是介导细胞外信号到细胞内反应的重要信号转导系统,对细胞生长、分化、凋亡和程序性死亡等多种生理过程具有重要调控作用(Sigoillot et al, 2002)。Notch信号通路可调控细胞的分化、增殖和凋亡,对于细胞生长发育具有重要意义,Notch信号的异常表达会诱发乳腺癌等多种癌症(Shimizu et al, 2011)。本研究发现,在FOXO信号通路、Toll和IMD信号通路、MAPK信号通路和Notch信号通路分别富集了66、15、15和99个下调miRNAs的靶基因,推测这些下调miRNAs通过下调其表达量,以解除对部分免疫相关基因的抑制,从而增强刺参对灿烂弧菌的抵抗能力。同时,分别有38和26个上调miRNA的靶基因注释到了泛素介导的蛋白水解和Notch信号通路等细胞免疫相关通路,推测这些miRNA及其靶基因广泛参与了上述信号通路的负调控过程,从而使刺参体壁组织免疫活性和新陈代谢过程受到灿烂弧菌的抑制。He等(2014)对灿烂弧菌侵染的刺参体腔细胞的研究表明,miRNAs对刺参先天免疫防御的调控主要是通过调控刺参体内补体系统(complement system)和Toll样受体(Toll like receptor, TLR)信号通路中的相关因子而实现的。本研究中,TLR信号通路富集程度较高,但富集靶基因数较少,其中大部分靶基因也富集在泛素介导的蛋白水解通路和Notch信号通路,推测可能与所研究的样本组织差异相关,其具体的作用机制尚需进一步的研究。
本研究通过miRNA-mRNA共表达网络分析发现,泛素介导的蛋白水解是富集基因数最多的通路,13个DEmiRNAs在刺参响应灿烂弧菌胁迫过程中全部参与调控泛素介导的蛋白水解,其中11个下调的DEmiRNAs (Aja-miR-1559-3p、Aja-miR-7550-5p、Aja- miR-2478和Aja-miR-9277-3p等)靶向结合115个mRNAs,2个上调的DEmiRNAs (Aja-miR-184和Novel-217)靶向结合19个mRNAs;多数miRNAs下调表达以减弱对mRNAs的抑制作用,从而增强相关免疫基因的表达,少数miRNAs上调表达以增强对mRNAs的抑制。泛素介导的蛋白水解是重要的细胞免疫通路,可以通过降解关键的调节蛋白来控制细胞的基本生命活动,调节细胞应激反应和对病原微生物的免疫反应(陈科等, 2012),例如,在青虾(Macrobrachium nipponensis)遭受霍乱弧菌(Vibrio cholerae)侵染时,体内的泛素化蛋白可以介导机体细胞免疫以应对霍乱弧菌的侵染(李席席, 2020)。上述实验结果表明,灿烂弧菌在参与胁迫时对泛素介导的蛋白水解通路产生影响,二者之间存在复杂的互作,但背后的分子机制仍需进一步探明。
综上所述,本研究对正常及灿烂弧菌胁迫的刺参体壁样品进行高通量测序,筛选到13个与病原胁迫相关的DEmiRNA,并对其靶基因进行分析,进而对2条关键通路进行了基因共表达分析。研究结果不仅为刺参响应灿烂弧菌胁迫的关键调控miRNAs的筛选及功能研究提供了重要的信息和线索,也为刺参–灿烂弧菌互作机制的阐明打下了基础。
AGGARWAL K, SILVERMAN N. Positive and negative regulation of the Drosophila immune response. BMB Reports, 2008, 41(4): 267-277 DOI:10.5483/BMBRep.2008.41.4.267 |
BETEL D, WILSON M, GABOW A, et al. The microRNA. org resource: Targets and expression. Nucleic Acids Research, 2008, 36(suppl 1): D149-D153 |
BOONCHUEN P, MARALIT B A, JAREE P, et al. MicroRNA and mRNA interactions coordinate the immune response in non-lethal heat stressed Litopenaeus vannamei against AHPND-causing Vibrio parahaemolyticus. Scientific Reports, 2020, 10(1): 787 DOI:10.1038/s41598-019-57409-4 |
CHEN H, XIN L, SONG X, et al. A norepinephrine-responsive miRNA directly promotes CgHSP90AA1 expression in oyster haemocytes during desiccation. Fish and Shellfish Immunology, 2017, 64: 297-307 DOI:10.1016/j.fsi.2017.03.020 |
CHEN K, CHENG H H, ZHOU R J. Molecular mechanisms and functions of autophagy and the ubiq-uitin-proteasome pathway. Hereditas, 2012, 34(1): 7-20 [陈科, 程汉华, 周荣家. 自噬与泛素化蛋白降解途径的分子机制及其功能. 遗传, 2012, 34(1): 7-20 DOI:10.3868/j.issn.2095-1558.2012.01.003] |
GUO R, DU Y, ZHOU N H, et al. Comprehensive analysis of differentially expressed microRNAs and their target genes in the larval gut of Apis mellifera ligustica during the late stage of Ascosphaera apis stress. Acta Entomologica Sinica, 2019, 62(1): 49-60 [郭睿, 杜宇, 周倪虹, 等. 意大利蜜蜂幼虫肠道在球囊菌胁迫后期的差异表达微小RNA及其靶基因分析. 昆虫学报, 2019, 62(1): 49-60] |
HE X B, JING Z Z, CHENG G F. MicroRNAs: New regulators of toll-like receptor signalling pathways. BioMed Research International, 2014, 945169 |
KROL J, LOEDIGE I, FILIPOWICZ W. The widespread regulation of microRNA biogenesis, function and decay. Nature Reviews Genetics, 2010, 11(9): 597-610 DOI:10.1038/nrg2843 |
LAN J F, ZHOU J, ZHANG X W, et al. Characterization of an immune deficiency homolog (IMD) in shrimp (Fenneropenaeus chinensis) and crayfish (Procambarus clarkii). Developmental and Comparative Immunology, 2013, 41(4): 608-617 DOI:10.1016/j.dci.2013.07.004 |
LI C H, FENG W, QIU L, et al. Characterization of skin ulceration syndrome associated microRNAs in sea cucumber Apostichopus japonicus by deep sequencing. Fish and Shellfish Immunology, 2012, 33(2): 436-441 DOI:10.1016/j.fsi.2012.04.013 |
LI C, XU D X. Understanding microRNAs regulation in heat shock response in the sea cucumber Apostichopus japonicus. Fish and Shellfish Immunology, 2018, 81: 214-220 DOI:10.1016/j.fsi.2018.07.034 |
LI C, ZHAO M, ZHANG C, et al. miR210 modulates respiratory burst in Apostichopus japonicus coelomocytes via targeting toll-like receptor. Developmental and Comparative Immunology, 2016, 65: 377-381 DOI:10.1016/j.dci.2016.08.008 |
LI X X. Study on pathogenicity of non-O1 Vibrio cholerae to Macrobrachium nipponensis, host immune response and probiotic effect of antagonistic bacteria. Doctoral Dissertation of Yangzhou University, 2020 [李席席. 非O1霍乱弧菌对青虾的致病性、宿主的免疫反应及拮抗菌的益生效果研究. 扬州大学博士研究生学位论文, 2020]
|
LIANG J R. Cloning and functional analysis of immune related microRNAs in Litopenaeus vannamei. Doctoral Dissertation of Guangdong Ocean University, 2020 [梁金荣. 凡纳滨对虾免疫相关microRNA的克隆与功能分析. 广东海洋大学博士学位研究生学位论文, 2020]
|
LIN D L. Absorption of milk-derived miRNAs in animals and effects of milk-derived exosomes on mice immune function. Doctoral Dissertation of Southern China Agriculture University, 2017 [林德麟. 乳源miRNAs在动物体内的吸收规律及乳exosome对小鼠免疫功能的影响. 华南农业大学博士研究生学位论文, 2017]
|
LIN F. Study on the rapid detection technology and characterization of Macrobrachium rosenbergii nodavirus and extra small virus isolated in China. Doctoral Dissertation of Jiangsu University, 2018 [林锋. 罗氏沼虾野田村病毒致病性及其快速检测技术研究. 江苏大学博士研究生学位论文, 2018]
|
LU M, ZHANG P J, LI C H, et al. MiR-31 modulates coelomocytes ROS production via targeting p105 in Vibrio splendidus challenged sea cucumber Apostichopus japonicus in vitro and in vivo. Fish and Shellfish Immunology, 2015a, 45(2): 293-299 DOI:10.1016/j.fsi.2015.04.024 |
LU M, ZHANG P J, LI C H, et al. miRNA-133 augments coelomocyte phagocytosis in bacteria-challenged Apostichopus japonicus via targeting the TLR component of IRAK-1 in vitro and in vivo. Scientific Reports, 2015b, 5: 12608 DOI:10.1038/srep12608 |
LV M, CHEN H H, SHAO Y N, et al. miR-137 modulates coelomocyte apoptosis by targeting 14-3-3ζ in the sea cucumber Apostichopus japonicus. Developmental and Comparative Immunology, 2017, 67: 86-96 DOI:10.1016/j.dci.2016.11.008 |
LV Z, LI C H, ZHANG P J, et al. miR-200 modulates coelo-mocytes antibacterial activities and LPS priming via targeting Tollip in Apostichopus japonicus. Fish and Shellfish Immunology, 2015, 45(2): 431-436 DOI:10.1016/j.fsi.2015.04.014 |
NAM J W, RISSLAND O S, KOPPSTEIN D, et al. Global analyses of the effect of different cellular contexts on microRNA targeting. Molecular Cell, 2014, 53(6): 1031-1043 DOI:10.1016/j.molcel.2014.02.013 |
OU J, LI Y, DING Z F, et al. Transcriptome-wide identification and characterization of the Procambarus clarkii microRNAs potentially related to immunity against Spiroplasma eriocheiris infection. Fish and Shellfish Immunology, 2013, 35(2): 607-617 DOI:10.1016/j.fsi.2013.05.013 |
SHAO Y N, LI C H, XU W, et al. miR-31 links lipid metabolism and cell apoptosis in bacteria-challenged Apostichopus japonicus via targeting CTRP9. Front Immunology, 2017, 8: 263 |
SHIMIZU T, TANAKA T, ISO T, et al. Notch signaling pathway enhances bone morphogenetic protein 2 (BMP2) responsiveness of Msx2 gene to induce osteogenic differentiation and mineralization of vascular smooth muscle cells. Journal of Biological Chemistry, 2011, 286(21): 19138-19148 DOI:10.1074/jbc.M110.175786 |
SIGOILLOT F D, EVANS DR, GUY H I. Growth-dependent regulation of mammalian pyrimidine biosynthesis by the protein kinase A and MAPK signaling cascades. Journal of Biological Chemistry, 2002, 277(18): 15745-15751 DOI:10.1074/jbc.M201112200 |
VALLET-GELY I, LEMAITRE B, BOCCARD F. Bacterial strategies to overcome insect defences. Nature Reviews Microbiology, 2008, 6(4): 302-313 DOI:10.1038/nrmicro1870 |
XIE S M. Expression characteristics and preliminary functional analysis of three genes in the lin-like growth factor system of razor clam Sinonovacula constricta. Doctoral Dissertation of Shanghai Ocean University, 2018 [谢淑媚. 缢蛏类胰岛素生长因子系统3个基因表达特征及功能初步分析. 上海海洋大学博士研究生学位论文, 2018]
|
XU P, GUO H, WANG H, et al. Identification and profiling of microRNAs responsive to cadmium toxicity in hepatopancreas of the freshwater crab Sinopotamon henanense. Hereditas, 2019, 156(1): 34 DOI:10.1186/s41065-019-0110-z |
ZHANG C F, TONG C, TIAN F, et al. Integrated mRNA and microRNA transcriptome analyses reveal regulation of thermal acclimation in Gymnocypris przewalskii: A case study in Tibetan Schizothoracine fish. PLoS One, 2017, 12(10): e0186433 DOI:10.1371/journal.pone.0186433 |
ZHANG C Y, WANG Y G, RONG X J. Isolation and identification of causative pathogen for skin ulcerative syndrome in Apostichopus japonicus. Journal of Fisheries of China, 2006, 30(1): 118-123 [张春云, 王印庚, 荣小军. 养殖刺参腐皮综合征病原菌的分离与鉴定. 水产学报, 2006, 30(1): 118-123] |
ZHUO M Q. Functional analysis of PI3Ks and its related signal pathway in insulin regulating lipid metabolism in yellow catfish Pelteobagrus fulvidraco. Doctoral Dissertation of Huazhong Agricultural University, 2018 [卓梅琴. 黄颡鱼PI3Ks功能解析及其通路在胰岛素调控脂类代谢中的作用机制. 华中农业大学博士学位研究生学位论文, 2018]
|



