2. 中国水产科学研究院淡水渔业研究中心 农业农村部稻鱼综合养殖生态重点实验室 江苏 无锡 214081
2. Key Laboratory of Integrated Rice-Fish Farming Ecology, Ministry of Agriculture and Rural Affairs, Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, China
自噬(autophagy)是继细胞凋亡之后生命科学领域的另一个研究热点。自噬是细胞在自噬相关基因(autophagy-related gene, Atg)的调控下通过溶酶体降解受损细胞器和大分子的一种生物学过程。它在维持细胞内物质循环、细胞内环境稳态和机体动态平衡方面发挥着重要作用(Farre et al, 2009; Mehrpour et al, 2010)。自噬过程中形成的自噬体具有双层膜结构,这些双层膜的囊泡能包裹待降解的物质和受损细胞器,随即将它们运输至溶酶体中进行降解。自噬在消化降解时也为细胞内其他细胞器的组装提供原材料,即细胞内物质再循环(Green et al, 2014; Mizushima et al, 2011)。迄今为止,在酵母中已经确定多达40个自噬相关基因和50多个其他相关基因调节自噬过程中复杂的膜动态变化,这些基因中的大部分在哺乳动物中都存在相应的同源基因(Nakatogawa et al, 2009)。自噬相关蛋白所形成的不同功能的复合体与其他调控因子相互作用共同参与自噬起始、自噬体的形成、自噬体与溶酶体的相互融合等过程(Cohen-Kaplan et al, 2016; Ge et al, 2014; Mizushima, 2007)。
Atg2家族蛋白是较早被发现的自噬相关蛋白(Barth et al, 2001; Shintani et al, 2001),酵母中只有Atg2这一种类型,但在哺乳动物细胞中存在Atg2A和Atg2B两个亚型。氨基酸预测得,Atg2家族蛋白的N端结构与液泡分拣蛋白13 (vacuolar protein sorting 13, VPS13)类似,但其功能未知,并且Atg2家族蛋白的C端在该家族中有1个保守的ATG-C结构域(Munoz-Braceras et al, 2015; Pfisterer et al, 2014)。通过透射电镜(TEM)观察发现,哺乳动物敲除Atg2基因后导致细胞内的自噬小体前体膜和分隔膜不断积聚(Kishi-Itakura et al, 2014)。推测Atg2基因在哺乳动物和酵母细胞中的保守性功能极可能是促进自噬前体膜的组装与融合(Zheng et al, 2017)。但Atg2在甲壳动物中的作用却极少报道。
克氏原螯虾(Procambarus clarkii)作为我国重要的淡水养殖虾类之一(Huang et al, 2021),由于其巨大的经济价值,已成为我国主要的水产经济甲壳物种(周剑等, 2021)。近年来,克氏原螯虾市场需求日益扩大,养殖规模也在不断扩大,集约化养殖和管理不善导致病毒性疾病频频发生。其中,白斑综合征病毒(white spot syndrome virus, WSSV)是一种极具破坏性的病原体,WSSV引起的暴发性疾病,严重制约着克氏原鳌虾养殖业的健康发展。WSSV病毒具有极强毒性,增殖速度快且致死率极高(徐婷婷等, 2022),已成为全球流行最广、传播最快的病毒之一,且宿主涵盖范围非常广泛(史晓丽等, 2019)。多项研究结果显示,自噬在病毒侵染中发挥着重要的作用(Levine et al, 2008; Shintani et al, 2004; 康恺等, 2014)。病毒侵染可以刺激机体细胞发生自噬,研究表明,细胞自噬可直接包裹胞内的病毒并运送至溶酶体中将其降解;此外,自噬可将病毒核酸呈递给胞内感受器进而激活天然免疫,也可将病毒抗原呈递给主要组织相容性复合物以激活适应性免疫。
本研究通过RACE技术克隆克氏原螯虾Atg2基因(PcAtg2)的全长序列,对其进行组织分布特征与在WSSV胁迫下的表达模式分析,通过RNA干扰(RNAi)技术追踪WSSV病毒在克氏原螯虾体内增殖情况,进一步观察WSSV胁迫下克氏原螯虾肝胰腺中自噬小体的形成,期望探究克氏原螯虾在WSSV胁迫下PcAtg2的分子功能和作用,以期为克氏原螯虾自噬基因对WSSV胁迫的应答分子机制提供更多的数据参考。
1 材料与方法 1.1 实验材料实验所用克氏原螯虾取自于中国水产科学研究院淡水渔业研究中心扬中养殖基地。本研究中使用的所有实验方案均经南京农业大学动物保护与伦理委员会批准[许可号:SYXK (Su) 2011-0036]。选取体长为7~10 cm、体重为8~12 g的克氏原螯虾,在玻璃缸(97 cm× 48 cm×63 cm)内暂养1周,同时充分曝气[养殖水温为(25±2) ℃, pH为6.73±0.18]。1周后,随机选取健康完整的雌雄克氏原螯虾各3尾,分别取肝胰腺、肌肉、心脏、中肠、鳃、胃、腹神经节、血淋巴、卵巢、精巢、表皮和眼柄组织,液氮速冻后置于–80 ℃冰箱保存备用。主要涉及实验耗材及试剂盒:氯仿、异丙醇、无水乙醇、NaCl、TRIzol试剂、cDNA逆转录试剂盒、SYBR qPCR Mixture、DNase/RNase-Free Water、HiScript-TS 5´/3´RACE Kit、Gel Extraction Kit、pMD19-T载体、大肠杆菌(Escherichia coli) DH5α感受态细胞、琼脂糖凝胶回收试剂盒等。实验主要仪器:恒温培养摇床、高速冷冻离心机(Eppendorf, 德国)、凝胶成像仪Beck-man Coulter CEQ 8000 (Thermo, 美国)、微量光度分析仪NanoDrop 2000 (Thermo, 美国)、荧光定量PCR仪(Bio-Rad, 美国)和电泳仪等。
1.2 总RNA的提取与cDNA的合成将上述所取的克氏原螯虾组织置于液氮中充分研磨以便进行RNA提取。利用1%琼脂糖凝胶电泳测定RNA产物的完整性,再利用微量光度分析仪测量RNA浓度和纯度(OD260 nm和OD280 nm的比值)。参照逆转录试剂盒指导说明书进行总RNA的逆转录,并保存在–80 ℃下备用。
1.3 克氏原螯虾Atg2基因克隆以克氏原螯虾肝胰腺组织cDNA为模板,根据本实验室构建的克氏原螯虾转录组文库所获得的Atg2基因保守序列(Shen et al, 2020),利用Primer Premier 5.0软件分别为5´RACE和3´RACE设计特异性引物并合成(表 1)。参照5´/3´-RACE操作说明将克氏原螯虾肝胰腺总RNA反转录成cDNA第一链,以5´/3´- RACE cDNA,5´/3´GSP为引物,分别进行3´端和5´端序列的快速扩增。5´/3´RACE-PCR产物进行DNA胶回收,随后与pMD19-T载体连接,37 ℃连接4 h,然后转化DH5α感受态细胞,经PCR鉴定筛选后,随即进行阳性菌液测序及拼接测序,最后,将测序结果与Atg2基因序列比对,获得克氏原螯虾Atg2 (PcAtg2)基因cDNA全序列。
|
|
表 1 相关引物的序列信息 Tab.1 Sequences of primers used in the present study |
利用ORF Finder在线平台进行开放阅读框预测;利用DNAMAN软件进行氨基酸序列翻译;PcAtg2基因的保守结构域在NCBI中CD-search工具进行预测;利用SOPMA和SWISS-MOLDEL在线工具分别进行PcAtg2基因编码蛋白的结构预测。以NCBI中BLAST工具进行PcAtg2基因氨基酸同源性分析;再进行PcAtg2蛋白信号肽预测,通过在线平台PcAtg2基因磷酸化位点和糖基化位点,使用Compute pI/Mw计算其理论等电点(pI)和相对分子量;最后,利用DNAMAN软件及多重序列对比工具进行多物种的氨基酸序列比对,并以MEGA 7.0软件构建PcAtg2氨基酸系统发育进化树。
1.5 PcAtg2基因在组织中的表达分析为了确定PcAtg2在不同组织中的表达特异性,依据以上RACE实验得到的PcAtg2基因全序列设计并合成实时荧光定量PCR (real-time quantitative PCR, RT-qPCR)扩增引物。克氏原螯虾β-actin基因做为内参基因并设计引物进行RT-qPCR实验,且每个样品及内参均设置3个重复。根据2−∆∆Ct算法计算各样品中PcAtg2基因相对表达量(Schmittgen et al, 2008)。
1.6 WSSV胁迫实验随机挑选120尾健康克氏原螯虾,用于WSSV感染实验。将实验虾随机分为2组,每组3个重复,每个重复20尾,分别于虾体第2背节注射PBS和WSSV悬液。实验组虾注射100 μL WSSV悬液(1.0× 108 CFU/mL),对照组注射100 μL无菌磷酸盐(PBS, pH 7.4)。注射后0、6、12、24、48和96 h时随机选取健康完整的3尾虾,获取克氏原螯虾肝胰腺和肌肉组织,置于–80℃保存。用于刺激克氏原螯虾的WSSV病毒悬液由厦门大学水产学院实验室馈赠。
1.7 RNA干扰(RNA interference, RNAi)实验PcAtg2基因和绿色荧光蛋白(green fluorescent protein, GFP)基因的双链RNA (double-stranded RNA, dsRNA)的合成均按照T7 RNAi Transcription Kit说明书进行。注射前,将合成的dsPcAtg2和dsGFP稀释至1 μg/μL。取健康的克氏原螯虾[平均体重为(5.0± 1.0) g],以3~5 μg/g虾的比例注射dsRNA (Bai et al, 2015)。注射实验共4组,分别为PBS注射组、dsGFP注射组、dsPcAtg2注射组和不注射组。每组3个重复,每个重复30尾。分别于注射实验后24、48、72和96 h后各组中随机挑选健康克氏原螯虾6尾,取肝胰腺组织置于RNAlater溶液中,用于检测dsRNA注射后PcAtg2基因的干扰效率。在dsRNA注射48 h后进行WSSV胁迫实验,在注射WSSV的0、6、12、24、48和96 h分别取其肝胰腺组织置于–80℃保存。同时,收集克氏原螯虾新鲜肝胰腺组织,并于2.5%戊二醛中预固定24 h,在1% OsO4中固定1 h,4 ℃下保存24 h,利用日本日立H-7500型透射电镜仪器观察WSSV感染下的自噬体。
1.8 统计分析所有数据均以平均值±标准差(Mean+SD)表示(组间重复n=3),采用SPSS 20.0软件进行单因素方差显著分析(one-way ANOVA),显著水平设为P < 0.05和P < 0.01。采用多重比较检验组间差异(T检验/Duncan检验),利用GraphPad Prism 8.0软件进行图形绘制。
2 结果与分析 2.1 PcAtg2基因全长及cDNA序列分析以克氏原螯虾肝胰腺总RNA为模板所克隆得到的PcAtg2基因序列全长为9 966 bp (GenBank登录号MZ425926),包括6 567 bp的开放阅读框、582 bp的5´端非编码区(untranslated region, UTR)和2 817 bp的3´UTR,其中3´UTR具有加尾信号(AATAAA)和典型的Poly(A)尾结构。推测编码氨基酸2 189个,编码蛋白的相对分子量为242.59 kDa,理论等电点(pI)为5.38;在该氨基酸序列上预测到了65个丝氨酸磷酸化位点和48个糖基化位点,信号肽结构域位列于第1~70位氨基酸,ATG结构域位于2 093~2 178位氨基酸(图 1)。

|
图 1 PcAtg2基因全长cDNA序列及推导的氨基酸序列 Fig.1 Nucleotides and deduced amino acid sequences of the PcAtg2 cDNAs 起始密码子(ATG)和终止密码子(TAA)用黑色方框表示,浅灰色阴影区域为VPS13-N端结构域序列,深灰色阴影区域为VPS13-C端结构域序列,箭头方向表示ATG-C端结构域,*表示氨基酸的终止,下划线表示poly A加尾信号(AATAAA)。 Black panes indicate the start codon (ATG) and the stop codon (TAA). N-terminal region and C-terminal region of VPS13 are shaded by light gray and dark gray, arrow below the sequences represents the autophagy-related protein C terminal domain. * indicates the termination codon. The poly (A) special sequences AATAAA is underlined. |
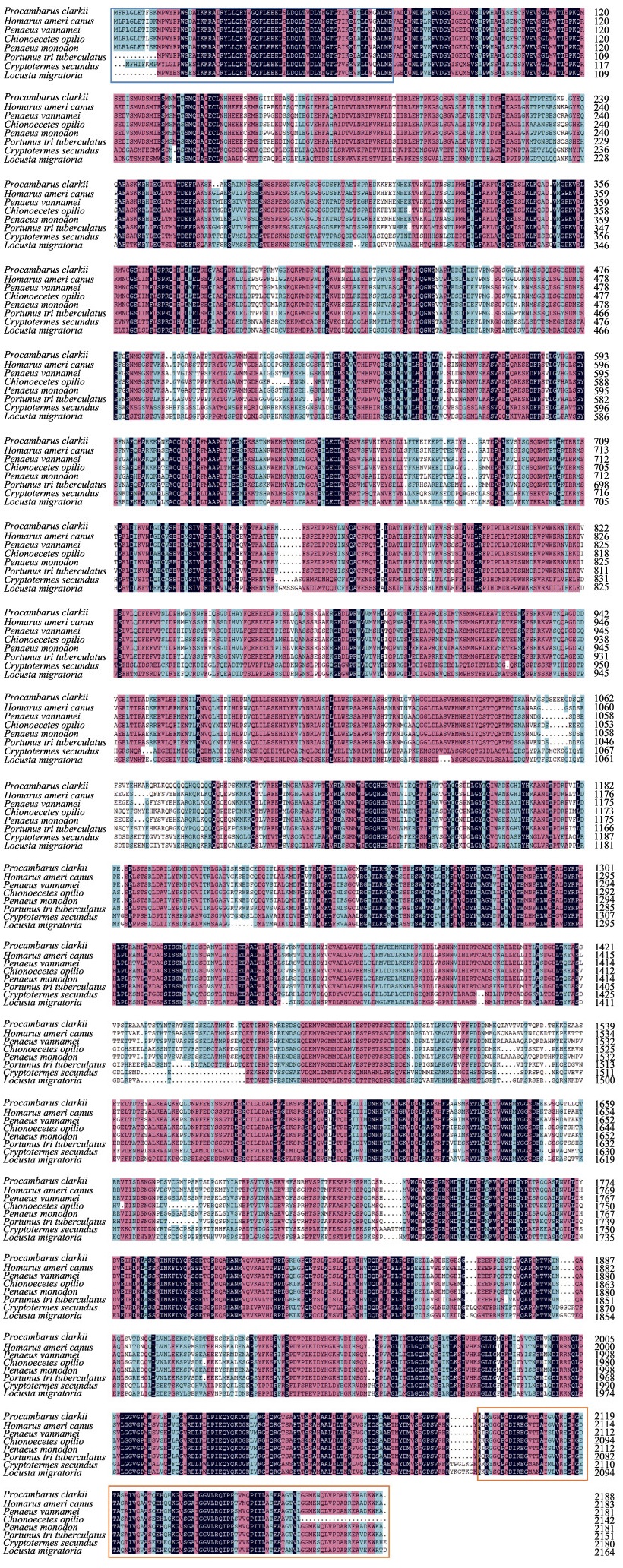
利用NCBI中BLASTp在线工具进行PcAtg2氨基酸序列的同源性分析。结果显示,PcAtg2与美洲螯龙虾(Homarus americanus)的Atg2氨基酸序列同源性最高(图 2),达88.26%,与凡纳对虾(Penaeus vannamei)、斑节对虾(P. monodon)、三疣梭子蟹(Portunus trituberculatus)、雪蟹(Chionoecetes opilio)、隐白蚁(Cryptotermes secundus)和东亚飞蝗(Locusta migratoria)的同源性分别为80.85%、80.67%、75.21%、74.60%、42.88%和43.37%。利用MEGA 7软件,基于邻接法构建了PcAtg2和其他物种的系统进化树(图 3A)。PcAtg2先与甲壳动物美洲螯龙虾聚为一支,随后与其他无脊椎动物聚为一类。
|
图 2 PcAtg2与其他已知物种Atg2氨基酸序列比对 Fig.2 Amino acid alignment of PcAtg2 with those of other known animals 基因登录号:克氏原螯虾,MZ425926;美洲螯龙虾,XP_042231353.1;凡纳对虾,XP_027223358.1;雪蟹,KAG0728919.1;斑节对虾,XP_037790247.1;三疣梭子蟹,MPC48873.1;隐白蚁,PNF21608.1;东亚飞蝗,ATX63054.1。红色方框表示信号肽序列,绿色方框为ATG结构域,下同。 Genbank accession number: P. clarkia, MZ425926; H. americanus, XP_042231353.1; P. vannamei, XP_027223358.1; C. opilio, KAG0728919.1; P. monodon, XP_037790247.1; P. trituberculatus, MPC48873.1; C. secundus, PNF21608.1; L. migratoria, ATX63054.1. The red box represents the signal peptide sequence, and the green box represents the ATG domain, the same below. |

|
图 3 PcAtg2氨基酸序列与其他物种Atg2氨基酸序列的系统发育进化树(A)和蛋白三级结构预测结果(B) Fig.3 Phylogenetic analysis of PcAtg2 amino acid sequences with other known species (A), and a tertiary structure prediction of PcAtg2 protein (B) |
PcAtg2蛋白二级结构预测结果显示,无规则卷曲占比最高(55.26%),其次是α-螺旋(26.97%)、延伸链(15.40%),β-折叠仅占2.38%。利用SWISS-MODEL在线工具构建了PcAtg2蛋白可能存在的三级结构(PDB DOI: 10.2210/pdb6A9J/pdb),结果如图 3B所示。
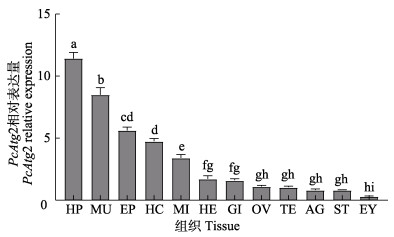
2.4 PcAtg2基因在各组织的表达情况采用RT-qPCR检测PcAtg2基因在肝胰腺、肌肉、心脏、中肠、鳃、胃、腹神经节、血淋巴、卵巢、精巢、表皮和眼柄组织中的表达情况。结果显示,PcAtg2基因在雌、雄性克氏原螯虾生殖器官组织中的相对表达量无显著差异,但在不同组织中的表达存在差异(P<0.05)。PcAtg2基因在肝胰腺组织中的相对表达量最高,显著高于其他组织,其次为肌肉和表皮,而在眼柄中的相对表达量最低(图 4)。
|
图 4 PcAtg2基因在不同组织中的表达 Fig.4 Expression of PcAtg2 gene in different tissues of P. clarkii HP:肝胰腺;MU:肌肉;EP:表皮;HC:血淋巴;MI:中肠;HE:心脏;GI:鳃;OV:卵巢;TE:精巢;AG:腹神经节;ST:胃;EY:眼柄。柱上字母不同表示组织间差异显著(P<0.05)。 HP: Hepatopancreas; MU: Muscle; EP: Epidermis; HC: Hemocyte; MI: Midgut; HE: Heart; GI: Gill; OV: Ovary; TE: Testis; AG: Abdominal ganglion; ST: Stomach; EY: Eyestalk. Different letters mean significant difference between different tissues (P<0.05). |
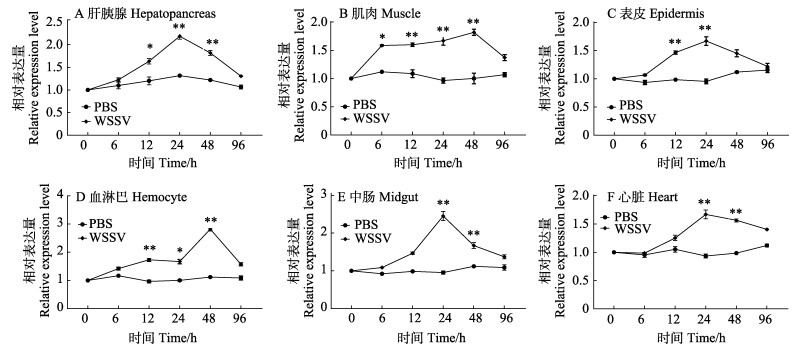
WSSV感染克氏原螯虾后的不同时间点,通过RT-qPCR检测了PcAtg2在克氏原螯虾肝胰腺、肌肉、表皮、血淋巴、中肠和心脏中的表达模式(图 5)。与PBS组相比,注射WSSV组的克氏原螯虾不同组织中PcAtg2的相对表达量呈先升高后降低趋势。PcAtg2在肝胰腺、表皮、中肠和心脏组织中的相对表达量在24 h达到峰值,然后恢复正常,而在肌肉和血淋巴组织中的相对表达量在48 h达到峰值,然后恢复正常水平。尽管在WSSV感染条件下,PcAtg2在不同组织中的表达模式不同,但均表现出一定程度的诱导作用。结果表明,PcAtg2在克氏原螯虾应对WSSV感染中发挥一定作用。
|
图 5 PcAtg2在WSSV感染克氏原螯虾各组织中的相对表达水平 Fig.5 Relative expression levels of PcAtg2 in tissues of P. clarkii after WSSV infection PBS注射组的表达模式作为对照。*表示组间差异显著(P<0.05), **表示组间差异极显著(P<0.01)。下同。 The expression pattern in the PBS injection group was used as a control. * indicated significant different with control group (P<0.05), ** indicated highly significant different with control group (P<0.01). The same as below. |
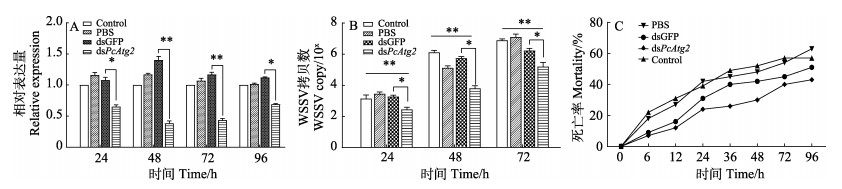
为验证dsRNA干扰效率,向克氏原螯虾注射体外合成的dsPcAtg2进行基因沉默。dsPcAtg2注射组在24、48、72和96 h的PcAtg2相对表达量分别仅为对照组(dsGFP注射组)的51.53%、26.61%、42.7%和62.73% (图 6A)。实验结果显示,RNAi有效抑制了PcAtg2基因的表达,并且在注射后48 h干扰效率达到最大值。
|
图 6 PcAtg2基因沉默和WSSV胁迫下的相关实验 Fig.6 Related experiments after PcAtg2 silencing and under WSSV stress A:注射dsPcAtg2的克氏原螯虾肝胰腺中PcAtg2基因的相对表达量;B:WSSV感染注射dsPcAtg2的克氏原螯虾后的增殖情况;C:WSSV胁迫下注射dsPcAtg2的克氏原螯虾的死亡率变化 A: Relative gene expression levels of PcAtg2 in hepatopancreas of P. clarkii after injection of dsPcAtg2; B: Proliferation of WSSV in the P. clarkii injected with dsPcAtg2 post WSSV challenging; C: Mortality changes of P. clarkii injected with dsPcAtg2 post WSSV challenging |
与对照组相比,dsPcAtg2注射组的WSSV拷贝数在WSSV胁迫后24—72 h出现上升趋势(图 6B),说明病毒增殖明显。然而,dsPcAtg2注射组的WSSV拷贝数显著低于对照组和dsGFP注射组,表明WSSV病毒复制在PcAtg2基因沉默期间受到一定程度的抑制。图 6C记录了WSSV感染期间每个实验组不同时间点的死亡率,其中PBS注射组总死亡率最高(63%),其次分别是对照组(57%)、dsGFP注射组(51%)和dsPcAtg2注射组(43%)。dsPcAtg2注射组的死亡率从24 h开始超过24%,低于dsGFP注射组(31%)、对照组(39%)和PBS注射组(63%)。dsPcAtg2注射组48 h的死亡率为30%,也低于dsGFP注射组(42%)、PBS注射组(48%)和对照组(52%)。死亡率结果显示,沉默PcAtg2基因能够降低WSSV对克氏原螯虾的致死率。
2.7 WSSV胁迫下克氏原螯虾肝胰腺超微结构分析为阐明WSSV胁迫对克氏原螯虾肝胰腺组织的影响,本研究检测了注射dsPcAtg2组在WSSV胁迫后克氏原螯虾肝胰腺组织超微结构的变化,采用透射电镜观察组织切片,在WSSV胁迫24和48 h后,对照组、dsPcAtg2注射组和dsGFP注射组克氏原螯虾肝胰腺组织中的溶酶体开始出现自噬空泡,细胞核附近溶酶体出现并聚集了更多自噬体(图 7),实验结果显示,克氏原螯虾在WSSV胁迫下能激活细胞自噬调节。其中,WSSV胁迫24和48 h后dsPcAtg2注射组的克氏原螯虾细胞中聚集的自噬体多于dsGFP注射组和对照组。在WSSV胁迫下,自噬标志基因(P62, LC3b, Atg5和Atg12)在克氏原螯虾肝胰腺组织中不同时间的表达情况见图 8。如图 8所示,dsPcAtg2注射组克氏原螯虾的LC3b、Atg5和Atg12基因相对表达量显著高于对照组。LC3b和Atg12基因相对表达量在48 h达到最大值,Atg5基因相对表达量在24 h时达到最大值。然而,P62基因的相对表达量显著低于对照组。
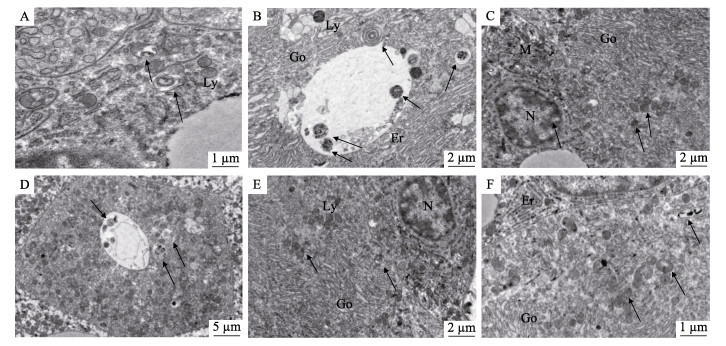
|
图 7 WSSV对克氏原螯虾肝胰腺组织超微结构的影响 Fig.7 Effects of WSSV on ultrastructure of hepatopancreas tissue of P. clarkia A~C分别为对照组、dsPcAtg2注射组、dsGFP注射组24 h的肝胰腺超微结构;D~F分别为对照组、dsPcAtg2注射组、dsGFP注射组48 h的肝胰腺超微结构M:线粒体;N:细胞核;Er:内质网;Ly:溶酶体;Go:高尔基体;箭头符号表示自噬小体。 A~C: Ultrastructure of hepatopancreas of control group, dsPcAtg2 injected group, and dsGFP injected group in 24 h; D~F: Ultrastructure of hepatopancreas of control group, dsPcAtg2 injected group, and dsGFP injected group in 48 h M: Mitochondria; N: Nucleus; Er: Endoplasmic reticulum; Ly: Lysosome, Go: Golgi apparatus; Arrow symbols indicate autophagosome. |
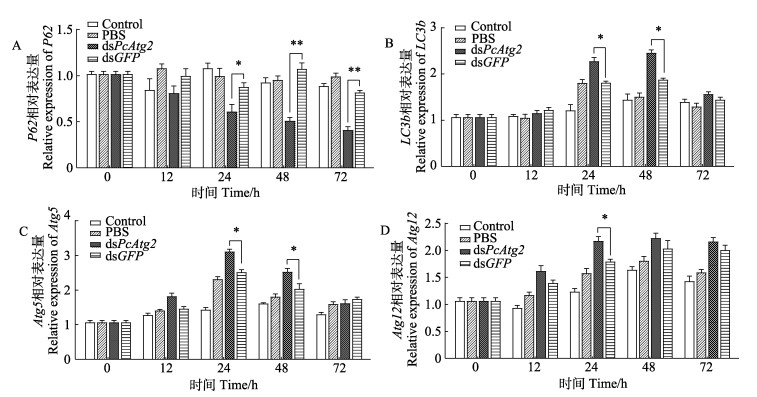
|
图 8 注射dsPcAtg2的克氏原螯虾感染WSSV后肝胰腺中自噬相关基因的相对表达量 Fig.8 Relative expression of autophagy related genes in hepatopancreas of dsPcAtg2 injected P. clarkii post WSSV challenging |
本研究利用RACE技术克隆得到了克氏原螯虾PcAtg2基因序列全长,氨基酸多序列比对结果显示,PcAtg2有65个丝氨酸磷酸化位点和48个糖基化位点,这些位点在众多生物反应中起到关键作用,如分子识别、信号转导和免疫调节等(范红弟等, 2020)。结构预测表明,PcAtg2含有ATG-C端和VPS13-N端和C端结构域,Atg2和VPS13的结构域具有相似的序列,推测Atg2可能也具有脂质转运活性(Saikat et al,2018)。Atg2至少可以使用2个独立的结合域与细胞膜相互作用(Kotani et al, 2018; Tamura et al, 2017)。一个膜结合结构域位于N端区域,与VPS13蛋白家族的N端区域具有弱序列同源性。虽然VPS13-N对于Atg2定位至自噬体前体不是至关重要的,但对于将Atg2固定到内质网非常重要(Kotani et al, 2018)。另一个膜结合域位于Atg2的C端,预计形成两亲性螺旋,对将Atg2靶向PAS至关重要(Kotani et al, 2018)。这2种膜结合结构域在细胞质到液泡的靶向与囊泡介导的分选以及细胞内蛋白运输中起作用(Leonzino et al, 2021)。系统进化树结果表明,PcAtg2基因与美洲螯龙虾氨基酸序列相似性最高,优先聚为一支,再与亲缘关系相近的虾蟹等甲壳类聚为一支,说明该基因在进化上相对保守。PcAtg2基因在克氏原螯虾不同组织分布研究发现,PcAtg2基因遍布所有组织,推测Atg2可能参与多种细胞代谢功能;然而,其在不同组织中的表达丰度不同,这也说明其具有组织偏好性,通常在免疫、发育组织中有较高的表达量。肝胰腺是甲壳动物重要的免疫器官,也是WSSV侵袭机体的重要靶器官,在调节生物体的代谢过程和免疫应答中起着重要作用(Shen et al, 2014; Jiao et al, 2019; Dai et al, 2017)。肝胰腺在甲壳动物物质代谢过程中发挥重要作用(Alexandre et al, 2013),在功能上与哺乳动物的肝脏和胰脏相类似(Röszer, 2014)。本研究中,PcAtg2在肝胰腺组织中的高表达也说明其在机体免疫反应中发挥重要作用。
目前已发现多种水生动物病毒感染也可引起细胞自噬,例如鲑传染性贫血病毒(infectious salmon anemia virus, ISAV)诱导细胞自噬,并能利用自噬进行复制(Schiotz et al, 2010)。而细胞自噬在牡蛎疱疹病毒(ostreid herpesvirus-1, OsHV-1)感染过程中发挥抗病毒作用(Moreau et al, 2015)。鲤春病毒血症病毒(spring viremiaof carp virus, SVCV)、乌鳢水泡病毒(snakehead vesiculovirus, SHVV)侵染细胞均能够诱发自噬活动,病毒与自噬的关系也因毒株和其靶细胞类型不同而存在差异(Liu et al, 2015; Wang et al, 2016)。近年来,越来越多的研究表明,Atgs在脊椎动物和无脊椎动物抵抗病原体感染的免疫反应中起着重要作用(Li et al, 2009; Richetta et al, 2013)。Atg1和Atg5沉默的果蝇(Drosophila)感染水泡性口炎病毒(vesicular stomatitis virus, VSV)后,实验组大量果蝇死亡,病毒浓度显著高于对照组,表明自噬在果蝇VSV感染中起着重要的拮抗作用(Shelly et al, 2009)。黄病毒科(Flaviviridae)的登革热病毒(dengue virus, DENV)可诱导自噬,促进自噬有助于病毒复制(Heaton et al, 2011)。柯萨奇病毒(Coxsackie virus, CV)中Atg7基因缺失后,病毒的复制水平显著降低,成熟病毒粒子的释放受到抑制(Meyers et al, 1998)。在新城疫病毒(Newcastle disease virus, NDV)感染胶质瘤细胞的实验中,发现用RNAi沉默Atg5和Beclin-1后,病毒浓度显著降低(Li et al, 2011)。斜带石斑鱼(Epinephelus coioides)感染红斑石斑鱼神经坏死病毒(red-spotted grouper nervous necrosis virus, RGNNV)可上调Atg6/Beclin-1的表达,斜带石斑鱼脾细胞中Atg6/Beclin-1的过度表达可通过调节干扰素和炎症反应促进病毒复制(Cai et al, 2019)。迄今为止,除了酵母、部分哺乳动物和真菌外,Atg2在甲壳动物的作用尚未见报道。此外,甲壳类动物自噬相关基因Atg2与病毒复制之间的潜在关系尚不清楚。
为揭示克氏原螯虾自噬相关基因PcAtg2及自噬在WSSV侵染中的作用,本研究检测了WSSV感染后不同时间PcAtg2基因相对表达量的变化,发现PcAtg2在WSSV胁迫下不同时间的表达各有差异,但PcAtg2基因的表达情况都出现先上调后下调的趋势,推测WSSV侵染调控或影响自噬相关基因PcAtg2及其参与的细胞自噬。病毒增殖可以利用细胞自噬,病毒不断增殖,调控自噬过程,使自噬水平上调,Atgs表达量出现上调(Sun et al, 2013)。为进一步探究PcAtg2基因在WSSV胁迫下起到的作用,本研究进行了RNAi实验。WSSV感染克氏原螯虾实验中dsPcAtg2注射组中的WSSV拷贝数在WSSV感染后24—72 h出现显著上升,说明病毒增殖明显。然而,WSSV拷贝数显著低于对照组和dsGFP注射组,表明WSSV病毒的复制在PcAtg2基因沉默期间受到一定程度的抑制。这也证明了PcAtg2基因是克氏原螯虾体内WSSV复制所必需的。一些病毒可以编码调节宿主因子的基因,从而干扰细胞自噬,延长受感染细胞的寿命,从而使病毒得以复制并持续感染(Clarke et al, 2003)。死亡率结果也表明,沉默PcAtg2基因能降低WSSV对克氏原螯虾的致死率。在本研究中,PcAtg2基因沉默后,通过透射电镜图像可以看出,对照组、dsPcAtg2注射组和dsGFP注射组的克氏原螯虾肝胰腺组织中的溶酶体开始出现自噬空泡,细胞核附近溶酶体出现并聚集了更多自噬体。实验结果显示,克氏原螯虾在WSSV胁迫下能激活细胞自噬调节。其中,WSSV胁迫24和48 h后注射dsPcAtg2组的克氏原螯虾细胞核附近聚集的自噬体多于注射dsGFP组和对照组。WSSV病毒侵染克氏原鳌虾细胞后,促使克氏原鳌虾细胞自噬增强,并利用细胞自噬促进自身增殖,通过自噬药物调控,发现WSSV病毒主要利用自噬体形成阶段,一旦形成自噬体,病毒增殖就会受到抑制(张广成等, 2020)。在WSSV胁迫下,自噬标志基因LC3b、Atg5和Atg12在克氏原螯虾肝胰腺组织中随着时间表现为上调表达模式,表明克氏原螯虾机体的自噬水平出现上调,然而P62基因的相对表达量趋于下调。LC3b是一种参与自噬体膜的延伸和成熟等过程的自噬相关因子,并与微管蛋白产生联系(Alemu et al, 2012; Nakatogawa et al, 2007),LC3b作为具有运输、调节、迁移等作用的微管蛋白,可能与克氏原螯虾体内的自噬体转运有关,这与红螯螯虾(Cherax quadricarinatus)的研究结论一致(孟凡娟等, 2019)。Atg5与Atg12会形成Atg5-Atg12复合物,在一些哺乳动物细胞中似乎所有Atg5和Atg12都表现为结合体的形式(Fan et al, 2016)。此外,Atg5-Atg12复合物还可以充当Atg8-磷脂酰乙醇胺(phosphatidylethanolamine, PE)结合系统的E3样连接酶。Atg5-Atg12的缺失会导致自噬缺陷(Hwang et al, 2012)。当PcAtg2基因沉默时,Atg5与Atg12相对表达量也出现上调趋势。说明,PcAtg2基因沉默能够上调自噬相关基因活性来参与机体自噬调节。然而,当PcAtg2基因沉默时,P62基因的相对表达上调。当自噬活性降低且自噬功能缺陷时,P62将继续在细胞质中积累。已知P62是自噬活性标志物,P62水平的增加通常被认为是自噬抑制的一个指标(Zhang et al, 2013)。研究发现,病毒增殖可以利用细胞自噬,细胞为避免病毒对其自身的利用,会相对下调自噬相关基因的表达,从而降低自噬水平。病毒侵染后期,病毒大量增殖,会反过来调控自噬过程,使自噬水平上调,自噬相关基因表达量上升(尹文娟等, 2021)。本研究中,沉默PcAtg2基因可能通过上调自噬相关基因表达来促进自噬小体的形成进而参与机体的自噬调节。PcAtg2通过抑制自噬来促进WSSV病毒复制,PcAtg2基因在克氏原螯虾抗病毒免疫防御中发挥重要作用。
综上所述,本研究通过RACE技术克隆获得了PcAtg2基因cDNA全长序列。同源性比对结果显示,该基因在进化上与甲壳动物美洲螯龙虾的同源性最高。PcAtg2基因遍布克氏原螯虾的各组织,且在肝胰腺组织中丰度最高。沉默PcAtg2基因可能通过上调自噬相关基因表达来促进自噬小体的形成,进而参与机体的自噬调节。PcAtg2可能通过抑制自噬来促进WSSV病毒复制,PcAtg2基因在克氏原螯虾抗病毒免疫防御中发挥重要作用。在细胞自噬调控的宿主防御机制开展更深一步的研究将有助于提供新的抗病毒策略。
ALEMU E A, LAMARK T, TORGERSEN K M, et al. ATG8 family proteins act as scaffolds for assembly of the ULK complex: Sequence requirements for LC3-interacting region (LIR) motifs. Journal of Biological Chemistry, 2012, 287(47): 39275-39290 DOI:10.1074/jbc.M112.378109 |
Alexandre V B L, GUERRA A L, Eduardo A D A, et al. Characterization of esterase patterns in hepatopancreas of three species of Macrobrachium (Palaemonidae). Biochemical Systematics and Ecology, 2013, 47(7): 132-138 |
BAI H K, QIAO H, LI F J, et al. Molecular characterization and developmental expression of vitellogenin in the oriental river prawn Macrobrachium nipponense and the effects of RNA interference and eyestalk ablation on ovarian maturation. Gene, 2015, 562(1): 22-31 DOI:10.1016/j.gene.2014.12.008 |
BARTH H, THUMM M. A genomic screen identifies AUT8 as a novel gene essential for autophagy in the yeast Saccharomyces cerevisiae. Gene, 2001, 274(1/2): 151-156 |
CAI J, ZOU Z H, WEI S N, et al. Identification of Beclin-1 from orange-spotted grouper (Epinephelus coioides) involved in viral infection. Fish and Shellfish Immunology, 2019, 94: 336-345 DOI:10.1016/j.fsi.2019.09.029 |
CLARKE T E, CLEM R J. Insect defenses against virus infection: The role of apoptosis. International Reviews of Immunology, 2003, 22(5/6): 401-424 |
COHEN-KAPLAN V, LIVNEH I, AVNI N, et al. The ubiquitin-proteasome system and autophagy: Coordinated and independent activities. International Journal of Biochemistry and Cell Biology, 2016, 79: 403-418 DOI:10.1016/j.biocel.2016.07.019 |
DAI L S, ABBAS M N, KAUSAR S, et al. Transcriptome analysis of hepatopancraes of Procambarus clarkii challenged with polyriboinosinic polyribocytidylic acid (poly I: C). Fish and Shellfish Immunology, 2017, 71: 144-150 DOI:10.1016/j.fsi.2017.10.010 |
FAN H D, LI Y D, YANG Q B, et al. Characterization and expression analysis of MKK7 in black tiger shrimp (Penaeus monodon) under different stressors. Journal of Fishery Sciences of China, 2020, 27(7): 748-758 [范红弟, 李运东, 杨其彬, 等. 斑节对虾MKK7基因的克隆及在不同胁迫条件下的表达分析. 中国水产科学, 2020, 27(7): 748-758] |
FAN X X, GAO Y, SHU L, et al. Transcriptome profiling indicating canine parvovirus type 2a as a potential immune activator. Virus Genes, 2016, 52(6): 1-12 |
FARRE J C, KRICK R, SUBRAMANI S, et al. Turnover of organelles by autophagy in yeast. Current Opinion in Cell Biology, 2009, 21(4): 522-530 DOI:10.1016/j.ceb.2009.04.015 |
GE L, BASKARAN S, SCHEKMAN R, et al. The protein-vesicle network of autophagy. Current Opinion in Cell Biology, 2014, 29(1): 18-24 |
GREEN D R, LEVINE B. To be or not to be? How selective autophagy and cell death govern cell fate. Cell, 2014, 157(1): 65-75 DOI:10.1016/j.cell.2014.02.049 |
HEATON N S, RANDALL G. Dengue virus and autophagy. Viruses, 2011, 3(8): 1332-1341 DOI:10.3390/v3081332 |
HUANG P D, SHEN G Q, GONG J, et al. A novel Dicistro-like virus discovered in Procambarus clarkii with "Black May" disease. Journal of Fish Diseases, 2021, 44(6): 803-811 DOI:10.1111/jfd.13309 |
HWANG S, MALONEY N S, BRUINSMA M W, et al. Nondegradative role of Atg5-Atg12/Atg16L1 autophagy protein complex in antiviral activity of interferon gamma. Cell Host and Microbe, 2012, 11(4): 397-409 DOI:10.1016/j.chom.2012.03.002 |
JIAO T, CHU X H, GAO Z Q, et al. New insight into the molecular basis of Fe (Ⅲ) stress responses of Procambarus clarkii by transcriptome analysis. Ecotoxicology and Environmental Safety, 2019, 182: 109388 DOI:10.1016/j.ecoenv.2019.109388 |
KANG K, LIN L, GAO H H, et al. Classical swine fever virus promotes cell autophagy which facilitates virus proliferation. Acta Veterinaria et Zootechnica Sinica, 2014, 45(9): 1481-1487 [康恺, 林鸷, 高海慧, 等. 猪瘟病毒促进细胞自噬并利于病毒增殖. 畜牧兽医学报, 2014, 45(9): 1481-1487] |
KISHI-ITAKURA C, KOYAMA-HONDA I, ITAKURA E, et al. Ultrastructural analysis of autophagosome organization using mammalian autophagy-deficient cells. Journal of Cell Science, 2014, 127(18): 4089-4102 |
KOTANI T, KIRISAKO H, KOIZUMI M, et al. The Atg2-Atg18 complex tethers pre-autophagosomal membranes to the endoplasmic reticulum for autophagosome formation. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(41): 10363-10368 |
LEONZINO M, REINISCH K M, De CAMILLI P. Insights into VPS13 properties and function reveal a new mechanism of eukaryotic lipid transport. Biochimica et Biophysica Acta-Molecular and Cell Biology of Lipids, 2021, 1866(10): 159003 DOI:10.1016/j.bbalip.2021.159003 |
LEVINE B, KROEMER G. Autophagy in the pathogenesis of disease. Cell, 2008, 132(1): 27-42 DOI:10.1016/j.cell.2007.12.018 |
LI B X, LI C Y, PENG R Q, et al. The expression of beclin-1 is associated with favorable prognosis in stage ⅢB colon cancers. Autophagy, 2009, 5(3): 303-306 DOI:10.4161/auto.5.3.7491 |
LI J H, LIU Y H, WANG Z K, et al. Subversion of cellular autophagy machinery by hepatitis B virus for viral envelopment. Journal of Virology, 2011, 85(13): 6319-6333 DOI:10.1128/JVI.02627-10 |
LIU L Y, ZHU B B, WU S S, et al. Spring viraemia of carp virus induces autophagy for necessary viral replication. Cellular Microbiology, 2015, 17(4): 595-605 DOI:10.1111/cmi.12387 |
MEHRPOUR M, ESCLATINE A, BEAU I, et al. Overview of macroautophagy regulation in mammalian cells. Cell Research, 2010, 20(7): 748-762 DOI:10.1038/cr.2010.82 |
MENG F J, ZHANG G C, YIN W J, et al. Study on interaction of autophagosome and microtubule during autophagy process in Cherax quadricarinatus. Journal of Fisheries of China, 2019, 43(12): 2494-2500 [孟凡娟, 张广成, 尹文娟, 等. 红螯光壳螯虾细胞自噬过程中自噬体与微管相互作用. 水产学报, 2019, 43(12): 2494-2500 DOI:10.11964/jfc.20180811406] |
MEYERS G, STOLL D, GUNN M. Insertion of a sequence encoding light chain 3 of microtubule-associated proteins 1A and 1B in a pestivirus genome: Connection with virus cytopathogenicity and induction of lethal disease in cattle. Journal of Virology, 1998, 72(5): 4139-4148 DOI:10.1128/JVI.72.5.4139-4148.1998 |
MIZUSHIMA N, KOMATSU M. Autophagy: Renovation of cells and tissues. Cell, 2011, 147: 728-741 DOI:10.1016/j.cell.2011.10.026 |
MIZUSHIMA N. Autophagy: Process and function. Genes Development, 2007, 21(22): 2861-2873 DOI:10.1101/gad.1599207 |
MOREAU P, MOREAU K, SEGARRA A, et al. Autophagy plays an important role in protecting Pacific oysters from OsHV-1 and Vibrio aestuarianus infections. Autophagy, 2015, 11(3): 516-526 DOI:10.1080/15548627.2015.1017188 |
MUNOZ-BRACERAS S, CALVO R, ESCALANTE R. TipC and the chorea-acanthocytosis protein VPS13A regulate autophagy in Dictyostelium and human HeLa cells. Autophagy, 2015, 11(6): 918-927 DOI:10.1080/15548627.2015.1034413 |
NAKATOGAWA H, ICHIMURA Y, OHSUMI Y. ATG8, a ubiquitin-like protein required for autophagosome formation, mediates membrane tethering and hemifusion. Cell, 2007, 130(1): 165-178 DOI:10.1016/j.cell.2007.05.021 |
NAKATOGAWA H, SUZUKI K, KAMADA Y, et al. Dynamics and diversity in autophagy mechanisms: Lessons from yeast. Nature Reviews Molecular Cell Biology, 2009, 10: 458-467 DOI:10.1038/nrm2708 |
PFISTERER S B G, BAKULA D, FRICKEY T, et al. Lipid droplet and early autophagosomal membrane targeting of Atg2A and Atg14L in human tumor cells. Journal of Lipid Research, 2014, 55(7): 1267-1278 DOI:10.1194/jlr.M046359 |
RICHETTA C, FAURE M. Autophagy in antiviral innate immunity. Cellular Microbiology, 2013, 15(3): 368-376 DOI:10.1111/cmi.12043 |
RÖSZER T. The invertebrate mid-intestinal gland ("hepatopancreas") is an evolutionary forerunner in the integration of immunity and metabolism. Cell and Tissue Research, 2014, 358(3): 685-695 DOI:10.1007/s00441-014-1985-7 |
SAIKAT C, CHINATSU O, ALEXANDER L, et al. Insights into autophagosome biogenesis from structural and biochemical analyses of the ATG2A-WIPI4 complex. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(42): E9792-E9801 |
SCHIOTZ B L, ROOS N, RISHOVD A L, et al. Formation of autophagosomes and redistribution of LC3 upon in vitro infection with infectious salmon anemia virus. Virus Research, 2010, 151(1): 104-107 DOI:10.1016/j.virusres.2010.03.013 |
SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative CT method. Nature Protocols, 2008, 3(6): 1101-1108 DOI:10.1038/nprot.2008.73 |
SHELLY S, LUKINOVA N, BAMBINA S, et al. Autophagy is an essential component of Drosophila immunity against vesicular stomatitis virus. Immunity, 2009, 30(4): 588-598 DOI:10.1016/j.immuni.2009.02.009 |
SHEN G Q, ZHANG X, GONG J, et al. Transcriptomic analysis of Procambarus clarkii affected by "Black May" disease. Scientific Reports, 2020, 10(1): 21225 DOI:10.1038/s41598-020-78191-8 |
SHEN H S, HU Y C, MA Y C, et al. In-depth transcriptome analysis of the red swamp crayfish Procambarus clarkii. PLoS One, 2014, 9(10): e110548 DOI:10.1371/journal.pone.0110548 |
SHI X L, ZHANG Y X, MENG X H, et al. cDNA cloning of the GLDC gene in Fenneropenaeus chinensis and its expression and functional analyses after WSSV infection. Progress in Fishery Sciences, 2019, 40(2): 74-82 [史晓丽, 张莹雪, 孟宪红, 等. 中国对虾甘氨酸脱羧酶的基因克隆、表达及其与抗WSSV的关联分析. 渔业科学进展, 2019, 40(2): 74-82 DOI:10.19663/j.issn2095-9869.20171219002] |
SHINTANI T, KLIONSKY D J. Autophagy in health and disease: A double-edged sword. Science, 2004, 306(5698): 990-995 DOI:10.1126/science.1099993 |
SHINTANI T, SUZUKI K, KAMADA Y, et al. Apg2p functions in autophagosome formation on the perivacuolar structure. Journal of Biological Chemistry, 2001, 276(32): 30452-30460 DOI:10.1074/jbc.M102346200 |
SUN Y M, LI F H, XIANG J H. Analysis on the dynamic changes of the amount of WSSV in Chinese shrimp Fenneropenaeus chinensis during infection. Aquaculture, 2013, 376/379: 124-132 DOI:10.1016/j.aquaculture.2012.11.014 |
TAMURA N, NISHIMURA T, SAKAMAKI Y, et al. Differential requirement for ATG2A domains for localization to autophagic membranes and lipid droplets. FEBS Letters, 2017, 591(23): 3819-3830 DOI:10.1002/1873-3468.12901 |
WANG Y, CHEN N, HEGAZY A M, et al. Autophagy induced by snakehead fish vesiculovirus inhibited its replication in SSN-1 cell line. Fish and Shellfish Immunology, 2016, 55: 415-422 DOI:10.1016/j.fsi.2016.06.019 |
XU T T, TENG G L, LI Y X, et al. Analysis of the effect of white spot syndrome virus (WSSV) on shrimp biomass in the Yellow Sea and the Northern East China Sea based on GFM and GAMM models. Progress in Fishery Sciences, 2022, 43(1): 46-55 [徐婷婷, 滕广亮, 李英瑕, 等. 基于GFM和GAMM模型分析对虾白斑综合征(WSSV)对黄海和东海北部水域虾类生物量的影响. 渔业科学进展, 2022, 43(1): 46-55] |
YIN W J, LIU F, MENG F J, et al. Cloning of ATG3 in Litopenaeus vannamei and research on its expression under WSSV infection. Journal of Tianjin Normal University (Natural Science Edition), 2021, 41(2): 51-55 [尹文娟, 刘芳, 孟凡娟, 等. 凡纳滨对虾(Litopenaeus vannamei) ATG3基因克隆及其在WSSV感染中的表达. 天津师范大学学报(自然科学版), 2021, 41(2): 51-55] |
ZHANG G C, YIN W J, LIU F, et al. Cloning and expression analysis of shrimp peritrophin-like gene during WSSV infection in Fenneropenaeus chinensis. Journal of Tianjin Normal University (Natural Science Edition), 2020, 40(1): 28-33 [张广成, 尹文娟, 刘芳, 等. 中国明对虾(Fenneropenaeus chinensis)类围食膜蛋白基因克隆及其在WSSV感染中的表达. 天津师范大学学报(自然科学版), 2020, 40(1): 28-33] |
ZHANG Y B, GONG J L, XING T Y, et al. Autophagy protein p62/SQSTM1 is involved in hamlet-induced cell death by modulating apotosis in U87MG cells. Cell Death and Disease, 2013, 4(3): e550 DOI:10.1038/cddis.2013.77 |
ZHENG J X, LI Y, DING Y H, et al. Architecture of the ATG2B-WDR45 complex and an aromatic Y/HF motif crucial for complex formation. Autophagy, 2017, 13(11): 1870-1883 DOI:10.1080/15548627.2017.1359381 |
ZHOU J, ZHAO Z M, HUANG Z P, et al. Comparison of nutrient components in muscles and hepatopancreas of pond-and paddy field-cultured Procambarus clarkii. Progress in Fishery Sciences, 2021, 42(2): 162-169 [周剑, 赵仲孟, 黄志鹏, 等. 池塘和稻田养殖模式下克氏原螯虾肌肉和肝脏营养成分比较. 渔业科学进展, 2021, 42(2): 162-169 DOI:10.19663/j.issn2095-9869.20191231001] |



