体色不仅是一种重要的表型性状,在动物的伪装、物种识别、配偶选择和紫外线防护等方面具有重要作用,而且是决定商品动物市场价值的一个重要质量标准(Tian et al, 2018)。皮肤色素沉着模式有着物种特异性特征,其取决于表皮和真皮层中色素细胞的类型、数量以及空间组合(蒋燕玲等, 2016)。与哺乳动物和两栖动物不同,在鱼类中已发现6种色素细胞(Wang et al, 2014),其中,黑色素细胞、黄色素细胞、红色素细胞和虹彩细胞广泛分布于硬骨鱼类中,如斑马鱼(Danio rerio) (Nagao et al, 2018)、橘色双冠丽鱼(Amphilophus citrinellus) (韦敏侠等, 2015)和曼龙鱼(Trichogaster trichopterus) (李小兵等, 2012),而白色素细胞和蓝色素细胞的分布较为有限。由于鱼类色素细胞的多样性,体色已成为高产与优质育种的关键性状。虽然鱼类体色的形成受生活环境、营养和生理因素的影响,但色素细胞的分化与发育主要受遗传因素的调控(Zhang et al, 2017)。与其他脊椎动物相比,研究发现,鱼类丰富的体色是由于特有基因组复制事件增加了30%色素相关基因所致,且色素相关基因的种类与表达差异是形成体色多样性的主要原因(Braasch et al, 2009; Xing et al, 2018)。因此,研究色素相关基因的差异表达有助于揭示鱼类体色差异的内在机理。
由于有着重要的生物学与医学意义,黑色素生物合成的分子机制已被深入研究,并阐明了脊椎动物中保守的黑色素形成途径(Zou et al, 2015)。黑色素是在黑色素小体的淀粉纤维样结构中合成,主要分真黑色素(黑色和褐色)和褐黑色素(黄色和红色),黑色素的沉积是由二者的比例和分布决定的(姚文成, 2018; Lamoreux et al, 2011)。黑色素的合成是一个复杂的色素调控系统,由多个基因位点编码产物共同调控(郑嫩珠等, 2015)。在脊椎动物中,已发现超过250种基因影响黑色素的形成,这些基因通过调控黑色素细胞的形成、迁移、增殖与基因转录等方式来调节皮肤黑色素沉积(Fontanesi et al, 2010)。前黑素小体蛋白(premelanosome protein, Pmel),又称Silv,是一种在内质网合成的跨膜运输糖蛋白,与黑色素小体的成熟有关,在黑色素形成和沉淀中发挥重要作用(潘家华等, 2020)。在黑色素合成信号通路中,pmel基因位于酪氨酸基因家族的下游,它不仅能催化5, 6-吲哚醌羧酸转化为真黑色素,而且当其被转运到黑色素小体内时,该蛋白裂解后聚集形成淀粉样纤维板结构以维持黑色素小体的正常形态并提供真黑色素附着场所,从而保障真黑色素的顺利合成(Jiang et al, 2014; 王磊等, 2019)。目前,对pmel基因的研究大多集中在哺乳动物和家禽中。在小鼠(Mus musculus)中发现,pmel基因是皮肤黑色素细胞和视网膜色素上皮细胞中正常黑色素小体发育所必需的,该基因的灭活导致表皮真黑色素沉积大量减少(Baxter et al, 2003)。Brunberg等(2006)和Kerje等(2004)研究表明,pmel基因突变分别抑制了马(Equus caballus)和鸡(Gallus gallus)毛发和皮肤中真黑色素的产生,导致表型颜色变淡。在鱼类中,Wang等(2022)在尼罗罗非鱼(Oreochromis niloticus)中发现,敲除pmela或pmelb基因都显著减少了皮肤黑素细胞的数量和大小,使鱼体颜色黄化;Schonthaler等(2005)在斑马鱼中也发现,敲除pmel基因会导致柱状黑素小体数量显著减少。另外,在半滑舌鳎(Cynoglossus semilaevis) (胡元日等, 2022)、鲤鱼(Cyprinus carpio) (Jiang et al, 2014)、乌鳢(Scatophagus argus) (Lin et al, 2021)、罗非鱼(Zhu et al, 2016)等鱼类中也利用qRT-PCR或转录组学方法对pmel基因在不同肤色的表达情况开展了研究,初步探讨该基因在鱼类体色形成中的作用。因此,pmel基因已成为研究鱼类体色变异的关键基因之一。
虹鳟(Oncorhynchus mykiss)属鲑科、大马哈鱼属,是世界上广泛养殖的冷水性经济鱼类,具有肉质鲜美、无腥味和营养全面等特点。目前,在我国养殖场中最常见2种表型,一种为背部皮肤呈灰黑色的野生型虹鳟,俗称虹鳟;另一种为背部皮肤呈金黄色的黄色突变型虹鳟,俗称金鳟,体色变异对虹鳟的市场价值有着重要影响。Zou等(2015)研究认为,鱼类黄色表型的产生是由于黑色素缺失造成的。pmela作为黑色素合成通路中的关键基因,但其在虹鳟体色变异中的研究尚未见报道。基于前期虹鳟与金鳟背部皮肤转录组数据(Wu et al, 2021b; Wu et al, 2022),本研究旨在获得虹鳟pmela基因cDNA全长序列,并通过qRT-PCR方法分析该基因在虹鳟、金鳟及其杂交F1代不同发育时期和不同组织中的表达差异,进而探讨pmela基因在虹鳟体色变异中的作用,为深入研究虹鳟体色变异的分子机制提供基础资料。
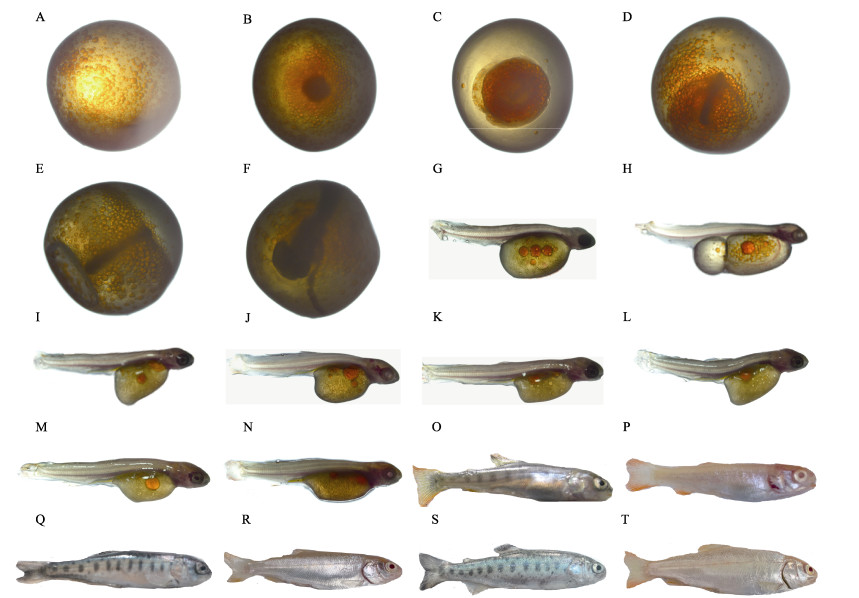
1 材料与方法 1.1 样品采集本实验所用的受精卵均来源于甘肃农业大学水产科学实训中心。首先,将受精卵置于水温[(12.0±0.5) ℃]和溶解氧(> 8.5 mg/L)相对恒定的环境中进行孵育,从而获得虹鳟、金鳟及其杂交F1代不同发育时期的胚胎及出膜后不同时期的背部皮肤,包括受精期、4细胞期、16细胞期、桑葚期、囊胚期、原肠期、神经期、体节期、心跳期、出膜后1 d、3 d、5 d、7 d、10 d、1个月、2个月、3个月(出膜后1~10 d采集的样品仅去除卵黄囊和头部) (n=3) (图 1)。待虹鳟、金鳟和杂交F1代长至12月龄时,分别各挑选3尾大小和体色基本一致的个体,并取背部皮肤、肌肉、脑、眼睛、鳃、头肾、肠、肝脏、脾脏和心脏(n=3),以上所有样品采集完立即投入液氮中速冻,后转入−80 ℃超低温冰箱保存备用。
|
图 1 虹鳟和金鳟不同发育时期形态 Fig.1 Morphology of wild-type and yellow mutant rainbow trout at different developmental stages A:受精期;B:桑葚期;C:原肠期;D:神经期;E:体节期;F:心跳期;G和H:出膜后1 d(前为虹鳟,后为金鳟,下同);I和J:出膜后5 d;K和L:出膜后7 d;M和N:出膜后10 d;O和P:出膜后1个月;Q和R:出膜后2个月;S和T:出膜后3个月 A: Fertilized-stage; B: Multi-cell; C: Gastrula; D: Neurula; E: Somites; F: Heartbeating; G and H: 1 day post-hatching (1 dph) (the former is wild-type rainbow trout, the latter is yellow mutant rainbow trout, the same below); I and J: 5 dph; K and L: 7 dph; M and N: 10 dph; O and P: 1 month post-hatching (1 M); Q and R: 2 M; S and T: 3 M |
取上述于−80 ℃保存的各组织样品,用液氮充分研磨后使用总RNA提取试剂盒(TIANGEN, 北京)提取RNA,通过1.0%的琼脂糖凝胶电泳和NanoDrop超微量分光光度计分别检测RNA的完整性和浓度,检测合格的RNA用PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis kit (TaKaRa, 大连)反转录生成cDNA第一链,−20 ℃保存。
1.2.2 pmela基因cDNA全长克隆根据转录组注释的序列,用Primer 5.0软件设计中间片段扩增引物pmela-1F和pmela-1R,以上述合成的cDNA第一链为模板进行pmela基因中间片段的克隆。PCR体系(50 μL):10×buffer 5 μL,dNTP 2.5 μL,Taq酶0.75 μL,pmela-1F和pmela-1R (10 μmol/L)各1.25 μL,模板2.5 μL,无菌ddH2O 36.75 μL。PCR参数:94 ℃,5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 150 s (35个循环);72 ℃,10 min。将PCR产物进行胶回收后与pMD19-T (TaKaRa, 大连)在16 ℃条件下连接5 h,转化到Trans 5α感受态细胞(TRANGEN, 北京),之后在37 ℃的生化培养箱中过夜培养,筛选阳性克隆进行菌液PCR检测,将含有目的片段的菌液送至上海生物科技有限公司(www.sangon.com)进行测序。
把测序后的序列经拼接比对后确认为中间序列,根据SMARTer® RACE 5′/3′ kit (Clontech, 美国)中的特异性引物的要求设计pmela-5′基因特异性引物(gene specific primer, GSP)、pmela-3′GSP、pmela-3′巢式基因特异性引物(nested gene specific primer, NGSP) (表 1),分别以生成的5'RACE-Ready cDNA和3'RACE-Ready cDNA为模板,通过降落PCR和巢式PCR相结合的方法进行pmela基因5′cDNA和3′cDNA末端的克隆。5′RACE和3′RACE依照SMARTer® RACE 5′/3′ kit (Clontech, 美国)进行。降落PCR体系(50 μL):ddH2O 15.5 μL,2×SeqAmp Buffer 25 μL,SeqAmp DNA polymerase 1 μL,5′ or 3′ RACE-Ready cDNA 2.5 μL,10×混合通用引物(universal primer mix, UPM) 5 μL,5′ or 3′ GSP (10 μmol/L) 1 μL。反应参数:94 ℃ 30 s,72 ℃ 3 min (5个循环);94 ℃ 30 s,70 ℃ 30 s,72 ℃ 3 min (5个循环);94 ℃ 30 s,65 ℃ 30 s,72 ℃ 3 min (25个循环)。巢式PCR体系(50 μL):ddH2O 17 μL,2×SeqAmp Buffer 25 μL,SeqAmp DNA polymerase 1 μL,5′ or 3′ RACE-Ready cDNA 5 μL (首次PCR产物稀释50倍),短通用引物(universal primer short, UPS) (10 μmol/L) 1 μL,5′或3′ NGSP (10 μmol/L) 1 μL。巢式PCR参数:94 ℃ 30 s,65 ℃ 30 s,72 ℃ 3 min (20个循环)。将产物经回收、纯化,之后连接转化培养,最后挑选阳性菌液测序。
|
|
表 1 虹鳟pmela基因克隆与表达分析所用引物 Tab.1 Primers for pmela gene cloning and expression analysis in rainbow trout |
DNAMAN 9.0软件用于拼接pmela基因全长cDNA序列,之后在NCBI中的ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)查找开放阅读框并翻译成氨基酸序列。使用在线软件ExPASy (https://web.expasy.org)分析蛋白的基本理化性质和亲疏水性。采用TMHMM软件(http://www.cbs. dtu.dk/services/tmhmm/)和SMART软件(https://smart.embl-heidelberg.de/)进行跨膜及蛋白质结构分析。二级结构使用在线软件SOPMA (https://npsa-prabi.ibcp. fr/)预测。从GenBank中下载其他物种Pmel氨基酸序列,并利用ClustalX 1.83软件进行序列同源比对以及MEGA 7.0软件中的邻接法(neighbor-joining)构建系统进化树。
1.2.4 pmela基因表达差异分析在pmela基因cDNA全长序列的基础上,设计qRT-PCR引物pmela-F和pmela-R,选取β-actin为内参基因(表 1)。将上述各组织的RNA经PrimeScriptTM RT reagent kit (TaKaRa, 大连)反转录成cDNA。首先,取金鳟出膜后3个月背部皮肤的cDNA (100 ng/μL)作为标准模板,以该模板进行5次5倍稀释,制成不同稀释倍数的标准模板。自每个稀释模板中取样1 μL进行qRT-PCR,经计算得到pmela基因和β-actin基因的扩增效率分别为0.84和0.91,且熔解曲线都为单一峰值,说明在该实验条件下检测得到的结果可较好反映样品中待测基因的表达水平。PCR体系为20 μL:SYBR Premix Ex Taq Ⅱ (Tli RNaseH Plus) 10.0 μL,pmela-F和pmela-R (10 μmol/L)各1 μL,cDNA 1 μL,RNase Free ddH2O 7 μL。反应参数:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s (40个循环)。其次,利用上述同样的qRT-PCR体系和程序,分析pmela基因在虹鳟、金鳟及其杂交F1代不同发育时期和组织中的表达差异。采用2–ΔΔCT法计算pmela基因在虹鳟、金鳟及其杂交F1代不同发育时期和组织中的相对表达量,之后运用SPSS 22.0的单因素方差分析(one-way ANOVA)和多重比较(Duncan)对数据进行显著性检验,P < 0.05为差异显著,P < 0.01时为差异极显著(吴垚磊等, 2021)。
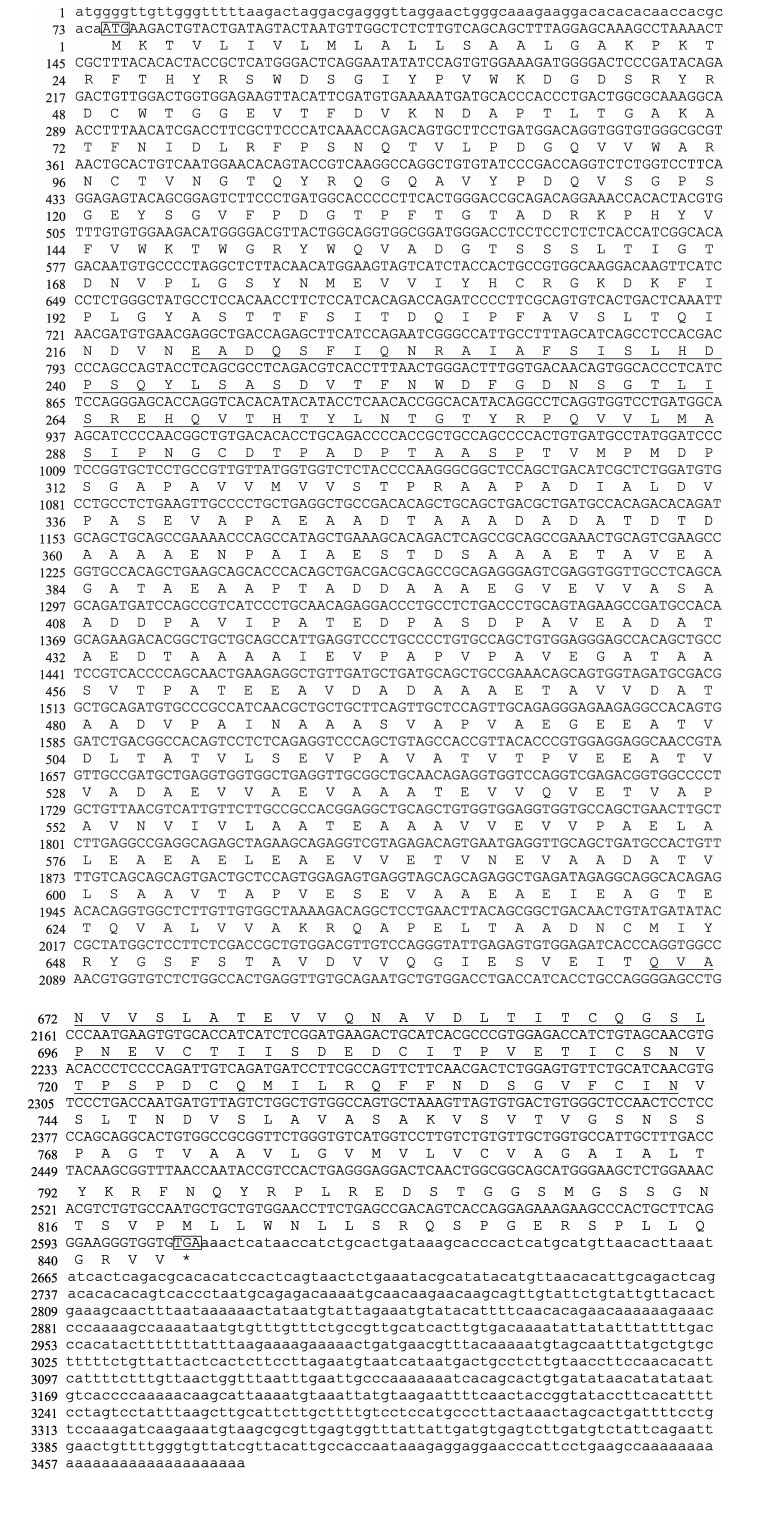
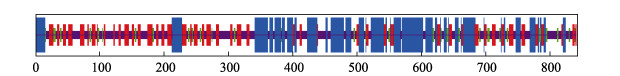
2 结果与分析 2.1 虹鳟pmela基因cDNA全长和氨基酸序列分析本研究获得虹鳟pmela基因cDNA全长3 476 bp(GenBank No. MW732713),包括75 bp 5′非编码区(untranslated region, UTR),2 532 bp开放阅读框(ORF) (编码843个氨基酸)以及869 bp 3′UTR,3′末端具有典型的poly(A)尾巴(图 2)。通过ProtParam软件分析可知,Pmela蛋白的分子式为C3810H6 008N1002O1282S24,理论分子质量约为87.13 kDa,理论等电点为4.02,总平均亲水系数为0.153,属于疏水性蛋白。使用ProtScale软件分析显示,Pmela蛋白第8位亮氨酸(Leu)的疏水性最强(3.533),第44位丝氨酸(Ser)的疏水性最弱(−2.989)。TMHMM分析显示,Pmela蛋白含1个跨膜区域,位于770~792位氨基酸。经过SMART分析发现,Pmela蛋白含有2个保守结构域,位于220~304位氨基酸的PKD结构域以及位于669~742位氨基酸的Tubulin_C结构域。二级结构分析显示,Pmela蛋白包含293个α螺旋(34.76%)、57个β折叠(6.76%)、298个无规卷曲(35.35%)和195个延伸(23.13%) (图 3)。
|
图 2 虹鳟pmela基因cDNA全长序列及编码氨基酸序列 Fig.2 The full-length cDNA and deduced amino acid sequences of pmela gene in rainbow trout 方框:起始密码子(ATG)和终止密码子(TGA);下划线:PKD (前)和Tubulin_C (后)结构域 Boxes: start codon (ATG) and stop codon (TGA); Underline: PKD (front) and Tubulin_C (behind) domains |
|
图 3 虹鳟Pmela蛋白的二级结构预测 Fig.3 The predicted secondary structure of Pmela protein in rainbow trout 紫色:无规卷曲;蓝色:α螺旋;红色:延伸;绿色:β折叠 Purple: Random coil; Blue: alpha-Helix; Red: Extended strand; Green: beta-Sheet |
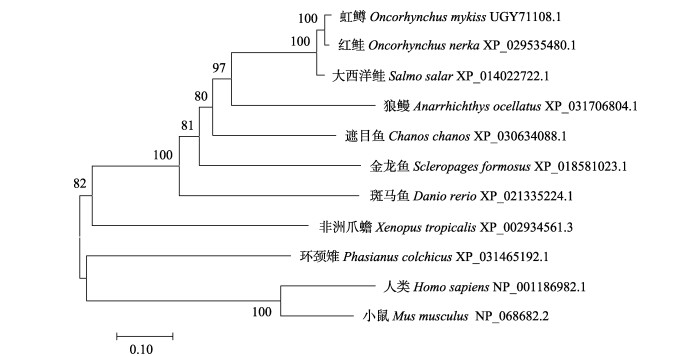
通过ClustalX 1.83软件将虹鳟与其他10个物种pmela基因编码的氨基酸序列进行同源比较,结果显示,虹鳟pmela基因编码的氨基酸序列与分类学地位相同的红鲑(Oncorhynchus nerka)和大西洋鲑(Salmo salar)的同源性最高,分别达到了97.75%和87.96%,与其他硬骨鱼类如遮目鱼(Chanos chanos)、狼鳗(Anarrhichthys ocellatus)、斑马鱼和金龙鱼(Scleropages formosus)的同源性在51%~61%之间,而与环颈雉(Phasianus colchicus)、非洲爪蟾(Xenopus tropicalis)、人类(Homo sapiens)和小鼠(Mus musculus) 的同源性较低(表 2);但Pmela蛋白中的PKD和Tubulin_C结构域的氨基酸序列在不同物种中的相似度较高(图 4)。系统进化树显示,虹鳟与红鲑的亲缘关系最近,与其他鱼类的亲缘关系次之,与环颈雉、非洲爪蟾、人类和小鼠的亲缘关系较远(图 5)。
|
|
表 2 虹鳟与其他物种Pmela氨基酸序列多重比对 Tab.2 Homology comparison of the amino acid sequences of Pmela between rainbow trout and other vertebrates |

|
图 4 虹鳟与其他物种Pmela蛋白中PKD和Tubulin_C结构域的氨基酸序列比对 Fig.4 Amino acid sequences comparison of PKD and Tubulin_C structural domains of Pmela protein between rainbow trout and other vertebrates 黑色区域:同源性=100%;粉红区域:同源性≥70%;蓝色区域:同源性≥50%;红色方框:PKD和Tubulin_C结构域 Black blocks: homology=100%; Pink blocks: homology≥70%; Blue blocks: homology≥50%; Red boxes: PKD and Tubulin_C domains |
|
图 5 Pmela氨基酸序列的系统进化分析 Fig.5 Phylogenetic tree based on Pmela amino acid sequences |
qRT-PCR结果显示,pmela基因在虹鳟与金鳟不同发育时期均有表达,总体上在出膜后时期的表达高于胚胎期(图 6)。在虹鳟中,pmela基因在受精期表达量最高,显著高于其他各时期(P < 0.05),其次是10 dph、3 M、7 dph、5 dph、16细胞期和2 M,其他时期中该基因的表达较低。在金鳟中,pmela基因表达量在3 M最高且显著高于其他各时期(P < 0.05),在1 dph至2 M过程中也有较高表达,在其他时期中的表达较低且差异不显著(P > 0.05)。虹鳟与金鳟同一发育时期的差异表达显示,pmela基因在受精期至16细胞期的表达为虹鳟高于金鳟,出膜后该基因的表达始终是金鳟高于虹鳟,其中,在4细胞期、16细胞期、原肠期、体节期、3 dph、5 dph、7 dph、10 dph和1 M中表达差异极显著(P < 0.01),在神经期、心跳期、1 dph和2 M中表达差异显著(P < 0.05),在受精期、桑葚期、囊胚期和3 M中表达差异不显著(P > 0.05)。
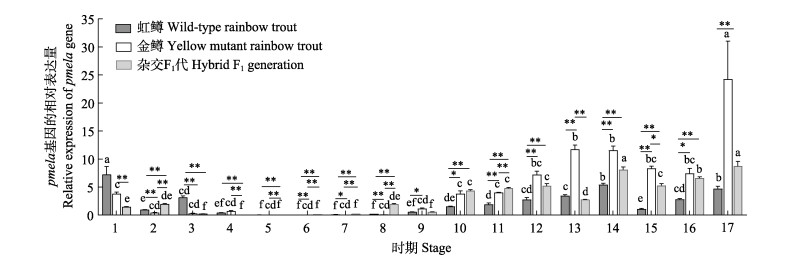
|
图 6 pmela基因在虹鳟、金鳟及其杂交F1代不同发育时期中的表达差异
Fig.6 Analysis of pmela gene expression at different developmental stages of wild-type rainbow trout, yellow mutant rainbow trout and their hybrid F1 generation
内参基因β-actin,n=3;pmela基因在虹鳟、金鳟及其杂交F1代不同发育时期的差异分析用小写字母表示,不同字母表示差异显著(P<0.05);pmela基因在虹鳟、金鳟及其杂交F1代同一发育时期的差异分析用*和**表示,*:P < 0.05,**:P < 0.01 1:受精期;2:4细胞期;3:16细胞期;4:桑葚期;5:囊胚期;6:原肠期;7:神经期;8:体节期;9:心跳期;10:出膜后1 d;11:出膜后3 d;12:出膜后5 d;13:出膜后7 d;14:出膜后10 d;15:出膜后1个月;16:出膜后2个月;17:出膜后3个月 Reference β-actin, n=3; Difference analysis of pmela gene expression at different developmental stages of wild-type rainbow trout, yellow mutant rainbow trout and their hybrid F1 generation are expressed in lowercase letters, different letters indicate significant difference (P < 0.05). Difference analysis of pmela gene expression at the same developmental stages among wild-type rainbow trout, yellow mutant rainbow trout and their hybrid F1 generation are indicated by * and **, * P < 0.05, ** P < 0.01 1: Fertilized-stage; 2: 4-cell; 3: 16-cell; 4: Multi-cell; 5: Blastula; 6: Gastrula; 7: Neurula; 8: Somites; 9: Heartbeating; 10: 1 dph; 11: 3 dph; 12: 5 dph; 13: 7 dph; 14: 10 dph; 15: 1 M; 16: 2 M; 17: 3 M |
与双亲类似,pmela基因在杂交F1代出膜后各时期中的表达都高于胚胎期,其中,在3 M和10 dph中表达最高,显著高于其他各时期(P < 0.05) (图 6)。杂交F1代与双亲同一发育时期的差异表达显示,pmela基因在杂交F1代4细胞期、桑葚期、囊胚期、原肠期、神经期、体节期、3 dph和1 M中的表达分别与双亲存在极显著或显著差异(P < 0.01或P < 0.05),在16细胞期、1 dph、5 dph、10 dph、2 M和3 M中的表达与虹鳟存在极显著差异(P < 0.01),在受精期和7 dph中的表达与金鳟存在极显著差异(P < 0.01)。
2.3.2 pmela基因在虹鳟、金鳟及其杂交F1代不同组织中的表达差异pmela基因在虹鳟与金鳟10个组织中均有表达,但主要集中在背部皮肤和眼睛(P < 0.05),在其他8个组织中的表达量较低(图 7)。虹鳟与金鳟相同组织间的差异表达显示,pmela基因在皮肤、肌肉、眼睛、鳃和心脏中表达差异极显著(P < 0.01),在肠和脾脏中表达差异显著(P < 0.05),其余组织中该基因的表达差异不显著(P > 0.05)。

|
图 7 pmela基因在虹鳟、金鳟及其杂交F1代不同组织中的表达差异 Fig.7 Analysis of pmela gene expression in different tissues of wild-type rainbow trout, yellow mutant rainbow trout and their hybrid F1 generation 内参基因:β-actin,n=3;pmela基因在虹鳟、金鳟及其杂交F1代不同组织的差异分析用小写字母表示,不同的字母表示差异显著(P < 0.05);pmela基因在虹鳟、金鳟及其杂交F1代相同组织的差异分析用*和**表示,*:P < 0.05,**:P < 0.01。 Reference gene β-actin, n = 3; Difference analysis of pmela gene expression in different tissues of wild-type rainbow trout, yellow mutant rainbow trout and their hybrid F1 generation are expressed in lowercase letters, different letters indicate significant difference (P < 0.05). Difference analysis of pmela gene expression in the same tissues among wild-type rainbow trout, yellow mutant rainbow trout and their hybrid F1 generation are indicated by * and **, * P < 0.05, ** P < 0.01. |
pmela基因在杂交F1代不同组织中均有表达,其中在眼睛中的表达最高(P < 0.05),其次是背部皮肤,其余组织中该基因的表达较低(图 7)。杂交F1代与双亲相同组织的表达结果显示,除肝脏外,pmela基因在杂交F1代其他组织中的表达与双亲均存在极显著或显著差异(P < 0.01或P < 0.05)。
3 讨论作为最大的脊椎动物类群,鱼类具有丰富的体色和令人眼花缭乱的斑纹,其不仅是一种重要的经济特征,更是生物发育和进化研究的重要内容(范云鹏等, 2019)。Pmel是一种黑色素细胞特异性糖蛋白,能够在细胞器中形成黑色素沉积的淀粉样纤维蛋白基质以及维持黑色素小体的环境稳态,从而在黑素小体发育中起到关键作用(Raposo et al, 2007)。大量研究表明,pmela基因对动物体表的黑色素沉积至关重要,其缺陷会导致动物体色异常(Hellström et al, 2011; Kerje et al, 2004)。本研究克隆了虹鳟pmela基因的cDNA全长序列,并利用qRT-PCR方法分析该基因在虹鳟、金鳟及其杂交F1代不同发育时期和组织中的表达差异,进而探讨pmela基因表达量与虹鳟体色变异的关系。
本研究获得虹鳟pmela cDNA全长为3 476 bp,ORF 2 532 bp,编码843个氨基酸。序列分析显示,Pmela属于疏水性蛋白、以无规卷曲为主且包含1个跨膜结构域,这与莆田黑鸭(Anas) Pmel蛋白的结构一致(潘家华等, 2020),但在凤头白鸭中发现该蛋白不存在跨膜结构域,说明Pmel蛋白结构有物种特异性特征(姚文成, 2018)。除此之外,虹鳟Pmela蛋白还存在PKD区,这与Theos等(2013)对人类Pmel蛋白的研究结果一致。已有研究表明,PKD区赋予了Pmel蛋白运输和淀粉样蛋白生成特性,从而保证后期黑色素小体中原纤维的形成(Theos et al, 2013),说明PDK区在黑色素生成中发挥着不可替代的作用。蛋白同源性和系统进化树分析结果显示,虹鳟与红鲑的氨基酸同源性最高且亲缘关系最近,其次是大西洋鲑,与其他动物同源性较低且亲缘关系较远,这一分析结果与虹鳟属于鲑科的分类学地位一致。
pmela基因在虹鳟、金鳟及其杂交F1代不同发育时期均有表达,其中,在受精期至16细胞期这一阶段的表达表现为虹鳟高于金鳟。黑色素细胞的前体是来自神经脊无色素沉积的黑色素母细胞,其通过背外侧或腹侧途径迁移到外胚层,最后分化为成熟的黑色素细胞(Vandamme et al, 2019)。研究表明,pmel基因的转录受小眼畸形相关转录因子(microphtalmia- associated transcription factor, mitf)直接调控(Du et al, 2003)。mitf作为神经脊细胞定向分化黑色素最早标记基因之一,其在黑色素母细胞整个迁移过程中持续表达(谢光跃等, 2014)。Baxter等(2003)在小鼠胚胎生长发育实验中也检测到pmel基因在黑色素母细胞中表达且能够识别早期发育阶段的黑色素母细胞。赵兵令等(2017)在小鼠毛囊生长的不同时期中发现,pmel基因在生长初期的表达量要明显高于中期和末期。课题组前期对mitfa基因在虹鳟与金鳟不同胚胎发育时期表达的研究中发现,该基因在受精期至16细胞期中的表达也表现为虹鳟高于金鳟(吴深基等, 2021a),因此,pmela基因在虹鳟受精期至16细胞期中的表达较金鳟高可能与mitfa基因有关。此外,在虹鳟中,pmela基因在心跳期的表达明显高于囊胚期至体节期,且该基因在出膜后1~10日龄这一阶段的表达呈逐渐升高的趋势。蒋燕玲等(2016)和周康奇等(2019)对橘色双冠丽鱼胚胎色素观察发现,黑色素在血液循环期开始出现且基因表达升高,说明pmela基因表达量升高可能由黑色素细胞增殖导致真黑色素增加引起。在出膜后的不同发育时期中,pmela基因在金鳟中的表达量始终高于虹鳟。目前,对pmela基因功能的研究局限于黑色素细胞,其在非黑色素细胞中的作用仍不清楚。在黑色素合成通路中,mitf作为中间枢纽基因,一方面其表达受黑素皮质素受体1基因(mc1r)直接调控,另一方面可以通过bHLHZip结构域与酪氨酸基因家族(tyr、tyrp1和dct)启动子的E-box结合来提高pmel基因的表达,进而促进黑色素的生成(Zhu et al, 2016; 陈旭东等, 2022)。在斑马鱼中,不仅发现黑色素细胞与黄色素细胞来至于同一前体干细胞,而且mitf和mc1r基因被证实在黄色素细胞中发挥着重要作用(Tu et al, 2010; Rawls et al, 2001; Cal et al, 2019)。除pmel基因外,本课题组在前期实验中发现,mitfa、mc1r、tyr、tyrp1和dct基因在绝大多数出膜后的不同发育时期中的表达都表现为金鳟高于虹鳟(吴深基等, 2021a、b; 王晓谰等, 2021; Wu et al, 2021a)。Ye等(2020)在对草鱼(Cyprinus carpio)体色进行研究时也猜测,tyr、dct和pmel基因参与黄色素的合成。综上,推测pmela基因在金鳟出膜后各时期中高表达可能与其直接或间接参与黄色素细胞的形成过程有关。有趣的是,pmela基因在杂交F1代5 dph至3 M过程中的表达位于金鳟与虹鳟之间,且在大部分时期中的表达均显著高于虹鳟。Zhang等(2017)在研究鲫鱼(Carassius auratus red var.)体色由灰转红过程中发现,黑色素合成关键基因(pmela、mitfa、tyr、tyrp1和dct)的表达水平逐渐下降,而黄色素合成关键基因的表达水平逐渐上升,说明pmela基因在F1代中的表达模式可能是由双亲体色差异造成的。
pmel基因在黑色素形成和沉积中发挥重要作用(潘家华等, 2020)。本研究中,pmela基因在虹鳟、金鳟和杂交F1代各组织中均有表达,但主要集中在背部皮肤和眼睛,说明pmela基因的高表达有助于不同组织黑色素沉积。其中在眼睛中高表达,主要是因为视网膜色素上皮中含有大量的黑色素细胞,这些细胞是实现视觉功能所必需的(Zou et al, 2006)。pmel在黑色素合成通路中属于正调控基因,大量研究发现,该基因主要在真黑色素沉着性组织中表达。在莆田黑鸭中,pmel基因只在毛囊和皮肤组织中高表达,在其他组织中几乎不表达(潘家华等, 2020);在牛(Bovine)中,皮肤是pmel基因的高表达组织(Kuehn et al, 2007)。这些研究均与本研究结果相吻合,揭示了pmela基因对真黑色素沉积具有重要的调节作用并与真黑色素沉积呈正相关。但pmela基因在金鳟背部皮肤中的表达高于虹鳟,这与该基因在大多数其他鱼类中的表达模式相反,例如,Jiang等(2014)在鲤鱼中发现,pmel基因在黑色皮肤中的表达高于黄色皮肤;该基因在乌鳢黑色斑点皮肤中的表达也高于无黑色斑点皮肤(Lin et al, 2021)。除pmel基因外,tyr-1、tyrp1和mitfa基因在大多数不同体色鱼类中的表达也有同样的规律(Zhang et al, 2017; Jiang et al, 2014; Zhu et al, 2016),但课题组前期实验中同样发现以上3个基因在12月龄背部皮肤中的表达也是金鳟高于虹鳟(王晓谰等, 2021; Wu et al, 2021a; 吴深基等, 2021a)。综合pmela基因在虹鳟与金鳟不同时期的表达规律,这些结果不仅说明了虹鳟体色变异的分子调控机制比其他鱼类更加复杂,而且再次验证了pmela基因的高表达可能与黄色素细胞相关。Kuehn等(2007)研究发现,pmel基因的表达并不局限于有真黑色素细胞的区域,在不含黑色素的皮肤组织中也发现了该基因的表达,认为pmel基因除了在黑素小体发育过程中发挥作用外,还发挥其他新的功能,具体原因还有待进一步研究。
4 结论本研究利用RACE技术,获得虹鳟pmela基因cDNA全长序列,通过生物信息学分析发现Pmela蛋白存在PKD功能结构域且该蛋白在鱼类中的保守性高于其他脊椎动物。qRT-PCR结果显示,pmela基因在出膜后各时期中的表达始终是金鳟高于虹鳟且大多数虹鳟与金鳟相同发育时期的表达均存在显著差异;在杂交F1代中,pmela基因在大多数出膜后时期的表达位于双亲之间;在不同的组织中,pmela基因主要在虹鳟、金鳟及其杂交F1代背部皮肤和眼睛中高表达。本研究结果表明,pmela基因的表达量高低与虹鳟体色具有一定的相关性,且可能参与其体色的形成过程。该结果可为进一步研究pmela基因在虹鳟体色变异中的作用提供基础资料。
BAXTER L L, PAVAN W J. Pmel17 expression is Mitf-dependent and reveals cranial melanoblast migration during murine development. Gene Expression Patterns, 2003, 3(6): 703-707 DOI:10.1016/j.modgep.2003.07.002 |
BRAASCH I, BRUNET F, VOLFF J N, et al. Pigmentation pathway evolution after whole-genome duplication in fish. Genome Biology and Evolution, 2009, 1: 479-493 DOI:10.1093/gbe/evp050 |
BRUNBERG E, ANDERSSON L, COTHRAN G, et al. A missense mutation in PMEL17 is associated with the silver coat color in the horse. BMC genetics, 2006, 7: 46 |
CAL L, SUAREZ-BREGUA P, BRAASCH I, et al. Loss-of- function mutations in the melanocortin 1 receptor cause disruption of dorso-ventral countershading in teleost fish. Pigment Cell and Melanoma Research, 2019, 32(6): 817-828 DOI:10.1111/pcmr.12806 |
CHEN X D, WU G Q, SONG H M, et al. Structure and expression analysis of body color-related mitf gene in Amphilophus citrinellus. Progress in Fishery Sciences, 2022, 43(2): 107-118 [陈旭东, 邬国强, 宋红梅, 等. 橘色双冠丽鱼体色相关基因mitf的结构及表达调控特性. 渔业科学进展, 2022, 43(2): 107-118] |
DU J, MILLER A J, WIDLUND H R, et al. MLANA/MART1 and SILV/PMEL17/GP100 are transcriptionally regulated by MITF in melanocytes and melanoma. American Journal of Pathology, 2003, 163(1): 333-343 DOI:10.1016/S0002-9440(10)63657-7 |
FAN Y P, ZHAO H, LUO S M, et al. Observation on body color development in red-white crucian carp and expression analysis of pigment genes in different color crucian carp. Acta Laser Biology Sinica, 2019, 28(4): 343-352 [范云鹏, 赵韩, 罗世民, 等. 花鲫早期体色发育和几个体色相关基因在不同鲫的表达分析. 激光生物学报, 2019, 28(4): 343-352] |
FONTANESI L, SCOTTI E, COLOMBO M, et al. A composite six bp in-frame deletion in the melanocortin 1 receptor (MC1R) gene is associated with the Japanese brindling coat colour in rabbits (Oryctolagus cuniculus). BMC Genetics, 2010, 11: 59 |
HELLSTRÖM A R, WATT B, FARD S S, et al. Inactivation of Pmel alters melanosome shape but has only a subtle effect on visible pigmentation. PLoS Genetics, 2011, 7(9): e1002285 DOI:10.1371/journal.pgen.1002285 |
HU Y R, YANG Q, WANG J L, et al. Expression analysis of lnc-XR_003049606.1 and its target gene pmelb in blind side skin melanization in Cynoglossus semilaevis. Journal of Fisheries of China, 2022, 46(4): 529-536 [胡元日, 杨倩, 王佳林, 等. 半滑舌鳎lnc-XR_003049606.1及其靶基因pmelb在无眼侧皮肤黑化过程中的表达. 水产学报, 2022, 46(4): 529-536] |
JIANG Y L, SONG H M, LIU Y, et al. Cloning and expression analysis of the developing sequence and tissue expression of TYR gene in Amphilophus citrinellus. Journal of Agricultural Biotechnology, 2016, 24(5): 697-707 [蒋燕玲, 宋红梅, 刘奕, 等. 橘色双冠丽鱼TYR基因的克隆及其发育时序和组织表达分析. 农业生物技术学报, 2016, 24(5): 697-707] |
JIANG Y, ZHANG S, XU J, et al. Comparative transcriptome analysis reveals the genetic basis of skin color variation in common carp. PLoS One, 2014, 9(9): e108200 DOI:10.1371/journal.pone.0108200 |
KERJE S, SHARMA P, GUNNARSSON U, et al. The Dominant white, Dun and Smoky color variants in chicken are associated with insertion/deletion polymorphisms in the PMEL17 gene. Genetics, 2004, 168(3): 1507-1518 DOI:10.1534/genetics.104.027995 |
KUEHN C, WEIKARD R. Multiple splice variants within the bovine silver homologue (SILV) gene affecting coat color in cattle indicate a function additional to fibril formation in melanophores. BMC Genomics, 2007, 8(1): 1-13 DOI:10.1186/1471-2164-8-1 |
LAMOREUX M L, WAKAMATSU K, ITO S. Interaction of major coat color gene functions in mice as studied by chemical analysis of eumelanin and pheomelanin. Pigment Cell Research, 2011, 14(1): 23-31 |
LI X B, ZHENG S M, WU Q. Microscopic observation on chromatophores of Trichogaster trichopterus. Sichuan Journal of Zoology, 2012, 31(4): 538-541 [李小兵, 郑曙明, 吴青. 曼龙鱼色素细胞的显微观察. 四川动物, 2012, 31(4): 538-541] |
LIN X, TIAN C, HUANG Y, et al. Comparative transcriptome analysis identifies candidate genes related to black-spotted pattern formation in spotted scat (Scatophagus argus). Animals, 2021, 11(3): 765 DOI:10.3390/ani11030765 |
NAGAO Y, TAKADA H, MIYADAI M, et al. Distinct interactions of Sox5 and Sox10 in fate specification of pigment cells in medaka and zebrafish. PLoS Genetics, 2018, 14(4): e1007260 DOI:10.1371/journal.pgen.1007260 |
PAN J H, ZHOU M, CHEN Q H, et al. Cloning, tissue expression and polymorphism analysis of PMEL17 gene in Putian black duck. China Poultry, 2020, 42(12): 19-24 [潘家华, 周劢, 陈巧慧, 等. 莆田黑鸭PMEL17基因的克隆、组织表达与多态性分析. 中国家禽, 2020, 42(12): 19-24] |
RAPOSO G, MARKS M S. Melanosomes-dark organelles enlighten endosomal membrane transport. Nature Reviews Molecular Cell Biology, 2007, 8(10): 786-797 DOI:10.1038/nrm2258 |
RAWLS J F, MELLGREN E M, JOHNSON S L. How the zebrafish gets its stripes. Developmental Biology, 2001, 240(2): 301-314 DOI:10.1006/dbio.2001.0418 |
SCHONTHALER H B, LAMPERT J M, VON LINTIG J, et al. A mutation in the silver gene leads to defects in melanosome biogenesis and alterations in the visual system in the zebrafish mutant fading vision. Developmental Biology, 2005, 284(2): 421-436 DOI:10.1016/j.ydbio.2005.06.001 |
TU S, JOHNSON S L. Clonal analyses reveal roles of organ founding stem cells, melanocyte stem cells and melanoblasts in establishment, growth and regeneration of the adult zebrafish fin. Development, 2010, 137(23): 3931-3939 DOI:10.1242/dev.057075 |
THEOS A C, WATT B, HARPER D C, et al. The PKD domain distinguishes the trafficking and amyloidogenic properties of the pigment cell protein PMEL and its homologue GPNMB. Pigment Cell and Melanoma Research, 2013, 26(4): 470-486 DOI:10.1111/pcmr.12084 |
TIAN X, PANG X, WANG L, et al. Dynamic regulation of mRNA and miRNA associated with the developmental stages of skin pigmentation in Japanese ornamental carp. Gene, 2018, 666: 32-43 DOI:10.1016/j.gene.2018.04.054 |
VANDAMME N, BERX G. From neural crest cells to melanocytes: Cellular plasticity during development and beyond. Cellular and Molecular Life Sciences, 2019, 76: 1919-1934 DOI:10.1007/s00018-019-03049-w |
WANG C, WACHHOLTZ M, WANG J, et al. Analysis of the skin transcriptome in two Oujiang color varieties of common carp. PLoS One, 2014, 9(3): e90074 DOI:10.1371/journal.pone.0090074 |
WANG C, XU J, KOCHER T D, et al. CRISPR knockouts of pmela and pmelb engineered a golden tilapia by regulating relative pigment cell abundance. Journal of Heredity, 2022, 6: esac018 |
WANG L, LIU J. Research progress on molecular mechanism in the formation of melanin. Journal of Xinjiang University, 2019, 36(4): 468-474 [王磊, 刘军. 黑色素形成分子机制研究进展. 新疆大学学报, 2019, 36(4): 468-474] |
WANG X L, WU S J, HUANG J Q, et al. Bioinformatics and expression analysis of tyrosinase gene at the different stages and tissues of rainbow trout (Oncorhynchus mykiss). Chinese Animal Husbandry and Veterinary Medicine, 2021, 48(5): 1516-1524 [王晓谰, 吴深基, 黄进强, 等. 虹鳟酪氨酸酶基因的生物信息学分析及其在不同发育阶段和组织中的表达分析. 中国畜牧兽医, 2021, 48(5): 1516-1524 DOI:10.16431/j.cnki.1671-7236.2021.05.002] |
WEI M X, SONG H M, QI B L, et al. Pigment cells development and body color variation of postembryonic development in Amphilophus citrinellus. Journal of Shanghai Ocean University, 2015, 24(1): 28-35 [韦敏侠, 宋红梅, 祁宝伦, 等. 橘色双冠丽鱼胚后色素细胞发育与体色变化. 上海海洋大学学报, 2015, 24(1): 28-35] |
WU S J, HUANG J Q, LI Y J, et al. Cloning and expression analysis of body colour-related gene mitfa in rainbow trout (Oncorhynchus mykiss). Journal of Agricultural Biotechnology, 2021a, 29(4): 753-763 [吴深基, 黄进强, 李永娟, 等. 虹鳟体色相关基因mitfa的克隆和表达分析. 农业生物技术学报, 2021a, 29(4): 753-763] |
WU S J, HUANG J Q, LI Y J, et al. Cloning, sequence analysis, and expression of tyrp1a and tyrp2 genes related to body colour in different developmental stages and tissues of rainbow trout Oncorhynchus mykiss. Aquaculture International, 2021a, 29: 941-961 DOI:10.1007/s10499-021-00666-w |
WU S J, HUANG J Q, LI Y J, et al. Analysis of yellow mutant rainbow trout transcriptomes at different developmental stages reveals dynamic regulation of skin pigmentation genes. Scientific Reports, 2022, 12(1): 256 DOI:10.1038/s41598-021-04255-y |
WU S J, HUANG J Q, LI Y J, et al. Integrative mRNA-miRNA interaction analysis reveals the molecular mechanism of skin color variation between wild-type and yellow mutant rainbow trout (Oncorhynchus mykiss). Comparative Biochemistry and Physiology Part D, Genomics and Proteomics, 2021b, 40: 100914 DOI:10.1016/j.cbd.2021.100914 |
WU S J, ZHAO L, HUANG J Q, et al. Cloning, sequencing and expression analysis of mc1r gene in different developmental stages and tissues of rainbow trout Oncorhynchus mykiss. Chinese Journal of Zoology, 2021b, 65(5): 746-755 [吴深基, 赵璐, 黄进强, 等. 虹鳟mc1r基因的克隆、序列分析及在不同发育阶段和组织的表达分析. 动物学杂志, 2021b, 65(5): 746-755] |
WU Y L, LI Y Z, WANG N, et al. Expression analysis of TYR and DCT genes related to body color in Cynoglossus semilaevis at different periods and in different tissues. Progress in Fishery Sciences, 2021, 42(6): 42-52 [吴垚磊, 李仰真, 王娜, 等. 半滑舌鳎酪氨酸酶基因(TYR)和多巴色素异构酶基因(DCT)的克隆表达与分析. 渔业科学进展, 2021, 42(6): 42-52] |
XIE G Y, JIANG X P, LIU G Q. Cellular basis and influencing factors of melanin synthesis. Animal Husbandry and Veterinary Medicine, 2014, 46(1): 109-112 [谢光跃, 姜勋平, 刘桂琼. 黑色素合成的细胞基础及影响因素. 畜牧与兽医, 2014, 46(1): 109-112] |
XING L, SUN L, LIU S, et al. De novo assembly and comparative transcriptome analyses of purple and green morphs of Apostichopus japonicus during body wall pigmentation process. Comparative Biochemistry and Physiology Part D, Genomics and Proteomics, 2018, 28: 151-161 DOI:10.1016/j.cbd.2018.09.001 |
YAO W C. Association between single nucleotide polymorphisms of PMEL17 gene and feather color traits in duck. Masterxs Thesis of Yangzhou University, 2018 [姚文成. PMEL17基因单核苷酸多态性与鸭羽色性状的关联研究. 扬州大学硕士研究生学位论文, 2018]
|
YE X, ZHOU L, JIA J, et al. ITRAQ proteomic analysis of yellow and black skin in Jinbian carp (Cyprinus carpio). Life (Basel), 2020, 10: 226 |
ZHANG Y, LIU J, PENG L, et al. Comparative transcriptome analysis of molecular mechanism underlying gray-to-red body color formation in red crucian carp (Carassius auratus, red var.). Fish Physiology and Biochemistry, 2017, 43(5): 1387-1398 DOI:10.1007/s10695-017-0379-7 |
ZHAO B L. GPNMB and its homolog PMEL regulate the formation of melanocytic pigment in mice. Master's Thesis of Shanxi Agricultural University, 2017 [赵兵令. GPNMB及其同系物PMEL调控小鼠黑色素细胞色素生成. 山西农业大学硕士研究生学位论文, 2017]
|
ZHENG N Z, LI L, XIN Q W, et al. Genetic effect of tyrosinase (TYR), microphthalmia-associated transcription factor (MITF) and agouti signaling protein (ASIP) geneson melanin deposition of white silky fowl (Gallus gallus domesticus Brisson). Journal of Agricultural Biotechnology, 2015, 23(8): 1076-1083 [郑嫩珠, 李丽, 辛清武, 等. 酪氨酸酶(TYR)、小眼畸形相关转录因子(MITF)和刺鼠信号蛋白(ASIP)基因对白绒乌骨鸡黑色素沉积的遗传效应. 农业生物技术学报, 2015, 23(8): 1076-1083] |
ZHOU K Q, SONG H M, PAN X H, et al. Expression analysis of mc1r gene relating to body color variation in Amphilophus citrinellus. Chinese Journal of Zoology, 2019, 54(1): 45-56 [周康奇, 宋红梅, 潘贤辉, 等. 橘色双冠丽鱼体色相关基因mc1r的组织表达分析. 动物学杂志, 2019, 54(1): 45-56] |
ZHU W, WANG L, DONG Z, et al. Comparative transcriptome analysis identifies candidate genes related to skin color differentiation in red tilapia. Scientific Reports, 2016, 6: 31347 DOI:10.1038/srep31347 |
ZOU J, BEERMANN F, WANG J, et al. The Fugu tyrp1 promoter directs specific GFP expression in zebrafish: Tools to study the RPE and the neural crest-derived melanophores. Pigment Cell Research, 2006, 19(6): 615-627 DOI:10.1111/j.1600-0749.2006.00349.x |
ZOU M, ZHANG X, SHI Z, et al. A comparative transcriptome analysis between wild and albino yellow catfish (Pelteobagrus fulvidraco). PLoS One, 2015, 10(6): e0131504 |



