2. 广东省珍珠科技创新中心 广东 湛江 524088;
3. 广东省海水养殖生物育种工程实验室 广东 湛江 524088;
4. 广东省水产动物病害防控与健康养殖重点实验室 广东 湛江 524088
2. Guangdong Science and Innovation Center for Pearl Culture, Zhanjiang 524088, China;
3. Guangdong Provincial Engineering Laboratory for Mariculture Organism Breeding, Zhanjiang 524088, China;
4. Guangdong Provincial Key Laboratory of Aquatic Animal Disease Control and Healthy Culture, Zhanjiang 524088, China
Rab (Ras-related proteins in brain)作为小分子GTP结合蛋白中最大的亚家族,约由200个氨基酸组成,分子量为20~25 kDa (Schwartz et al, 2007),广泛分布于各种真核生物中,是膜泡运输过程中的重要调节因子,镶嵌在几乎所有与膜相关的细胞器上,调节作用主要通过GTP结合和GTP水解两种方式,在囊泡运输的各个阶段行使重要功能(Jordens et al, 2005; 聂宇等, 2008; 黄薇等, 2016)。目前,在水产动物中有关Rab蛋白功能的研究主要集中在免疫方面,例如:Rab11a和Rab11b可以通过胞吞途径参与斑点叉尾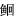
SNP是指物种基因组中由单个核苷酸突变所引起的DNA序列多态性,往往与生物体机能的改变和疾病的发生有关(陈昆平等, 2018)。在罗非鱼(Oreochromis niloticus)抗病选育研究中,高风英等(2014)和Fu等(2014)分别在MHC IIB和MCP-8基因中找到了与链球菌病抗性相关的SNP标记;付瑞雪等(2015)对牙鲆(Paralichthys olivaceus)的抗感染鳗弧菌病群体和易感群体进行了部分SNP位点的分型,发现丝氨酸蛋白酶基因在牙鲆抗鳗弧菌感染中发挥了重要作用;Zhao等(2012)报道了凡纳滨对虾(Litopenaeus vannamei)血蓝蛋白的C末端的SNP位点可能与对不同病原菌的抗性相关;邢贺飞等(2016)筛选了半滑舌鳎(Cynoglossus semilaevis) Nramp基因与抗鳗弧菌感染相关的SNP位点,发现Nramp基因不同基因型对半滑舌鳎的抗病能力有着极其重要的影响。目前,SNP作为最理想的DNA分子标记,被广泛应用于水产动物免疫相关基因与抗逆关联分析的研究中,为水产动物的育种奠定了基础。
马氏珠母贝是我国培育海水珍珠的主要贝种,具有重要经济价值,主要分布在我国广东、广西和海南沿海,低温耐受能力较弱,严重限制了海水珍珠的养殖区域和产量(赖卓欣, 2020)。为了开展珍珠贝北移养殖,本实验室持续构建了马氏珠母贝耐低温选育系(low temperature resistant line, R) (Wang et al, 2018),并利用基因组重测序技术比较分析了耐低温选育系F3和马氏珠母贝北部湾野生群体(Beibu Gulf wild population, W)的遗传结构(赖卓欣, 2020),获得了Rab基因在内的一批在选育过程中受到强烈正选择的候选基因(Lai et al, 2021; 陈琨等, 2022)。本研究运用RACE技术克隆了马氏珠母贝Rab7基因的全长cDNA序列,对马氏珠母贝低温胁迫后的鳃组织进行了时序表达研究,借助前述全基因组重测序数据库,对Rab7基因进行外显子SNP位点的筛选和遗传多态性、单倍型、频率的分析及计算,以期深入了解Rab7基因在马氏珠母贝低温响应中的作用,为进一步研究马氏珠母贝对温度胁迫的应答机制提供基础数据。
1 材料与方法 1.1 实验材料本实验所用的马氏珠母贝均来自广东省雷州市后洪村海区(20°29′N,109°54′E)。把表面附着物清理干净的马氏珠母贝运回实验室后,暂养于22 ℃,2 d后从中选取规格相似、活力正常且鳞片旺盛、生理状态良好的158个个体用于基因克隆及基因表达分析,实验贝壳长为(69.23±3.80) mm。随机取8只马氏珠母贝,剪取闭壳肌、鳃、性腺、肝胰腺、足和外套膜,经液氮速冻,置于–80 ℃冰箱备用,其他个体用于温度胁迫实验。
温度胁迫下鳃组织中时序表达样品制备:以3个300 L的大白桶为容器,分别设置海水温度为17 ℃(低温组)、22 ℃(对照组)和32 ℃(高温组)(赖卓欣等, 2019),在水温调到对应的温度后将150只马氏珠母贝随机分成3组,分别放进3个桶内养殖,在6 h、12 h、1 d、2 d和3 d从每个组随机取8只马氏珠母贝,剪取鳃组织,液氮急冻,–80 ℃冰箱保存,用于后续实验。养殖过程中,每天更换50%相同温度的海水,投喂等量的单胞藻,保持微充气。
1.2 Pm-Rab7基因全长克隆采用Trizol法提取总RNA,3' RACE和5' RACE模板的制备参考SMARTTM RACE cDNA Amplification试剂盒说明书。利用Primer Premier 5.0软件设计实验所需引物(表 1),利用巢式PCR扩增获得5′端和3′端序列,PCR产物用1%的琼脂糖凝胶电泳检测,将与目的片段长度一致的PCR产物和载体pMD18-T连接,连接产物转化到DH5α感受态细胞中,用氨苄选择培养基培养后进行菌液PCR,挑取阳性菌送生工生物工程(上海)股份有限公司测序。将测序获得的成功克隆序列与马氏珠母贝基因组已知的部分进行拼接,最终获得基因全长序列。
|
|
表 1 引物序列 Tab.1 Primer sequences |
DNAMAN软件进行序列拼接及氨基酸序列推导;ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder)找到Pm-Rab7的开放阅读框(ORF);利用SMART 4 (http://smart.embl-heidelberg.de/)预测序列保守结构域;对氨基酸的理化性质分析、信号肽预测和跨膜结构分析分别利用ExPASy-ProtParam、SignalP 5.1和TMHMM Server软件完成;利用NCBI COBALT (https://www.ncbi.nlm.nih.gov/tools/cobalt/)和DNAMAN 9进行多序列比对;蛋白质三级结构由Phyre 2 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测;功能位点在氨基酸序列中的分布情况利用SoftBerry-Psite(http://www.softberry.com/berry.phtml?topic=psite&group=programs&subgroup=proloc)预测;MEGA 11用于生物系统发育树构建。
1.4 Pm-Rab7的组织表达及温度胁迫下的差异分析本研究以β-actin为内参基因(赖卓欣等, 2019; 陈琨等, 2022),利用2–ΔΔCt法计算Pm-Rab7基因在不同组织的相对表达量以及在实验温度下各时间点的相对表达量,以各组织的反转录cDNA为模板,反应体系(10 μL):上下游引物各0.4 μL,cDNA 0.4 μL,2×PerfectStart Green qPCR SuperMix 5 μL,灭菌ddH2O 3.8 μL。反应条件:95 ℃预变性5 min,95 ℃变性10 s,60 ℃退火15 s,72 ℃延伸15 s,45个循环;熔解曲线为95 ℃ 10 s,65 ℃ 60 s,95 ℃ 1 s。每个实验组共8个样品,每个样品重复3次。利用SPSS 22.0软件以进行单因素方差分析(one-way ANOVA)组织间表达量和温度胁迫后各时间点表达量的均值比较,显著性水平为P<0.05。
1.5 Pm-Rab7基因SNP分析利用课题组前期完成的马氏珠母贝耐低温选育群体(R)和北部湾野生群体(W)各30个个体的全基因组重测序文库(赖卓欣, 2020),对Pm-Rab7基因外显子区的SNP位点进行筛选。利用软件Haploview 4.2计算D′值和R2值及进行连锁不平衡分析和单倍型分析;PopGen 32软件统计分析SNP的基因型、基因型频率、观测杂合度(Ho)、期望杂合度(He)以及哈迪–温伯格平衡(HWE)等遗传参数;PIC软件计算SNP位点的多态信息含量;使用SPSS 22.0软件的卡方检验进行2个群体的SNP差异统计分析。
2 结果与分析 2.1 Pm-Rab7基因克隆及序列分析本研究利用RACE技术克隆获得马氏珠母贝Pm-Rab7基因cDNA序列,Pm-Rab7总长为1 153 bp,5′ UTR为30 bp,3′ UTR为505 bp,ORF为618 bp,编码205个氨基酸(图 1),理论分子量为23.04 kDa,等电点为5.40。带负电的残基总数(Asp+Glu)为27个,带正电的残基总数(Arg+Lys)为25个;脂溶指数为77.02,平均亲水性(grand average of hydropathicity, GRAVY)为–0.343,为亲水性蛋白。结构域分析结果显示,Pm-Rab7具有Rab典型的结构域RAB,其中第15~22号氨基酸(GDSGVGKT),第37~45号氨基酸(YKATIGADF),第62~67号氨基酸(WDTAGQ),第124~128号氨基酸(GNKID),第153~157号氨基酸(ETSAK)是Rab家族的保守序列。

|
图 1 Pm-Rab7的核苷酸序列分析 Fig.1 Nucleotide sequence analysis of Pm-Rab7 小写字母表示5′和3′非编码区;大写字母表示编码区以及推导的氨基酸序列;方框内为起始密码子与终止密码子;阴影部分为结构域。 5′ and 3′ non-coding regions are indicated by lowercase letters; coding regions and deduced amino acid sequences are indicated by uppercase letters; within the box are the start codon and stop codon; and the shaded region is the domain. |
为了确定Pm-Rab7与其他物种的同源性,将Pm-Rab7与普氏野马(Equus przewalskii)、牛(Bos taurus)、野生火鸡(Meleagris gallopavo)、斑尾咬鹃(Apaloderma vittatum)、锦龟(Chrysemys picta bellii)、中华鳖(Pelodiscus sinensis)、大蟾蜍(Bufo bufo)、高山倭蛙(Nanorana parkeri)、新西兰背唇隆头鱼(Notolabrus celidotus)、象鼻鱼(Paramormyrops kingsleyae)、台湾家白蚁(Coptotermes formosanus)、多胚跳小蜂(Copidosoma floridanum)、笠贝(Lottia gigantea)、太平洋牡蛎(Crassostrea gigas)、棘冠海星(Acanthaster planci)和紫色球海胆(Strongylocentrotus purpuratus)的Rab基因序列进行多序列比对。结果显示,Pm-Rab7与笠贝、太平洋牡蛎等其他软体动物的RAB序列相似,程度最高的是太平洋牡蛎,达92.79% (图 2)。
|
图 2 Rab7氨基酸序列多序列对比 Fig.2 Multiple alignment of Rab7 amino acid sequences 红色到灰色代表保守性从高到低。 Red to gray represents conservatism from high to low. |
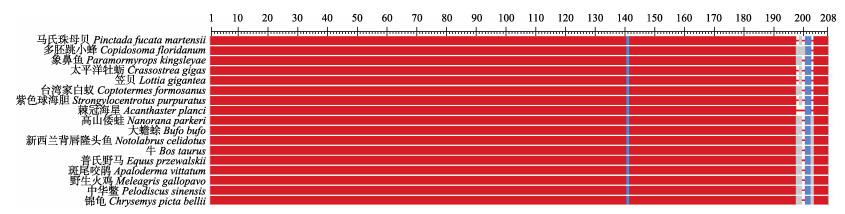
对马氏珠母贝、太平洋牡蛎、新西兰背唇隆头鱼和普氏野马的Rab7基因利用在线软件进行蛋白三维结构的构建,预测的具体结果见图 3,从图 3可以看出,不同物种的三维结构十分相似,说明Rab7基因在生物进化过程中相对比较保守。
|
图 3 马氏珠母贝(a)、太平洋牡蛎(b)、新西兰背唇隆头鱼(c)和普氏野马(d) Rab7蛋白分子结构 Fig.3 Molecular structure of Rab7 protein structure in P. f. martensii (a), C.gigas (b), N. celidotus (c), and E. przewalskii (d) |
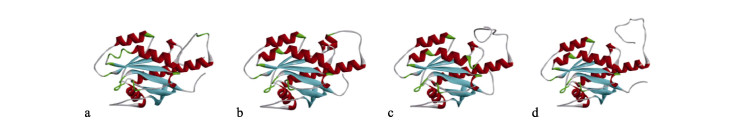
将Pm-Rab7与其他物种的RAB序列建立系统进化树,结果显示,进化树呈现出3大支,其中,哺乳类、鸟类、爬行类、两栖类与鱼类同为脊椎动物,聚为一大支;节肢动物与软体动物同为原口动物,聚为一大支;棘皮动物另聚为一大支(图 4),这与传统的分类基本保持一致。
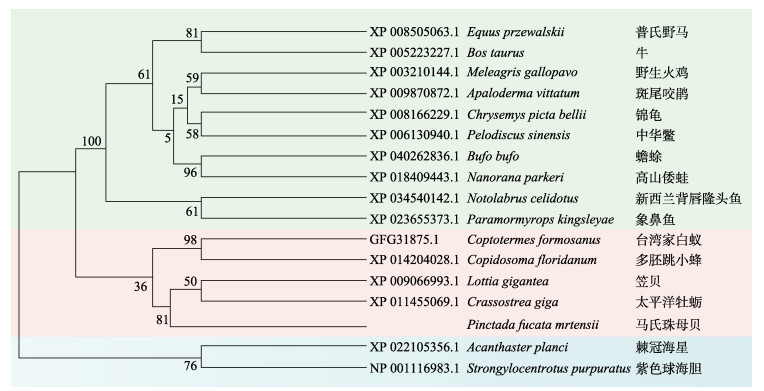
|
图 4 Rab7的系统进化树 Fig.4 The phylogenetic tree of Rab7 |
采用实时荧光定量PCR (qRT-PCR)技术检测Rab7在马氏珠母贝闭壳肌、鳃、性腺、足、肝胰腺和外套膜的表达模式,结果显示,Pm-Rab7基因在所有马氏珠母贝检测组织中均有不同程度的表达,在性腺和鳃中表达量显著高于其他组织(P<0.05)(图 5)。

|
图 5 Pm-Rab7在马氏珠母贝不同组织中的表达分布 Fig.5 Pm-Rab7 gene expression profiles in different tissues of P. f. martensii Gi:鳃;Go:性腺;A:闭壳肌;He:肝胰腺;M:外套膜;F:足。不同字母代表差异显著(P<0.05)。 Gi: Gill; Go: Gonad; A: Adductor muscle; He: Hepatopancreas; M: Mantle; F: Foot. Different letters represent significant difference (P<0.05). |
利用qRT-PCR技术检测了Pm-Rab7在温度胁迫下马氏珠母贝鳃组织中的时序表达。结果显示,在17 ℃低温组,Pm-Rab7的表达量呈现先上升再下降的趋势,在6 h~3 d内均显著高于22 ℃对照组(P<0.05),在1 d时达到峰值;在32 ℃高温组,Pm-Rab7的表达量总体稳定,仅12 h时显著高于对照组(P<0.05),其他时间点与对照组无显著性差异(P>0.05),说明该基因的表达与马氏珠母贝对低温胁迫的响应密切相关(图 6)。
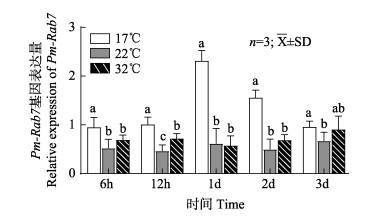
|
图 6 17 ℃、22 ℃和32 ℃下马氏珠母贝鳃中Pm-Rab7基因的时序表达 Fig.6 Temporal expression of the Pm-Rab7 gene in the gills of P. f. martensii at 17 ℃, 22 ℃, and 32 ℃ 同一时间点相同字母代表无显著性差异(P>0.05)。 The same letters at the same time point represent no significant difference (P>0.05). |
通过R和W群体的全基因组重测序数据,在Pm-Rab7基因的外显子共发现了7个SNP位点。对这些SNP位点进行遗传信息分析,结果显示,在R群体PIC范围为0.091~0.352,平均值为0.251,3个位点为低度多态性(PIC<0.25),4个为中度多态性(0.25<PIC<0.5),Ho范围为0.033~0.533,平均值为0.310;He范围为0.097~0.463,平均值为0.311 (表 2)。Pm-Rab7的7个SNP位点连锁不平衡分析结果显示:位点完全连锁不平衡(D′=1, R2=1)有0对,强连锁不平衡(0.8≤D′<1, 0.3≤R2<1)有1对,弱连锁不平衡(D′<0.8, R2<0.3)有8对(图 7A)。在W群体中PIC范围为0.062~0.357,平均值为0.211,4个位点为低度多态性(PIC<0.25),3个为中度多态性(0.25<PIC<0.5),Ho范围为0.067~0.533,平均值为0.291;He的范围为0.066~0.472,平均值为0.260 (表 2)。Pm-Rab7的7个SNP位点连锁不平衡分析结果显示:位点完全连锁不平衡(D′=1, R2=1)有0对,强连锁不平衡(0.8≤D′<1, 0.3≤R2<1)有0对,弱连锁不平衡(D′<0.8, R2<0.3)有13对(图 7B)。
|
|
表 2 Pm-Rab7的SNP多态性分析 Tab.2 Analysis of the SNP polymorphisms of Pm-Rab7 |
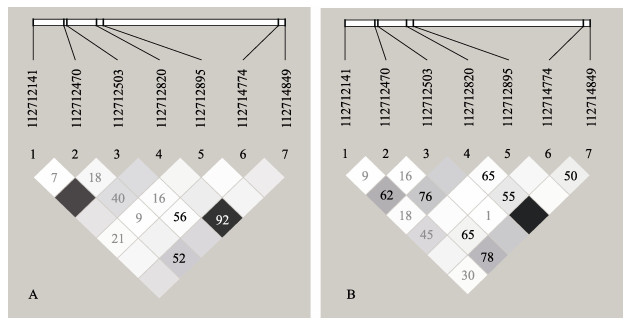
|
图 7 Pm-Rab7 SNP位点的连锁不平衡分析 Fig.7 Linkage disequilibrium analysis for the SNPs in Pm-Rab7 A:耐低温选育系;B:北部湾野生群体 A: Low temperature resistant line; B: Beibu Gulf wild population |
在Pm-Rab7基因外显子区域共检测到7个SNP位点,其中,g.112712470、g.112712503、g.112714774这3个SNP位点的基因型频率和等位基因频率在两个群体间差异显著(P<0.05) (表 3),g.112712470的AG基因型在R和W中的频率分别为53.3%和26.7%,g.112712503的AG基因型在R和W中的频率分别为40.0%和13.3%,g.112714774的GG基因型在R和W中的频率分别为83.3%和36.7%,表明这3个位点与耐低温性状相关(P<0.05)。值得注意的是,位点g.112712895的基因型CC只在R群体中被检测到,说明这个位点在低温选育群体中受到了强烈的正选择。
|
|
表 3 Pm-Rab7在耐低温选育系(R)和北部湾野生群体(W)间分布频率差异显著的SNP位点及其基因型 Tab.3 The characterization and significance of Pm-Rab7 SNP sites in low temperature resistant line (R) and Beibu Gulf wild population (W) |
具有Mg2+作用位点、共有的5个保守序列、GTP/GDP开关Ⅰ和开关Ⅱ是Rab家族成员共同具有的特征,C端最后几个氨基酸通常以固定模式存在,半胱氨酸会进行异戊二烯化修饰,在细胞膜的可逆定位中有很重要的作用(Stroupe et al, 2000; Pereira-Leal et al, 2001; Takai et al, 2001; 吴文林等, 2006)。RAB结构域是Rab基因家族所具有的保守区域,已有研究表明,利用5个保守的氨基酸序列来鉴定Rab蛋白是一种已确定的验证方法(Pereira-Leal et al, 2000),本研究利用RACE技术成功克隆获得的马氏珠母贝Rab7基因中包含5个保守的氨基酸序列,预测蛋白结果显示,Pm-Rab7具有Rab蛋白的保守结构域RAB,这些都印证了同源Rab基因结构上高度保守(Kelly et al, 2012)。此外,Rab7有2个区域,分别称为开关Ⅰ和开关Ⅱ,这2个区域是Rab7构象在与GDP和GTP结合的2种状态之间变化的位置。当Rab和GTP相结合时,Rab处于激活状态,可以自动识别其中的效应影响因子并执行各种效应功能。GTP在被水解后,Rab7变得不活跃,这在抑制Rab与其他能调节免疫蛋白之间的相互作用、免疫病毒防御等领域发挥着重要作用(Pasqualato et al, 2004)。多序列比对结果表明,Pm-Rab7与其他物种的Rab7的氨基酸序列相似性高,与太平洋牡蛎Rab相似度最高,为92.79%。由此可推测,本研究克隆获得的序列为Rab7基因。各物种的Rab7基因进化树构建结果显示,原口动物、棘皮动物和脊椎动物分别聚为三大支。在原口动物中,Pm-Rab7首先与其他软体动物聚类,软体动物再与节肢动物聚为一大支,这说明Pm-Rab7在进化过程中相对保守。
除少数物种的Rab结合蛋白仅在特定的组织中可以表达,大多数的Rab结合蛋白不具有组织特异性(Stenmark et al, 2001; Uno et al, 2010)。本研究结果显示,Rab7在马氏珠母贝鳃和性腺的表达量相对较高,在外套膜、闭壳肌、肝胰腺等组织均有表达,没有明显的组织特异性,这与Rab基因在日本对虾的各个组织中均有表达的研究结果相似(Wu et al, 2007)。鳃组织是马氏珠母贝与外界环境接触的主要器官,也是双壳贝类抵御外界刺激的第一道防线(Tiscar et al, 2004),Pm-Rab7基因在鳃组织中的高表达暗示Rab7在抵抗外界各种环境刺激时发挥重要作用。温度胁迫后的Pm-Rab7时序表达结果显示:在32 ℃高温组,Pm-Rab7的表达总体稳定;而在17 ℃低温组,Pm-Rab7在所有时间点的表达量均显著高于对照组(P<0.05),这说明Rab基因主要参与了马氏珠母贝的低温适应过程的响应。在多种水产动物中的研究中已表明,Rab基因参与了生物的免疫应答,而低温会引起马氏珠母贝的免疫反应(赖卓欣, 2020),推测Pm-Rab7可能是通过参与马氏珠母贝的免疫过程而对温度胁迫作出响应。
遗传多样性是生命进化和适应的基础,广义上指物种群体内及群体间的遗传变异度,其种内变异程度决定物种的进化潜力(马静等, 2010),通常可利用PIC、杂合度以及有效等位基因数等指标来衡量物种的遗传多样性,其丰富程度能反映物种对环境变化的适应能力(李浩等, 2019)。本研究中,R群体Pm-Rab7多个SNP位点的多态位点信息低于W群体,说明经过连续多代的高强度人工选择,降低了R群体的遗传多态性。其中,g.112712895位点在R群体中偏离哈迪–温伯格平衡(P>0.05)(表 2),而偏离哈迪–温伯格平衡现象的出现可作为在抗性群体的构建过程中与性状相关的初步判断(曹建萌等, 2017)。根据生物遗传性状影响,可将基因编码区SNP分为同义SNP和非同义SNP,而同义SNP可能通过影响少数密码子共翻译折叠时间,从而影响蛋白质的结构和功能(Komar et al, 2007)。本研究发现,Pm-Rab7基因3个SNP位点的基因型频率在W群体和R群体间差异显著(P<0.05),这3个位点的变异类型中A→G变异最多,均属于同义突变,但这些SNP位点与表型性状间的关联还需要进一步的实验验证。以上研究结果可为进一步深入研究马氏珠母贝对环境温度变化的适应机制提供重要参考资料。
CAO J M, HU X X, LU M X, et al. SNPs of C9 gene and their association with resistance to the infection of Streptococcus agalactiae in Oreochromis niloticus. Journal of Agricultural Biotechnology, 2017, 25(3): 354-365 [曹建萌, 胡欣欣, 卢迈新, 等. 尼罗罗非鱼补体C9基因单核苷酸多态性及其与抗无乳链球菌感染的关联分析. 农业生物技术学报, 2017, 25(3): 354-365] |
CHEN K P, LU M X, LIU Z G, et al. Molecular cloning analysis of the 5' regulatory region of Ikaros gene from Oreochromis niloticus and screening of its SNP markers for Streptococcus agalactiae resistance. Journal of Fishery Sciences of China, 2018, 25(2): 237-250 [陈昆平, 卢迈新, 刘志刚, 等. 吉富尼罗罗非鱼Ikaros基因5'调控区的克隆序列分析及抗无乳链球菌相关SNP位点筛选. 中国水产科学, 2018, 25(2): 237-250] |
CHEN K, LAI Z X, LIU Y, et al. Sequence characteristics of PEPCK gene of Pinctada fucata martensii and its selection in low temperature-resistant line. Journal of Fishery Sciences of China, 2022, 29(1): 58-69 [陈琨, 赖卓欣, 刘雅, 等. 马氏珠母贝PEPCK基因序列特征及其在耐低温品系的选择印记分析. 中国水产科学, 2022, 29(1): 58-69] |
FU G H, WAN Z Y, XIA J H, et al. The MCP-8 gene and its possible association with resistance to Streptococcus agalactiae in tilapia. Fish and Shellfish Immunology, 2014, 40(1): 331-336 DOI:10.1016/j.fsi.2014.07.019 |
FU R X, ZHOU N Y, LIU J X, et al. The polymorphism of serine protease Ⅰ-1 in Japanese flounder (Paralichthys olivaceus). Progress in Fishery Sciences, 2015, 36(5): 55-62 [付瑞雪, 周纳宇, 刘金相, 等. 牙鲆(Paralichthys olivaceus)丝氨酸蛋白酶Ⅰ-1基因多态性. 渔业科学进展, 2015, 36(5): 55-62] |
GAO F Y, PANG J C, LU M X, et al. Major histocompatibility complex MHCHB gene polymorphism and its association with resistance/susceptibility to Streptococcus agalactiae in Nile tilapia (Oreochromis niloticus). Chinese Agricultural Science Bulletin, 2014, 30(2): 76-83 [高风英, 庞纪彩, 卢迈新, 等. 尼罗罗非鱼MHCHB基因多态性及其与链球菌病抗性的关系. 中国农学通报, 2014, 30(2): 76-83] |
HUANG W, KANG Y J. The role of Rabs in glucose transport. Chinese Journal of Biochemistry and Molecular Biology, 2016, 32(3): 274-280 [黄薇, 康雁君. Rab蛋白在葡萄糖转运中的作用. 中国生物化学与分子生物学报, 2016, 32(3): 274-280 DOI:10.13865/j.cnki.cjbmb.2016.03.06] |
JORDENS I, MARSMAN M, KUIJL C, et al. Rab proteins, connecting transport and vesicle fusion. Traffic, 2005, 6(12): 1070-1077 DOI:10.1111/j.1600-0854.2005.00336.x |
KELLY E E, HORGAN C P, GOUD B, et al. The Rab family of proteins: 25 years on. Biochemical Society Transactions, 2012, 40(6): 1337-1347 DOI:10.1042/BST20120203 |
KOMAR A A. SNPs, silent but not invisible. Science, 2007, 315(5811): 466-467 DOI:10.1126/science.1138239 |
LAI Z X, LIU Y, WANG Q H, et al. cDNA cloning of FBP gene in Pinctada fucata martensii and its response to temperature stress. Progress in Fishery Sciences, 2019, 40(2): 106-114 [赖卓欣, 刘雅, 王庆恒, 等. 马氏珠母贝(Pinctada fucata martensii) FBP基因的克隆及其对温度胁迫的响应. 渔业科学进展, 2019, 40(2): 106-114] |
LAI Z X. Genome selection sweep of low temperature resistant line from Pinctada fucata martensii. Master's Thesis of Guangdong Ocean University, 2020 [赖卓欣. 马氏珠母贝耐低温选育系的选择印记分析. 广东海洋大学硕士研究生学位论文, 2020]
|
LAI Z, ADZIGBLI L, CHEN Q, et al. Identification and allelic variants associated with cold tolerance of PmPIAS in Pinctada fucata martensii. Frontiers in Physiology, 2021, 12: 634838 DOI:10.3389/fphys.2021.634838 |
LI H, MENG D L, XUE B B, et al. SNPs detection of α-amylase exon region and its association with growth traits in Sinonovacula constricta. Marine Fisheries, 2019, 41(2): 214-223 [李浩, 孟德龙, 薛宝宝. 缢蛏α淀粉酶基因外显子区域SNPs筛选及其与生长性状关联性. 海洋渔业, 2019, 41(2): 214-223] |
LIU S, ZHANG Y, ZHOU Z, et al. Efficient assembly and annotation of the transcriptome of catfish by RNA-Seq analysis of a doubled haploid homozygote. BMC Genomics, 2012, 13(1): 1-18 DOI:10.1186/1471-2164-13-1 |
MA J, AN Y P, WANG C F, et al. Research progress of genetic diversity. Shannxi Journal of Agricultural Sciences, 2010(1): 126-130 [马静, 安永平, 王彩芬, 等. 遗传多样性研究进展. 陕西农业科学, 2010(1): 126-130] |
NIE Y, LI J J, LIANG A H, et al. EoRab43 is an atypical Rab gene in Euplotes octocarinatus. Acta Zoologica Sinica, 2008, 54(1): 128-135 [聂宇, 李江姣, 梁爱华, 等. EoRab43为八肋游仆虫中编码非典型Rab的基因. 动物学报, 2008, 54(1): 128-135] |
ONGVARRASOPONE C, CHANASAKULNIYOM M, SRITUNYALUCKSANA K, et al. Suppression of PmRab7 by dsRNA inhibits WSSV or YHV infection in shrimp. Marine Biotechnology, 2008, 10(4): 374-381 DOI:10.1007/s10126-007-9073-6 |
PASQUALATO S, SENIC-MATUGLIA F, RENAULT L, et al. The structural GDP/GTP cycle of Rab11 reveals a novel interface involved in the dynamics of recycling endosomes. Journal of Biological Chemistry, 2004, 279(12): 11480-11488 DOI:10.1074/jbc.M310558200 |
PEREIRA-LEAL J B, SEABRA M C. Evolution of the Rab family of small GTP-binding proteins. Journal of Molecular Biology, 2001, 313(4): 889-901 DOI:10.1006/jmbi.2001.5072 |
PEREIRA-LEAL J B, SEABRA M C. The mammalian Rab family of small GTPases: Definition of family and subfamily sequence motifs suggests a mechanism for functional specificity in the Ras superfamily. Journal of Molecular Biology, 2000, 301(4): 1077-1087 |
SCHWARTZ S L, CAO C, PYLYPENKO O, et al. Rab GTPases at a glance. Journal of Cell Science, 2007, 120(Pt 22): 3905-3910 |
STENMARK H, OLKKONEN V M. The Rab GTPase family. Genome Biology, 2001, 2(5): 1-7 |
STROUPE C, BRUNGER A T. Crystal structures of a Rab protein in its inactive and active conformations. Journal of Molecular Biology, 2000, 304(4): 585-598 |
SUN F, PEATMAN E, LI C, et al. Transcriptomic signatures of attachment, NF-κB suppression and IFN stimulation in the catfish gill following columnaris bacterial infection. Developmental and Comparative Immunology, 2012, 38(1): 169-180 |
TAKAI Y, SASAKI T, MATOZAKI T. Small GTP-binding proteins. Physiological Reviews, 2001, 81(1): 153-208 |
TISCAR P G, MOSCA F. Defense mechanisms in farmed marine molluscs. Veterinary Research Communications, 2004, 28: 57-62 |
UNO T, HATA K, HIRAGAKI S, et al. Small GTPases of the Rab family in the brain of Bombyx mori. Histochemistry and Cell Biology, 2010, 134(6): 615-622 |
WANG L, LI L, WANG L, et al. Two Rab GTPases, EsRab-1 and EsRab-3, involved in anti-bacterial response of Chinese mitten crab Eriocheir sinensis. Fish and Shellfish Immunology, 2013, 35(3): 1007-1015 |
WANG Q, LIU Y, ZHENG Z, et al. Adaptive response of pearl oyster Pinctada fucata martensii to low water temperature stress. Fish and Shellfish Immunology, 2018, 78: 310-315 |
WU W L, WU S J. Rab GTPase family: Structure, function and its research prospects. Journal of Applied Oceanography, 25(4): 599–605 [吴文林, 吴穗洁. Rab蛋白的结构、功能与研究展望. 应用海洋学学报, 2006, 25(4): 599–605]
|
WU W, ZHANG X. Characterization of a Rab GTPase up-regulated in the shrimp Peneaus japonicus by virus infection. Fish and Shellfish Immunology, 2007, 23(2): 438-445 |
XING H F, GAO F T, ZHANG Y Z, et al. Molecular cloning, expression and SNP screening of natural resistance-associated macrophage protein (Nramp) gene cDNA from half smooth tongue sole (Cynoglossus semilaevis). Progress in Fishery Sciences, 2016, 37(4): 116-127 [邢贺飞, 高峰涛, 张永珍, 等. 半滑舌鳎(Cynoglossus semilaevis) Nramp基因克隆与表达分析及SNP筛选. 渔业科学进展, 2016, 37(4): 116-127] |
ZHAO X, GUO L, ZHANG Y, et al. SNPs of hemocyanin C-terminal fragment in shrimp Litopenaeus vannamei. FEBS Letters, 2012, 586(4): 403-410 |
ZONG R, WU W, XU J, et al. Regulation of phagocytosis against bacterium by Rab GTPase in shrimp Marsupenaeus japonicus. Fish and Shellfish Immunology, 2008, 25(3): 258-263 |



