2. 中国水产科学研究院 农业农村部水生生物基因组学重点实验室 北京 100141;
3. 中国水产科学研究院渔业工程研究所 北京 100141
2. Key Laboratory of Aquatic Genomics, Ministry of Agriculture and Rural Affairs, Beijing Key Laboratory of Fishery Biotechnology, Chinese Academy of Fishery Sciences, Beijing 100141, China;
3. Fisheries Engineering Institute, Chinese Academy of Fishery Sciences, Beijing 100141, China
鲤(Cyprinus carpio)是欧洲和亚洲地区重要的大宗淡水养殖鱼类,具有肉质好、耐寒性强、食物转化效率高等优良性状,在全球100多个国家广泛养殖。FAO发布报告显示,全球鲤年产量为418.95万t,约占全球淡水水产养殖总量的7.7% (FAO, 2020)。近年来,随着高密度集约化养殖模式的大范围推广,集约化养殖对水产动物产生的环境压力导致鱼类容易感染各种疾病病原体,包括病毒、细菌、真菌和寄生虫等,导致各类传染病的频繁暴发,给水产养殖业造成了巨大的经济损失(Talwar et al, 2018)。嗜水气单胞菌(Aeromonas hydrophila)是水产养殖中最为常见的致病菌之一,可引发多种淡水鱼类暴发细菌性败血症,包括草鱼(Ctenopharyngodon idella) (Yin et al, 2021)、罗非鱼(Oreochromis niloticus) (Pachanawan et al, 2008)、鲤(Stratev et al, 2015)等,且流行地域广、致死率高,严重危害水产养殖业的发展(Jones et al, 1995)。
甘露糖受体C1型基因(mannose receptor C-type 1, MRC1)是C型凝集素超家族(C-type lectin superfamily)中甘露糖受体家族(MR family)的成员之一,主要表达于巨噬细胞和树突状细胞的细胞膜表面(Stahl et al, 1998)。其结构包含胞外区、跨膜区和胞质区,其中胞外区由3个典型的结构域组成:8~10个串联排列的C型碳水化合物识别域(C-type carbohydrate recognition domain, CRD),1个Ⅱ型纤连蛋白重复区(Fibronectin repeats type Ⅱ, FNⅡ)和1个富含半胱氨酸的胞外区(Cysteine-rich, CR) (Napper et al, 2001)。MRC1基因编码的甘露糖受体是一种模式识别受体(pattern recognition receptor, PRR),通过胞外结构域能够广泛识别并结合多种配体,通过与配体之间的相互作用促进或抑制TNF-α (Xu et al, 2010)、IL-1β、1L-6 (Yamamoto et al, 1997; Zhang et al, 2005)等多种促炎症细胞因子的表达,同时,甘露糖受体参与介导吞噬细胞对病原体的吞噬,在抗病原感染和维持内环境稳态中发挥重要作用(Kerrigan et al, 2009)。由于其在先天免疫反应中发挥的关键作用,在人(Homo sapiens)、小鼠(Mus musculus)等哺乳动物中已有深入研究,但在鱼类中所做的工作相对较少。目前,在斑马鱼(Danio rerio) (Zheng et al, 2015)、草鱼(Wang et al, 2014)、大黄鱼(Larimichthys crocea) (Dong et al, 2016)、大菱鲆(Scophthalmus maximus) (Fontenla et al, 2016)、团头鲂(Megalobrama amblycephala) (Liu et al, 2014)等鱼类中已成功克隆出甘露糖受体基因。Liu等(2014)发现,甘露糖受体在团头鲂的主要免疫相关组织脾脏和头肾中具有明显较高的表达水平;Dong等(2016)在对弧菌(Vibrio)侵染大黄鱼的研究中同样发现,MRC1基因在感染弧菌的大黄鱼肝脏、脾脏和肾脏中均显著上调表达,表明甘露糖受体可能在鱼类免疫反应中发挥关键作用。Zhang等(2005)研究促炎细胞因子的释放调控发现,甘露糖受体对人类肺泡巨噬细胞促炎细胞因子的释放有负调节作用。另外,有报道显示,促炎因子IL-1β和TNF-α的释放需要MRC1和TLR4 (Toll样受体4)的协同作用(Sulek et al, 2007; Tachado et al, 2007)。Wu等(2018)在检测嗜水气单胞菌感染后团头鲂主要免疫组织中甘露糖受体基因MRC1、促炎细胞因子TNF-α和抗炎因子AGR的时间表达时发现,感染细菌后MRC1在组织中出现高表达,促炎细胞因子TNF-α在前6 h的浓度也明显更高,随后TNF-α的表达降低,但同时抗炎细胞因子ARG2表达逐渐升高并在12 h达到峰值。这些研究表明,MRC1可能参与调控促炎因子TNF-α和抗炎因子AGR的释放,进而在鱼类抵抗病原感染的免疫反应中发挥作用。
本研究首次从鲤全基因组资源中鉴定出MRC1基因的11个拷贝,开展基因结构预测、系统进化分析和共线性分析,并进一步检测和比较鲤MRC1基因各拷贝在多个健康鱼组织以及感染嗜水气单胞菌后不同时间点的脾脏中的表达情况,旨在为今后深入研究鲤MRC1基因的抗病相关功能及分子选育抗病新品系提供必要的数据基础。
1 材料与方法 1.1 MRC1基因序列获得和蛋白结构预测利用本课题组已有的鲤全基因组资源并结合转录组数据(NCBI收录号为SRP174892、SRP072018) (Xu et al, 2019),鉴定出全部MRC1基因,并提取出基因所在位置上下游±2 000 bp的基因组DNA序列,利用FGENESH (Salamov et al, 2000)和BLAST (Beyer et al, 2007)软件,逐一重新进行基因结构预测和注释,并获得对应的氨基酸序列。本研究用到的其他物种包括人、小鼠、原鸡(Gallus gallus)、绿头鸭(Anas platyrhynchos)、蜥蜴(Lacerta agilis)、非洲爪蟾(Xenopus tropicalis)、青鳉(Oryzias latipes)、三刺鱼(Gasterosteus aculeatus)、大黄鱼、河鲀(Tetraodon nigroviridis)、尼罗罗非鱼、大西洋鳕鱼(Gadus morhua)、红鳍东方鲀(Takifugu rubripes)、斑马鱼等的MRC1的氨基酸序列通过NCBI数据库(https://www.ncbi.nlm.nih.gov/protein/)和Ensembl数据库(http://www.ensembl.org/index.html?redirect=no)获得。鲤MRC1蛋白的结构预测以氨基酸序列作为输入文件,利用TBtools的Batch SMART软件完成(Chen et al, 2020)。
1.2 共线性分析共线性指不同物种间同源基因按保守的顺序排列,主要是用来描述同一染色体上基因的位置关系,也就是指由同一祖先型分化而来的不同物种间基因的类型以及相对顺序的保守性(宋驰等, 2010; 周显臻等, 2021)。为理解不同物种基因组间MRC1基因在进化上的保守性,通过NCBI和Ensembl数据库查询不同物种MRC1上下游基因获得共线性数据。
1.3 多序列比对和系统进化分析使用NCBI在线多序列比对工具COBALT (https://www.ncbi.nlm.nih.gov/tools/cobalt/cobalt.cgi)对14个物种的MRC1氨基酸序列进行比对,比较不同物种与鲤MRC1蛋白的一致性。使用鲤MRC1的11条拷贝蛋白序列与从NCBI和Ensembl数据库检索到的代表性脊椎动物的MRC1氨基酸序列进行系统发育分析,包括人、小鼠、原鸡、绿头鸭、蜥蜴、非洲爪蟾、青鳉、三刺鱼、大黄鱼、河鲀、尼罗罗非鱼、大西洋鳕鱼、红鳍东方鲀、斑马鱼和鲤15个物种的28条氨基酸序列。利用MEGA 11软件ClustalW2程序的默认参数对氨基酸序列补齐并进行多序列比对,使用基于JTT矩阵的方法计算进化距离,位点间的速率变化采用γ分布构建Neighbor-Joining (NJ)树,并采用1 000次重复抽样来评估系统发生树(Tamura et al, 2021)。
1.4 MRC1基因在健康鲤不同组织中的表达为探究MRC1基因各拷贝在健康鲤组织中的表达情况,采集40尾黄河鲤的脑、肌肉、肝脏和脾脏组织,使用RNeasy试剂盒(Qiagen)提取RNA,并委托测序公司利用HiSeq2000测序平台进行150PE的转录组测序。原始转录组测序数据以fastaq.gz的文件格式储存,对测序序列进行质量控制便于后续差异基因及表达量的分析,首先使用Bowtie2软件(https://bowtie-bio.sourceforge.net/index.shtml)对所选的鲤参考基因组建立索引,使用Tophat2 (http://ccb.jhu.edu/software/tophat/index.shtml)将质控后的reads比对到参考基因组上,使用Cufflinks (http://cole-trapnell-lab.github.io/cufflinks/)进行基因表达值(FPKM)的计算(Trapnell et al, 2012),随后提取每个样本中MRC1各拷贝的表达数据,使用R软件的ggplot2扩展包可视化展示(http://cran.r-project.org/package=ggplot2)。
1.5 MRC1基因在鲤感染嗜水气单胞菌后不同时期脾脏中的表达分析本课题组在前期进行了鲤感染嗜水气单胞菌的攻毒实验(Jiang et al, 2016),具体过程如下:将体重为(180±25) g、体长为(15±3) cm的健康鲤随机分为实验组和对照组,对实验组腹腔注射100 μL浓度为1×108 CFU/mL的嗜水气单胞菌菌液,对照组仅注射同等剂量的无菌PBS。注射完成后开始计时,分别在4、12、24 h采集脾脏组织。实验组和对照组各设3组重复,每个时间点分别随机取9条鱼。使用RNeasy试剂盒(Qiagen)提取RNA,并委托测序公司进行转录组测序。转录组数据已提交到NCBI SRA数据库,收录号为SRP072018。数据通过质量控制后进行基因表达值(FPKM)的计算。利用Cuffdiff (http://cole-trapnell-lab.github.io/cufflinks/announcements/cuffdiff2-paper/)对测序reads进行计数并计算各拷贝的表达量,同时Cuffdiff还可以计算出不同时间点各拷贝的统计学差异,其主要是通过RSEM (RNA-Seq by Expectation-Maximization,期望最大化)算法对转录本进行水平定量,筛选|log2(fold change)|>1.5且校正后P<0.05的拷贝进行保留。
2 结果 2.1 鲤MRC1基因的鉴定和蛋白结构预测通过搜索基因组数据并重新注释,在鲤全基因组范围共鉴定出11条MRC1基因序列,其中,在鲤3、4、7、8、14、31号染色体上各找到1条序列,42号染色体上找到2条,13号染色体上找到3条。鲤MRC1基因各拷贝的信息详见表 1。进而对MRC1基因11个拷贝的蛋白结构域进行了预测,结果如图 1所示,其中,HHLG3g0497、HHLG4g0565和HHLG13g0210的蛋白结构高度相似,包含甘露糖受体蛋白的关键结构域,即8个串联排列的CRD结构域和1个FN2结构域,该结果与大黄鱼MRC1结构域预测相一致;HHLG42g0001的预测结构包含1个信号肽、12个CRD结构域、1个FA58C结构域和1个LCCL结构域;而其他各拷贝预测到2~8个数目不等的CRD结构域。
|
|
表 1 鲤MRC1基因各拷贝具体信息 Tab.1 Detailed information of MRC1 gene in Cyprinus carpio |

|
图 1 鲤MRC1基因不同拷贝蛋白结构域分析 Fig.1 Protein domain prediction of MRC1 in C. carpio |
MRC1基因在鲤基因组中存在多拷贝现象,与人、小鼠、原鸡、绿头鸭、蜥蜴、非洲爪蟾、青鳉、红鳍东方鲀、斑马鱼等15个代表性物种的MRC1基因拷贝数进行比较,发现在不同物种中MRC1基因出现了不同程度的多拷贝现象。如表 2所示,MRC1基因在大部分的物种中为单拷贝,在硬骨鱼中的三刺鱼、大西洋鳕鱼和斑马鱼中存在2个拷贝,而在鲤中则存在多达11个拷贝。
|
|
表 2 鲤和其他物种MRC1基因拷贝数比较分析 Tab.2 Copy number of MRC1 gene of C. carpio and other species |
为了更好地理解不同物种MRC1基因的同源性以及在基因组上的保守性,对人、小鼠、原鸡、爪蟾、蜥蜴、红鳍东方鲀、斑马鱼和鲤8个物种的MRC1基因进行共线性分析(图 2)。由于鲤染色体和斑马鱼染色体呈现典型的2∶1同源共线性关系,优先选择对应斑马鱼2号染色体MRC1-b基因所在染色体的鲤3、4号染色体上结构完整且氨基酸序列较长的HHLG3g0497和HHLG4g0565这2个拷贝来分析。从分析结果可以看出,MRC1基因在进化上具有相对较高的保守性。与MRC1基因关联性最强的是钙离子通道B2亚基基因(cacnb2),存在于人、小鼠、原鸡、爪蟾、蜥蜴、红鳍东方鲀、斑马鱼和鲤8个物种中,并且cacnb2基因在鲤和斑马鱼基因组上处于一致的位置,都位于MRC1基因上游。而在其他物种中cacnb2基因位于MRC1基因下游,这可能是在进化过程中基因出现了易位。C型凝集素受体12基因(colec12)、cacnb2、非典型趋化因子受体4a基因(ackr4a)、转录因子f5基因(e2f5)均存在于鲤和斑马鱼MRC1基因上下游。另外,位于鲤4号染色体上的MRC1上下游的基因分布和斑马鱼上基因分布高度相似。
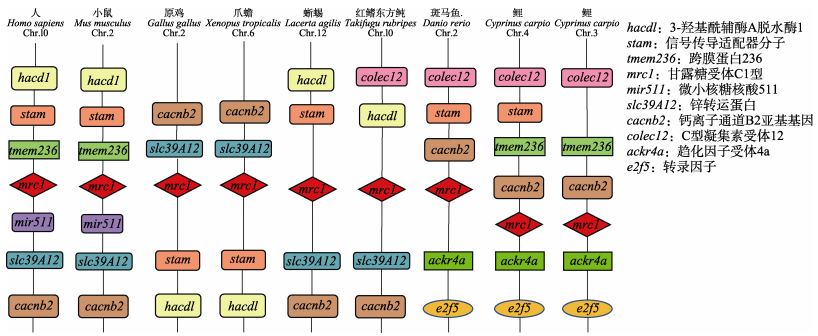
|
图 2 MRC1基因共线性分析 Fig.2 Synteny of genes neighboring MRC1 |
从NCBI和Ensembl数据库检索到的几种代表性物种的MRC1氨基酸序列进行多序列比对和系统发育分析,包括人、小鼠、原鸡、绿头鸭、蜥蜴、非洲爪蟾、青鳉、三刺鱼的MRC1a,大黄鱼、河鲀、尼罗罗非鱼、大西洋鳕鱼的MRC1a,红鳍东方鲀、斑马鱼的MRC1a,及鲤鱼的HHLG3g0497,总计15个物种的28条蛋白序列。多序列比对结果显示(图 3),15个物种的MRC1氨基酸长度大体一致,其中,与鲤MRC1蛋白一致性最高的是斑马鱼(81.98%),一致性最低的是蜥蜴(47.58%)。

|
图 3 鲤MRC1蛋白与其他物种的序列比对 Fig.3 Alignment of MRC1 among C. carpio and other species |
系统进化分析一定程度上反映MRC1基因在整个物种进化过程中的差异,利用MEGA 11.0软件构建系统进化树。如图 4所示,进化树包括哺乳类、鸟类、爬行类、两栖类和鱼类5个分支,各分支依次聚集在一起。其次鸟类中的鸡和绿头鸭、哺乳类中的人和小鼠亲缘关系最近聚在一起。硬骨鱼类中,鲤3、4号染色体上结构完整的HHLG3g0497、HHLG4g0565两个拷贝与大西洋鳕鱼的MRC1b拷贝、斑马鱼的MRC1b拷贝、三刺鱼的MRC1b拷贝聚集为一个亚分支。鲤HHLG13g0210拷贝与斑马鱼的MRC1a拷贝聚为一支,同时和其他硬骨鱼类聚成一个亚分支。
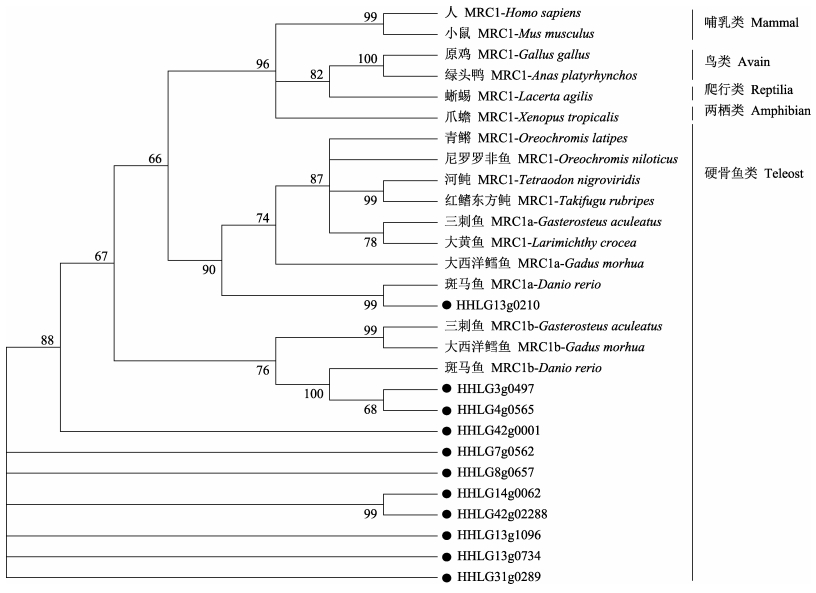
|
图 4 MRC1蛋白的系统进化树 Fig.4 Phylogenetic tree of MRC1 proteins |
MRC1在健康黄河鲤脑和肌肉组织中各拷贝表达情况高度相似,表达较高的分别是HHLG3g0497、HHLG4g0565、HHLG13g0210和HHLG42g0001,其他7个拷贝在健康鲤脑和肌肉组织中表达量非常低或者不表达。在肝脏组织中,表达量较高的拷贝分别为HHLG3g0497、HHLG4g0565、HHLG13g0210和HHLG31g0289。在脾脏组织中,HHLG3g0497、HHLG4g0565、HHLG13g0210和HHLG13g0734拷贝的表达量较高(图 5)。总体来说,MRC1基因在健康黄河鲤脾脏中的表达量相对高于在其他3种组织中的表达量。

|
图 5 MRC1基因在健康鲤脑(A)、肝脏(B)、肌肉(C)和脾脏(D)组织中的表达 Fig.5 Expression of MRC1 genes in healthy C. carpio brain (A), liver (B), muscle (C) and spleen (D) |
通过使用Cuffdiff计算转录本的表达量,同时计算出MRC1基因各拷贝在感染嗜水气单胞菌4、12、24 h后的表达差异,筛选出显著差异表达的拷贝。结果发现(图 6),HHLG13g0734在感染细菌4 h后显著上调(P<0.05),感染24 h后表现出极显著的上调(P<0.001);而HHLG3g0497在感染细菌4、12、24 h后表现出极显著的下调(P<0.001)。
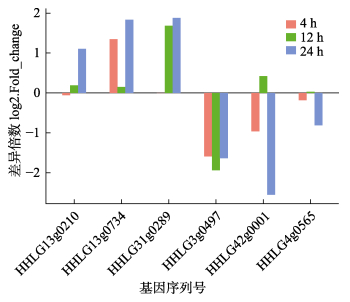
|
图 6 感染嗜水气单胞菌4、12、24 h后不同拷贝的表达分析 Fig.6 Expression analysis of MRC1 genes at 4 h, 12 h and 24 h after Aeromonas hydrophila infection |
嗜水气单胞菌是一种常见的革兰氏阴性菌,也是典型的人–畜–鱼共患病病原菌,可以侵染人类、爬行动物和鱼类,引起多种水产动物的败血症(Boulanger et al, 1977; Pasquale et al, 1994)和人类的腹泻(Davis et al, 1978)。甘露糖受体(MR)是一种C型凝集素,也是一种模式识别受体,能够识别各种病原体的表面多糖,在先天免疫和获得性免疫中都发挥重要作用(Gazi et al, 2009)。Sallusto等(1995)证明人类外周血单核细胞会发展成树突状细胞向T细胞提供可溶性抗原,在确定抗原捕获机制时发现MR在低密度配体情况下连续向细胞输送大量配体,表明MR的特异性和对炎症刺激的反应能力提高了免疫过程中抗原呈递的能力。同时有研究表明,人类甘露糖受体C1型(hMRC1)可以增强HIV-1与巨噬细胞的结合并有助于抑制HIV-1病毒的释放(Nguyen et al, 2003; Sukegawa et al, 2018)。目前,在鱼类中克隆了模式物种和几种经济物种的MRC1基因全长,但对其抗病机制以及在抗炎症过程中的作用研究较少。为了深入探究鲤MRC1基因的结构和功能特点及其在感染嗜水气单胞菌过程中的表达情况,本研究从鲤基因组中鉴定得到11个MRC1基因拷贝,并分析了序列结构、系统进化及其在进化过程中的保守程度,同时还探究了各拷贝在健康鲤各组织以及受嗜水气单胞菌感染后不同阶段的表达情况。
不同物种MRC1的多拷贝现象和共线性结果体现了硬骨鱼类和其他物种之间的进化差异。基因多拷贝是基因和基因组复制的结果。在物种进化过程中,基因和基因组复制被认为对物种多样性起着重要作用。脊椎动物早期进化经历了两轮全基因组复制,为多样性提供了原材料(Zhang, 2003)。比较基因组学研究发现,硬骨鱼的辐鳍鱼纲发生了第3次基因组复制即鱼类特异性基因组重复(Meyer et al, 2005)。鲤是一种异源四倍体硬骨鱼类,除经历了硬骨鱼类共有的第3轮全基因组复制事件外,还经历了特有的第4轮全基因组复制事件。最新的研究表明,鲤第4轮全基因组复制事件大约发生在820万年前,全基因组复制直接导致基因拷贝的增加(Xu et al, 2019)。本研究发现,MRC1基因在多种硬骨鱼包括其他鲤科鱼类中出现多拷贝现象,支持硬骨鱼类较四足动物多经历了一轮基因组加倍的观点。其中,鲤MRC1基因在全基因组范围多达11个拷贝,与异源四倍体鲤鱼经历了特有的第4轮全基因组复制事件的观点相一致。共线性分析结果发现,位于鲤4号染色体的MRC1基因HHLG4g0565上下游的基因分布与斑马鱼几乎一致,colec12、cacnb2、ackr4a和e2f5均存在于鲤和斑马鱼MRC1基因上下游,距离鲤MRC1基因上游最近的是cacnb2和tmem236,cacnb2也是人、小鼠、蜥蜴、红鳍东方鲀、斑马鱼共有的基因,表明基因在基因组复制过程中可能会出现一定程度的差异保留和丢失(Brunet et al, 2006)。与MRC1基因关联较强的基因中,ackr4a同样具有免疫相关功能,参与鱼类抵御细菌感染的先天免疫反应,能有效抑制下游炎症因子(IL-1β和TNF-α)的表达(陈亚, 2022),而另一基因colec12也参与宿主对病原体的防御反应(Suzuki et al, 1997)以及细菌感染后宿主的免疫识别和免疫清除(Feng et al, 2016),表明MRC1基因所在区域可能形成了一定的基因功能区,参与机体的免疫反应。
鲤MRC1基因各个拷贝在相同组织中的表达各有差异,表明各拷贝可能具有不同的功能。其中,HHLG3g0497、HHLG4g0565、HHLG13g0210等拷贝在不同组织中均高表达,推测这几个基因可能广泛存在于鲤各组织中,参与维持机体内环境稳态。此外,各基因拷贝在脾脏中的表达量相对高于其他组织。脾脏是鱼类主要的免疫器官之一,由此推测,鲤MRC1基因极有可能参与机体的免疫反应。在其他鱼类中也得到相似结果,比如团头鲂MRC1在主要免疫相关组织脾脏和头肾中的表达明显高于其他组织(Liu et al, 2014),感染嗜水气单胞菌后,MRC1在脾脏中显著上调(Wu et al, 2018)。MRC1基因在感染了弧菌的大黄鱼脾脏中同样显著上调表达(Dong et al, 2016)。本研究进一步检测了鲤MRC1基因各拷贝在感染嗜水气单胞菌后不同时期的脾脏中的表达,并进行了差异比较分析。结果发现,不同的拷贝表现不同的表达特征,比如HHLG3g0497在感染细菌4、12、24 h持续表现出极显著下调,而HHLG13g0734在感染细菌4 h后显著上调,12 h时表达差异不明显,24 h时再次表现出极显著上调。这个结果暗示HHLG3g0497和HHLG13g0734两个基因拷贝可能参与了鲤抵抗嗜水气单胞菌感染的免疫反应。我们推测,这可能是由于在进化过程中MRC1经全基因组复制产生的各个基因拷贝出现了亚功能化或去功能化(Guo et al, 2011),其中只有部分拷贝保留了免疫相关功能。
本研究初步揭示了鲤MRC1的多拷贝现象,为更深入地了解MRC1基因在鲤感染嗜水气单胞菌后的免疫反应中的作用提供了分子基础,为嗜水气单胞菌引起的鱼类疾病防控策略的制定提供了基础理论参考。但由于对各拷贝在细菌感染过程中具体引起了哪些抗炎症因子的变化并不清楚,因此,后续需要进一步探究感染过程中各拷贝和抗炎症因子(如IL-1β和TNF-α等)的时空表达及相互作用。
BEYER D, THOMAS A H, RANJIT J, et al. The software model checker Blast. International Journal on Software Tools for Technology Transfer, 2007, 9(5/6): 505-525 |
BOULANGER Y, LALLIER R, COUSINEAU G. Isolation of enterotoxigenic Aeromonas from fish. Canadian Journal of Microbiology, 1977, 23(9): 1161-1164 DOI:10.1139/m77-174 |
BRUNET F G, HUGUES R C, PARIS M, et al. Gene loss and evolutionary rates following whole-genome duplication in teleost fishes. Molecular Biology and Evolution, 2006, 23(9): 1808-1816 DOI:10.1093/molbev/msl049 |
CHEN C J, CHEN H, ZHANG Y, et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202 DOI:10.1016/j.molp.2020.06.009 |
CHEN Y. Regulation mechanisms of the Miichthys miiuy eIF3k and ackr4a-mediated autophagy in NF-κB signaling. Doctoral Dissertation of Shanghai Ocean University, 2022 [陈亚.  鱼eIF3k和ackr4a介导的自噬在调控NF-κB信号通路中的机制研究. 上海海洋大学博士研究生学位论文, 2022] 鱼eIF3k和ackr4a介导的自噬在调控NF-κB信号通路中的机制研究. 上海海洋大学博士研究生学位论文, 2022]
|
DAVIS W A, KANE J G, GARAGUSI V F, et al. Human Aeromonas infections: A review of the literature and a case report of endocarditis. Medicine, 1978, 57(3): 267-277 DOI:10.1097/00005792-197805000-00006 |
DONG X L, LI J J, HE J Y, et al. Anti-infective mannose receptor immune mechanism in large yellow croaker (Larimichthys crocea). Fish and Shellfish Immunology, 2016, 54: 257-265 DOI:10.1016/j.fsi.2016.04.006 |
FAO. The state of world fisheries and aquaculture 2020. Sustainability in Action, 2020
|
FENG S S, JIANG Y L, ZHANG S H, et al. Genome wide identification of scavenger receptors class A in common carp (Cyprinus carpio) and their expression following Aeromonas hydrophila infection. Fish and Shellfish Immunology, 2016, 54: 60-67 DOI:10.1016/j.fsi.2016.03.156 |
FONTENLA F, NOIA M, LEIRO J M, et al. The turbot macrophage mannose receptor: Phylogenetic analysis, functional characterization and changes in gene expression during vaccination and infection with Philasterides dicentrarchi. Fish and Shellfish Immunology, 2016, 53: 121-122 |
GAZI U, MARTINEZ-POMARES L. Influence of the mannose receptor in host immune responses. Immunobiology, 2009, 214(7): 554-561 DOI:10.1016/j.imbio.2008.11.004 |
GUO B C, WAGNER A, HE S. Duplicated gene evolution following whole-genome duplication in teleost fish. Gene Duplication, 2011, 27–36
|
JIANG Y L, FENG S S, ZHANG S H, et al. Transcriptome signatures in common carp spleen in response to Aeromonas hydrophila infection. Fish and Shellfish Immunology, 2016, 57: 41-48 DOI:10.1016/j.fsi.2016.08.013 |
JONES B L, WILCOX M H. Aeromonas infections and their treatment. Journal of Antimicrobial Chemotherapy, 1995, 35(4): 453-461 DOI:10.1093/jac/35.4.453 |
KERRIGAN A M, BROWN G D. C-type lectins and phagocytosis. Immunobiology, 2009, 214(7): 562-575 DOI:10.1016/j.imbio.2008.11.003 |
LIU X L, TANG X C, WANG L, et al. Molecular cloning and expression analysis of mannose receptor in blunt snout bream (Megalobrama amblycephala). Molecular Biology Reports, 2014, 41(7): 4601-4611 DOI:10.1007/s11033-014-3331-2 |
MEYER A, VAN DE PEER Y. From 2R to 3R: Evidence for a fish-specific genome duplication (FSGD). Bioessays, 2005, 27(9): 937-945 DOI:10.1002/bies.20293 |
NAPPER C E, DYSON M H, TAYLOR M E. An extended conformation of the macrophage mannose receptor. Journal of Biological Chemistry, 2001, 276(18): 14759-14766 DOI:10.1074/jbc.M100425200 |
NGUYEN D G, HILDRETH J E K. Involvement of macrophage mannose receptor in the binding and transmission of HIV by macrophages. European Journal of Immunology, 2003, 33(2): 483-493 DOI:10.1002/immu.200310024 |
PACHANAWAN A, PHUMKHACHORN P, RATTANACHAI-KUNSOPON P. Potential of Psidium guajava supplemented fish diets in controlling Aeromonas hydrophila infection in tilapia (Oreochromis niloticus). Journal of Bioscience and Bioengineering, 2008, 106(5): 419-424 DOI:10.1263/jbb.106.419 |
PASQUALE V, BALODA S B, DUMONTET S, et al. An outbreak of Aeromonas hydrophila infection in turtles (Pseudemis scripta). Applied and Environmental Microbiology, 1994, 60(5): 1678-1680 DOI:10.1128/aem.60.5.1678-1680.1994 |
SALAMOV A A, SOLOVYEV V V. Ab initio gene finding in Drosophila genomic DNA. Genome Research, 2000, 10(4): 516-522 DOI:10.1101/gr.10.4.516 |
SALLUSTO F, CELLA M, DANIELI C, et al. Dendritic cells use macropinocytosis and the mannose receptor to concentrate macromolecules in the major histocompatibility complex class Ⅱ compartment: Downregulation by cytokines and bacterial products. Journal of Experimental Medicine, 1995, 182(2): 389-400 DOI:10.1084/jem.182.2.389 |
SONG C, WANG Y. Microsynteny analysis of tomato and peach genome. Hereditas, 2010, 32(9): 966-973 [宋弛, 王瑛. 番茄和桃基因组的微同线性研究. 遗传, 2010, 32(9): 966-973] |
STAHL P D, EZEKOWITZ R A B. The mannose receptor is a pattern recognition receptor involved in host defense. Current Opinion in Immunology, 1998, 10(1): 50-55 DOI:10.1016/S0952-7915(98)80031-9 |
STRATEV D, DASKALOV H, VASHIN I. Characterisation and determination of antimicrobial resistance of beta-haemolytic Aeromonas spp isolated from common carp (Cyprinus carpio L.). Revue De Medecine Veterinaire, 2015, 166(1/2): 54-61 |
SUKEGAWA S, MIYAGI E, BOUAMR F, et al. Mannose receptor 1 restricts HIV particle release from infected macrophages. Cell Reports, 2018, 22(3): 786-795 DOI:10.1016/j.celrep.2017.12.085 |
SULEK J, WAGENAAR R A, SHIREMAN J, et al. Increased expression of the collagen internalization receptor uPARAP/ Endo180 in the stroma of head and neck cancer. Journal of Histochemistry and Cytochemistry, 2007, 55(4): 347-353 DOI:10.1369/jhc.6A7133.2006 |
SUZUKI H, KURIHARA Y, TAKEYA M, et al. A role for macrophage scavenger receptors in atherosclerosis and susceptibility to infection. Nature, 1997, 386(6622): 292-296 DOI:10.1038/386292a0 |
TACHADO S D, ZHANG J M, ZHU J P, et al. Pneumocystis-mediated IL-8 release by macrophages requires coexpression of mannose receptors and TLR2. Journal of Leukocyte Biology, 2007, 81(1): 205-211 DOI:10.1189/jlb.1005580 |
TALWAR C, NAGAR S, LAL R, et al. Fish gut microbiome: Current approaches and future perspectives. Indian Journal of Microbiology, 2018, 58(4): 397-414 DOI:10.1007/s12088-018-0760-y |
TAMURA K, STECHER J, KUMAR S. MEGA11: Molecular evolutionary genetics analysis version 11. Molecular Biology and Evolution, 2021, 38(7): 3022-3027 DOI:10.1093/molbev/msab120 |
TRAPNELL C, POBERTS A, GOFF L, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature Protocols, 2012, 7(3): 562-578 DOI:10.1038/nprot.2012.016 |
WANG L, LIU L C, ZHOU Y, et al. Molecular cloning and expression analysis of mannose receptor C type 1 in grass carp (Ctenopharyngodon idella). Developmental Comparative Immunology, 2014, 43(1): 54-58 DOI:10.1016/j.dci.2013.10.006 |
WU C S, ZHAO X H, BABU V S, et al. Distribution of mannose receptor in blunt snout bream (Megalobrama amblycephala) during the embryonic development and its immune response to the challenge of Aeromonas hydrophila. Fish and Shellfish Immunology, 2018, 78: 52-59 DOI:10.1016/j.fsi.2018.03.049 |
XU P, XU J, LIU G J, et al. The allotetraploid origin and asymmetrical genome evolution of the common carp Cyprinus carpio. Nature Communications, 2019, 10: 4625 DOI:10.1038/s41467-019-12644-1 |
XU X L, XIE Q M, SHEN Y H, et al. Involvement of mannose receptor in the preventive effects of mannose in lipopolysaccharide-induced acute lung injury. European Journal of Pharmacology, 2010, 641(2/3): 229-237 |
YAMAMOTO Y, KLEIN T W, FRIEDMAN H. Involvement of mannose receptor in cytokine interleukin-1beta (IL-1beta), IL-6, and granulocyte-macrophage colony-stimulating factor responses, but not in chemokine macrophage inflammatory protein 1beta (MIP-1beta), MIP-2, and KC responses, caused by attachment of Candida albicans to macrophages. Infection and Immunity, 1997, 65(3): 1077-1082 DOI:10.1128/iai.65.3.1077-1082.1997 |
YIN L C, XU W T, LIU X L, et al. Autophagy promotes innate defense against Aeromonas hydrophila in grass carp (Ctenopharyngodon idella) monocytes or macrophages. Aquaculture, 2021, 535: 736391 DOI:10.1016/j.aquaculture.2021.736391 |
ZHANG J M, TACHADO S D, PATEL N, et al. Negative regulatory role of mannose receptors on human alveolar macrophage proinflammatory cytokine release in vitro. Journal of Leukocyte Biology, 2005, 78(3): 665-674 DOI:10.1189/jlb.1204699 |
ZHANG J Z. Evolution by gene duplication: an update. Trends in Ecology and Evolution, 2003, 18(6): 292-298 DOI:10.1016/S0169-5347(03)00033-8 |
ZHENG F F, ASIM M, LAN J F, et al. Molecular cloning and functional characterization of mannose receptor in zebrafish (Danio rerio) during infection with Aeromonas sobria. International Journal of Molecular Sciences, 2015, 16(5): 10997-11012 |
ZHOU X Z, YAO S H, LI J W, et al. Analyses of gene duplication and synteny in Melampsora larici-populina (Pucciniales, Basidiomycota). Mycosystema, 2021, 40(3): 580-591 [周显臻, 姚诗幻, 李嘉雯. 落叶松-杨栅锈菌基因复制事件及共线性分析. 菌物学报, 2021, 40(3): 580-591] |



