2. 中国水产科学研究院黄海水产研究所 海水养殖生物育种与可持续产出全国重点实验室 青岛海洋科技中心海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, State Key Laboratory of Mariculture Biobreeding and Sustainable Goods; Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao Marine Science and Technology Center, Qingdao 266071, China
生物钟包括视交叉上核(suprachiasmatic nucleus, SCN)中枢生物钟与各组织外周生物钟(Yu, 2017; Khan et al, 2016)。生物钟基因(BioClock genes)广泛存在于真核生物的神经中枢和外周组织中,参与调节生物机体众多生理活动。真核生物的诸多生理活动存在周期性,这是由于内源性节律变化的存在,这种节律变化统称为生物学节律。生物钟核心环路由Clock: Bmal组成的正调节因子和Per: Cry组成的负调节因子构成,呈现出核心生物钟的负反馈环路调节,继而影响松果体(pineal gland, PG)中褪黑素(melatonin, MLT)的合成节律,MLT又通过影响“脑/下丘脑–脑垂体–性腺”轴(brain/hypophysis-pituitary-gonad axis, BPG轴)中的下丘脑促性腺激素释放激素(gonadotropin-releasing hormone, GnRH)的分泌,GnRH调节垂体前叶远端部(pars distalis, PD)促黄体激素(luteinizing hormone, LH)和促卵泡激素(follicle-stimulating hormone, FSH)的分泌,最终导致鱼类及其他动物的生殖机能呈现周期性变化(Keisuke et al, 2016; Saha et al, 2019; Zhang D et al, 2022)。
随着生物钟基因在生物机体内的作用逐渐被揭示,关于生物钟基因在生物繁殖过程中的作用引起了国内外相关领域研究者的重视。目前,对生物钟基因节律变化的研究以日变化规律为主。诸多研究发现,生物钟基因在哺乳类、禽类、鱼类和甲壳类的性腺中的表达存在着日节律(Takashi et al, 2000; Chen et al, 2017; Saha et al, 2019)。随着季节的更替,一年中的日照时长、温度等季节性因素有着规律性的变化,生物钟基因的表达也存在着相应的季节性变化规律。在旗鱼(Xiphias gladius)、大西洋鲑鱼(Salmo salar)、日本沼虾(Macrobrachium nipponense)、日本鹌鹑(Coturnix japonica)、马岗鹅、小鼠(Mus musculus)、苏尼特羊(Ovis aries)的卵巢中,均发现生物钟基因的表达存在季节性变化规律(Davie et al, 2009; Wharfe et al, 2011; Chen et al, 2017; Takashi et al, 2017; 劳永聪等, 2019; 向光明等, 2019a、b; Basili et al, 2021)。Wharfe等(2011)研究发现,非繁殖期小鼠卵巢中Clock、Bmal、Cry1、Cry2、Per1和Per2基因表达量高于繁殖期,并证明了生物钟基因在性成熟小鼠卵巢中的表达存在月周期性规律,同时,发现母体皮质酮的分泌与Clock、Bmal1和Per2的表达密切相关,推测生物钟基因与蛋白质、脂肪和糖类的代谢存在关联。向光明等(2019a、b)通过对比季节性发情的苏尼特羊和常年发情的小尾寒羊(O. aries)卵巢中Per1、Per2基因的表达差异发现,生物钟基因在苏尼特羊卵巢中存在季节性表达规律,同时,推测Per1、Per2基因的高表达不利于绵羊的发情。Basili等(2021)通过对繁殖季节(6—8月)和非繁殖季节(10—12月)捕获的雌性旗鱼进行研究,发现非繁殖季节的旗鱼卵巢组织中Clock、Bmal1、Cry2和Per2基因的表达量高于繁殖季节,且生物钟基因的季节性表达可调节MLT的表达量,证明生物钟基因可参与调节旗鱼卵巢的成熟,同时证明生物钟基因在鱼类卵巢组织中的表达也存在着季节性变化规律,但受到旗鱼习性影响,该研究未能对性成熟旗鱼卵巢组织的完整发育周期中生物钟基因的表达规律进行探究。
半滑舌鳎(Cynoglossus semilaevis)是我国北方名贵的海水养殖鱼类。众多研究表明,鱼类的繁殖主要受具有周期性的光照和温度等因素的影响;在繁殖过程中,生物钟基因也存在周期性表达。关于生物钟基因与鱼类繁殖调控机制之间的研究逐渐兴起(Herrero et al, 2014; Pérez et al, 2019)。本研究以半滑舌鳎为研究对象,克隆其生物钟核心环路的正负调节因子(Clock1a、Bmal1a、Cry1a、Cry2和Per2)基因,并分析其在Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ期卵巢组织中的表达变化规律,以期丰富半滑舌鳎等海水硬骨鱼类繁育理论,并更好地提高半滑舌鳎的繁育技术与苗种生产效率。
1 材料与方法 1.1 实验用鱼及样品处理实验用半滑舌鳎取自本实验室在山东省日照市的培育基地,卵巢发育不同时期所用半滑舌鳎选自300尾人工养殖达到性成熟(3龄以上)的雌鱼,体重为(1 795.7±82.5) g。实验用鱼的培育条件:在室内水泥池中(5 m ×5 m×1 m)流水培育(日换水率达到600%),水温为8~25 ℃,盐度为28~31,pH为7.8~8.4,溶解氧(DO)为5 mg/L以上,每日光照14 h,每日饲喂2次虾和干饲料。人工控制水温和光照周期来调控亲鱼性腺发育成熟,不同繁殖期在相同亲鱼培育池中随机选取4尾半滑舌鳎雌鱼,连续采集1年。解剖前先用240 mg/L的MS-222麻醉,通过将塑料管插入泄殖孔取少量卵母细胞进行活检,初判卵巢的发育阶段(Shi et al, 2016)。在卵巢发育的各个阶段,选取符合采样要求的4尾半滑舌鳎雌鱼,采用MS-222 (240 mg/L)将半滑舌鳎雌鱼麻醉后迅速进行解剖,切取适量半滑舌鳎卵巢组织放入无RNA酶EP管中并立即放入液氮中,转至实验室置于超低温冰箱(−80 ℃)中保存留用。留取每个个体适量的卵巢中段,固定在Bouin´s固定液中。根据本实验室半滑舌鳎卵巢发育期判定标准对所取卵巢组织样品所处发育阶段进行判断(Shi et al, 2015、2016)。
1.2 总RNA提取和cDNA第一链的合成取保存于–80 ℃的Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ期半滑舌鳎卵巢组织,参照艾科瑞生物的RNAex Pro RNA提取试剂(AG21101)操作说明提取卵巢组织的总RNA,通过NanoDrop 2000C超微量分光光度计测定RNA的纯度和浓度,确保OD260 nm/OD280 nm值在1.8~2.0之间,RNA浓度在300~500 ng/μL之间。通过1%琼脂糖凝胶电泳检验RNA的完整性,28S条带亮度达到18S条带的2倍则为纯度高且完整的RNA提取样品。将装有符合要求的RNA样品的离心管短暂离心后置于–80 ℃保存。
取1 μg Ⅲ期卵巢组织的总RNA,参照TaKaRa公司的PrimerScripyTM 1st Strand cDNA Synthesis反转录试剂盒(6110A)说明,反转录成cDNA模板,置于–20 ℃保存备用。
1.3 Clock1a、Bmal1a、Cry1a、Cry2和Per2基因克隆和序列测定在NCBI的GenBank数据库中搜索鱼类Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的同源cDNA序列,并分析Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的编码区和保守区,采用Primer 6.0设计特异性引物(表 1)。使用Bio-Rad S1000 PCR仪(伯乐,新加坡),按照TaKaRa公司的TaqTM HS Perfect Mix (R300A)操作说明进行克隆。PCR扩增体系:TaKaRa Taq HS Perfect Mix (2×) 12 μL,cDNA模板1 μL,上下游引物各1 μL,灭菌水补至25 μL。PCR扩增条件:95 ℃ 5 min,之后35个循环(95 ℃ 5 s,Tm 30 s,72 ℃,1.5 min),72 ℃ 5 min,最后4℃保存。Clock1a、Bmal1a、Cry1a、Cry2和Per2的Tm分别为51、54、52、53和53 ℃。
|
|
表 1 半滑舌鳎生物钟基因克隆所用引物 Tab.1 Cloning primer of BioClock genes in C. semilaevis |
半滑舌鳎Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的扩增产物经1%琼脂糖凝胶电泳分离后,使用北京天根生化的TIANgel Midi Purification Kit (DP209)对目的片段进行回收并纯化。回收的Clock1a、Bmal1a、Cry1a、Cry2和Per2的PCR产物与TaKaRa公司的pMDTM19-T Vector载体(D102A)连接,转化至T感受态细胞中,挑取其中的阳性克隆,送至睿博兴科生物技术有限公司测序。
1.4 基因序列分析使用DNAMAN 6.0软件将测序所得的序列片段进行比对拼接,获得半滑舌鳎Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的完整编码区(ORF)序列并推导其氨基酸序列;利用NCBI数据库(https://www.ncbi.nlm.nih.gov/)进行结构域的预测;利用Jalview进行多重序列比对;利用DNAMAN 6.0进行序列同源性分析;利用在线软件ExPASy (http://web.expasy.org/compute_pi/)推断氨基酸序列的分子量及等电点;利用软件MEGA7通过邻接法(NJ)构建进化树,1 000次自举(bootstrap)重复检验进化树的置信度。
1.5 Clock1a、Bmal1a、Cry1a、Cry2和Per2基因在繁殖周期的表达分析以克隆获得的半滑舌鳎Clock1a、Bmal1a、Cry1a、Cry2和Per2基因序列为模板,设计定量引物(表 2),同时,以半滑舌鳎18S基因作为内参基因,合成其特异性引物(表 2),引物由北京睿博兴科生物技术有限公司合成。使用Mastercycler ep realplex Real-time PCR仪(Eppendorf),按照TaKaRa Ex Taq®(Mg2+ free Buffer) (TaKaRa)操作说明进行PCR扩增。qRT-PCR扩增体系:TaKaRa Ex Taq (5 U/μL) 0.125 μL,10×Ex Taq Buffer (Mg2+ free) 2.5 μL,MgCl2 (25 mmol/L) 2 μL,dNTP Mixture (各2.5 mmol/L) 2 μL,cDNA模板1 μL,上下游引物各1 μL,灭菌水补至25 μL。qRT-PCR扩增条件:95 ℃预变性30 s,之后40个循环(95 ℃ 5 s,Tm 30 s,72 ℃ 30 s),Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的Tm分别为50.5、53.6、52.3、53和52.5 ℃。以18S作为内参基因对样品中RNA的量进行校正。程序运行结束后,对熔解曲线进行分析,检验产物特异性。实验样品设3个平行,并设阴性对照(cDNA模板用灭菌水代替),以确定实验结果的可靠性。
|
|
表 2 半滑舌鳎生物钟基因表达分析所用引物 Tab.2 Primers for expression of BioClock genes in C. semilaevis |
基因相对表达量参照2–ΔΔCt (Livak et al, 2001)法计算,结果以平均值±标准误(Mean±SE)表示。采用SPSS 26进行单因素方差分析(one-way ANOVA)与Duncan多重比较,P < 0.05为差异显著。
2 结果与分析 2.1 半滑舌鳎Clock1a和Bmal1a的序列和结构特征从半滑舌鳎卵巢内分离得到了Clock1a和Bmal1a基因cDNA序列,并分别获得了NCBI登录号OQ054330和OQ054331。Clock1a基因的编码区序列长为2 139 bp,编码712个氨基酸,分子量为81.9 kDa,等电点为6.35,序列中存在保守性较强的PAS11 (由271~373位的103个氨基酸组成)、PASD1 (由28~91位的64个氨基酸组成)结构域(图 1)。Bmal1a基因的编码区序列长为1 881 bp,编码626个氨基酸,分子量为68.9 kDa,等电点为6.35,序列中存在保守性较强的PAS11 (由71~134位的63个氨基酸组成)、PASD3 (由343~446位的103个氨基酸组成)结构域(图 2)。
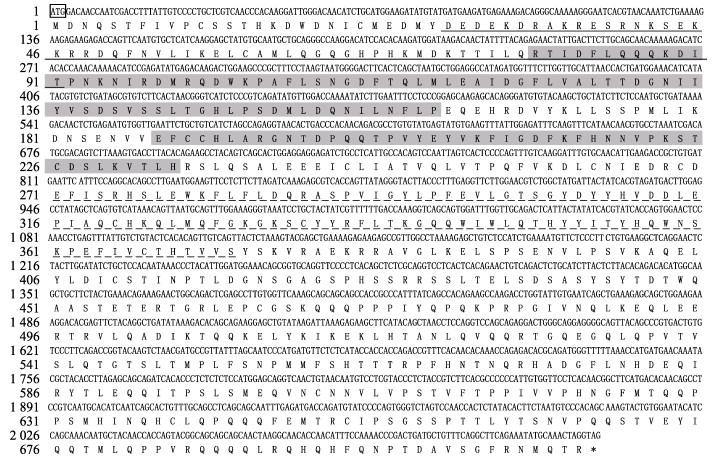
|
图 1 半滑舌鳎Clock1a基因ORF序列及推导的氨基酸序列 Fig.1 Open reading frame sequence of C. semilaevis Clock1a gene and the deduced amino acid sequence 方框部分:起始密码子;下划线:功能结构域PASD1;虚下划线:功能结构域PAS11;阴影部分:保守序列;终止密码子用*表示。 Start codon is marked with frame. Functional domain PASD1 is marked with underline. Functional domain PAS11 is indicated with virtual underline. Conserved sequence is marked in gray, and the termination codon is denoted by *. |

|
图 2 半滑舌鳎Bmal1a基因ORF序列及推导的氨基酸序列 Fig.2 Open reading frame sequence of C. semilaevis Bmal1a gene and the deduced amino acid sequence 方框部分:起始密码子;下划线:功能结构域PASD3;虚下划线:功能结构域PAS11;阴影部分:保守序列;终止密码子用*表示。 Start codon is marked with frame. Functional domain PASD3 is marked with underline. Functional domain PAS11 is indicated with virtual underline. Conserved sequence is marked in gray, and the termination codon is denoted by *. |
从半滑舌鳎卵巢得到Cry1a、Cry2和Per2基因cDNA序列,NCBI登录号分别为OQ054332、OQ054333和OQ054334。Cry1a基因的编码区序列长为1 896 bp,编码631个氨基酸,分子量为71.4 kDa,等电点为8.12,序列中存在功能性结构域FAD7 (由288~486的199个氨基酸组成) (图 3)。Cry2基因的编码区序列长为2 007 bp,编码669个氨基酸,分子量为76.0 kDa,等电点为6.18,序列中存在功能性结构域FAD7 (由288~486位的199个氨基酸组成)(图 4)。Per2基因的编码区序列长为4 248 bp,编码1 415个氨基酸,分子量为154.0 kDa,等电点为5.99,序列中存在功能性结构域PAS11 (由373~474位的102个氨基酸组成)和保守性较强的PeriodC (由1 190~1 384位的295个氨基酸组成)(图 5)。

|
图 3 半滑舌鳎Cry1a基因ORF序列及推导的氨基酸序列 Fig.3 Open reading frame sequence of C. semilaevis Cry1a gene and the deduced amino acid sequence 方框部分:起始密码子;下划线:功能结构域FAD7;阴影部分:保守序列;终止密码子用*表示。 Start codon is marked is framed. Functional domain FAD7 is marked with underline. Conserved sequence is marked in gray, and the termination codon is denoted by *. |
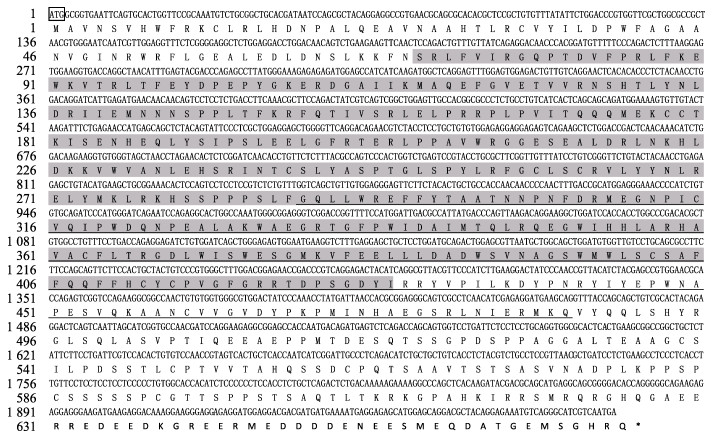
|
图 4 半滑舌鳎Cry2基因ORF序列及推导的氨基酸序列 Fig.4 Open reading frame sequence of C. semilaevis Cry2 gene and the deduced amino acid sequence 方框部分:起始密码子;下划线:功能结构域FAD7;阴影部分:保守序列;终止密码子用*表示。 Start codon is marked is framed. Functional domain FAD7 is marked with underline. Conserved sequence is marked in gray, and the termination codon is denoted by *. |

|
图 5 半滑舌鳎Per2基因ORF序列及推导的氨基酸序列 Fig.5 Open reading frame sequence of C. semilaevis Per2 gene and the deduced amino acid sequence 方框部分:起始密码子;下划线:功能结构域PAS11;虚下划线:功能性结构域PeriodC;阴影部分:保守序列;终止密码子用*表示。 Start codon is marked is framed. Functional domain PAS11 is marked with underline. Functional domain PeriodC indicated with virtual underline. Conserved sequence is marked in gray, and the termination codon is denoted by *. |
对半滑舌鳎Clock1a氨基酸序列与塞内加尔鳎(Solea senegalensis: KAG 7515747.1)、大菱鲆(Scophthalmus maximus: XP 047185380.1)和大黄鱼(Larimichthys crocea: XP 027129473.1)的Clock1a氨基酸序列进行比对,并绘制了多个物种基于氨基酸序列的NJ系统进化树(图 6)。发现半滑舌鳎的Clock1a氨基酸序列与其他鱼类的Clock1a氨基酸序列较为保守,且在进化树上聚为一簇。对半滑舌鳎的Clock1a氨基酸序列与多个物种的Clock1a氨基酸序列同源性进行分析,结果显示,半滑舌鳎Clock1a与塞内加尔鳎的同源性为75%,与大菱鲆的同源性为73%,与大黄鱼的同源性为73%,与爪蟾(Xenopus tropicalis: NP 001122127.1)同源性为47%,与日本鹌鹑(NP 001310116.1)同源性为46%,与人(Homo sapiens: NP 004889.1)同源性为46%。半滑舌鳎的Clock1a与其他硬骨鱼类同源性较高,但与哺乳类、禽类、两栖类等同源性较低,说明Clock1a在进化过程中出现了进化差异(图 6)。
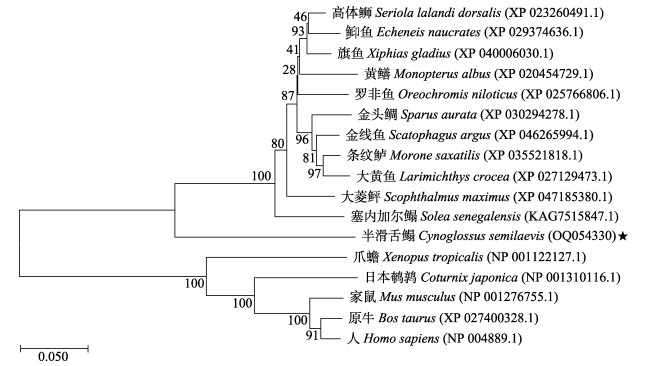
|
图 6 Clock1a氨基酸序列的系统进化树分析(枝上数字表示自展值) Fig.6 Phylogenetic tree based on Clock1a amino acid sequences (Numbers on branches indicate bootstrap support values) |
对半滑舌鳎Bmal1a氨基酸序列与斑马鱼(Danio rerio: AFV 39705.1)、团头鲂(Megalobrama amblycephala: XP 048015598.1)、尖吻欧雅鱼(Squalius pyrenaicus: VAX 53699.1)的Bmal1a氨基酸序列进行比对,并绘制了多个物种基于氨基酸序列的NJ系统进化树(图 7)。结果显示,半滑舌鳎的Bmal1a氨基酸序列与其他鱼类的Bmal1a氨基酸序列较为保守,且在进化树上聚为一簇。对半滑舌鳎的Bmal1a氨基酸序列与多个物种的Bmal1a氨基酸序列同源性进行分析,结果显示,半滑舌鳎Bmal1a与斑马鱼的同源性为100%,与团头鲂的同源性为99%,与尖吻欧雅鱼的同源性为99%,与爪蟾(BAI 82610.1)同源性为84%,与原鸡(Gallus gallus: NP 001384605.1)同源性为88%,与人同源性为88% (BAA 19968.1)。半滑舌鳎的Bmal1a与其他硬骨鱼类的同源性极高,同时与哺乳类、禽类、两栖类等同源性也较高,说明Bmal1a在进化进程中保守性较高(图 7)。

|
图 7 Bmal1a氨基酸序列的系统进化树分析(枝上数字表示自展值) Fig.7 Phylogenetic tree based on Bmal1a amino acid sequences (Numbers on branches indicate bootstrap support values) |
对半滑舌鳎与塞内加尔鳎(XP 043877840.1)、䲟鱼(Echeneis naucrates: XP 029350587.1)、龟壳攀鲈(Anabas testudineus: XP 026217745.1)的Cry1a氨基酸序列进行比对并绘制了多个物种基于氨基酸序列的NJ系统进化树(图 8)。发现半滑舌鳎的Cry1a氨基酸序列与其他鱼类的Cry1a氨基酸序列较为保守,且在进化树上聚为一簇。对半滑舌鳎的Cry1a氨基酸序列与多个物种的Cry1a氨基酸序列同源性进行分析,结果显示,半滑舌鳎Cry1a与塞内加尔鳎的同源性为88%,与䲟鱼的同源性为89%,与龟壳攀鲈的同源性为88%,与爪蟾(XP 031753830.1)的同源性为78%,与日本鹌鹑(XP 015719355.1)的同源性为78%,与人(AAH 30519.1)的同源性为82%。半滑舌鳎的Cry1a与其他硬骨鱼类及哺乳类、禽类、两栖类等同源性均较高,说明Cry1a在进化进程中保守性较高(图 8)。
|
图 8 Cry1a氨基酸序列的系统进化树分析(枝上数字表示自展值) Fig.8 Phylogenetic tree based on Cry1a amino acid sequences (Numbers on branches indicate bootstrap support values) |
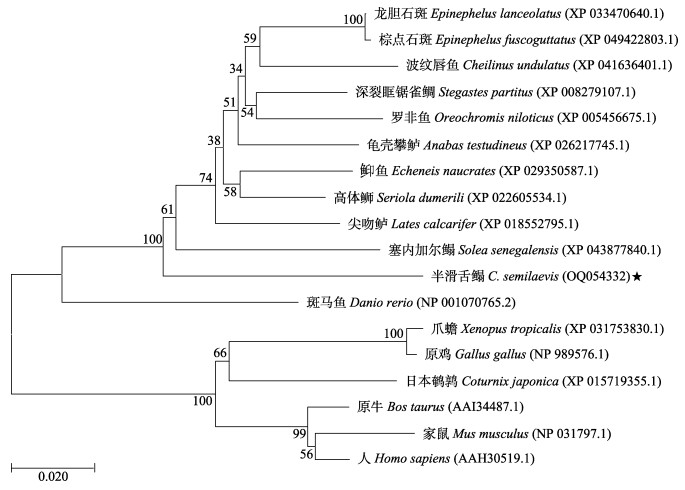
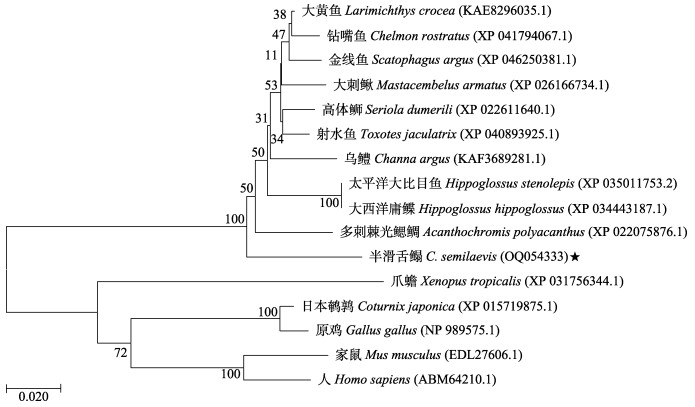
对半滑舌鳎Cry2氨基酸序列与高体

|
图 9 Cry2氨基酸序列的系统进化树分析(枝上数字表示自展值) Fig.9 Phylogenetic tree based on Cry2 amino acid sequences (Numbers on branches indicate bootstrap support values) |
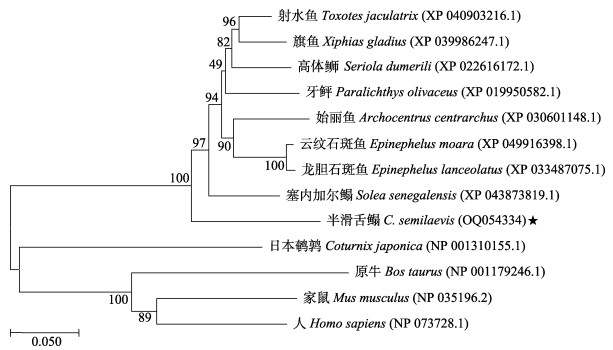
对半滑舌鳎Per2氨基酸序列与尖吻鲈鱼(Lates calcarifer: XP 018539634.1)、射水鱼(XP 040903216.1)、大刺鳅(XP 026175360.1)的Per2氨基酸序列进行比对并绘制了多个物种基于氨基酸序列的NJ系统进化树(图 10)。发现半滑舌鳎的Per2氨基酸序列与其他鱼类的Per2氨基酸序列较为保守,且在进化树上聚为一簇。对半滑舌鳎的Per2氨基酸序列与多个物种的Per2氨基酸序列同源性进行分析,结果显示,半滑舌鳎Per2与尖吻鲈鱼和射水鱼的同源性为84%,与大刺鳅的同源性为82%,与爪蟾(XP 031757399.1)的同源性为61%,与日本鹌鹑(NP 001310155.1)的同源性为67%,与人(NP 073728.1)的同源性为71%。半滑舌鳎的Per2与其他硬骨鱼类的同源性极高,与哺乳类、禽类、两栖类等同源性也较高,说明Per2在进化进程中保守性较强(图 10)。
|
图 10 Per2氨基酸序列的系统进化树分析(枝上数字表示自展值) Fig.10 Phylogenetic tree based on Per2 amino acid sequences (Numbers on branches indicate bootstrap support values) |
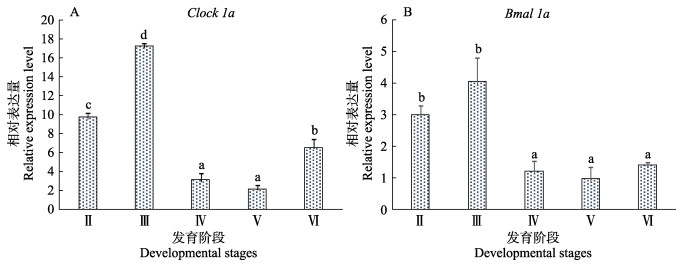
在性成熟半滑舌鳎卵巢的不同发育时期,Clock1a mRNA在卵巢Ⅱ和Ⅲ期表达量显著增加,在Ⅲ期达到最大值(P < 0.05),随后急剧下降,在Ⅴ期达到最小值(P < 0.05),在Ⅵ期又显著增加(P < 0.05)。Clock1a mRNA在卵巢Ⅲ期的表达量是Ⅱ期的1.78倍,是Ⅴ期的8.31倍(图 11A)。Bmal1a mRNA从卵巢Ⅱ期至Ⅲ期表达量增加至最大值,随后显著下降,在Ⅴ期达到最小值(P < 0.05),随后逐渐增加。Bmal1a mRNA在卵巢Ⅲ期的表达量是Ⅱ期的1.36倍,是Ⅴ期的4.28倍(图 11B)。
|
图 11 半滑舌鳎卵巢不同发育时期的Clock1a和Bmal1a mRNA表达分析 Fig.11 Expression profiles of Clock1a and Bmal1a mRNA in the different developmental stages of ovary in C. semilaevis 不同字母间差异显著(P < 0.05)。下同。 Different letters represent significant difference (P < 0.05). The same as below. |
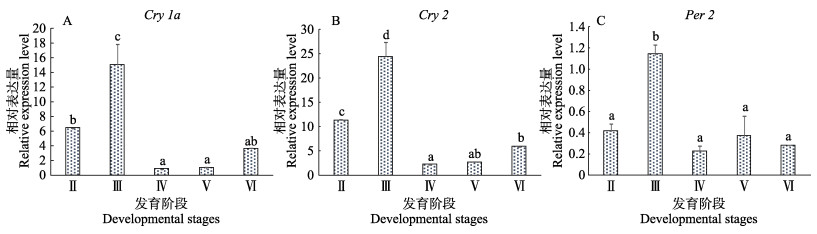
在性成熟半滑舌鳎卵巢的不同发育时期,Cry1a mRNA在卵巢Ⅱ~Ⅲ期表达量显著增加,在Ⅲ期达到最大值(P < 0.05),随后急剧下降,在Ⅴ期达到最小值(P < 0.05),在Ⅵ期略有升高。Cry1a mRNA在卵巢Ⅲ期的表达量是Ⅱ期的2.35倍,是卵巢Ⅴ期的16.29倍(图 12A)。Cry2 mRNA在卵巢Ⅱ~Ⅲ期表达量显著增加,在Ⅲ期达到最大值(P < 0.05),随后急剧下降,在Ⅳ期达到最小值(P < 0.05),随后逐渐增加。Cry2 mRNA在卵巢Ⅲ期的表达量是Ⅱ期的2.16倍,是Ⅴ期的9.50倍(图 12B)。Per2 mRNA在卵巢Ⅱ~Ⅲ期表达量显著增加,在Ⅲ期达到最大值(P < 0.05),随后急剧下降,在Ⅳ期达到最小值(P < 0.05),随后无明显变化。Per2 mRNA在卵巢Ⅲ期的表达量是Ⅱ期的2.76倍,是Ⅴ期的3.09倍(图 12C)。
|
图 12 半滑舌鳎卵巢不同发育时期的Cry1a、Cry2、Per2 mRNA表达分析 Fig.12 Expression profiles of Cry1a, Cry2, and Per2 mRNA in the different developmental stages of ovary in C. semilaevis |
本研究利用克隆技术获得了半滑舌鳎的生物钟相关基因Clock1a、Bmal1a、Cry1a、Cry2和Per2的编码区序列。经预测均未发现跨膜结构域和信号肽。Clock1a氨基酸序列中发现功能性结构域PASD1和PAS11。Bmal1a氨基酸序列中发现功能性结构域PASD3和PAS11。PAS11、PASD1、PASD3是PAS (PER-ARNT-SIM)同源结构域,结构域PAS可与结构域bHLH形成的功能性DNA结合复合物bHLH: PAS,与顺式元件E-box结合激活靶基因的转录,形成Clock: Bmal异二聚体的转录因子(Marco et al, 2004; Bian et al, 2017)。在青鳉(Oryzias latipes)的Clock1和Bmal1序列中也存在功能性结构域PAS-A和PAS-B,且在功能性结构域PAS-A上发现了参与形成Clock: Bmal异二聚体的氨基酸残基Ala174、Ala175和Leu179 (Teika et al, 2019)。半滑舌鳎Clock1a氨基酸序列中的功能性结构域PASD1、PAS11以及Bmal1a氨基酸序列中的功能性结构域PASD3、PAS11可能与E-box结合,在半滑舌鳎生物钟基因环路调节过程中起到促进Clock: Bmal异二聚体形成的作用,正向调节生物钟基因环路。氨基酸同源性分析显示,半滑舌鳎Clock1a、Bmal1a与其他硬骨鱼类的同源性分别达到60%~79%、94%~100%,与爪蟾同源性分别为47%、84%,Clock1a与日本鹌鹑同源性为46%,Bmal1a与原鸡同源性为88%,与人同源性分别为46%、88%。在构建的进化树中,半滑舌鳎Clock1a、Bmal1a序列分别与其他硬骨鱼类聚为一簇,其他脊椎动物的Clock1a、Bmal1a序列分别聚为一簇。说明,半滑舌鳎Clock1a基因在进化进程中出现了进化差异,但在硬骨鱼类中保守性较强,Bmal1a基因则在脊椎动物进化进程中保守性强。
Cry1a氨基酸序列中发现功能性结构域FAD7。Cry2氨基酸序列发现功能性结构域FAD7;并且Cry1a和Cry2序列中FAD7同源性达到75%。Crys包含一个光解酶同源区(PHR),PHR由初级口袋(与功能性结构域FAD结合)、次级口袋和扩展的C端区域组成,Crys通过PHR结构域上2个不同的区域直接与Clock和Bmal相互作用(Gul et al, 2020)。当Cry: Per异二聚体与Clock: Bmal异二聚体结合后,Cry氨基酸序列中的FAD7结构域通过切除Clock: Bmal异二聚体氨基酸序列中的胸腺嘧啶二聚体使其序列发生改变,从而不能与E-box结合,实现负反馈调节(Saha et al, 2019)。在欧洲海鲈(Dicentrarchus labrax)的Cry1和Cry2序列中发现了功能性结构域FAD7 (Pozo et al, 2012)。在牙鲆(Paralichthys olivaceus)的Cry1序列中发现了功能性结构域FAD (Zhang et al, 2022)。在哺乳动物黑线仓鼠(Cricetulus barabensis)的Cry1和Cry2序列中也发现了功能性结构域FAD (朱涵毅等, 2019)。半滑舌鳎Cry1a和Cry2氨基酸序列中的功能性结构域FAD7可能通过抑制Clock: Bmal异二聚体与E-box结合,在半滑舌鳎生物钟基因环路调节过程中起到抑制Clock: Bmal异二聚体形成的作用,逆向调节生物钟基因环路。氨基酸同源性分析显示,半滑舌鳎Cry1a和Cry2与其他硬骨鱼类的同源性分别达到85%~91%和84%~94%,与爪蟾的同源性分别为78%和68%,Cry1a与日本鹌鹑的同源性为78%,Cry2与原鸡的同源性为70%,与人的同源性分别为82%和70%。在构建的进化树中,半滑舌鳎Cry1a和Cry2分别与其他硬骨鱼类聚为一簇,其他脊椎动物的Cry1和Cry2聚为一簇。说明半滑舌鳎Cry1a基因在进化进程中保守性较强,Cry2基因在进化进程中出现了进化差异,但在硬骨鱼类中保守性较强。同时,半滑舌鳎Cry1a与Cry2的同源性达62%,表明2种半滑舌鳎Cry基因在进化进程中出现了进化差异。
Per2氨基酸序列中发现功能性结构域PeriodC和PAS11。在卡氏欧雅鱼(Squalius carolitertii)、尖吻欧雅鱼、勐欧雅鱼(S. torgalensis)、阿拉丁欧雅鱼(S. aradensis)等4种硬骨鱼生物钟基因适应与趋同的研究中发现,Per序列中有功能性结构域PAS3或PAS11,以及末端区域的功能性结构域PeriodC。功能性结构域PeriodC可与Clock1a和Bmal1a氨基酸序列中的功能性结构域PAS结合,功能性结构域PAS具有光接收活性且在蛋白质形成二聚体的过程中发挥作用,功能性结构域PeriodC可能在FAD7切除Clock: Bmal异二聚体氨基酸序列中的胸腺嘧啶二聚体的过程中发挥作用(Moreno et al, 2021)。在瓦氏黄颡鱼(Pelteobagrus vachellii)的Per2氨基酸序列中存在功能性结构域PAS(Qin et al, 2021)。王明勇等(2012)研究发现,斑马鱼Per2氨基酸序列中的功能性结构域PAS11能与E-box结合,使限速酶AANAT2的表达受到抑制,同时,PAS11可通过RORE增强Bmal1b的表达,从而使Per2基因在斑马鱼生物钟中发挥双重作用。半滑舌鳎Per2氨基酸序列中的功能性结构域PeriodC可能通过与Clock1a或Bmal1a氨基酸序列中的功能性结构域PAS结合,使Clock: Bmal异二聚体的形成受到抑制。而半滑舌鳎Per2氨基酸序列中的功能性结构域PAS可能与Clock1a或Bmal1a的氨基酸序列中的功能性结构域PAS形成竞争,抑制Clock: Bmal异二聚体的形成,半滑舌鳎Per2氨基酸序列中的功能性结构域PeriodC与功能性结构域PAS11共同逆向调节生物钟基因环路。同时,功能性结构域PAS11可能通过抑制限速酶AANAT2的表达,达到调节MLT表达的作用。氨基酸同源性分析显示,半滑舌鳎Per2与其他硬骨鱼类的同源性达到70%~84%,与爪蟾的同源性为61%,与日本鹌鹑的同源性为67%,与人的同源性为71%;半滑舌鳎Per2在构建的进化树中与其他硬骨鱼类聚为一簇,其他脊椎动物的Per2也聚为一簇,说明Per2基因在进化进程中保守性较强。
生物钟核心环路由Clock和Bmal组成的正调节因子Clock: Bmal和Per、Cry组成的负调节因子Per: Cry构成。二聚化的Clock: Bmal蛋白能结合位于启动子上游的E/E´-box核苷酸序列,从而启动Per和Cry以及钟控制基因(circadian controlled genes, CCGs)等基因的表达,表达产物中的Per和Cry蛋白在细胞质内形成异二聚体Per: Cry,Per: Cry入核后,通过与Clock: Bmal异二聚体结合,抑制该二聚体与启动子上游的E/E´-box的结合,从而呈现出核心生物钟的负反馈环路调节(Saha et al, 2019)。生物钟基因可抑制MLT合成过程的限速酶AANAT2的表达,从而参与调节PG中MLT的合成节律,继而影响BPG轴中GnRH的分泌(Saha et al, 2019)。在大菱鲆季节性繁殖过程中,MLT受体Mtnr1和Mtnr2的表达量随着卵巢的发育成熟逐渐增加,在产卵期达到最高值,说明MLT是调节大菱鲆卵巢发育的一种有效激素(Zhao et al, 2022)。在非繁殖季节和繁殖季节的点带石斑鱼(Epinephelus coioides)脑和垂体中以及三刺鱼(Gasterosteus aculeatus)和旗鱼的性腺中,MLT的表达也存在着季节性规律,繁殖季节的旗鱼卵巢组织中MLT表达量高于非繁殖季节的卵巢组织,同时,在非繁殖季节的旗鱼卵巢组织中Clock、Bmal1、Cry2和Per2基因的表达量高于繁殖季节,说明Clock、Bmal1、Cry2h和Per2基因是旗鱼卵巢发育成熟过程中的重要基因(Sokoyowska et al, 2004; Chai et al, 2013; Basili et al, 2021)。本研究通过qRT-PCR技术检测了半滑舌鳎性成熟过程中Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ期卵巢组织中的Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的表达变化规律,它们在卵巢发育过程中的表达均从Ⅱ期到Ⅲ期显著升高,并在Ⅲ期达到最大值(P < 0.05),随后显著性降低,在Ⅳ期到Ⅵ期呈相对低的表达量。说明半滑舌鳎的Clock1a、Bmal1a、Cry1a、Cry2和Per2基因是参与调节卵巢发育成熟的重要基因。
在本研究中,半滑舌鳎Clock1a、Bmal1a、Cry1a、Cry2和Per2基因的表达变化趋势与卵巢发育成熟过程呈负相关,与季节性光照周期呈正相关并呈现出周年表达规律。生物钟基因的周期性表达能使生物体产生食物摄入周期,从而影响脂肪累积和热量消耗(Oike et al, 2014);孙学亮等(2012)通过模拟不同光照周期对半滑舌鳎生长发育的影响进行研究,发现长光照周期条件下饲料系数低。半滑舌鳎亲鱼培育过程中,卵巢Ⅱ期和Ⅲ期的光照条件为长光照,Ⅳ期和Ⅴ期的光照条件为相对短光照,生物钟基因可能通过在卵巢Ⅳ期和Ⅴ期的低表达影响半滑舌鳎的摄食和能量平衡,有助于满足卵巢发育过程中的能量需求。生物钟基因对硬骨鱼类卵巢发育周期影响的具体机制目前仍不明确,徐倏豪(2016)通过细胞转染实验发现,斑马鱼胚胎组织中Clock1a: Bmal1b异二聚体可激活骨形态发生蛋白2a (Bmp2a)的表达,Bmp2a可通过影响Per2的表达参与生物钟的调控,且Bmp2a参与脂肪的生成与代谢。Shi等(2016)研究发现,半滑舌鳎卵巢组织中的孕激素和脂联素受体Paqr7b在卵巢发育Ⅱ~Ⅴ期的卵母细胞中表达量逐渐增加,在Ⅴ期达到最大值,在Ⅵ期急剧下降。生物钟基因可能与以上蛋白共同作用,达到调节半滑舌鳎卵巢周期性发育成熟。Khan等(2016)通过模拟不同光照条件发现,斑马鱼卵巢组织中Clock1a、Bmal1a、Cry2a和Per1b在长光照条件下的表达量高于短光照条件。在日本鹌鹑卵巢中,Clock、Per2、Per3基因在长光照条件下的表达量高于短光照条件(Takashi et al, 2000)。本研究中,半滑舌鳎生物钟基因的表达变化规律与上述斑马鱼和日本鹌鹑卵巢中表达变化特征的报道相似。
受光照条件等具有季节性变化规律的外在条件以及外围生物钟机制的共同影响,卵巢组织中生物钟基因的表达也呈现出年周期性表达规律。在人工控制培育水温和光照周期的条件下,对半滑舌鳎亲鱼卵巢的发育成熟过程研究发现,半滑舌鳎亲鱼卵巢发育与水温和光周期具有显著的相关关系(柳学周等, 2009)。在非发情期的苏尼特羊卵巢中,Per1和Per2基因的表达量高于发情期(向光明等, 2019a、b);Basili等(2021)通过对繁殖季节和非繁殖季节的旗鱼性成熟个体和非成熟个体的卵巢中重要基因表达特征进行研究,发现卵巢中生物钟基因(Clock、Bmal1、Cry2和Per2)的表达存在季节性差异,且非繁殖季节Clock、Bmal1、Cry2和Per2基因的表达量高于繁殖季节,性成熟个体和非成熟个体间在非繁殖季节的表达差异不明显,说明了生物钟基因季节性表达在旗鱼卵巢的成熟过程中具有潜在的调控作用。本研究中,对半滑舌鳎卵巢组织中Clock1a、Bmal1a、Cry1a、Cry2和Per2基因在卵巢发育的Ⅱ~Ⅵ期的表达量变化分析贯穿了一个年周期,且半滑舌鳎卵巢组织中的5个生物钟基因表达量相对高的Ⅱ期和Ⅲ期处于非繁殖季节,而5个生物钟基因表达量相对低的Ⅳ期和Ⅴ期处于繁殖季节,因此,半滑舌鳎卵巢组织中Clock1a、Bmal1a、Cry1a、Cry2和Per2的表达也存在季节性规律。生物钟基因表达随着半滑舌鳎卵巢发育经历Ⅱ~Ⅵ期后再度处于Ⅱ期并开始新的发育周期时,即表现为年周期性的表达规律。在生产实践中,可以结合半滑舌鳎生物钟表达特征,通过调控半滑舌鳎亲鱼培育期的光照长短,促进其性腺发育成熟,有望获取繁殖期提前的半滑舌鳎亲鱼,将有助于半滑舌鳎苗种繁育效率的提升。
综上分析,本研究克隆了半滑舌鳎生物钟相关基因Clock1a、Bmal1a、Cry1a、Cry2和Per2的序列,通过构建的系统进化树,发现半滑舌鳎Clock1a、Bmal1a、Cry1a、Cry2和Per2分别与其他硬骨鱼类的Clock1a、Bmal1a、Cry1a、Cry2和Per2聚为一簇,说明其在长期进化过程中具有较近的亲缘关系。半滑舌鳎Clock1a与哺乳类、禽类和两栖类同源性较低,说明Clock1a在进化过程中出现了进化差异。半滑舌鳎Bmal1a、Cry1a、Cry2和Per2与哺乳类、禽类和两栖类同源性较高,说明其在进化进程中保守性较强。本研究初步探明了Clock1a、Bmal1、Cry1a、Cry2和Per2基因在性成熟半滑舌鳎卵巢组织的Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ期的表达变化规律,为进一步揭示生物钟相关基因参与调控半滑舌鳎繁殖机理奠定了基础。
BASILI D, GIOACCHINI G, TODISCO V, et al. Opsins and gonadal circadian rhythm in the swordfish (Xiphias gladius) ovary: Their potential roles in puberty and reproductive seasonality. General and Comparative Endocrinology, 2021, 303: 113707 DOI:10.1016/j.ygcen.2020.113707 |
BIAN S S, ZHENG X L, SUN H Q, et al. Clock1a affects mesoderm development and primitive hematopoiesis by regulating Nodal-Smad3 signaling in the zebrafish embryo. Journal of Biological Chemistry, 2017, 292(34): 14165-14175 DOI:10.1074/jbc.M117.794289 |
CHAI K, LIU X C, ZHANG Y, et al. Day-night and reproductive cycle profiles of melatonin receptor, kiss, and gnrh expression in orange-spotted grouper (Epinephelus coioides). Molecular Reproduction and Development, 2013, 80(7): 535-548 DOI:10.1002/mrd.22191 |
CHEN S H, QIAO H, FU H T, et al. Molecular cloning, characterization, and temporal expression of the clock genes period and timeless in the oriental river prawn Macrobrachium nipponense during female reproductive development. Comparative Biochemistry and Physiology, 2017, 207: 43-51 DOI:10.1016/j.cbpa.2017.02.011 |
DAVIE A, MINGHETTI M, MIGAUD H. Seasonal variations in Clock-gene expression in Atlantic salmon (Salmo salar). Chronobiology International, 2009, 26(3): 379-395 DOI:10.1080/07420520902820947 |
GUL S, AYDIN C, OZCAN O, et al. The Arg-293 of Cryptochrome1 is responsible for the allosteric regulation of Clock-Cry1 binding in circadian rhythm. Journal of Biological Chemistry, 2020, 295(50): 17187-17199 DOI:10.1074/jbc.RA120.014333 |
HERRERO M J, LEPESANT J M J. Daily and seasonal expression of clock genes in the pituitary of the European sea bass (Dicentrarchus labrax). General and Comparative Endocrinology, 2014, 208: 30-38 DOI:10.1016/j.ygcen.2014.08.002 |
KEISUKE I, TAKASHI Y. Comparative analysis reveals the underlying mechanism of vertebrate seasonal reproduction. General and Comparative Endocrinology, 2016, 227: 64-68 DOI:10.1016/j.ygcen.2015.05.009 |
KHAN Z A, YUMNAMCHA T, RAJIV C, et al. Melatonin biosynthesizing enzyme genes and clock genes in ovary and whole brain of zebrafish (Danio rerio): Differential expression and a possible interplay. General and Comparative Endocrinology, 2016, 233: 16-31 DOI:10.1016/j.ygcen.2016.05.014 |
LAO Y C, SHI Y F, CHEN Y H, et al. Tissue expression of clock gene in female Magang goose at different reproductive stages. Journal of Zhongkai University of Agriculture and Engineering, 2019, 32(2): 11-16 [劳永聪, 石逸夫, 陈永浩, 等. Clock基因在不同繁殖时期雌性马岗鹅中的组织表达. 仲恺农业工程学院学报, 2019, 32(2): 11-16 DOI:10.3969/j.issn.1674-5663.2019.02.003] |
LIU X Z, XU Y J, LIU N Z, et al. Study on histological and morphometric characters of gonad development of Cynoglossus semilaevis Günther. Progress in Fishery Sciences, 2009, 30(6): 25-35 [柳学周, 徐永江, 刘乃真, 等. 半滑舌鳎卵巢发育的组织学和形态学数量特征研究. 渔业科学进展, 2009, 30(6): 25-35 DOI:10.3969/j.issn.1000-7075.2009.06.004] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method. Methods, 2001, 25(4): 402-408 DOI:10.1006/meth.2001.1262 |
MARCO H, KEES F, SACCO C, et al. The PAS fold a redefinition of the PAS domain based upon structural prediction. European Journal of Biochemistry, 2004, 271: 1198-1208 DOI:10.1111/j.1432-1033.2004.04023.x |
MORENO J M, JESUS T F, COELHO M M, et al. Adaptation and convergence in circadian-related genes in Iberian freshwater fish. BMC Ecology and Evolution, 2021, 21(38): 1-15 |
OIKE H, OISHI K, KOBORI M. Nutrients, clock genes, and chrononutrition. Current Nutrition Reports, 2014, 3(3): 204-212 DOI:10.1007/s13668-014-0082-6 |
PEREZ J H, TOLLA E D, DUNN I C, et al. A comparative perspective on extra-retinal photoreception. Trends in Endocrinology and Metabolism, 2019, 30(1): 39-53 DOI:10.1016/j.tem.2018.10.005 |
POZO A, VERA L M, SÁNCHEZ J A, et al. Molecular cloning, tissue distribution and daily expression of cry1 and cry2 clock genes in European seabass (Dicentrarchus labrax). Comparative Biochemistry and Physiology, 2012, 3: 364-371 |
QIN C J, SHAO T, LIAO X F, et al. Diurnal expression of circadian clock genes period1 and period3 in Pelteobagrus vachellii. Journal of Oceanology and Limnology, 2021, 39(2): 652-660 DOI:10.1007/s00343-020-9267-6 |
SAHA S, SINGH K M, GUPTA B B P. Melatonin synthesis and clock gene regulation in the pineal organ of teleost fish compared to mammals: Similarities and differences. General and Comparative Endocrinology, 2019, 279: 27-34 DOI:10.1016/j.ygcen.2018.07.010 |
SHI B, LIU X Z, THOMAS P, et al. Identification and characterization of a progestin and adipoQ receptor (PAQR) structurally related to Paqr7 in the ovary of Cynoglossus semilaevis and its potential role in regulating oocyte maturation. General and Comparative Endocrinology, 2016, 237: 109-120 DOI:10.1016/j.ygcen.2016.08.008 |
SHI B, LIU X Z, XU Y J. Molecular characterization of three gonadotropin subunits and their expression patterns during ovarian maturation in Cynoglossus semilaevis. International Journal of Molecular Sciences, 2015, 16(2): 2767-2793 DOI:10.3390/ijms16022767 |
SOKOYOWSKA E, KALAMARZ H, KULCZYKOWSKA E. Seasonal changes in brain melatonin concentration in the three-spined stickleback (Gasterosteus aculeatus): towards an endocrine calendar. Comparative Biochemistry and Physiology, 2004, 139: 365-369 DOI:10.1016/j.cbpb.2004.10.001 |
SUN X L, YANG S Y, CHEN C X, et al. Effects of photoperiod on growth and antioxidant indices in half-smooth tongue-sole (Cynoglossus semilaevis). Chinese Journal of Fisheries, 2012, 25(2): 23-27 [孙学亮, 杨树元, 陈成勋, 等. 光照周期对半滑舌鳎生长和抗氧化指标的影响. 水产学杂志, 2012, 25(2): 23-27] |
TAKASHI Y, YOSHIKAZU S, ERI M, et al. Molecular analysis of avian circadian clock genes. Molecular Brain Research, 2000, 78: 207-215 DOI:10.1016/S0169-328X(00)00091-7 |
TEIKA O, GOSHI N, JUN-ICHI H, et al. Circadian oscillation of TNF-α gene expression regulated by clock gene, Bmal1 and Clock1, in the Japanese medaka (Oryzias latipes). International Immunopharmacology, 2019, 70: 362-371 DOI:10.1016/j.intimp.2019.02.004 |
WANG M Y, HUANG G D, WANG H. Advances in the zebrafish circadian clock mechanisms. Hereditas, 2012, 34(9): 1133-1143 [王明勇, 黄国栋, 王晗. 斑马鱼生物钟研究进展. 遗传, 2012, 34(9): 1133-1143] |
WHARFE M D, MARK P J, WADDELL B J. Circadian variation in placental and hepatic clock genes in rat pregnancy. Endocrinology, 2011, 152(9): 3552-3560 DOI:10.1210/en.2011-0081 |
XIANG G M, LIU Q Y, WANG X Y, et al. Tissue expression and polymorphism of Per1 gene and their association with seasonal reproduction in sheep (Ovis aries). Journal of Agricultural Biotechnology, 2019a, 27(7): 1215-1223 [向光明, 刘秋月, 王翔宇, 等. Per1基因组织表达及多态性与绵羊季节性繁殖的相关性研究. 农业生物技术学报, 2019a, 27(7): 1215-1223] |
XIANG G M, LIU Q Y, WANG X Y, et al. Tissue expression and polymorphism of Per2 gene and their association with seasonal reproduction in sheep (Ovis aries). Chinese Journal of Animal Science, 2019b, 55(11): 74-78 [向光明, 刘秋月, 王翔宇, 等. Per2基因组织表达及多态性与绵羊季节性繁殖的相关性研究. 中国畜牧杂志, 2019b, 55(11): 74-78] |
XU S H. Bmp2a signaling plays a role in the circadian clock through directil facilitating Per2 expression in zebrafish. Masterxs Thesis of Soochow University, 2016 [徐倏豪. 骨形态发生蛋白Bmp2a调控斑马鱼生物钟研究. 苏州大学硕士研究生学位论文, 2016]
|
YU Q. Biological clock: The oscillator of gene expression. Science China: Life Science, 2017, 61(1): 128-130 |
ZHANG D Q, SHI B, SHAO P, et al. The identification of miRNAs that regulate ovarian maturation in Cynoglossus semilaevis. Aquaculture, 2022, 555: 738250 DOI:10.1016/j.aquaculture.2022.738250 |
ZHANG P, SUN B G. Cryptochrome 1 is involved in anti-bacterial immunity in Japanese flounder (Paralichthys olivaceus). Aquaculture, 2022, 561: 738675 DOI:10.1016/j.aquaculture.2022.738675 |
ZHAO C Y, XU S H, LIU Y F, et al. Changes of melatonin and its receptors in synchronizing turbot (Scophthalmus maximus) seasonal reproduction and maturation rhythm. Acta Oceanologica Sinica, 2022, 41(1): 84-98 DOI:10.1007/s13131-021-1923-y |
ZHU H Y, XUE H L, XU J H, et al. Clone and bioinformatics analysis of Cry1 and Cry2 in Criecetulus barabensis. Journal of Qufu Normal University, 2019, 45(4): 74-82 [朱涵毅, 薛慧良, 徐金会, 等. 黑线仓鼠Cry1、Cry2克隆及生物信息学分析. 曲阜师范大学学报, 2019, 45(4): 74-82] |



