2. 昆明海关技术中心 云南 昆明 650201
2. Kunming Customs Technology Center, Kunming 650201, China
肠道微生物群被认为是影响宿主健康和营养的重要因素(Wang et al, 2018)。鱼肠道中复杂的微生物群落在鱼类消化代谢(Valdes et al, 2018)、免疫调节(Tran et al, 2018)等过程中发挥重要作用。寄生在消化道中的共生细菌,可以极大地影响宿主的消化功能(Douglas, 2015)。无菌的斑马鱼(Danio rerio)不能吸收大分子蛋白质,但可以通过构建微生物群来帮助其恢复蛋白质的摄取能力,表明肠道微生物群有助于宿主的营养吸收和同化(Bates et al, 2006)。环境因素包括饮食、药物和应激,可以调节肠道微生物的组成和多样性,其中,饮食习惯(食性)被认为是影响鱼类肠道微生物的一个重要因素(Rimoldi et al, 2021)。已有研究表明,食物组成是引起虹鳟(Oncorhynchus mykiss) (Desai et al, 2012; Ingerslev et al, 2014)、浅色黄姑鱼(Nibea coibor)和双棘黄姑鱼(Nibea diacanthus)(Li et al, 2019)、大口黑鲈(Micropterus salmoides) (Zhou et al, 2021)肠道微生物组成变化的主要因素。肠道微生物在很大程度上依赖于宿主的饮食组成,因此,不少研究聚焦营养变化对肠道微生物的调节(Fontinha et al, 2021; Yao et al, 2021; 翟少伟等, 2016)。
随着宏基因组测序方法的发展,肠道微生物与宿主的互生作用也逐渐被重视,肠道菌群的功能被广泛研究。虽然鱼类肠道菌群与宿主消化的关系已基本为人所知,如肠道微生物群可以通过细菌消化酶的产生影响宿主的消化功能(Ghanbari et al, 2015);肠道微生物可以产生多种消化酶,如脂肪酶、蛋白酶和纤维素酶,促进鱼类对食物的消化(Ray et al, 2012);Kuang等(2020)对2种滤食性鱼类肠道微生物群落进行比较分析,发现肠道微生物和周围环境相比具有独特的核心菌群;比较入侵的亚洲鲤鱼和美洲本土鲤鱼的肠道微生物发现,菌群的定植与摄食有关,不同食物类型是造成肠道特异菌群差异化定植的主要原因(Huang et al, 2020)。但目前鲜有将不同食性鱼类肠道微生物进行系统比较的研究报道。
近几年来,云南滇池许多土著鱼类随环境污染、外来物种入侵等因素影响濒临灭绝,急需对其保护。本实验选取滇池高背鲫(Carassius auratus)、昆明裂腹鱼(Shizothorax grahami)、草鱼(Ctenopharyngodon idellus)、鳜鱼(Siniperca chuatsi)分别作为杂食、滤食、草食和肉食性鱼类代表,比较分析4种鱼类肠道微生物群落组成及其多样性。通过探讨食性与肠道优势菌群之间的关联,揭示4种食性鱼类肠道微生物群落在组成、核心类群和功能预测等方面的差异,有助于对鱼类肠道微生物多样性及其生态效应的理解,建立饮食习惯、微生物和宿主(即食性–微生物–鱼类)之间的功能连接。通过探讨鱼类营养代谢的微生物效应,充分利用微生物功能,为通过饮食控制肠道菌群来改善鱼类生长及健康提供科学依据和理论基础。
1 材料与方法 1.1 实验材料及样品采集本实验所采用的草鱼[(1 323.6±40.20) g]、鳜鱼[(841.3±34.54) g]、滇池高背鲫[(350.4±25.98) g]、昆明裂腹鱼[(471.1±23.94) g]于2019年10月10―21日,用双船围网(材料为PA23tex5×3;缩结系数ET/EN为0.60/0.75;网目尺寸为8 cm)捕捞于云南滇池(东经120°42',北纬25°04'),将活鱼运输到云南农业大学水产养殖实验室进行解剖,每种鱼随机选择6尾规格一致(体质量和健康状况等)的个体,将新鲜活鱼用麻醉剂(40 mg/L丁香酚)浸泡后,用75%酒精冲洗鱼类体表,在无菌条件下解剖取肠道并将肠道内容物挤出,每种鱼2尾混合进行收集。12个肠道内容物样品编号为草鱼CI1~CI3、鳜鱼SC1~SC3、滇池高背鲫CA1~CA3、昆明裂腹鱼SG1~SG3。将样品存于−20 ℃冰箱直至后续提取DNA。
1.2 样品DNA的提取使用Power Food微生物DNA分离试剂盒(QIAGEN Srl,意大利)对这些样品进行DNA提取,详细操作步骤参照Power Food Microbial DNA isolation Kit说明书,使用1.2%琼脂糖凝胶电泳和Qubit 4荧光仪(Thermo Fisher Scientific, 美国)检测DNA的质量和测定其浓度。
1.3 PCR扩增和16S rRNA测序利用引物341F: ACTCCTACGGGAGGCAGCAG和806R: GGACTACHVGGGTATCTAAT扩增细菌16S rRNA基因的V3~V4区域。PCR采用rTaq DNA Polymerase 20 μL反应体系:10× Buffer 2 μL,2.5 mmoL/L dNTPs 2 μL,引物341F/806R各0.8 μL,Taq酶0.2 μL,BSA 0.2 μL,模板DNA 10 ng,补ddH2O至20 μL。反应条件:95 ℃ 3 min,然后95 ℃ 45 s,56 ℃ 45 s和72 ℃ 45 s循环30次,最后72 ℃延伸10 min。利用2%琼脂糖凝胶分离扩增产物,使用AxyPrep DNA凝胶提取试剂盒(Axygen Biosciences, 美国)并根据操作说明书提取扩增产物,利用Qubit 4荧光仪(Thermo Fisher Scientific, 美国)进行定量。按照Igene Code生物技术有限公司(北京)的说明书,在Illumina (HiSeq 2500)测序平台上将纯化的扩增产物等摩尔量混合并进行末端测序。
1.4 生物信息学和数据分析根据Fadrosh等(2014)的方法对原始reads进行过滤,获得洁净的高质量reads。使用FLSAH将洁净后reads对端合并获得有效标签(Chen et al, 2018),使用UPARSE(v7.0.1090)将相似性为97%的有效标签聚类成操作分类单元(OTUs)(Edgar, 2013),从丰度最高的标签序列中选取每个OTU的代表性序列,然后使用RDP Classifier (v2.2)对每个OTU的代表性序列进行分类(置入度阈值为80%)。采用主成分分析(PCA)评估样品间的差异,采用Mothur v1.31.2 (http://www.mothur.org/wiki/Calculators)生成alpha多样性指数(ACE、Chao1、Sobs、Shannon和Simpson)。使用R (v3.1.1)统计包进行线性判别并分析效应大小。这些分析使用在线云平台(https://www.omicsmart.com/)进行。利用分析软件SPSS 20.0进行数据统计,数据以平均值±标准差表示;利用单因素方差分析(one-way ANOVA)检验食性操纵的主效应,通过Tukey多重比较确定各实验组间的差异显著水平,P < 0.05为差异显著;用Origin 7.5软件作图。
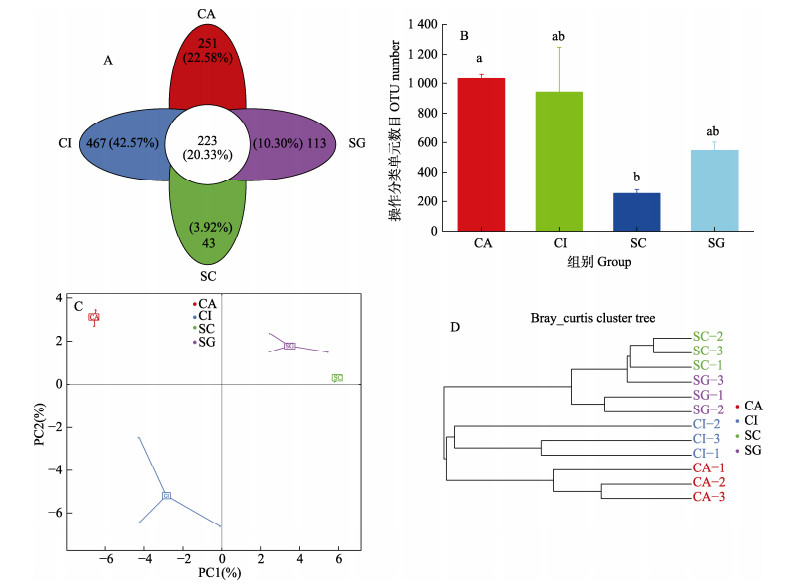
2 结果 2.1 16S rRNA基因测序结果分析所有鱼类共获得2 300个操作分类单元(OTU),4种鱼类OTU交叉分布情况见图 1A。4种鱼类共有/特有的OTU总数为1 079个,其中,223个OTU是4种鱼类所共有,仅占20.33%,其余大部分OTU为不同群体所特有,如草鱼(CI)、滇池高背鲫(CA)、昆明裂腹鱼(SG)和鳜鱼(SC)分别特有467 (42.57%)、251(22.88%)、113(10.30%)和43(3.92%)个OTU。对4种鱼类获得的OTU数比较分析发现(图 1B),滇池高背鲫OTU数量最多,而鳜鱼最少,且鳜鱼OTU数量显著少于滇池高背鲫(P < 0.05),而与其他组别之间未达到显著水平。基于OTU组成(97%相似性)的PCA分析可以反映样品的差异和距离,即2个样品距离越近,则表示这2个样品的组成越相似。由图 1C可以看出,不同来源样品(4种鱼类)表现出分散的分布情况,而来源相同的样品组成具有相似性,其中,鳜鱼和昆明裂腹鱼样品组成相似性较高。根据样品间物种组成聚类分析判断各样品物种组成的相似性,图中相同颜色的样品表示属于同一个分组,样品越靠近,枝长越短,说明2个样品的物种组成越相似。所有样品的聚类分析结果如图 1D所示,鳜鱼与昆明裂腹鱼样品物种组成相似性较高,而滇池高背鲫与草鱼样品中物种组成相似性较高。
|
图 1 4种不同食性鱼类OTU分析 Fig.1 OTU analysis of four fishes with different feeding habits 图1A中的数字为各鱼类的OTU数量和百分比;各柱形图上不同字母代表差异显著(P < 0.05);CI:草鱼;CA:滇池高背鲫;SC:鳜鱼;SG:昆明裂腹鱼。下同。 The number in Fig.1A are the OTU amount and percentage of each fish species. Different letters in each bar chart of Fig.1B indicate statistical difference (P < 0.05). CI: Ctenopharyngodon idellus; CA: Carassius auratus; SC: Siniperca chuatsi; SG: Shizothorax graham. The same below. |
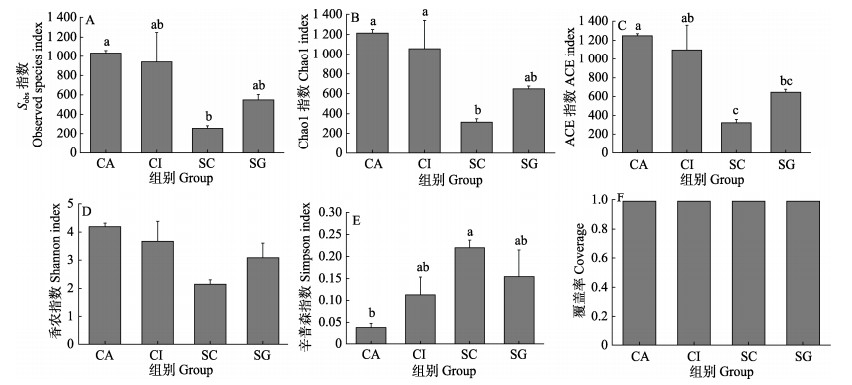
覆盖率(coverage)常被用来评估抽样的完整性,本研究中各样品检测到的操作分类单元(OTU)的覆盖率都在98%以上(图 2F),表明各样品中几乎所有微生物种类都被检测到了。Alpha多样性是对单个样品中物种多样性的分析,包括observed species (Sobs)指数、Chao1指数、ACE指数,Shannon(香农)指数以及Simpson指数等。其中,Sobs指数、Chao1指数和ACE指数反映样品中群落的丰富度,由图 2A~C可以看出较一致的规律,4种鱼类群落的丰富度由大到小依次为滇池高背鲫鱼、草鱼、昆明裂腹鱼和鳜鱼。香农指数和辛普森指数反映群落的多样性,香农指数越大表明群落多样性越丰富,而辛普森指数却相反,越小说明群落多样性越丰富。通过香农指数和辛普森指数也共同反映了一个趋势,4种鱼类群落多样性由高到低依次为滇池高背鲫鱼、草鱼、昆明裂腹鱼和鳜鱼。
|
图 2 4种不同食性鱼类肠道菌群α-多样性指数 Fig.2 Alpha diversity indices of intestinal microbiota of the four fishes with different feeding habits |
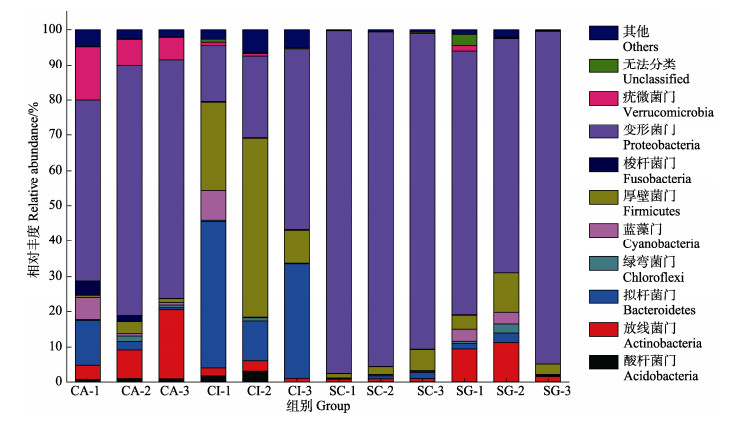
基于门水平对样品进行物种注释分析,共获得37个菌门。位于前9位的细菌组成如图 3所示,其他物种合并为others。在所有样品中,变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)的丰度最高,分别为66.60%、9.82%、9.04%和5.18%。组间肠道微生物丰度在门水平上的差异性如表 1所示,草鱼(CI)组中变形菌门的丰度最低,显著低于鳜鱼(SC)和昆明裂腹鱼(SG)组(P < 0.05),相反,厚壁菌门和拟杆菌门在草鱼中的丰度最高,显著高于其他3种鱼(P < 0.05)。滇池高背鲫(CA)组中的疣微菌门(Verrucomicrobia)的丰度最高,显著高于其他3种鱼(P < 0.05)。
|
图 3 鱼类肠道优势菌群在门水平的分布情况 Fig.3 Distribution of fish intestinal dominant flora at phylum level |
|
|
表 1 4种鱼类肠道内容物中的优势菌门的相对丰度/% Tab.1 Relative abundance of predominant phylum in intestinal contents of the four fish species/% |
基于属水平对样品进行物种注释分析,共获得324个菌属,除去其中未被鉴定的小部分菌属,并将丰度小于0.5%的菌属合并为others后,得到41种菌属(图 4),通过统计分析筛选出13种具有显著差异的菌属(表 2)。由表 2可知,4种鱼类中部分菌属丰度差异显著,鳜鱼和昆明裂腹鱼中乳酸杆菌属(Limnobacter) (25.05%, 23.56%)和假单胞杆菌属(Pseudomonas) (26.07%, 8.16%)丰度较大;而滇池高背鲫中红杆菌属(Rhodobacter) (14.99%)、发酵单胞菌属(Zymomonas) (10.63%)、棒状杆菌属(Clavibacter) (8.57%)和苍黄杆菌属(Luteolibacter) (6.68%)丰度较大;草鱼中拟杆菌属(Bacteroides) (22.56%)和枸橼酸杆菌属(Citrobacter) (9.43%)丰度较大。显然,不动杆菌属(Acinetobacter) 为鳜鱼的优势菌群;厌氧菌属(Anaerospora)、单胞菌属(Arenimonas)、脱氯单胞菌属(Dechloromonas)、苍黄杆菌属和发酵单胞菌属为滇池高背鲫的优势菌群;拟杆菌属为草鱼的优势菌群(P < 0.05);埃希氏杆菌属(Escherichia)、枝动杆菌属(Mycoplana)和乳酸杆菌属为昆明裂腹鱼与鳜鱼共有的优势菌群;假单胞杆菌属为昆明裂腹鱼与滇池高背鲫共有的优势菌群。整体上看,鳜鱼和昆明裂腹鱼中优势菌群组成较相似,而草鱼与滇池高背鲫中优势菌群组成和丰度呈现多样化。

|
图 4 鱼类肠道优势菌群在属水平的分布情况 Fig.4 Distribution of fish intestinal dominant flora at genus level |
|
|
表 2 4种鱼类肠道内含物中的优势菌属的相对丰度/% Tab.2 Relative abundance of predominant genus in intestinal contents of the four fish species/% |
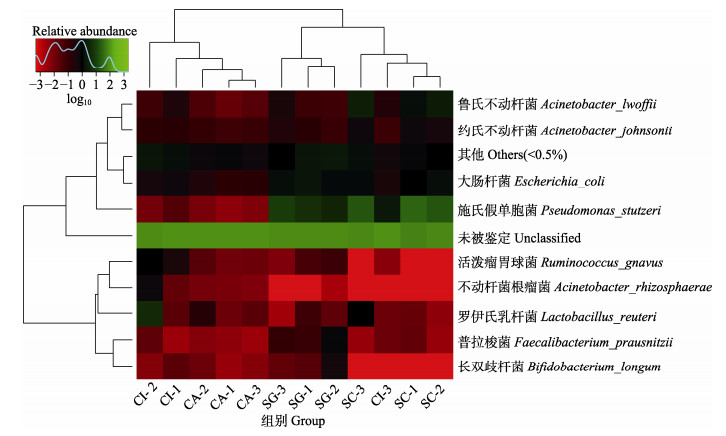
为了探讨食性对肠道优势菌群组成和多样性差异化的影响,进一步在种水平分析获得2 048种差异拷贝,对差异拷贝进行物种注释分析,其中绝大部分拷贝未被鉴定,将鉴定出的9种拷贝(细菌)相对丰度和组成见图 5。整体上看,施氏假单胞菌(Pseudomonas stutzeri)、大肠杆菌(Escherichia coli)和鲁氏不动杆菌(Acinetobacter lwoffii)含量相对丰富,且约氏不动杆菌(Acinetobacter johnsonii)、鲁氏不动杆菌、大肠杆菌和施氏假单胞菌在4种鱼类中分布具有显著性差异(P < 0.05),其中,鳜鱼中的约氏不动杆菌、鲁氏不动杆菌和施氏假单胞菌显著高于其他3种鱼类(P < 0.05),可以判定为鳜鱼的优势菌,同理,大肠杆菌为鳜鱼和昆明裂腹鱼共有的优势菌。
|
图 5 被鉴定出的鱼类肠道优势菌的分布情况 Fig.5 Distribution of identified intestinal dominant bacteria in fish |
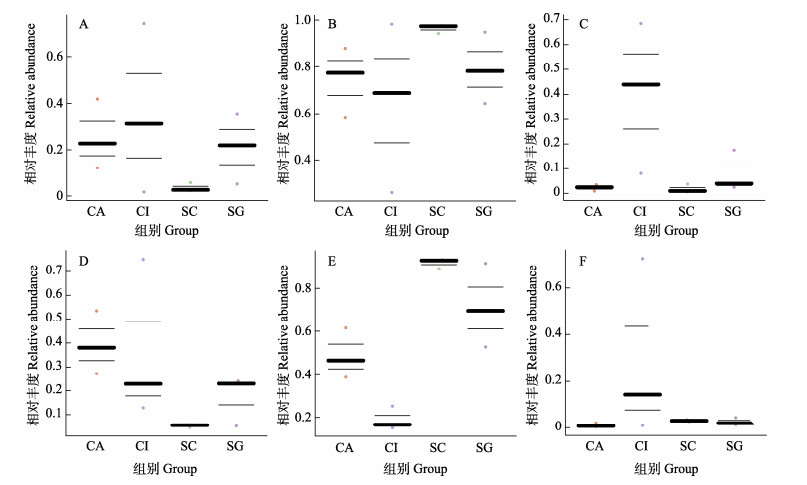
BugBase是一款分析和预测微生物组样品表型的工具,可以基于样品OTU丰度预测大量信息,并按组进行分类统计与可视化。本研究主要预测了革兰氏阳性(Gram-positive)(图 6A)、革兰氏阴性(Gram-negative) (图 6B)、抗病潜力(resistant potential) (图 6C)、碳水化合物消化潜力(carbohydrate digestion potential) (图 6D)、蛋白质消化潜力(protein digestion potential)(图 6E)和脂肪消化潜力(fat digestion potential) (图 6F)。整体上看,所有鱼,特别是鳜鱼(SC)肠道中以革兰氏阴性为主;草鱼(CI)抗病潜力略高于其他3种鱼类;4种鱼类的碳水化合物消化潜力由大到小依次为滇池高倍鲫、草鱼、昆明裂腹鱼、鳜鱼;蛋白质消化潜力由大到小依次为鳜鱼、昆明裂腹鱼、滇池高背鲫鱼、草鱼;4种鱼类脂肪消化潜力都较低。
|
图 6 鱼类肠道微生物的表型预测 Fig.6 Phenotypic prediction of fish intestinal microbes A:革兰氏阳性;B:革兰氏阴性;C:抗病潜力;D:碳水化合物消化潜力;E:蛋白质消化潜力;F:脂肪消化潜力。 A: Gram-positive; B: Gram-negative; C: Resistant potential; D: Carbohydrate digestion potential; E: Protein digestion potential; F: Fat digestion potential. |
脊椎动物的肠道微生物菌群在宿主营养、免疫和健康方面发挥着重要作用(Liu et al, 2016; Valdes et al, 2018)。研究表明,饮食习惯(Miyake et al, 2015; Zhou et al, 2021)和宿主物种(Li et al, 2019; Youngblut et al, 2019)是影响鱼类肠道菌群微生态的主要因素。食性影响肠道菌群变化,但并不是导致鱼类肠道微生物多样性和结构变化的唯一原因(李建柱等, 2016)。本研究也发现,食性是影响鱼类肠道微生物多样性和结构变化的重要因素,与鱼类对不同营养物质的消化代谢潜力相关。还有研究发现,肠道菌群的多样性决定了宿主肠道微生态的稳定性,也与宿主的健康和抗病潜力相关(Li et al, 2019; Kuang et al, 2020)。Alpha(α-)多样性指数是反映单个样品组成和肠道菌群多样性的有效指标,α-多样性指数越高,越有利于鱼类的健康(Huang et al, 2020)。也有研究指出,香农指数越高,表明细菌群落的稳定性越好,对疾病的抵抗力越强(郁维娜等, 2018)。Li等(2015)研究发现,同一栖息地的滤食性大头鱼(Aristichys nobilis)肠道内的细菌多样性高于其他2种草食性鲤鱼。本研究也发现,鱼类肠道菌群多样性受到食性和宿主物种的显著影响,具体表现为除香农指数外,Sobs、chao1、ACE和辛普森指数均受食性影响,综合表现为杂食性(滇池高倍鲫) > 草食性(草鱼) > 滤食性(昆明裂腹鱼) > 肉食性(鳜鱼)。结合肠道微生物表型预测,发现草鱼抗病能力优于其他3种鱼,而滇池高背鲫鱼与鳜鱼的抗病潜力无显著差异,表明单一的香农指数不足以判别宿主抗病能力大小。
益生菌的相对丰度可以反映鱼类的健康状况,也已成为肠道菌群失调和疾病发生的潜在微生物特征(郁维娜等, 2016)。变形菌门和厚壁菌门是虹鳟(Ingerslev et al, 2014)、浅色黄姑鱼和双棘黄姑鱼(Li et al, 2019)、黑鲷(Megalobrama terminalis) (Liu et al, 2021)、大口黑鲈(Zhou et al, 2021)和七彩神仙鱼(Symphysodon haraldi) (Zhang et al, 2021)等许多鱼类肠道中的2种典型优势菌群。本研究结果与前人报道一致,变形菌门和厚壁菌门是4种实验鱼肠道中的典型优势菌群。有研究指出,厚壁菌门能够参与控制宿主的能量平衡(Semova et al, 2012);变形菌门的相对丰度与宿主健康状况密切相关(Blandford et al, 2018);放线菌可维持宿主体内平衡,被广泛用作益生菌(Binda et al, 2018)。本研究的4种鱼类肠道中放线菌门含量较丰富,且食性对放线菌门丰度的影响不显著,表明不能仅凭食性影响鱼类肠道菌群来控制机体内环境平衡和健康。另外,4种鱼类之间的微生物区系存在一些重叠,同时分离明显,如在属水平上,不动杆菌属为鳜鱼的优势菌群,拟杆菌属为草鱼的优势菌群,埃希氏杆菌属、枝动杆菌属和乳酸杆菌属为昆明裂腹鱼与鳜鱼共有的优势菌群,假单胞杆菌属为昆明裂腹鱼与滇池高背鲫共有的优势菌群等。另外,约氏不动杆菌、鲁氏不动杆菌、大肠杆菌和施氏假单胞菌在4种鱼类中分布差异显著,其中,约氏不动杆菌、鲁氏不动杆菌和施氏假单胞菌为鳜鱼的优势菌,大肠杆菌为鳜鱼和昆明裂腹鱼共有的优势菌。这些鱼类携带不同的共生细菌可能是食性和宿主物种选择性地富集不同类群的微生物所致,正如之前的研究表明,在进化过程中,宿主倾向于通过细胞表面识别粘附机制,获得合适的环境细菌(McFall-Ngai, 1998)。这种现象可能为促进类似环境下鱼类肠道细菌群落的分化提供了一种额外的机制,这种选择机制也是宿主和微生物之间长期的生态适应性结果,且对宿主的健康至关重要(Sullam et al, 2012)。另外,特异菌群在不同食性条件下差异富集也可能与菌群的功能相适应,如芽孢杆菌、鲸杆菌可以作为益生菌的潜在候选菌(Larsen et al, 2014);不动杆菌可能诱发动物感染和炎症(Joly-Guillou, 2005);假单胞菌生产维生素B12,梭杆菌生产丁酸,弧菌是潜在的病原菌(Zhou et al, 2019),这些常见菌群的多样性受食性影响,并在鱼类消化和免疫系统中发挥不同的功能。本研究通过BugBase微生物表型预测发现,肉食性的鳜鱼肠道微生物表现出较高的蛋白质消化潜力,同时碳水化合物的消化潜力较高,而草鱼肠道微生物呈现相反的消化潜力,这与鳜鱼和草鱼的食性和食物组成高度统一。结合不同食性鱼类肠道核心菌群多样性差异的结果,推测鳜鱼肠道优势菌属(不动杆菌属)中某些菌群,如约氏不动杆菌、鲁氏不动杆菌和施氏假单胞菌可能有助于宿主对蛋白质的消化,而草鱼肠道中拟杆菌属中的某些菌群可能有助于宿主消化纤维素。通过揭示不同菌群对特定营养物质的潜在利用能力,有助于我们通过饮食控制肠道菌群来改善鱼类生长及健康。
4 结论食性是影响鱼类肠道微生物多样性的主要因素之一,其中草食性和杂食性鱼类肠道微生物较肉食性鱼类丰富。另外,不同食性鱼类肠道微生物区系有重叠也有差异,这是宿主和微生物之间长期的生态适应性结果。这种适应机制与微生物在不同食性鱼类肠道中差异化组成有关,也与肠道微生物中特异菌群的生物学功能有关。本研究通过高通量测序技术和生物学信息学分析方法,系统比较了4种食性鱼类的肠道微生物群落结构和多样性;筛选出与食性相关的优势菌群,并预测其功能,旨在建立食性–微生物–鱼类之间的功能连接,为利用调控微生物技术来推动鱼类健康养殖快速可持续发展提供理论基础。
BATES J M, MITTGE E, KUHLMAN J, et al. Distinct signals from the microbiota promote different aspects of zebrafish gut differentiation. Developmental Biology, 2006, 297(2): 374-386 DOI:10.1016/j.ydbio.2006.05.006 |
BINDA C, LOPETUSO L R, RIZZATTI G, et al. Actinobacteria: A relevant minority for the maintenance of gut homeostasis. Digestive and Liver Disease, 2018, 50(5): 421-428 DOI:10.1016/j.dld.2018.02.012 |
BLANDFORD M I, TAYLOR B A, SCHLACHER T A, et al. Epitheliocystis in fish: An emerging aquaculture disease with a global impact. Transboundary and Emerging Diseases, 2018, 65(6): 1436-1446 DOI:10.1111/tbed.12908 |
CHEN S, ZHOU Y, CHEN Y, et al. FASTP: An ultra-fast all-in-one FASTQ preprocessor. Bioinformatics, 2018, 34(17): i884-i890 DOI:10.1093/bioinformatics/bty560 |
DESAI A R, LINKS M G, COLLINS S A, et al. Effects of plant-based diets on the distal gut microbiome of rainbow trout (Oncorhynchus mykiss). Aquaculture, 2012, 350: 134-142 |
DOUGLAS A E. Multiorganismal insects: Diversity and function of resident microorganisms. Annual Review of Entomology, 2015, 60: 17-34 DOI:10.1146/annurev-ento-010814-020822 |
EDGAR R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998 DOI:10.1038/nmeth.2604 |
FADROSH D W, MA B, GAJER P, et al. An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumina MiSeq platform. Microbiome, 2014, 2(1): 1-7 DOI:10.1186/2049-2618-2-1 |
FONTINHA F, MAGALHAES R, MOUTINHO S, et al. Effect of dietary poultry meal and oil on growth, digestive capacity, and gut microbiota of gilthead seabream (Sparus aurata) juveniles. Aquaculture, 2021, 530: 735879 DOI:10.1016/j.aquaculture.2020.735879 |
GHANBARI M, KNEIFEL W, DOMIG K J. A new view of the fish gut microbiome: Advances from next-generation sequencing. Aquaculture, 2015, 448: 464-475 DOI:10.1016/j.aquaculture.2015.06.033 |
HUANG Q, SHAM R C, DENG Y, et al. Diversity of gut microbiomes in marine fishes is shaped by host-related factors. Molecular Ecology, 2020, 29(24): 5019-5034 DOI:10.1111/mec.15699 |
INGERSLEV H C, VON-GERSDORFF J L, STRUBE M L, et al. The development of the gut microbiota in rainbow trout (Oncorhynchus mykiss) is affected by first feeding and diet type. Aquaculture, 2014, 424: 24-34 |
JOLY-GUILLOU M L. Clinical impact and pathogenicity of Acinetobacter. Clinical Microbiology and Infection, 2005, 11(11): 868-873 DOI:10.1111/j.1469-0691.2005.01227.x |
KUANG T, HE A, LIN Y, et al. Comparative analysis of microbial communities associated with the gill, gut, and habitat of two filter-feeding fish. Aquaculture Reports, 2020, 18: 100501 DOI:10.1016/j.aqrep.2020.100501 |
LARSEN A M, MOHAMMED H H, ARIAS C R. Characterization of the gut microbiota of three commercially valuable warmwater fish species. Journal of Applied Microbiology, 2014, 116(6): 1396-1404 DOI:10.1111/jam.12475 |
LI J Z, HOU J, ZHANG P F, et al. Comparative study of intestinal microbial community structure in different species of carp in aquaponics system. South China Fisheries Science, 2016, 12(6): 42-50 [李建柱, 侯杰, 张鹏飞, 等. 鱼菜共生模式中不同鱼类肠道微生物群落结构的比较. 南方水产科学, 2016, 12(6): 42-50] |
LI T, LONG M, GATESOUPE F J, et al. Comparative analysis of the intestinal bacterial communities in different species of carp by pyrosequencing. Microbial Ecology, 2015, 69(1): 25-36 DOI:10.1007/s00248-014-0480-8 |
LI Z, ZHANG X, AWEYA J J, et al. Formulated diet alters gut microbiota compositions in marine fish Nibea coibor and Nibea diacanthus. Aquaculture Research, 2019, 50(1): 126-138 DOI:10.1111/are.13874 |
LIU H, GUO X, GOONERATNE R, et al. The gut microbiome and degradation enzyme activity of wild freshwater fishes influenced by their trophic levels. Scientific Reports, 2016, 6(1): 1-12 DOI:10.1038/s41598-016-0001-8 |
LIU Y, LI X, LI J, et al. The gut microbiome composition and degradation enzymes activity of black Amur bream (Megalobrama terminalis) in response to breeding migratory behavior. Ecology and Evolution, 2021, 11(10): 5150-5163 DOI:10.1002/ece3.7407 |
MCFALL-NGAI M J. The development of cooperative associations between animals and bacteria: Establishing détente among domains. American Zoologist, 2015, 38(4): 593-608 |
MIYAKE S, NGUGI D K, STINGL U. Diet strongly influences the gut microbiota of surgeonfishes. Molecular Ecology, 2015, 24(3): 656-672 DOI:10.1111/mec.13050 |
RAY A K, GHOSH K, RINGO E. Enzyme-producing bacteria isolated from fish gut: A review. Aquaculture Nutrition, 2012, 18(5): 465-492 DOI:10.1111/j.1365-2095.2012.00943.x |
RIMOLDI S, ANTONINI M, GASCO L, et al. Intestinal microbial communities of rainbow trout (Oncorhynchus mykiss) may be improved by feeding a Hermetia illucens meal/low-fishmeal diet. Fish Physiology and Biochemistry, 2021, 47(2): 365-380 DOI:10.1007/s10695-020-00918-1 |
SEMOVA I, CARTEN J D, STOMBAUGH J, et al. Microbiota regulate intestinal absorption and metabolism of fatty acids in the zebrafish. Cell Host and Microbe, 2012, 12(3): 277-288 DOI:10.1016/j.chom.2012.08.003 |
SULLAM K E, ESSINGER S D, LOZUPONE C A, et al. Environmental and ecological factors that shape the gut bacterial communities of fish: A meta-analysis. Molecular Ecology, 2012, 21(13): 3363-3378 DOI:10.1111/j.1365-294X.2012.05552.x |
TRAN N T, ZHANG J, XIONG F, et al. Altered gut microbiota associated with intestinal disease in grass carp (Ctenopharyngodon idellus). World Journal of Microbiology and Biotechnology, 2018, 34(6): 1-9 |
VALDES A M, WALTER J, SEGAL E, et al. Role of the gut microbiota in nutrition and health. British Medical Journal, 2018, 361: k2179 |
WANG A R, RAN C, RING E, et al. Progress in fish gastrointestinal microbiota research. Reviews in Aquaculture, 2018, 10(3): 626-640 DOI:10.1111/raq.12191 |
YAO W, LI X, ZHANG C, et al. Effects of dietary synbiotics supplementation methods on growth, intestinal health, non-specific immunity and disease resistance of Pacific white shrimp, Litopenaeus vannamei. Fish and Shellfish Immunology, 2021, 112: 46-55 |
YOUNGBLUT N D, REISCHER G H, WALTERS W, et al. Host diet and evolutionary history explain different aspects of gut microbiome diversity among vertebrate clades. Nature Communications, 2019, 10(1): 1-15 |
YU W N, DAI W F, TAO Z, et al. Characterizing the compositional and functional structures of intestinal micro-flora between healthy and diseased Litopenaeus vannamei. Journal of Fisheries of China, 2018, 42(3): 399-409 [郁维娜, 戴文芳, 陶震, 等. 健康与患病凡纳滨对虾肠道菌群结构及功能差异研究. 水产学报, 2018, 42(3): 399-409] |
ZHAI S W, SHI Q C, CHEN X H. Effect of dietary antimicrobial peptides-surfactin supplementation on parameters of intestinal health indices of genetically improved farmed tilapia (GIFT, Oreochromis niloticus). Acta Hydrobiologica Sinica, 2016, 40(4): 823-829 [翟少伟, 史庆超, 陈学豪. 饲料中添加抗菌肽Surfactin对吉富罗非鱼肠道健康的影响. 水生生物学报, 2016, 40(4): 823-829] |
ZHANG Y, WEN B, MENG L J, et al. Dynamic changes of gut microbiota of discus fish (Symphysodon haraldi) at different feeding stages. Aquaculture, 2021, 531: 735912 |
ZHOU L, LIN K, GAN L, et al. Intestinal microbiota of grass carp fed faba beans: A comparative study. Microorganisms, 2019, 7(10): 465 |
ZHOU Y L, HE G L, JIN T, et al. High dietary starch impairs intestinal health and microbiota of largemouth bass, Micropterus salmoides. Aquaculture, 2021, 534: 736261 |



