2. 广西壮族自治区水产引育种中心 广西 南宁 530001
2. Aquatic Cultivation and Breeding Centre of Guangxi Zhuang Autonomous Region, Nanning 530001, China
胡子鲇(Clarias fuscus),隶属鲇形目(Siluriformes)、胡子鲇科(Clariidae)、胡子鲇属(Clarias),俗称塘虱鱼、土虱、角鱼,该物种具有适应性强、营养价值高、肉质鲜嫩的特点,是我国南方大面积养殖的淡水经济鱼类之一,市场潜力巨大(李广丽等, 2013a)。胡子鲇的生长过程中存在明显的性别生长二态性,在同等养殖条件下,雄鱼的生长速度要远远高于雌鱼;且存在雌鱼上市时性腺指数过高的问题,导致同等规格的雄性个体更受青睐(王文达等, 2012)。因此,研究胡子鲇性腺分化与发育的分子调控机制,具有重要的理论价值和现实意义。研究人员对胡子鲇的形态学(程玮玮等, 2010)、生理学(王文达等, 2012; 李广丽等, 2013b; 赵莎莎等, 2014)、养殖学(吴莉芳等, 2010; 陈晓庆等, 2020)、遗传学(邓思平等, 2012b; 孙晶等, 2012)以及基因组学(林星桦等, 2023)等展开了研究,为探究该物种的性别决定与分化相关基因的分子机制研究提供了良好的基础。
实时荧光定量PCR技术(qRT-PCR)实现了对基因从PCR定性检测到实时定量检测的突破,因其有较高的敏感性、准确性和特异性,已成为当今研究基因表达的有效工具之一(苏晓燕等, 2022)。然而,在鱼类的研究中,许多变量会影响qRT-PCR结果的准确性,如RNA质量、完整性、转录效率及参考基因的选择等。合适的标准化方法至关重要,许多研究者使用稳定表达的内参基因来标准化靶基因的表达数据。在早年的研究中,常以gapdh (甘油醛-3-磷酸脱氢酶)、18S rRNA (18S小亚基单位核糖体核酸)、RPL13 (核糖体蛋白L13)等作为内参基因,而随着技术的日益进步,越来越多的实验证明,内参基因的稳定性受到实验条件以及物种间种类的影响,至今未发现适用于所有物种的内参基因,因此,寻找合适的内参基因至关重要(Yang et al, 2020)。
目前,在已有研究报道中,研究者直接选取在其他物种已报道的β-actin基因作为胡子鲇内参基因开展基因表达分析(孙晶等, 2012; 李广丽等, 2013a),尚未对内参基因在胡子鲇性腺发育和不同组织中的表达稳定性和准确性进行系统评估。因此,本实验以胡子鲇为研究对象,结合两种方式挑选候选基因:一是来源于已经发表的其他相近物种内参基因,二是来源于实验室胡子鲇性腺不同发育时期转录组中稳定表达的候选基因。本研究共筛选出13个内参候选基因,利用BestKeeper、GeNorm、NormFinder和RefFinder软件从13个候选内参基因中挑选适合作为胡子鲇性腺不同发育阶段和成鱼不同组织qRT-PCR的内参基因,以期为后续功能基因的研究提供稳定可靠的内参基因。
1 材料与方法 1.1 实验材料胡子鲇样品取自广东海洋大学淡水养殖实验基地。经过MS-222麻醉后,分别取Ⅱ、Ⅲ、Ⅳ期精巢和Ⅱ、Ⅲ、Ⅳ期卵巢;同时,取性成熟脑、肌肉、皮肤、精巢、卵巢、头肾、肝脏、脾脏、肾脏、肠、心脏、胃和鳃等13个组织。每个样品进行3个技术重复,统一收集组织–80 ℃保存,用于后续RNA提取。
1.2 总RNA的提取和cDNA的合成TRIZOL法提取上述胡子鲇成鱼不同组织和不同性腺发育时期(Ⅱ、Ⅲ和Ⅳ期)的总RNA,通过1%琼脂糖凝胶电泳评估总RNA完整性,并使用NanoDrop2000C (Thermo Scientific)测定总RNA浓度和纯度,总RNA的260 nm/280 nm处的吸光度比值均在1.8~2.1之间的样品用于后续实验。使用耐高温全预混第一链cDNA合成试剂盒(北京全式金生物技术有限公司),将总RNA反转录成cDNA,等量混合所有组织cDNA模板。将cDNA样品放置于–20 ℃冰箱保存备用。
1.3 引物设计本研究从其他物种已知内参基因和实验室转录组文库中挑选了13个候选内参基因,其中,6个基因来源于已发表的其他相近物种内参基因:肌动蛋白β1 (actin beta 1, actb1)、肌动蛋白β2 (actin beta 2, actb2)、真核翻译延长因子1α1 (eukaryotic translation elongation factor 1 alpha 1, ef1a)、真核翻译延长因子1β1 (eukaryotic translation elongation factor 1 beta 2, eef1b2)、核糖体蛋白L13a (ribosomal protein L13a, rpl13a)、核糖体蛋白L13 (ribosomal protein L13, rpl13) (Huggett et al, 2005; Wang et al, 2012; Chen et al, 2017; Zhang et al, 2019; 张宁等, 2018),7个基因来源于实验室胡子鲇性腺不同发育时期转录组(登录号:CRA008348)中稳定表达的候选基因:卷曲螺旋结构域蛋白124 (coiled-coil domain containing 124, ccdc124)、丝切蛋白(cofilin 1, cfl1)、核苷二磷酸激酶3 (nucleoside diphosphate kinase 3, nm23)、真核翻译起始因子3亚基G (eukaryotic translation initiation factor 3 subunit G, eif3g)、RAS相关蛋白-1a (RAS-related protein-1a, rap1a)、核因子κb激酶调节亚基γ抑制剂(inhibitor of nuclear factor kappa B kinase regulatory subunit gamma, ikbkg)和酪氨酸3-单加氧酶/色氨酸5-单加氧酶活化蛋白β (tyrosine 3-monooxygenase/ tryptophan 5-monooxygenase activation protein beta, ywhab)。在胡子鲇基因组(Tian et al, 2023)中分别获取上述13个候选基因的目的序列,遵循qRT-PCR引物设计的原则,采用Primer Premier 6.0设计出13对荧光定量特异引物(表 1),由广州生工生物科技有限公司合成。
|
|
表 1 候选内参基因引物 Tab.1 Candidate internal reference gene primer |
使用LightCycler480实时定量PCR仪,按照染料法荧光定量预混试剂盒进行PCR扩增。以胡子鲇不同时期性腺组织样品逆转录得到的cDNA混合液为模板,以所得的13对引物进行qRT-PCR。qRT-PCR
体系(10 μL):2×PerfectStart Green qPCR SuperMix 5 μL、Nuclease-free水3 μL、cDNA模板1 μL、正反向引物各0.5 μL。qRT-PCR程序:95 ℃预变性2 min;95 ℃变性30 s,57 ℃退火30 s,72 ℃延伸20 s,45个循环;最后4 ℃保存。
PCR产物经克隆测序后,用DNASTAR-Lasergene v6软件分析测序结果,将不同组织的cDNA稀释10倍,各取等体积混合,连续4倍稀释7次,得到8个不同梯度的cDNA,并以此为模板,用罗氏LightCycler 480实时荧光定量仪对候选内参基因进行标准曲线扩增。反应体系:2×SYBR PreMix Ex Taq (TliRNaseH Plus) 8 μL,上下游引物(10 μmol/L)各0.5 μL,cDNA 1 μL;反应程序:50 ℃ 120 s;95 ℃ 30 s;95 ℃ 10 s,60 ℃ 60 s,40个循环,收集荧光信号;熔解曲线95 ℃ 30 s,65 ℃ 1 min,95 ℃ 1 s。根据标准曲线结果,将所有组织cDNA稀释200倍,对所有基因进行qRT-PCR扩增,每个样品重复3次,反应体系及条件同上。
1.5 实验数据处理和分析根据4种不同的算法(BestKeeper、NormFinder、geNorm和RefFinder模型)预测内参基因的表达稳定性。BestKeeper根据Ct值的变异系数(CV)和标准差(SD)评估内参基因的表达稳定性(Pfaffl et al, 2004)。对于NormFinder和geNorm,利用公式Q=2Cqmin -Cqsample计算得到Q值(Cqmin为基因在样品中的最小Cq值;Cqsample为基因在相关样品中的Cq值)。NormFinder引入一个ANOV导向模型来计算Q值组内和组间变异,并根据稳定值(SV)对内参基因进行排序(Andersen et al, 2004)。geNorm通过计算内参基因相对于同一分析中包含的所有其他基因的平均成对变异值,来确定基因表达稳定性指数M。综合分析工具RefFinder (https://www.heartcure.com.au/for-researchers/),根据几何平均值(GM)对内参基因的稳定性进行分析,对13个候选内参基因的稳定性进行评估,从而筛选出在胡子鲇性腺不同发育阶段和成鱼不同组织中表达相对稳定的内参基因。
1.6 内参基因稳定性的验证为进一步验证候选内参基因的稳定性,选取稳定性分析结果前二的基因作为候选内参基因,随机挑选胡子鲇不同发育时期性腺转录组中4个差异表达基因(zp3、atp2b3、slc13a5和parp8),通过qRT-PCR检测其表达水平,并与转录组差异基因结果进行对比,以评估差异表达量的准确性。
2 结果 2.1 定量引物质量检测候选内参基因引物PCR扩增后,均可获得单一的PCR产物,无引物二聚体和非特异性扩增(图 1),扩增效果良好。qRT-PCR定量实验标准曲线分析显示,其相关系数R2值为0.83~1.00 (不同组织)、0.88~1.00 (不同时期性腺)(表 2),符合qRT-PCR实验对引物的要求。进一步分析13个候选内参基因的熔解曲线,峰值(Tm)均为单一值,扩增曲线重复性好,引物特异性高(图 2),qRT-PCR结果准确可靠,可用于表达稳定性分析。
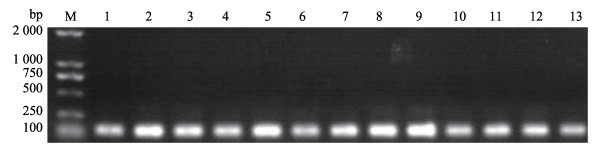
|
图 1 13个候选内参基因PCR扩增产物 Fig.1 PCR amplification products by 13 candidate internal reference genes M: DL 2000 DNA Marker; 1: actb1; 2: actb2; 3: ef1a; 4: rpl13a; 5: rpl13; 6: eef1b2; 7: ccdc124; 8: cfl1; 9: nm23; 10: rap1a; 11: eif3g; 12: ikbkg; 13: ywhab |
|
|
表 2 13个候选内参基因验证结果 Tab.2 Verification results of 13 candidate internal reference genes |
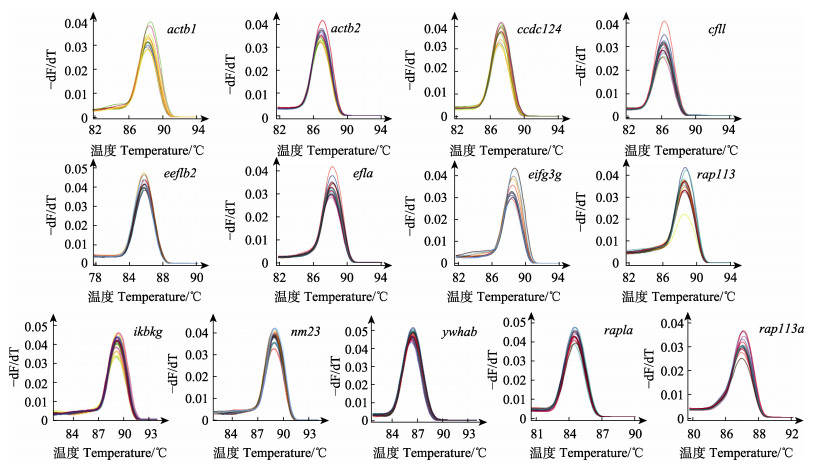
|
图 2 13个候选内参基因熔解曲线分析 Fig.2 Melting curve analysis of 13 candidate internal reference genes |
qRT-PCR的Ct值可直接反映基因的表达丰度,Ct值的大小与基因表达的丰度呈负相关。如表 2所示,所有13个内参基因在不同组织中的平均Ct值为17.16~21.28,在雌雄不同时期性腺的Ct值为16.68~ 21.54。其中,actb2的Ct值在不同组织与雌雄不同时期性腺中均最小,说明该基因的表达丰度最高。
2.3 候选内参基因稳定性分析 2.3.1 Bestkeeper软件分析结果BestKeeper分析结果如图 3所示。在胡子鲇成鱼不同组织中,基因稳定性依次为actb1 (SD=0.08; CV=3.84) > actb2 (SD=0.13; CV=1.73)=ef1a (SD=0.13; CV=2.59) > rpl13 (SD=0.16; CV=1.97) > ywhab (SD=0.22; CV=6.00) > eef1b2 (SD=1.76; CV=5.66) > rpl13a (SD=1.90; CV=2.83) > nm23 (SD=1.91; CV=3.64) > eif3g (SD=1.92; CV=3.46) > ccdc124 (SD=2.07; CV=6.09) > rap1a (SD=2.11; CV=2.38) > cfl1 (SD=2.14; CV=2.11) > ikbkg (SD=2.66; CV=3.13)。actb1、actb2、ef1a、rpl13和ywhab5个基因的SD值小于1.0,说明上述基因在13个不同组织中表达较为稳定。
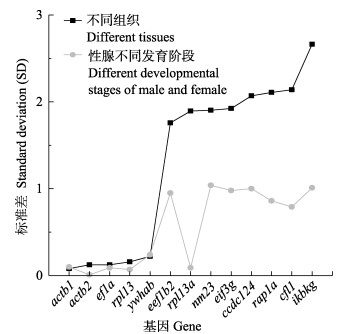
|
图 3 BestKeeper分析不同组织和雌雄性腺不同发育时期的候选内参基因表达稳定值 Fig.3 BestKeeper analysis of the expression stability of candidate reference genes in different tissues and different developmental stages of male and female gonads |
在胡子鲇雌雄性腺不同发育时期中,基因稳定性依次为actb2 (SD=0.01; CV=1.73) > rpl13 (SD=0.07; CV=1.97) > ef1a (SD=0.09; CV=2.67) > rpl13a (SD=0.09; CV=3.01) > actb1 (SD=0.10; CV=2.01) > ywhab (SD=0.24; CV=5.78) > cfl1 (SD= 0.79; CV=2.35) > rap1a (SD=0.86; CV=2.49) > eef1b2 (SD=0.95; CV=5.51) > eif3g (SD=0.98; CV=3.56) > ccdc124 (SD=1.00; CV=5.90) > ikbkg (SD=1.01; CV=3.23) > nm23 (SD=1.04; CV=3.70)。有10个基因的SD值小于1.0,说明actb2、rpl13、ef1a、rpl13a、actb1、ywhab、cfl1、rap1a、eef1b2和eif3g基因在雌雄性腺不同发育时期中表达稳定。
2.3.2 NormFinder软件分析结果NormFinder软件分析结果如图 4所示,在胡子鲇成鱼不同组织中,13个候选内参基因的稳定性依次为actb2 (SV=0.04) = rpl13 (SV=0.04) > actb1 (SV=0.17) > rpl13a (SV=0.32) > ywhab (SV=0.37) > nm23 (SV=0.47) > rap1a (SV=0.47) > cfl1 (SV=0.47) > eif3g (SV=0.62) > ef1a (SV=0.65) > ccdc124(SV=0.71) > ikbkg (SV=0.73)>eef1b2 (SV=0.84)。雌雄性腺不同发育时期中,13个候选内参基因稳定性依次为actb2 (SV=0.04) = actb1 (SV=0.04) > rpl13 (SV=0.07) > ef1a (SV=0.12) > rpl13a (SV=0.22) > cfl1 (SV=0.31) > ywhab (SV=0.37) > rap1a (SV=0.47) > nm23 (SV=0.47) > eif3g (SV=0.62) > ccdc124 (SV= 0.71) > ikbkg (SV=0.73) > eef1b2 (SV=0.75)。其中,actb2、rpl13基因与actb2、actb1基因的SV值分别在成鱼不同组织与雌雄性腺不同发育时期中比其他基因的SV值低,即稳定性比其他基因强,同时可观察到eef1b2基因的稳定性最差。
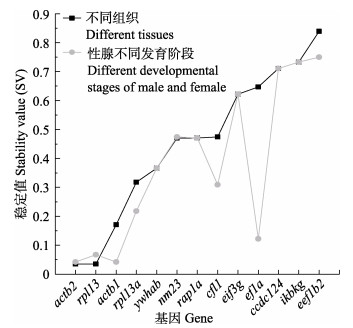
|
图 4 NormFinder分析不同组织和雌雄性腺不同发育时期的候选内参基因表达稳定值 Fig.4 NormFinder analysis of the expression stability of candidate reference genes in different tissues and different developmental stages of male and female gonads |
在胡子鲇成鱼不同组织中,13个候选内参基因的稳定性如图 5所示,依次为actb2 (M=0.07) =rpl13 (M=0.07) > rpl13a (M=0.17) > actb1 (M=0.26) > ef1a (M=0.39) > ccdc124 (M=0.47) > ywhab (M=0.50) > rap1a (M=0.64) > eif3g (M=0.72) > nm23 (M=0.76) > eef1b2 (M=0.80) > ikbkg (M=0.84) > cfl1 (M=0.89),其中,actb2和rpl13基因的M值最小,其基因稳定性最高,其次为rpl13a、actb1和ef1a,cfl1基因稳定性最差。
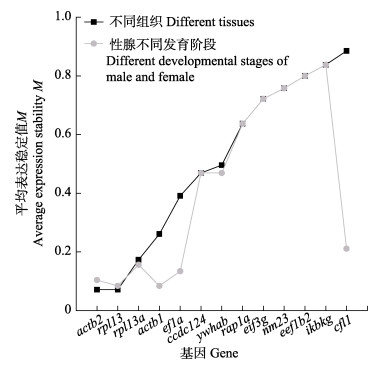
|
图 5 GeNorm分析不同组织和雌雄性腺不同发育时期的候选内参基因表达稳定值 Fig.5 GeNorm analysis of the expression stability of candidate reference genes in different tissues and different developmental stages of male and female gonads |
雌雄性腺不同发育时期中13个候选内参基因的稳定性依次为actb1 (M=0.08)=rpl13 (M=0.08) > actb2 (M=0.10) > ef1a (M=0.13) > rpl13a (M=0.16) > cfl1 (M=0.21) > ywhab (M=0.47)= ccdc124 (M=0.47) > rap1a (M=0.64) > eif3g (M=0.72) > nm23 (M=0.76) > eef1b2 (M=0.80) > ikbkg (M=0.84),其中,actb1和rpl13基因的M值最小,其基因稳定性最高,其次为actb2、ef1a和rpl13a,ikbkg基因稳定性最差。
2.4 RefFinder分析使用RefFinder对Bestkeeper、geNorm、NormFinder 3种模型进行整合分析,综合比较和排名内参基因的表达稳定性(Xie et al, 2011)。RefFinder分析结果如图 6所示,在胡子鲇成鱼不同组织中各基因的稳定性依次为actb2 (GM=1.73) > rpl13 (GM=1.97) > cfl1 (GM=2.11) > rap1a (GM=2.38) > ef1a (GM=2.59) > rpl13a (GM=2.83) > ikbkg (GM=3.13) > eif3g (GM=3.46) > nm23 (GM=3.64) > actb1 (GM=3.84) > eef1b2 (GM=5.66) > ywhab (GM=6.00) > ccdc124 (GM=6.09),actb2的表达稳定性最好。在胡子鲇雌雄性腺不同发育时期各基因的稳定性为actb2 (GM=1.73) > rpl13 (GM=1.97) > actb1 (GM=2.01) > cfl1 (GM=2.35) > rap1a (GM=2.49) > ef1a (GM=2.67) > rpl13a(GM=3.01) > ikbkg (GM=3.23) > eif3g (GM=3.56) > nm23(GM=3.70) > eef1b2 (GM=5.51) > ywhab (GM=5.78) > ccdc124(GM=5.90),actb2表达稳定性最好。
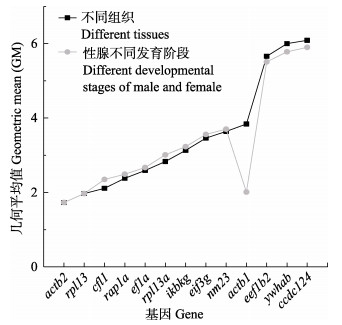
|
图 6 RefFinder分析不同组织和雌雄性腺不同发育时期的候选内参基因表达几何平均值 Fig.6 RefFinder analysis of geometric means of candidate endogenous gene expression in different tissues and at different developmental periods in male and female gonads |
为进一步验证4种分析软件对结果的准确性,以actb2和rpl13作为内参基因分析zp3、atp2b3、slc13a5和parp8四个差异表达基因在雌、雄性腺中的相对表达量(图 7)。结果显示,以actb2基因作为内参基因获得的差异倍数值更接近于转录组分析结果,说明actb2相比于rpl3更稳定,更适于作为胡子鲇性腺不同发育阶段和成鱼不同组织的内参基因。
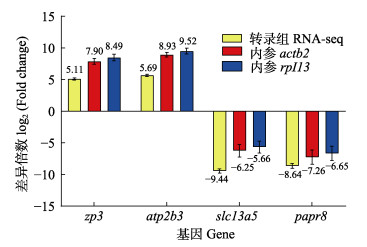
|
图 7 不同内参基因分析4种不同基因在性腺组织中的相对表达水平 Fig.7 Relative expression levels of 4 different genes in gonadal tissues analyzed by different internal reference genes |
当前,qRT-PCR是研究基因调控机制的重要方法,是准确测定基因表达量的首选方法(Forlenza et al, 2012; Gadkar et al, 2014)。在基因表达分析中,通常会使用合适的内参基因对实验数据进行校对,以确保实验结果的可信程度(Dheda et al, 2005)。理想的内参基因应满足:在不同的组织中表达水平中等或较高,不是过高或过低;在不同组织或者不同时间表达基本一致;表达水平不受内源性因素或外源性因素影响(Schmid et al, 2003)。并不是所有物种都适用同一套内参基因(Yang et al, 2020),如:在大西洋鲑(Salmo salar) (Olsvik et al, 2005)、斑马鱼(Danio rerio) (McCurley et al, 2008)、黄鳝(Monopterus albus) (Hu et al, 2014)以及黄带拟鲹(Pseudocaranx dentex)(李杰锋等, 2023)中,不同组织间最为稳定表达的内参基因为EF-1α;而在多鳞
理想的内参基因表达丰度应在15~30之间(Wan et al, 2011),过高或过低的Ct值会使qRT-PCR数据分析时很难准确减去基线值(Vandesompele et al, 2002),而本研究所使用的13个候选内参基因的Ct值均在理想的Ct值范围内,可以作为候选内参基因进行实验研究。
基于上述结论,本研究采用了GeNorm、NormFinder和BestKeeper软件对13个候选内参基因在胡子鲇性腺不同发育阶段和成鱼不同组织的表达稳定性进行评估,结果显示,上述候选基因的表达稳定性排序各不相同。以ef1a基因为例,该基因作为多数鱼类的内参基因,在胡子鲇成鱼不同组织中的稳定性基于不同软件的分析评价结果有较大的差异,基于BestKeeper和GeNorm软件的结果显示ef1a基因较为稳定,而NormFinder软件的结果显示其稳定性较差,这可能是由于不同软件采用了不同的统计模型所致(Ward et al, 2015),这种现象也在方格星虫(Sipuculus nudus)(彭冰冰等, 2022)、多鳞
本研究基于RefFinder综合评估发现,actb2基因在胡子鲇性腺发育不同阶段和成鱼不同组织中表达最为稳定,可作为后续研究胡子鲇分子表达调控的内参基因。本研究actb2基因在不同组织使用NormFinder软件分析得到的SV值为0.35,与青海湖裸鲤(Gymnocypris przewalskii)(刘思嘉等, 2023)的研究结果接近;相比达氏鲟(Acipenser dabryanus)不同性腺发育时期β-actb基因的GM值区间在2~4 (武梦斌等, 2020),本研究使用RefFinder分析actb2基因的GM更低,即表达稳定性更高,且表达丰度适中,符合理想内参基因条件(朱芷葳等, 2006)。为了进一步验证4个软件对于预测结果的准确性,本研究以actb2与rpl13为内参基因,挑选了4个基因研究在雌、雄性腺中的相对表达量,实验结果进一步证实actb2的结果更靠近转录组,是适合胡子鲇不同性腺发育时期的内参基因。
肌动蛋白(actin)是一种高度保守的蛋白,其基因也具有高度保守性,富含于许多细胞之中,尤其在肌肉细胞中含量较高,对于细胞的结构以及运动有着重要的影响,因此,肌动蛋白家族的基因是在qRT-PCR实验中使用频率较高的内参基因家族,尤以actb1基因为主(Huggett et al, 2005)。本研究所获得的内参基因actb2,属于β-肌动蛋白的异构体之一,鉴于其自身的性质,符合作为内参基因的要求。目前,使用actb2作为内参基因已在斑马鱼中有相关研究报道,斑马鱼6个不同胚胎发育阶段和6个成年个体的组织中,actb2表达变异性较低,适合作为斑马鱼的内参基因(Casadei et al, 2011);但由于肌动蛋白是一个庞大的基因家族,actb2与actb1具有较高的同源性,为89%,目前设计其特异性引物较为困难(Sun et al, 2012),因此,是否能广泛地作为其他物种的内参基因仍待进一步研究。
4 结论本研究采用qRT-PCR技术,利用GeNorm、NormFinder、BestKeeper和RefFinder等软件对胡子鲇13个候选基因进行表达稳定性分析,以筛选在胡子鲇不同性腺发育时期及成鱼不同组织中稳定表达的内参基因。结果表明,在胡子鲇性腺发育不同阶段和成鱼不同组织,actb2表达最稳定,可作为胡子鲇性腺发育不同阶段和成鱼不同组织qRT-PCR分析的最适单内参基因,研究结果为后续开展胡子鲇分子生物学的研究提供了数据支撑和基础资料。
ANDERSEN C L, JENSEN J L, ØRNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Research, 2004, 64(15): 5245-5250 DOI:10.1158/0008-5472.CAN-04-0496 |
CASADEI R, PELLERI M C, VITALE L, et al. Identification of housekeeping genes suitable for gene expression analysis in the zebrafish. Gene Expression Patterns, 2011, 11(3/4): 271-276 |
CHEN X J, ZHANG X Q, HUANG S, et al. Selection of reference genes for quantitative real-time RT-PCR on gene expression in golden pompano (Trachinotus ovatus). Polish Journal Veterinary Sciences, 2017, 20(3): 583-594 DOI:10.1515/pjvs-2017-0071 |
CHEN X Q, ZHANG Y, TAN J H, et al. Effect of flocs concentration on the performance of African catfish (Clarias gariepinus) in bioflocs aquaculture systems. Journal of Fishery Sciences of China, 2020, 27(7): 779-788 [陈晓庆, 张扬, 谭竟宏, 等. 絮团浓度对革胡子鲇零换水养殖效果的影响. 中国水产科学, 2020, 27(7): 779-788] |
CHENG W W, FANG Q. Study on morphological variations Silurus meridionalis and Clarias lazera. Anhui Agricultural Science Bulletin, 2010, 16(9): 194-195 [程玮玮, 方遒. 大口鲇和革胡子鲇形态特征研究. 安徽农学通报(上半月刊), 2010, 16(9): 194-195] |
DENG S P, WANG J J, WU T L, et al. cDNA cloning and expression analysis of Dmrt1 in Clarias fuscus. Acta Hydrobiologica Sinica, 2012a, 36(4): 610-617 [邓思平, 王静杰, 吴天利, 等. 胡子鲇Dmrt1基因全长cDNA克隆及其表达分析. 水生生物学报, 2012a, 36(4): 610-617] |
DENG S P, ZHANG Y Y, ZHU C H, et al. Analysis on genetic structure of three cultured populations Claris fuscu with AFLP markers. Journal of Guangdong Ocean University, 2012b, 32(3): 24-28 [邓思平, 张艳艳, 朱春华, 等. 胡子鲇3个养殖群体遗传结构的AFLP分析. 广东海洋大学学报, 2012b, 32(3): 24-28] |
DHEDA K, HUGGETT J F, CHANG J S, et al. The implications of using an inappropriate reference gene for real-time reverse transcription PCR data normalization. Analytical Biochemistry, 2005, 344(1): 141-143 DOI:10.1016/j.ab.2005.05.022 |
FORLENZA M, KAISER T, SAVELKOUL H F J, et al. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Cytokine Protocols. Totowa: Humana Press, 2012, 820: 7-23 |
GADKAR V Y, FILION M. New developments in quantitative real-time polymerase chain reaction technology. Current Issues in Molecular Biology, 2014, 16: 1-6 |
HU Q, GUO W, GAO Y, et al. Reference gene selection for real-time RT-PCR normalization in rice field eel (Monopterus albus) during gonad development. Fish Physiology and Biochemistry, 2014, 40(6): 1721-1730 DOI:10.1007/s10695-014-9962-3 |
HU X Q, WANG J H, SONG H, et al. Screening of internal reference genes in flower bud and different organs during sex differentiation of Trichosanthes kirilowii Maxim. Molecular Plant Breeding, 2023, 1-10 [胡秀芹, 王婧华, 宋晗, 等. 栝楼性别分化期花芽及不同器官内参基因的筛选. 分子植物育种, 2023, 1-10] |
HUGGETT J, DHEDA K, BUSTIN S, et al. Real-time RT-PCR normalisation; strategies and considerations. Genes and Immunity, 2005, 6(4): 279-284 DOI:10.1038/sj.gene.6364190 |
LI G L, DENG S P, SUN J, et al. Effects of aromatase inhibitor letrozole on sex differentiation and related gene expression in Clarias fuscus. Journal of Fishery Sciences of China, 2013a, 20(5): 911-917 [李广丽, 邓思平, 孙晶, 等. 芳香化酶抑制剂来曲唑对胡子鲇性腺分化及相关基因表达的影响. 中国水产科学, 2013a, 20(5): 911-917] |
LI G L, DENG S P, WANG W D, et al. Effects of sex steroid hormones on sex differentiation of Clarias fuscus. Acta Hydrobiologica Sinica, 2013, b, 37(6): 1020-1027 [李广丽, 邓思平, 王文达, 等. 性类固醇激素对胡子鲇性分化的影响. 水生生物学报, 2013, b, 37(6): 1020-1027] |
LI J F, WANG H, LI B S, et al. Screening and evaluating reference genes for quantitative real-time PCR in striped Jack (Pseudocaranx dentex). Progress in Fishery Sciences, 2023, 44(6): 107-115 [李杰锋, 王焕, 李步苏, 等. 黄带拟鲹qRT-PCR内参基因筛选及验证. 渔业科学进展, 2023, 44(6): 107-115] |
LIN X H, ZHOU D Y, SHEN Y J, et al. The genome survey analysis of male and female Hongkong catfish (Clarias fuscus). Genomics and Applied Biology, 2023, 42(6): 610-619 [林星桦, 周大颜, 沈奕君, 等. 胡子鲇(Clarias fuscus)雌雄基因组Survey分析. 基因组学与应用生物学, 2023, 42(6): 610-619] |
LIU S J, QI D L, QI H F, et al. Selection and validation of reference genes in Gymnocypris przewalskii by quantitative real-time PCR. Fisheries Science, 2023, 42(3): 413-423 [刘思嘉, 祁得林, 祁洪芳, 等. 青海湖裸鲤实时荧光定量PCR内参基因筛选与验证. 水产科学, 2023, 42(3): 413-423] |
LUO S S, GAO Z X, FENG B, et al. Preferential selection of internal reference genes in qRT-PCR analysis of diploid and tetraploid loach, Misgurnus anguillicaudatus. Journal of Huazhong Agricultural University, 2019, 38(5): 1-8 [罗双双, 高泽霞, 冯兵, 等. 二倍体和四倍体泥鳅qRT-PCR分析中内参基因优选. 华中农业大学学报, 2019, 38(5): 1-8] |
MCCURLEY A T, CALLARD G V. Characterization of housekeeping genes in zebrafish: Male-female differences and effects of tissue type, developmental stage and chemical treatment. BMC Molecular Biology, 2008, 9(1): 102 DOI:10.1186/1471-2199-9-102 |
OLSVIK P A, LIE K K, JORDAL A E O, et al. Evaluation of potential reference genes in real-time RT-PCR studies of Atlantic salmon. BMC Molecular Biology, 2005, 6: 21 DOI:10.1186/1471-2199-6-21 |
PENG B B, BING D C, ZHOU Y N, et al. Selection of reference genes for real-time quantitative PCR of peanut worm Sipuculus nudus. Fisheries Science, 2022, 41(2): 304-310 [彭冰冰, 宾东超, 周于娜, 等. 方格星虫实时荧光定量PCR内参基因的选择与验证. 水产科学, 2022, 41(2): 304-310] |
PFAFFL M W, TICHOPAD A, PRGOMET C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations. Biotechnology Letters, 2004, 26(6): 509-515 DOI:10.1023/B:BILE.0000019559.84305.47 |
SCHMID H, COHEN C D, HENGER A, et al. Validation of endogenous controls for gene expression analysis in microdissected human renal biopsies. Kidney International, 2003, 64(1): 356-360 DOI:10.1046/j.1523-1755.2003.00074.x |
SU J G, ZHANG R F, DONG J, et al. Evaluation of internal control genes for qRT-PCR normalization in tissues and cell culture for antiviral studies of grass carp (Ctenopharyngodon idella). Fish and Shellfish Immunology, 2011, 30(3): 830-835 DOI:10.1016/j.fsi.2011.01.006 |
SU X Y, HAN B Y, MENG Y Q, et al. Stability of reference genes in different tissues of triploid rainbow trout Oncorhynchus mykiss by quantitative real-time PCR. Fisheries Science, 2022, 41(1): 35-43 [苏晓燕, 韩步鹰, 孟玉琼, 等. 三倍体虹鳟实时荧光定量PCR内参基因稳定性分析. 水产科学, 2022, 41(1): 35-43] |
SUN J, LI G L, ZHU C H, et al. Molecular cloning and expression of Cyp19a1b cDNA in Clarias fuscus. Journal of Fishery Sciences of China, 2012, 19(3): 408-415 [孙晶, 李广丽, 朱春华, 等. 胡子鲇脑型芳香化酶基因全长cDNA克隆及表达. 中国水产科学, 2012, 19(3): 408-415] |
SUN Y, LI Y, LUO D, et al. Pseudogenes as weaknesses of ACTB (Actb) and GAPDH (Gapdh) used as reference genes in reverse transcription and polymerase chain reactions. Public Library of Science One, 2012, 7(8): e41659 |
TIAN C X, LIN X H, ZHOU D Y, et al. A chromosome-level genome assembly of Hongkong catfish (Clarias fuscus) uncovers a sex-determining region. BMC Genomics, 2023, 24(1): 291 DOI:10.1186/s12864-023-09394-2 |
VANDESOMPELE J, DE P K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biology, 2002, 3(7): 0034 |
WARD D S, DERN-WIELOCH J, WEIGEL R, et al. Reference gene validation for RT-qPCR, a note on different available software packages. PLoS One, 2015, 10(3): e0122515 DOI:10.1371/journal.pone.0122515 |
WAN H, YUAN W, RUAN M, et al. Identification of reference genes for reverse transcription quantitative real-time PCR normalization in pepper (Capsicum annuum L. ). Biochemical and Biophysical Research Communications, 2011, 416(1/2): 24-30 |
WANG W D, ZHU C H, DENG S P, et al. Occurrence and differentiation of gonad in Clarias fuscus. Acta Hydrobiologica Sinica, 2012, 36(5): 993-997 [王文达, 朱春华, 邓思平, 等. 胡子鲇性腺发生与分化组织学研究. 水生生物学报, 2012, 36(5): 993-997] |
WANG Y F, ZHANG S C. EF-1α is a useful internal reference for studies of gene expression regulation in amphioxus Branchiostoma japonicum. Fish and Shellfish Immunology, 2012, 32(6): 1068-1073 DOI:10.1016/j.fsi.2012.03.001 |
WU L F, QIN G X, SUN Z W, et al. Effect of dietary dehulled soybean meal replacing fish meal on the activity of digestive enzyme and the intestinal tissue of Clarias lazera. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2010, 49(4): 99-105 [吴莉芳, 秦贵信, 孙泽威, 等. 饲料中去皮豆粕替代鱼粉对埃及胡子鲇消化酶活力和肠道组织的影响. 中山大学学报(自然科学版), 2010, 49(4): 99-105] |
WU M B, YE H, YUE H M, et al. Identification of reference genes for qRT-PCR in Dabry´s sturgeon, Acipenser dabryanus. Journal of Fishery Sciences of China, 2020, 27(7): 759-770 [武梦斌, 叶欢, 岳华梅, 等. 达氏鲟实时荧光定量PCR内参基因的筛选. 中国水产科学, 2020, 27(7): 759-770] |
XIE F L, SUN G L, STILLER J W, et al. Genome-wide functional analysis of the cotton transcriptome by creating an integrated EST database. Public Library of Science One, 2011, 6(11): e26980 |
YANG C L, YUAN X Y, ZHANG J, et al. Comprehensive transcriptome analysis of reference genes for fruit development of Euscaphis konishii. PeerJ, 2020, 8: e8474 DOI:10.7717/peerj.8474 |
ZHANG C N, SAMAD R, LU K L, et al. Molecular characterization of p38 MAPK from blunt snout bream (Megalobrama amblycephala) and its expression after ammonia stress, and lipopolysaccharide and bacterial challenge. Fish and Shellfish Immunology, 2019, 84: 848-856 DOI:10.1016/j.fsi.2018.10.074 |
ZHANG N, DU W W, WANG Z D, et al. Screening of reference genes for real-time PCR in different tissues from Sillago sihama. Journal of Guangdong Ocean University, 2018, 38(5): 8-14 [张宁, 杜文文, 王中铎, 等. 多鳞  不同组织荧光定量PCR内参基因筛选. 广东海洋大学学报, 2018, 38(5): 8-14 DOI:10.3969/j.issn.1673-9159.2018.05.002] 不同组织荧光定量PCR内参基因筛选. 广东海洋大学学报, 2018, 38(5): 8-14 DOI:10.3969/j.issn.1673-9159.2018.05.002] |
ZHANG Z B, HU J Y. Development and validation of endogenous reference genes for expression profiling of medaka (Oryzias latipes) exposed to endocrine disrupting chemicals by quantitative real-time RT-PCR. Toxicological Sciences, 2007, 95(2): 356-368 DOI:10.1093/toxsci/kfl161 |
ZHAO S S, DING L J. Composition, microscopy and ultrastructure of peripheral blood cells in Clarias fuscus. Jiangsu Agricultural Sciences, 2014, 42(10): 220-223 [赵莎莎, 丁丽军. 胡子鲇外周血细胞的组成及显微与超微结构. 江苏农业科学, 2014, 42(10): 220-223 DOI:10.3969/j.issn.1002-1302.2014.10.074] |
ZHU Z R, DONG C S. The stability comparison of housekeeping genes as internal standards. Letters in Biotechnology, 2006, 17(5): 807-809 [朱芷葳, 董常生. 持家基因作为相对定量内标物的稳定性比较. 生物技术通讯, 2006, 17(5): 807-809 DOI:10.3969/j.issn.1009-0002.2006.05.038] |



