2. 上海海洋大学 上海 201306;
3. 青岛海洋科学与技术国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266237
Different Parts of China in 2014
2. Shanghai Ocean University, Shanghai 201306 ;
3. 3. Function Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266200
随着全球对虾养殖产业的不断发展,其病害也日趋严重,给养殖户造成了严重的经济损失(Nakano et al, 1994; Wongteerasupaya et al, 1995; Karunasagar et al, 1997)。据Nakano等(1994)不完全统计,自20世纪90年代白斑综合征病毒(White spot syndrome virus, WSSV)病害暴发以来,出现了大幅度的上升趋势,并且已经成为遏制养殖产业健康发展的致命点(蔡生力等, 1995; Lo et al, 1999; Flegel, 1997)。探究对虾病害的流行变异特点和规律,显得愈发重要和突出。
引起全球范围对虾病害暴发,并导致巨大经济损失的主要病因为病毒类疾病。到目前为止,在已经发现的数十种病毒中,白斑综合征病毒(White spot syndrome virus, WSSV)造成的灾害最为严重。WSSV可感染40余种甲壳纲动物以及水生浮游动物,在全球养殖区均有流行,它是一种大型、双链、环状的DNA病毒,其基因组大小约为300 kb,隶属于线头病毒科(Nimaviridae)、白斑病毒属(Whispovirus)(Marks et al, 2005; Mayo, 2002)。
同已经在GenBank上公布的中国株(WSSV-CN)、台湾株(WSSV-TW)和泰国株(WSSV-TH)基因组全序列相比较,发现有约99%的核苷酸保持一致性(于力等, 2008; Marks et al, 2004)。虽然多数基因高度保守,但在有些位点上会出现差异,主要表现在大片段插入/缺失、单核苷酸替换和在开放性阅读框(Open Reading Frames, ORFs)出现的重复序列单元数目的差异(Variable Number Tandem Repeat, VNTR) (van Hulten et al, 2001; Yang et al, 2001)。WSSV毒株间最大的差异往往表现在ORF75、ORF94和ORF125 VNTR区的变化,VNTR已被应用于WSSV分子流行病学的研究(Gudkovs et al, 2014; Dieu et al, 2004; Musthaq et al, 2006; 于洪涛, 20081))。
1) 于洪涛. 中国境内白斑综合征病毒(WSSV)分离株的序列差异分析. 中国海洋大学硕士研究生学位论文, 2008, 32−63
本研究对中国境内2014年1−8月病害暴发期间采集到的48份WSSV阳性样本,进行ORF75、ORF94和ORF125重复片段的VNTR分析,以此获取2014年WSSV病毒ORF75、ORF94和ORF125重复片段变化情况。
1 材料与方法 1.1 WSSV样本来源实验用患病样本取自中国各地2014年1−8月间出现发病症状的养殖池(表 1)。所有样本采集后置于−20℃保存备用。
|
|
表 1 WSSV样本采集信息及ORF75、ORF94和ORF125重复数 Table 1 WSSV samples and repeat units (RUs) present in ORF75, ORF94 and ORF125 |
取约30 mg鳃组织,按照海洋动物组织基因组DNA提取试剂盒(离心柱型)的说明书进行DNA提取。对WSSV DNA样本的检测采用GB/T28630的套式PCR方法。PCR产物通过用1×TBE电泳缓冲液配制的1%琼脂糖凝胶进行电泳分析。
1.3 ORF75、ORF94和ORF125的PCR扩增将所有WSSV核酸样本进行ORF75、ORF94和ORF125的PCR扩增。主要混合物与体积在3种PCR扩增中均一致:10× PCR反应缓冲液(含Mg2+) 2.5 μl,双蒸水17.3 μl,脱氧核酸混合物2 μl,正向与反向引物各0.5 μl,Ex Taq DNA聚合酶0.2 μl,WSSV模板1 μl,总体积为25 μl。PCR扩增中使用的引物与反应条件见表 2 (Dieu et al, 2004; 于洪涛等, 20081); Kathy et al, 2013; Wongteerasupaya et al, 2003)。
1)于洪涛.中国境内白斑综合征病毒(WSSV)分离株的序列差异分析. 中国海洋大学硕士研究生学位论文, 2008, 32-63
|
|
表 2 PCR扩增引物与反应条件 Table 2 PCR primers and cycling conditions |
VNTR扩增后的PCR产物用胶回收试剂盒获得纯化目的片段DNA,用NanoDrop 2000测得DNA浓度后,通过pMD®18-T Vector连接转化后的菌液送至上海生工进行测序。采用DNAMAN软件分析ORF75、ORF94和ORF125重复单元数目的差异。
2 结果与分析 2.1 套式PCR检测结果在WSSV病害暴发区采集到的48份样本中,共有6份在第1轮检测中出现阳性条带,其余42份样本均在第2轮检测中出现阳性条带。
2.2 ORF75、ORF94和ORF125扩增结果在48份WSSV样本中,3次PCR扩增均仅有部分地区的样本可检出。其中,共有12份(2#、3#、5#、6#、7#、13#、24#、27#、28#、34#、44#、48#)出现ORF75检测条带,未检出的有36份,包括山东即墨、日照、青岛,浙江宁波、湖州,江苏如东,辽宁,福建漳浦,广东湛江、江门的样本。ORF75的检出率为25.0%,其中,24#、44#与48#样本扩增条带比其他样本扩增条带略小(图 1)。
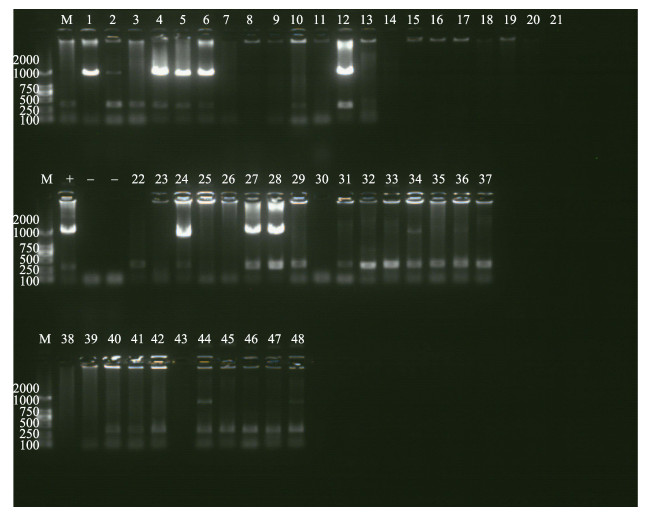
|
图 1 ORF75扩增 Figure 1 Amplification of ORF75 M: DNA Marker DL2000;+:阳性对照;-:阴性对照;1-48:样本编号 M: DNA Marker DL2000; +: Positive control; -: Negative control; 1-48: No. of samples |
PCR扩增ORF94共有18个样本(1#、12#、13#、14#、23#、24#、28#、29#、30#、31#、32#、33#、34#、35#、36#、37#、40#、47#)出现检测条带,有30份样本未检出,包括山东即墨、日照、青岛,浙江宁波、湖州,福建漳浦,广东湛江、江门的样本。ORF94检出率为37.5%,其中,前6个样本与之后的条带相比,扩增片段略大(图 2)。
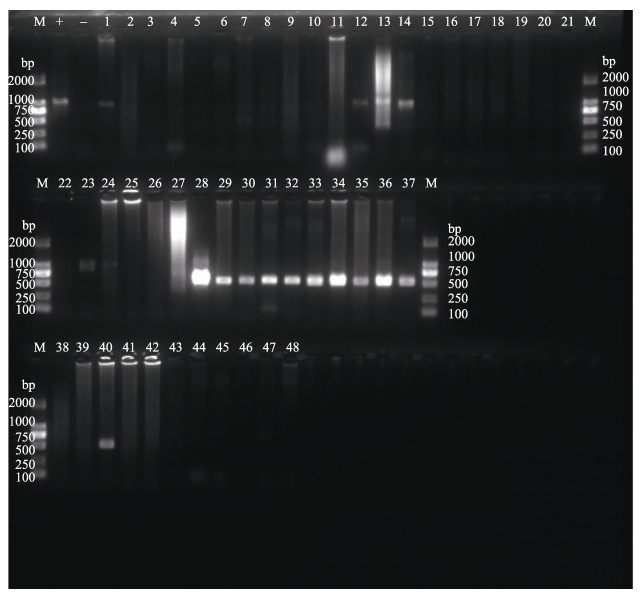
|
图 2 ORF94扩增 Figure 2 Amplification of ORF94 M: DNA Marker DL2000;+:阳性对照;-:阴性对照;1-48:样本编号 M: DNA Marker; +: Positive control; -: Negative control; 1-48: No. of samples |
ORF125的PCR扩增中,共有28份(2#、3#、4#、5#、6#、7#、12#、13#、14#、24#、25#、26#、27#、28#、30#、31#、32#、33#、34#、35#、36#、37#、40#、44#、45#、46#、47#、48#)样本出现条带,未检出的样本主要来自山东即墨、日照,浙江宁波、湖州,辽宁,广东湛江、江门。检出率为58.3%,其中,样本24#、25#较之其他样本的扩增片段略小(图 3)。
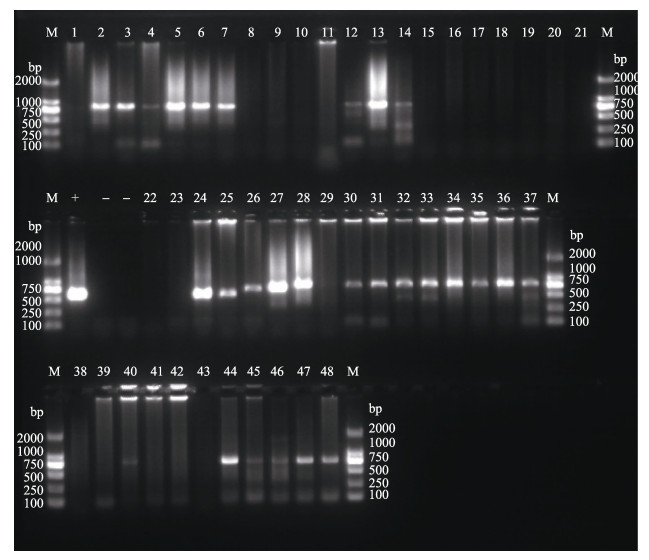
|
图 3 ORF125的扩增 Figure 3 Amplification of ORF125 M: DNA Marker DL2000;+:阳性对照;-:阴性对照;1-48:样本编号 M: DNA Marker DL2000; +: Positive control, -: Negative control; 1-48: No. of samples |
ORF75主要扩增出6种不同大小的片段,分别为2205 bp (河北曹妃甸)、2308 bp (江苏赣榆)、2013 bp (福建厦门)、2431 bp (江苏南京)、2352 bp (江苏南京、浙江温州)和1738 bp (河北黄骅),将测得的目的片段与重复单元序列比对,结果显示,45 bp的75RU数目对应为8、8、4、11、9和3,而102 bp的75 RU数目对应为2、3、1、2、3和1。在ORF94的扩增中,主要有两种不同大小的片段,902 bp (河北曹妃甸、江苏赣榆和如东、辽宁、福建厦门)和519 bp (江苏南京、浙江温州、河北黄骅),其中的重复单元数目分别为14和4。而对于ORF125的扩增,扩增出的片段长度有720 bp (河北曹妃甸)、851 bp (江苏赣榆、如东)、633 bp (福建厦门、漳州)、726 bp (山东青岛、江苏南京)、789 bp (江苏南京、浙江温州)以及812 bp (河北黄骅),其中的重复单元数目对应为5、7、3、3、6和0 (表 1)。
3 讨论本研究分析2014年采集的48份WSSV检测阳性样本,样本涉及从北向南分布的7个省份,即辽宁、河北、山东、江苏、浙江、福建和广东,样本采集时间为当地白斑病暴发期间。ORF75、ORF94和ORF125可扩增出来的概率分别为25.0%、37.5%和58.3%,但不同地区、不同样本之间还是存在明显的差异,其中,浙江的湖州、宁波,广东的湛江、江门,山东的即墨、日照地区的样本3种重复单元均未扩增出,并且一致性较高。仅在江苏的赣榆、南京部分样本和福建厦门地区的样本中,3种重复单元序列同时被扩增出。其他地区的样本有1个或者两个ORFs可以被扩增出来,但不同样本之间差异性较大。
在各地区样本的ORF75中,重复单元数目差异比较明显,在ORF75中或许存在相对活跃的变异位点或者序列,江苏南京的样本中含有的重复片段最多,且南京样本之间也有差异,而河北黄骅样本含有的重复片段最少,推测是由于DNA序列缺失较多;从ORF94的扩增结果中可以看出,仅存在两种重复单元数目,序列相对比较保守;在ORF125的扩增结果中,出现了4种重复单元数目,在这4种情况中,福建厦门、漳浦、山东青岛和江苏南京的样本序列中重复片段较之其他样本有差异,且其片段偏小,河北黄骅的样本中,片段大小虽有812 bp,但却无重复片段,猜测此样本序列有很大程度的变异。
在Kathy等(2013)对非洲马达加斯加岛、莫桑比亚和沙特阿拉伯王国地区采集的13个样本进行的研究中发现,3个地区ORF75的重复单元数目均为3,没有出现差异的情况;而ORF94大多数序列缺失,仅有沙特阿拉伯地区的两个样本出现了重复单元数目,分别为7和13;ORF125的重复单元数目为6、7、8不等(Kathy et al, 2013)。Gudkovs等(2014)研究表明,ORF94的重复单元数目在海水与淡水虾类及淡水鳌虾中也有相对的稳定性。然而,在Waikhom等(2006)的实验中却显示,在相同或不同对虾物种以及其他少量敏感甲壳纲动物宿主的不同株中,表现出了ORF94的不稳定性(Sindhuprriya et al, 2014; Waikhom et al, 2006)。通过实验研究,发现3种ORFs的重复单元数目并不确定,出现了隔离区上的差异,不同地区之间的差异性比较明显,推测这可能与不同WSSV毒株的致病力强弱有关。
不同WSSV毒株在核苷酸序列上的差异必然会造成开放性阅读框和蛋白编码的改变,差异序列的存在,可能会使病毒自身更适应环境,更利于自身复制并建立感染。因此,为了更好地做好病害的防御工作以及确保养殖产业的健康发展,研究WSSV不同毒株之间的核苷酸序列差异,可为WSSV的流行病学研究提供重要的参考依据。
| 于力, 李庆章. 虾白斑综合征病毒的研究进展. 中国预防兽医学报 , 2008, 30 (6) : 486-489 | |
| 蔡生力, 王崇明. 1993-1994年对虾暴发病的流行病学研究. 水产学报 , 1995, 19 (2) : 112-119 | |
| Dieu BTM, Marks H, Siebenga JJ, et al. Molecular epidemiology of white spot syndrome virus within Vietnam. J Gen Virol , 2004, 85 : 3607-3618 DOI:10.1099/vir.0.80344-0 | |
| Flegel TW. Major viral diseases of the black tiger prawn (Penaeus monodon) in Thailand. World J Microbiol Biotechnol , 1997, 13 (4) : 433-442 DOI:10.1023/A:1018580301578 | |
| Gudkovs N, Murwantoko M, Walker PJ. Stability of the WSSV ORF94 VNTR genotype maker during passage in marine shrimp, freshwater crayfish and freshwater prawns. Dis Aquat Organ , 2014, 111 (3) : 249-257 DOI:10.3354/dao02780 | |
| Karunasagar I, Otta SK, Karunasagar I. Histopathological and bacteriological study of white spot syndrome of Penaeus monodon along the west coast of India. Aquaculture , 1997, 153 (1-2) : 9-13 DOI:10.1016/S0044-8486(97)00011-2 | |
| Lo CF, Hsu HC, Tsai MF, et al. Specific genomic DNA fragment analysis of different geographical clinical samples of shrimp white spot syndrome virus. Dis Aquat Organ , 1999, 35 (3) : 175-185 | |
| Marks H, Goldbach RW, Vlak JM, et al. Genetic variation among isolates of white spot syndrome virus. Arch Virol , 2004, 149 (4) : 673-697 DOI:10.1007/s00705-003-0248-9 | |
| Marks H, van Duijse JA, Zuidema D, et al. Fitness and virulence of an ancestral white spot syndrome virus isolate from shrimp. Virus Res , 2005, 110 (1-2) : 9-20 DOI:10.1016/j.virusres.2005.01.002 | |
| Mayo MA. A summary of taxonomic changes recently approved by ICTV. Arch Virol , 2002, 147 (8) : 1655-1656 DOI:10.1007/s007050200039 | |
| Musthaq SS, Sudhakaran R, Ahmed VPI, et al. Variability in the tandem repetitive DNA sequences of white spot syndrome virus (WSSV) genome and suitability of VP28 gene to detect different isolates of WSSV from India. Aquaculture , 2006, 256 : 34-41 DOI:10.1016/j.aquaculture.2006.01.036 | |
| Nakano H, Koube H, Umezaea S, et al. Mass mortalities of cultured Kuruma shrimp, Penaeus japonicus, in Japan in 1993: epizootiological survey and infection trials. Fish Pathol , 1994, 29 : 135-139 DOI:10.3147/jsfp.29.135 | |
| Sindhuprriya M, Saravanan P, Otta SK, et al. White spot syndrome virus (WSSV) genome stability maintained over six passages through three different penaeid shrimp species. Dis Aquat Organ , 2014, 111 (1) : 23-29 DOI:10.3354/dao02786 | |
| van Hulten MC, Witteveldt J, Peters S, et al. The white spot syndrome virus DNA genome sequence. Virology , 2001, 286 (1) : 7-22 DOI:10.1006/viro.2001.1002 | |
| Waikhom G, John KR, George MR, et al. Differential host passaging alters pathogenicity and induces genomic variation in white spot syndrome virus. Aquaculture , 2006, 261 (1) : 54-63 DOI:10.1016/j.aquaculture.2006.07.031 | |
| Wongteerasupaya C, Pungchai P, Withyachumnarnkul B, et al. High variation in repetitive DNA fragment length for white spot syndrome virus (WSSV) isolates in Thailand. Dis Aquat Organ , 2003, 54 (3) : 253-257 | |
| Wongteerasupaya C, Vickers JE, Sriurairatana S, et al. A non-occluded, systemic baculovirus that occurs in cells of ectodermal and mesodermal origin and causes high mortality in the black tiger prawn Penaeus monodon. Dis Aquat Organ , 1995, 21 (1) : 69-77 | |
| Yang F, He J, Lin X, et al. Complete genome sequence of the shrimp white spot bacilliform virus. J Virol , 2001, 75 (23) : 11811-11820 DOI:10.1128/JVI.75.23.11811-11820.2001 |



