2. 上海海洋大学水产与生命学院 上海 201306
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
水产养殖行业发展迅猛,而养殖中出现的病害却十分严峻(Nakano et al, 1994; Wongteerasupaya et al, 1995; Karunasagar et al, 1997)。其中,作为危害最为严重之一的白斑病,自20世纪90年代被发现后,给全球各地的水产养殖行业造成了难以估量的损失。白斑病由对虾白斑综合征病毒(White spot syndrome virus, WSSV)引起。WSSV基因组约为300 kb,为环状、双链DNA病毒,能够感染数10种甲壳纲动物和水生浮游动物(Mayo, 2002; Hossain et al, 2001),可使被感染动物在3~10 d内死亡(Lightner, 1996)。
有大约99%的核苷酸会在不同毒株的基因组序列之间表现出一致性,而导致差异存在的主要原因一般为大序列的缺失或者插入、易于发生基因重组的可变区、开放型阅读框(Open reading frames, ORFs)内重复单元差异(Variable number tandem repeat, VNTR)、单核苷酸突变等(童桂香等, 2004; Marks et al, 2004)。有4种毒株已经在GenBank上公布了基因组的全序列,分别是中国台湾株(TW, AF440570) (307287 bp)、泰国株(TH, AF369029)(292967 bp)、中国株(CN, AF332093)(305107 bp)和韩国株(KR, JX515788)。具有最大基因组的WSSV毒株TH-96-Ⅱ(AY864666, 312 kb)被视为祖先株(Balakrishnan et al, 2008)。
同已知毒株基因组序列的比较,可以研究某些基因片段(ORF14/15、ORF23/24、ORF75、ORF94和ORF125)的缺失情况以及VNTR数目和单核苷酸多态性(Single nucleotide polymorphisms, SNPs),这些方面均已被应用到分子流行病学研究(Gudkovs et al, 2014; Dieu et al, 2004; Musthaq et al, 2006)。在ORF75上有2种重复单元(Repeat units, RUs),分别为45 bp和102 bp;ORF94上有1种RU,大小为54 bp;而存在于ORF125的1种RU有69 bp。根据之前报道的大多数流行病学研究,从这些RUs的数量上的差异(Dieu et al, 2004、2010; Pradeep et al, 2008; Tan et al, 2011; Shekar et al, 2012),可以更好地解释分子流行病学的调查结果。
本研究应用2015年4~10月期间在山东、江苏、天津、浙江、海南、广东6省市采集到的57份WSSV阳性的样本,通过特异性扩增目的片段,比较不同地区、不同分离株之间在ORF14/15、ORF23/24上的缺失变异情况,以及ORF75、ORF94和ORF125上的RU数目差异,以此了解我国WSSV在2015年的分子流行变异情况。
1 材料与方法 1.1 实验材料实验样本主要涉及中国明对虾(Fenneropenaeus chinensis)、凡纳滨对虾(Litopenaeus vannamei)和日本对虾(Penaeus japonicus)(表 1),所有样本采集后置于–80℃冰箱保存。
|
|
表 1 WSSV样本采集信息及ORF75、ORF94和ORF125RU数目 Table 1 WSSV samples and repeat units (RUs) of ORF75, ORF94 and ORF125 |
从样本中取鳃组织约30 mg,提取DNA[海洋动物组织基因组DNA提取试剂盒(离心柱型)],保存于–20℃备用。
1.2.2 PCR检测按照GB/T 28630.2-2012白斑综合征(WSD)诊断规程第2部分套式PCR检测法进行DNA样本检测,然后用1%的琼脂糖凝胶电泳分析。
1.2.3 PCR扩增将检测呈阳性的DNA样本通过特定的引物进行扩增,体系配置见表 2,实验过程中引物及反应条件见表 3(于洪涛, 2008; Dieu et al, 2004、2010; Marks et al, 2005; Tang et al, 2013; Wongteerasupaya et al, 2003)。
|
|
表 2 反应体系及用量 Table 2 Reaction system and amount |
|
|
表 3 PCR扩增引物以及反应条件 Table 3 PCR primers and reaction conditions |
PCR产物胶回收后用NanoDrop 2000检测DNA浓度,通过pMD®18-T连接转化,将连接成功菌液测序。ORF14/15和ORF23/24片段分别与TH-96-Ⅱ株和中国台湾株(TW)比对(Dieu et al, 2004; Zwart et al, 2010),分析序列缺失情况。ORF75、ORF94和ORF125测序结果分别与其相应的RU序列比对(Gudkovs et al, 2014)。
2 结果与分析 2.1 PCR检测结果样本核酸提取后,通过套式PCR检测,除10份样本在第二轮中呈现阳性目的条带,47份样本均在第一轮中出现阳性目的条带。
2.2 扩增结果通过特定引物扩增,在ORF14/15中,除1#、2#、3#、5#、6#、7#、8#、12#、21#、23#、25#、26#、27#、28#、29#、30#、31#、40#、52#、53#、54#和55#外,其余35份样本均有目的条带出现,检出率为61.40%。13#、14#、15#、16#、17#、35#、36#和37#样本的条带较之其他条带略大,其中,山东利津3个样本均未检出条带(图 1)。在ORF23/24扩增中,共有14份样本(9#、24#、32#、33#、34#、39#、40#、45#、46#、47#、48#、51#、56#和57#)出现目的条带(图 2),检出率为24.56%。目的条带之间的大小基本没有差别。山东利津、昌邑、威海、江苏赣榆、天津汉沽、宝坻和广东广州没有目的条带出现。而在ORF75中,共有10份样本(4#、9#、32#、33#、34#、45#、46#、47#、48#和57#)检出(图 3),检出率为17.54%,山东利津、昌邑威海、潍坊、天津汉沽、宝坻、海南海口和广东广州无样本检出。其中,57#山东潍坊的样本在所有目的条带中,片段最大。在ORF94中,共有40份样本检出,检出率为70.18%,其中,1#和3#的目的片段最小(图 4)。江苏赣榆、天津宝坻和广东广州无目的条带出现。在ORF125,共有44份样本检出,检出率为77.19%,在所有地区的样本中均有发现。仅有1#、2#、19#、20#、21#、23#、24#、25#、27#、28#、29#、30#和31#样本无相应目的条带出现(图 5)。
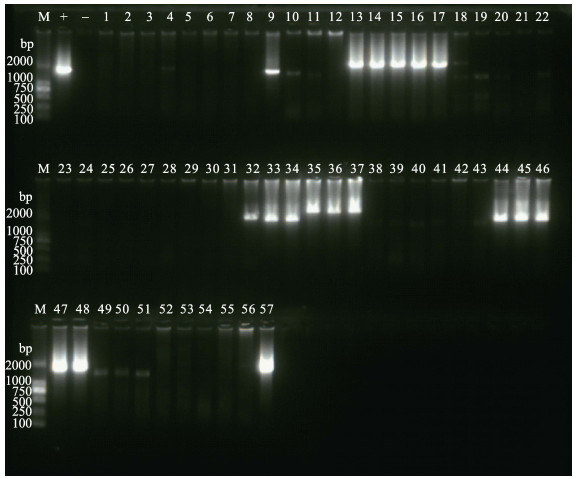
|
图 1 引物ORF14/15的扩增结果 Figure 1 Amplification of primer ORF14/15 M: DNA Marker DL 2000; +:阳性对照; –:阴性对照; 1~57:样本编号。下图同 M: DNA Marker DL 2000; +: Positive control; –: Negative control; 1~57: Sample number. The same as below |

|
图 2 引物ORF23/24的扩增结果 Figure 2 Amplification of primer ORF23/24 |
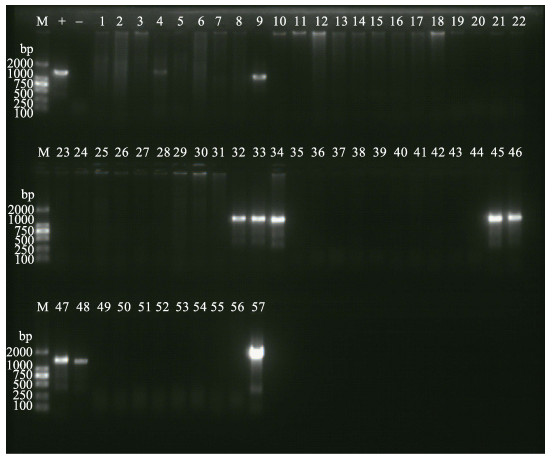
|
图 3 引物ORF75的扩增结果 Figure 3 Amplification of primer ORF75 |
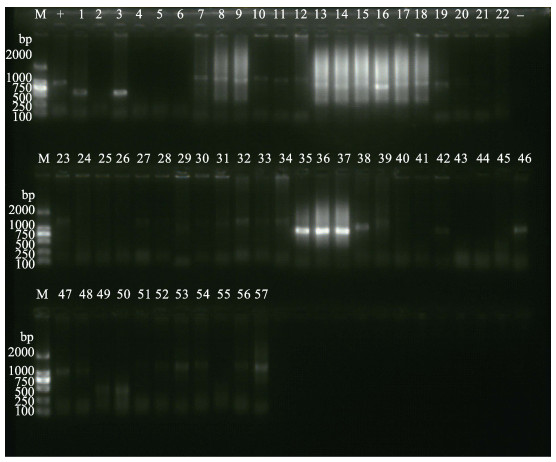
|
图 4 引物ORF94的扩增 Figure 4 Amplification of primer ORF94 |

|
图 5 ORF125扩增 Figure 5 Amplification of primer ORF125 |
在ORF14/15扩增中,共扩增3种不同的片段,分别为1270 bp(山东即墨)、1892 bp(山东即墨、潍坊、寿光、浙江舟山、江苏赣榆、天津汉沽、北辰、宝坻、海南海口和广东广州)、2075 bp(山东威海、昌邑),通过与TH-96-Ⅱ比对,发现分别缺失6530、5908、5725 bp (图 6)。而在ORF23/24中,所有样本片段大小均为1140 bp,与台湾株相比对,缺失12070 bp (图 7)。

|
图 6 WSSV不同毒株ORF14/15区域的序列比对 Figure 6 Scheme of deletion region of ORF14/15 in different WSSV strains 左右两边的矩形表示用引物ORF14/15扩增出的序列,中间的虚线表示与TH-96-Ⅱ毒株进行序列比对缺失的部分 Rectangles mean sequences amplified by ORF14/15. The imaginary lines in the middle mean sequences deletion compared to TH-96-Ⅱ strain |

|
图 7 WSSV不同毒株ORF23/24区域的序列比对 Figure 7 Scheme of deletion region of ORF23/24 in different WSSV strains 左右两边的矩形表示用引物扩增出的序列, 中间的虚线表示与TW毒株进行序列比对缺失的部分 The right and left rectangles mean sequences which can be amplified. The imaginary line in the middle means sequence deletion compared to TW strain |
在ORF75扩增中,有3种片段大小出现,分别为1246 bp(江苏赣榆、天津北辰)、985 bp(山东即墨、寿光、天津北辰和浙江舟山)和1738 bp(山东潍坊),通过与重复片段比对,45 bp的重复片段数目分别为2、1、3,而102 bp的重复片段均为1(表 1)。在ORF94扩增中,共有5种大小不一的片段出现,分别为523 bp(山东利津)、742 bp(山东即墨、威海)、769 bp(海南海口)、1024 bp(山东即墨、天津北辰)和1135 bp(山东潍坊、即墨、寿光、天津汉沽、浙江舟山),经比对,重复片段数目分别为4、5、5、10、12(表 1)。在ORF125扩增中,出现5种不同的片段,分别为491 bp(山东利津、昌邑、浙江舟山)、518 bp(山东即墨)、703 bp(山东威海、海南海口、天津汉沽、北辰)、722 bp(山东即墨、浙江舟山)、746 bp(山东昌邑、寿光、潍坊、天津北辰、宝坻、广东广州),经过比对,其重复片段的数目分别为3、3、5、5、6(表 1)。
3 讨论本研究所用样本来自于山东利津、即墨、昌邑、寿光、威海、潍坊、江苏赣榆、天津汉沽、北辰、宝坻、浙江舟山、海南海口和广东广州,采集时间分布于2015年4~10月WSSV病害暴发期间。ORF14/15和ORF23/24的检出率分别为61.40%和24.56%,大部分扩增ORF23/24成功的样本在ORF14/15中均有目的条带出现,但除去40#样本(海南海口),其仅在ORF23/24扩增中出现。ORF75、ORF94和ORF125的检出率分别为17.54%、70.18%和77.19%,其中,2#(山东利津)、21#(山东即墨)、24#(山东即墨)和25#(山东即墨)样本在3种扩增中均无条带出现,而9#(山东即墨)、32#(浙江舟山)、33#(浙江舟山)、34#(山东寿光)、46#(天津北辰)、47#(天津北辰)、48#(天津北辰)和57#(山东潍坊)样本却在3种扩增中均有条带出现。在5次PCR扩增中,9#(山东即墨)、32#(浙江舟山)、33#(浙江舟山)、34#(山东寿光)、46#(天津北辰)、47#(天津北辰)、48#(天津北辰)和57#(山东潍坊)样本都出现目的条带,而2#(山东利津)、21#(山东即墨)和25#(山东即墨)样本则在5次扩增中均无扩增出目的片段。
对于ORF14/15,TH-96-Ⅱ最为完整,为7800 bp。与TH-96-Ⅱ相比较,韩国株有5721 bp的缺失,泰国株缺失5316 bp,台湾株缺失5138 bp,而中国株缺失5132 bp。本研究中出现3种缺失程度不一样的片段,且相互之间差别较大,即其中2075 bp的片段与韩国株KR的缺失程度相近。6530 bp和5908 bp的2种缺失情况也出现在之前的研究中,而与6530 bp相近的6533 bp和6540 bp的缺失情况分别在2013年和2014年的研究中出现(孙新颖等, 2016a、2016b)。在Tang等(2013)研究中也有相近程度的情况,即缺失5950 bp,而印度的1株和墨西哥地区的3株WSSV毒株有更接近的缺失程度,4株分离株均缺失5892 bp。从现有研究发现,WSSV在ORF14/15上的缺失均在5000 bp以上,相较于完整的7800 bp,缺失程度较大,可能越短的DNA片段在复制中占据更多优势。
报道中的中国株ORF23/24片段缺失最小,仅为1169 bp;泰国株的缺失程度最大,为13120 bp;而韩国株的缺失程度为中等,为5654 bp。Lan等(2002)发现,在中国厦门发现3种不同宿主感染的WSSV,相对中国株的ORF23/24序列分别缺失5717、5926和9319 bp。而Dieu等(2004)发现,在越南中部和南部采集的3种不同的WSSV分离株,相较于台湾株分别缺少8539、11450和12166 bp。相较于其他地区或大或小的缺失情况,本研究中所有样本在ORF23/24上缺失程度一致,均缺失12070 bp,属于是大片段的缺失,这与2013年样本研究中的缺失情况一致,而2014年样本有2种缺失,即12064 bp和12070 bp,二者仅是在中间位置相差6个碱基(孙新颖等, 2016a、2016b)。ORF23/24的这种大片段缺失在普遍范围上比较稳定,结合ORF14/15较大的缺失情况,推测WSSV的进化可能通过逐渐稳定的缺失和更小的基因组来完成,使得基因组更适合复制,以适应环境且WSSV自身具有更强的毒性。
除了ORF75之外,ORF94和ORF125的检出率都比较高。在是否检出方面,不同地区样本之间的RU数目差异比较大,即使是出自于同一地区的样本之间也依然有差异,如山东即墨的样本。ORF75中的45 bp的RUs数目为3以及102 bp的RUs数目为1的情况在之前2014年样本研究中有出现,新出现了45 bp RU为1和2两种类型;ORF94中RUs为4的情况在2013年和2014年样本中均存在,而RUs为12只出现在2013年样本中,且样本中的RUs或为小数目重复,或为大数目重复,RUs为5和10两种属于新类型;ORF125的RUs数目与2014年样本检测中的数据部分一致(孙新颖等, 2016a、c)。Tang等(2013)研究中仅有1种ORF75的VNTR情况:共3个RU,2个45 bp RU和1个102 bp RU;Durán-Avelar等(2015)发现,ORF75总RU有5~20之间共12种情况,而ORF94包含有13种数目不同的RU,ORF125则只有1种总RU数目为8的情况;此外,Sindhupriya等(2014)的实验中,ORF94上有4、6、7、8和11五种不同的VNTR,而ORF125上有3种(4、5和6)不同的VNTR,部分VNTR情况在不同研究中存在交叉情况。相较于ORF14/15和ORF23/24,ORF75、ORF94和ORF125VNTR的研究更多应用于WSSV类型的归划(Durán-Avelar et al, 2015; Tang et al, 2013),从而确定WSSV的地理归属,揭示WSSV分子流行情况。
2015年样本在重复片段数目上的差异依然很大,ORF14/15上的缺失出现了新的片段,与韩国株极其相近,在ORF23/24上,虽然出现大片段缺失,却表现出了缺失的稳定性。而各个样本在ORF75、ORF94和ORF125上的RU数目则出现差异性和部分稳定性。因此,推测不同地区WSSV毒株为了更好复制遗传以适应环境,出现了适应性的变异,同时本实验也为系统的分子流行病的研究提供了数据基础。
| Balakrishnan P, Malathi S, Iddya K, et al. Characterization of variable genomic regions of Indian white spot syndrome virus. Virology, 2008, 376(1): 24-30 DOI:10.1016/j.virol.2008.02.037 | |
| Dieu BTM, Marks H, Siebenga JJ, et al. Molecular epidemiology of white spot syndrome virus within Vietnam. Journal of General Virology, 2004, 85(12): 3607-3618 DOI:10.1099/vir.0.80344-0 | |
| Dieu BTM, Marks H, Zwart MP, et al. Evaluation of white spot syndrome virus variable DNA loci as molecular markers of virus spread at intermediate spatiotemporal scales. Journal of General Virology, 2010, 91(5): 1164-1172 DOI:10.1099/vir.0.018028-0 | |
| Durán-Avelar MDJ, Pérez-Enríquez R, Zambrano-Zaragoza JF, et al. Genotyping WSSV isolates from northwestern Mexican shrimp farms affected by white spot disease outbreaks in 2010-2012. Diseases of Aquatic Organisms, 2015, 114(1): 11-20 DOI:10.3354/dao02844 | |
| Gudkovs N, Murwantoko, Walker PJ. Stability of the WSSV ORF94 VNTR genotype maker during passage in marine shrimp, freshwater crayfish and freshwater prawns. Diseases of Aquatic Organisms, 2014, 111(3): 249-257 DOI:10.3354/dao02780 | |
| Hossain MS, Chakraborty A, Joseph B, et al. Detection of new hosts for white spot syndrome virus of shrimp using nested polymerase chain reaction. Aquaculture, 2001, 198(1-2): 1-11 DOI:10.1016/S0044-8486(00)00571-8 | |
| Karunasagar I, Otta SK, Karunasagar I. Histopathological and bacteriological study of white spot syndrome of Penaeus monodon along the west coast of India. Aquaculture, 1997, 153(1-2): 9-13 DOI:10.1016/S0044-8486(97)00011-2 | |
| Lan Y, Lu W, Xu X. Genomic instability of prawn white spot bacilliform virus (WSBV) and its association to virus virulence. Virus Research, 2002, 90(1-2): 269-274 DOI:10.1016/S0168-1702(02)00231-9 | |
| Lightner DV. A handbook of pathology and diagnostic procedures for diseases of penaeid shrimp. Special publication of the World Aquaculture Society. LA, Baton Rouge, 1996, 305 | |
| Marks H, Goldbach RW, Vlak JM, et al. Genetic variation among isolates of white spot syndrome virus. Archives of Virology, 2004, 149(4): 674-697 | |
| Marks H, van Duijse JJA, Zuidema D, et al. Fitness and virulence of an ancestral white spot syndrome virus isolate from shrimp. Virus Research, 2005, 110(1-2): 9-20 DOI:10.1016/j.virusres.2005.01.002 | |
| Mayo MA. A summary of taxonomic changes recently approved by ICTV. Archives of Virology, 2002, 147(8): 1655-1663 DOI:10.1007/s007050200039 | |
| Musthaq SS, Sudhakaran R, Ahmed VPI, et al. Variability in the tandem repetitive DNA sequences of white spot syndrome virus (WSSV) genome and suitability of VP28 gene to detect different isolates of WSSV from India. Aquaculture, 2006, 256(1-4): 34-41 DOI:10.1016/j.aquaculture.2006.01.036 | |
| Nakano H, Koube H, Umezaea S, et al. Mass mortalities of cultured Kuruma shrimp, Penaeus japonicus, in Japan in 1993: Epizootiological survey and infection trials. Fish Patholog, 1994, 29(2): 135-139 DOI:10.3147/jsfp.29.135 | |
| Pradeep B, Shekar M, Gudkovs N, et al. Genotyping of white spot syndrome virus prevalent in shrimp farms of India. Diseases of Aquatic Organisms, 2008, 78(3): 189-198 | |
| Shekar M, Pradeep B, Karunasagar I. White spot syndrome virus: Genotype, epidemiology and evolutionary studies. Indian Journal of Virology, 2012, 23(2): 175-183 DOI:10.1007/s13337-012-0078-z | |
| Sindhupriya M, Saravanan P, Otta SK, et al. White spot syndrome virus (WSSV) genome stability maintained over six passages through three different penaeid shrimp species. Diseases of Aquatic Organisms, 2014, 111(1): 23-29 DOI:10.3354/dao02786 | |
| Sun XY, Wan XY, Liu QH, et al. Deletion and variation sequence comparison of prawns WSSV from different parts of China in 2013. Journal of Fishery Sciences of China, 2016a, 23(3): 693-703 [孙新颖, 万晓媛, 刘庆慧, 等. 2013年中国典型对虾养殖区白斑综合征病毒流行株高变异区序列的分析比较. 中国水产科学, 2016a, 23(3): 693-703] | |
| Sun XY, Liu QH, Wan XY, et al. Comparison of the missing sequences of ORF14/15 and ORF23/24 of WSSV from different regions of China in 2014. Progress in Fishery Sciences, 2016b, 37(4): 140-146 [孙新颖, 刘庆慧, 万晓媛, 等. 2014年中国不同地区对虾白斑综合征病毒ORF14/15和ORF23/24缺失区序列比较. 渔业科学进展, 2016b, 37(4): 140-146] | |
| Sun XY, Wan XY, Liu QH, et al. Sequence comparison of WSSV variable regions from different parts of China in 2014. Progress in Fishery Sciences, 2016c, 37(2): 127-133 [孙新颖, 万晓媛, 刘庆慧, 等. 白斑综合征病毒2014年中国毒株变异区的序列比较. 渔业科学进展, 2016c, 37(2): 127-133] | |
| Tan YW, Shi ZL. Genotyping of white spot syndrome virus in Chinese cultured shrimp during 1998-1999. Virologica Sinica, 2011, 26(2): 123-130 DOI:10.1007/s12250-011-3181-1 | |
| Tang KFJ, Groumellec ML, Lightner DV. Novel, closely related, white spot syndrome virus (WSSV) genotypes from Madagascar, Mozambique and the Kingdom of Saudi Arabia. Diseases of Aquatic Organisms, 2013, 106(1): 1-6 DOI:10.3354/dao02645 | |
| Tong GX, Li XZ, Wei XX, et al. Comparative analysis of deletion region of white spot syndrome virus genome among isolates in Guangxi. Journal of Shanghai Ocean University, 2004, 23(1): 8-13 [童桂香, 黎小正, 韦信贤, 等. 白斑综合征病毒广西株缺失区基因的比较分析. 上海海洋大学学报, 2004, 23(1): 8-13] | |
| Wongteerasupaya C, Pungchai P, Withyachumnarnkul B, et al. High variation in repetitive DNA fragment length for white spot syndrome virus (WSSV) isolates in Thailand. Diseases of Aquatic Organisms, 2003, 54(3): 253-257 | |
| Wongteerasupaya C, Vickers JE, Sriurairatana S, et al. A non-occluded, systemic baculovirus that occurs in cells of ectodermal and mesodermal origin and causes high mortality in the black tiger prawn Penaeus monodon. Diseases of Aquatic Organisms, 1995, 21(1): 69-77 | |
| Yu HT. Analysis of sequence variability among white spot syndrome virus (WSSV) isolates within China. Masterxs Thesis of Ocean University of China, 2008: 32-63. [于洪涛. 中国境内白斑综合征病毒(WSSV)分离株的序列差异分析. 中国海洋大学硕士研究生学位论文, 2008: 32-63.] | |
| Zwart MP, Dieu BTM, Hemerik L, et al. Evolutionary trajectory of white spot syndrome virus (WSSV) genome shrinkage during spread in Asia. PLoS One, 2010, 5(10): e13400 DOI:10.1371/journal.pone.0013400 |



