2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 农业农村部海洋渔业可持续发展重点实验室 山东省渔业资源与生态环境重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
4. 上海海洋大学水产与生命学院 上海 201306
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071;
3. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs; Shandong Provincial Key Laboratory of Fishery Resources and Eco-Environment, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
4. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
保护多样性是生态学和保护生物学的主要目标(Vermeulen et al, 2002),也是政府制定相关保护措施与政策的根本依据。然而,精确评价生物多样性、掌握目标种的分布与生物量需要花费大量的人力、物力和财力,尤其对于水生生物而言,其特殊生存环境更是增加了研究的困难。
随着分子生物学的高速发展,可以直接从环境样品中提取DNA片段,然后利用高通量测序及荧光定量PCR等技术进行物种定性或定量分析,即eDNA (Environmental DNA, eDNA)技术(Ficetola et al, 2008; Haile et al, 2009; Bohmann et al, 2014; Evans et al, 2017; 单秀娟等, 2018),这种技术作为一种新的物种监测方法应用到水生生物调查中。然而,由于物种之间的差异,其释放的eDNA量的多少与eDNA片段的大小各不相同(Geerts et al, 2018)。因此,针对不同的研究对象应采用不同的eDNA富集与提取方法,以期达到最佳的研究效果。
自20世纪80年代以来,中国对虾(Fenneropenaeus chinensis)资源严重衰退,目前其捕捞产量主要来源于增殖放流(袁伟等, 2015)。放流初期,其个体较小、游泳能力较弱,其群体数量相对较少,传统的方法难以准确监测。因此,为了能够准确掌握中国对虾的分布与资源量状况,合理开发利用其资源,本研究以渤海中国对虾为研究对象,采用滤膜法富集eDNA,结合DNeasy Blood and Tissue kit提取eDNA,最后应用实时荧光定量PCR(绝对定量)分析DNA样品,建立了一套适于中国对虾研究的eDNA技术操作流程,旨在为中国对虾分布监测及其资源评估提供一种新的方法。
1 材料与方法 1.1 特异性引物设计针对中国对虾的线粒体细胞色素酶氧化亚基Ⅰ(COⅠ)基因设计特异性引物。在GenBank数据库中检索中国对虾COⅠ基因序列,利用BioEdit和MEGA7软件进行序列比对,使用Primer Premier 6与Beacon Designer 8软件设计引物与探针,并在NCBI网站上进行引物特异性测试。引物由生工生物工程(上海)股份有限公司合成,详细信息见表 1。
|
|
表 1 中国对虾mtDNA COⅠ基因PCR扩增引物信息 Tab.1 Primer pairs for mtDNA COⅠ of F. chinensis |
中国对虾肌肉组织取自2016年7月渤海渔业资源调查捕获的中国对虾,-20℃保存。
DNA提取采取传统的酚-氯仿-异戊醇方法,具体参照闫晗等(2012)的方法并做相应的改进,最后将提取的DNA溶液稀释到50 ng/μl,用1.5 ml无菌离心管-20℃保存备用。PCR 25 μl体系:10×Taq Buffer 2.5 μl,dNTPs(各2.5 mmol/L)0.5 μl,正反向引物(COⅠPF/COⅠPR) (10 mmol/L)各0.5 μl,Taq DNA Polymerase (5 U/μl) 0.5 μl,模板DNA(50 ng/μl) 1 μl, MgCl2 (25 mmol/L) 1.5 μl,ddH2O 18 μl。PCR反应程序:94℃预变性3 min;94℃变性30 s,60℃退火30 s,72℃延伸1 min,35个循环;72℃复延伸10 min。引物对COⅠDF/COⅠDR的反应体系与反应条件与引物对COⅠPF/COⅠPR相同。
1.3 重组质粒标准品的制备将1.2中的PCR产物纯化,连接到pMD-18-T质粒载体上,转化入大肠杆菌感受态细胞中,在LB固体平板培养基上过夜培养,挑取单菌落扩大培养进行质粒DNA提取,将质粒DNA稀释到特定浓度,-80℃保存备用。
1.3.1 PCR产物纯化将1.2中PCR产物用2%的琼脂糖凝胶电泳进行检测,选择电泳条带单一且明亮的产物使用Gel Extraction Kit(OMEGA)试剂盒进行切胶回收,纯化的PCR产物-20℃保存。具体实验步骤参照说明书。
1.3.2 质粒连接转化将经过纯化的PCR产物连接到pMD-18-T质粒载体(TaKaRa)上(具体操作详见说明书);将10 μl连接产物加入100 μl DH5α感受态细胞(TaKaRa)中,冰浴30 min;42℃热激1 min,将重组质粒转化入感受态细胞;加入1 ml SOC培养基,37℃ 150 r/min振荡培养1 h;取150 μl菌液将其涂布在LB固体培养基上(含Amp、X-gal、IPTG)上,37℃倒置过夜培养;挑取6个白色单菌落分别置于1.5 ml离心管中培养,菌液送生工生物工程(上海)股份有限公司测序;选取连接转化成功的菌液扩大培养。
1.3.3 标准品制备将经过扩大培养的菌液使用Plasmid Mini Kit(OMEGA)试剂盒进行质粒DNA提取(具体操作参照说明书),提取完成后,用紫外分光光度计检测DNA浓度,并将其稀释为108 copies/μl的标准品,-80℃保存。
1.4 eDNA样品采集实验水样取自中国水产科学研究院黄海水产研究所水产遗传育种中心的中国对虾养殖池,养殖池长5.5 m、宽3.6 m、高1.2 m,养殖水体体积为14 m3。其中,对虾平均体重为27 g,每池180尾虾(中国对虾的体重及数量数据由基地工作人员收集)。
实验水样采集方案设计如下:选取直径为47 mm的玻璃纤维膜、硝酸纤维膜、聚碳酸酯膜、尼龙膜共4种材质的滤膜,每种滤膜根据其孔径大小设置0.45、0.8、1.2、5 μm共4个梯度,取水量设置500 ml、1 L、2 L共3个梯度,同一样本(即使用同一孔径与同一材质的滤膜过滤相同体积的水样)做3次技术重复。此外,在养殖池入水口取6个平行样本作为阴性对照,共计150个样本。取养殖池表层水,采用无菌真空抽滤装置进行过滤,每次过滤之前用无菌水冲洗过滤漏斗,防止交叉污染。过滤完成后,将每张滤膜单独卷起来,放入1.5 ml离心管中并做好标记,-20℃保存直至进行DNA提取。
1.5 eDNA提取利用DNeasy Blood and Tissue Kit(Qiagen, 德国)试剂盒提取DNA,参照Renshaw等(2015)的方法并加以改进。将滤膜取出解冻后,用剪刀将滤膜剪成细条状,放入2 ml的离心管中,加入570 μl Buffer ATL和60 μl蛋白酶K,涡旋混匀,恒温水浴3 h (期间每隔15 min轻轻颠倒混匀);加入630 μl Buffer AL,涡旋震荡混匀;加入630 μl无水乙醇,涡旋震荡混匀;将2 ml离心管中的混合液分3次转移到DNeasy离心柱中(离心柱每次能承载液体为700 μl),室温8000 g离心1 min,弃滤液及收集管;将离心柱放入新收集管,加入500 μl Buffer AW1。室温8000 g离心1 min,弃滤液及收集管;将离心柱放入新收集管中,加入500 μl Buffer AW2。室温12000 g离心3 min,弃滤液及收集管;将离心柱放入新1.5 ml离心管中,在离心柱中部加入50 μl Buffer TE,室温孵育1 min,室温8000 g离心1 min;重复上一步骤以增大DNA产量。
1.6 eDNA的定量分析所有提取的eDNA样品采用BBI生命科学有限公司的2× TaqMan Fast qPCR Master Mix (Low Rox)实时荧光定量PCR试剂盒进行定量分析。PCR反应体系采用20 μl体系:10 μl 2×TaqMan Fast qPCR Master Mix,0.4 μl正向引物(10 μmol/L),0.4 μl反向引物(10 μmol/L),0.4 μl探针(10 μmol/L),2 μl模板DNA,6.8 μl PCR水。扩增反应程序采用两步法:(1)94℃预变性3 min;(2)94℃变性5 s,60℃退火延伸34 s,40个循环。
标准品及每个eDNA样品做3个重复,即每一采样类型9个重复(即使用同一孔径与同一材质的滤膜过滤相同体积的水样)(3个qPCR重复×3次取样重复),每个96孔板做3个无模板空白对照以检测污染问题,标准品浓度从107 copies/μl以10倍为梯度稀释到102 copies/μl。最终的eDNA拷贝数取平均值,实验数据采用绝对定量法分析。使用Applied Biosystems ABI 7500型定量PCR仪和96孔板(Thermo Fisher)进行qPCR扩增,应用系统软件SDS1.4.0.25自动计算Ct值及生成标准曲线与扩增曲线。
1.7 数据分析数据分析与处理在R.3.0.2软件上进行,使用GAM进行曲线拟合,误差控制在95%的置信区间以内。
2 结果与分析 2.1 引物特异性本实验设计的引物成功扩增出了597 bp和106 bp的特异性目的片段,与预期结果完全一致,其测序结果在NCBI网站上经过Blast比对分析,其结果显示与原始序列片段同源性达100%。同时,引物特异性通过对虾肌肉组织DNA的PCR扩增与琼脂糖凝胶电泳检测得到验证,其目的条带单一且明亮,结果如图 1所示。
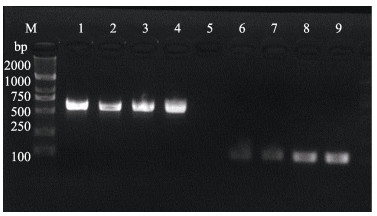
|
图 1 中国对虾mtDNA COⅠ的扩增结果 Fig.1 Amplification of mtDNA COⅠ of F. chinensis M: DNA Marker DL 2000;泳道1~4:引物COⅠPF/PR的PCR产物; 泳道6~9:引物COⅠDF/DR的PCR产物 M: DNA Marker DL 2000; Lane 1~4: PCR products of COⅠPF/PR; Lane 6~9: PCR products of COⅠDF/DR |
共采集132个样本:养殖池入水口出6个;玻璃纤维膜与硝酸纤维膜均取36个,无堵塞现象出现;聚碳酸酯膜为30个,此材质的孔径大小为0.45 μm,在过滤300 ml水样后便被堵塞;尼龙膜为24个,此材质的孔径大小为0.45 μm,在过滤150 ml水样后堵塞,而孔径大小为0.8 μm的滤膜,在过滤350 ml水样后堵塞。
在整个环境DNA水样采集过程发现,用不同材质、相同孔径的滤膜过滤同一体积的水样,用玻璃纤维膜过滤所用时间最短,主要原因在于玻璃纤维膜的通透性最强。
2.3 DNA检测提取的中国对虾肌肉组织DNA纯度及浓度均较高,其PCR产物电泳检测目的条带单一(图 1)。
eDNA提取完成后,紫外分光光度计检测显示,DNA浓度最小值为12.23 ng/μl,最大为173.68 ng/μl,且大部分DNA样品的A260 nm/A280 nm值在1.8~2.0之间。此结果与已有研究结果相比,eDNA浓度及纯度均较高,这与取样的养殖池中国对虾密度较大且物种单一有关。此外,物种之间的差异也导致了不同的eDNA释放速率与释放量。
2.4 标准曲线的制备通过实时荧光定量PCR扩增,系统根据荧光值的变化自动生成中国对虾COⅠ基因的标准曲线与扩增曲线(图 2)。曲线的相关系数R2=0.993,回归方程为Y=-3.531x+40.975,说明本研究在稀释的质粒标准品浓度范围内具有良好的线性关系,所建立的标准曲线能够正确反映出中国对虾COⅠ基因的扩增。
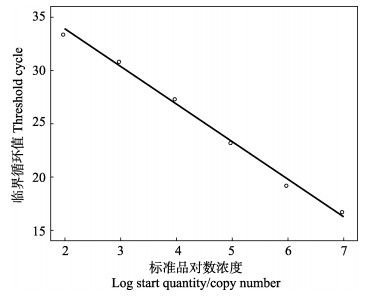
|
图 2 中国对虾COⅠ基因的qPCR标准曲线 Fig.2 The standard curve of qPCR of F. chinensis COⅠ gene |
通过实时荧光定量PCR的检测发现(图 3、图 4),在滤膜未出现堵塞的情况下,用同一材质、同一孔径的滤膜过滤不同体积的水样,过滤的水样体积越大,所提取的DNA浓度(copies/μl)及产量(copies)越大。在滤膜未出现堵塞的情况下,用同一材质、不同孔径的滤膜过滤相同体积的水样,滤膜孔径越大,提取的DNA浓度及产量越小。通过对所有样品的检测,结果显示,使用0.45 μm的玻璃纤维滤膜过滤2 L的水样能够提取到的DNA的浓度最高,为1750 copies/μl,且其eDNA产量也最大。理论上,随着取样水量的成倍增加,所提取eDNA的浓度及产量的平均值均应该呈倍数关系增加,但由于在实验操作的过程中存在一定的误差,导致最终提取的eDNA浓度及产量并非呈严格的倍数关系递增。
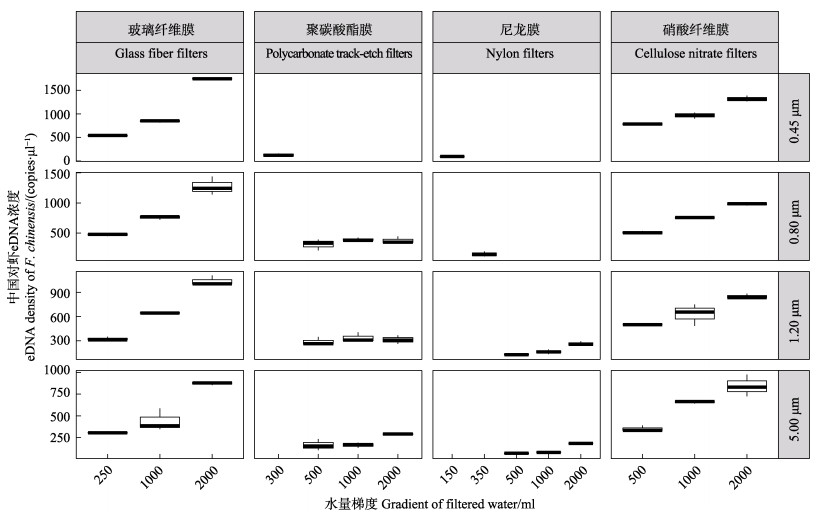
|
图 3 不同滤膜类型及过滤水量所富集的eDNA的浓度 Fig.3 Concentration of eDNA enriched by different membrane and volume of filtered water |
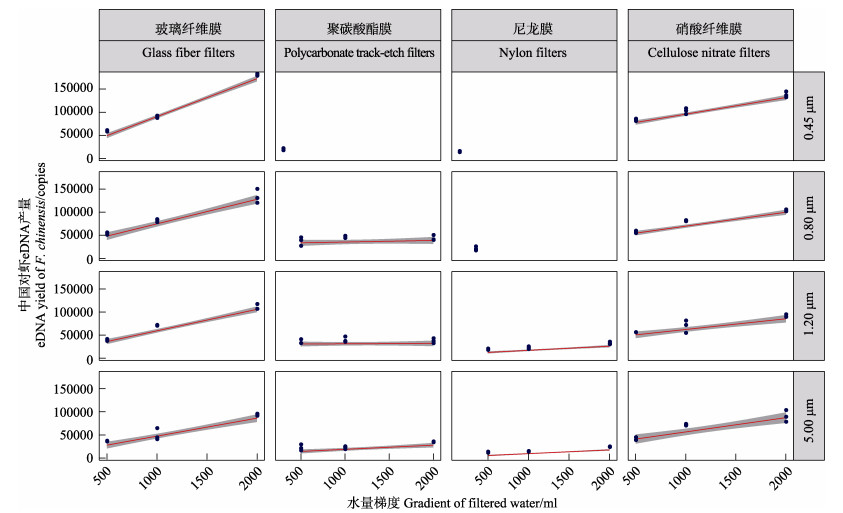
|
图 4 不同滤膜类型及过滤水量所富集的eDNA的产量 Fig.4 Output of eDNA enriched with different membrane and volume of filtered water |
与传统调查方法相比,eDNA技术具有经济高效、省时省力、灵敏度高等特点,对于资源衰退严重的物种,此方法能更准确地反应其分布与资源量状况,但不同水样采集方法、eDNA富集方法及eDNA的提取方法均会对物种定性与定量分析产生影响(Minamoto et al, 2016; Geerts et al, 2018)。
3.1 水样采集Moyer等(2014)对池塘表层水、中层水、底层水分析发现,表层水与底层水的效果较好,但海洋生态环境复杂,不同于池塘,已有研究采集的水样从15 ml至10 L不等,以1 L与2 L为主(Rees et al, 2014)。本研究发现,在滤膜没有堵塞之前,采集水样体积越大,获得DNA拷贝越多,后期分析越有利,由于海上调查时受时间限制,建议过滤2 L水样为宜。
3.2 eDNA富集方法eDNA富集方法通常有酒精沉淀(Thomsen et al, 2012)、离心(Klymus et al, 2015)及过滤(Jerde et al, 2011)三种方法。酒精沉淀法和离心法适合于小体积水样的eDNA富集,主要用于养殖池及其他人工水体等静水生态系统。由于目标种检出率受生物量影响,相对于人工水体,自然生态系统中目标种密度相对较小,为了避免假阳性结果产生,建议采用滤膜法采集水样,以增加目标种的检出率,尤其适合中国对虾这种资源衰退严重的物种。此外,选用滤膜法更有利于样品保存,有效防止eDNA的降解。
水生生物释放到环境中的有效DNA片段集中在0.1~10 μm之间(Turner et al, 2014),选取合适的滤膜孔径对于富集eDNA至关重要。目前,最常用的滤膜有4种:玻璃纤维膜(Jerde et al, 2011)、硝酸纤维膜(Goldberg et al, 2011)、聚碳酸酯膜(Takahara et al, 2012)、尼龙膜(Thomsen et al, 2012)。各种滤膜对eDNA的富集效果取决于具体研究对象,不同研究对象的最适合滤膜也不同。本研究发现,使用同一孔径、不同材质的滤膜过滤同一体积水样的时间不同,其中,0.45 μm的玻璃纤维膜通透性最强,时间最短,对中国对虾eDNA的富集效果最佳。
3.3 eDNA的提取目前,eDNA提取主要采用酚-氯仿-异戊醇法(Costas et al, 2007; Deiner et al, 2014)与商业化的试剂盒提取(Minamoto et al, 2012; Sigsgaard et al, 2017)。酚-氯仿-异戊醇法与试剂盒法相比价格低,由于其在提取过程中DNA损耗较多,用于生物量丰富的物种定性检测尚可,定量分析则不能正确反映物种生物量。对于濒危物种及稀有物种而言,其在自然水域中密度非常小,释放到水环境中的DNA属于微量,为了更有效地对目标种进行物种监测与生物量评估,建议使用商业化试剂盒提取eDNA。对于试剂盒的选用,使用最多的主要有DNeasy Blood and Tissue Kit (Qiagen, 德国)与PowerWater DNA Isolation Kit (MOBIO Laboratories, 美国)两种试剂盒。有学者就各类试剂盒对eDNA的提取效果做过对比分析(Deiner et al, 2015; Eichmiller et al, 2016),普遍认为DNeasy Blood and Tissue Kit效果较好,同一试剂盒对不同目标种可能存在差异。因此,研究者应针对所选取的研究对象做eDNA技术操作流程各个步骤的优化。
3.4 eDNA的定量分析目的基因的选择及荧光定量PCR中荧光标记方法均是影响荧光定量PCR结果准确性的主要因素。Hebert等(2005)研究表明,在eDNA浓度较低的情况下,细胞中的线粒体拷贝数要远大于核DNA,更易于被检测到。基于长度差异与进化速率的考虑,mtDNA中COⅠ基因更适于用来分析亲缘关系较近的分类类群(焦明超等, 2011)。同时,荧光标记方法有扩增序列非特异和序列特异两类检测,其中,TaqMan探针法较染料法具有更强的特异性,能够排除假阳性结果的产生。考虑到渤海虾类组成及周边对虾养殖业可能产生的影响,建议选择COⅠ基因作为扩增的目的基因,同时采用TaqMan探针法进行荧光定量PCR。
4 小结本研究通过实时荧光定量PCR进行检测,确定采用0.45 μm的玻璃纤维滤膜过滤2 L水样结合DNeasy Blood and Tissue Kit试剂盒提取eDNA,能够检测到的DNA拷贝数最多,初步建立了一套针对中国对虾eDNA技术的操作流程。
致谢: 感谢中国水产科学研究院黄海水产研究所水产遗传育种中心陈宝龙老师与课题组项目聘用人员王惠宾在取样工作中给予的莫大帮助!
Bohmann K, Evans A, Gilbert MTP, et al. Environmental DNA for wildlife biology and biodiversity monitoring. Trends in Ecology & Evolution, 2014, 29(6): 358-367 |
Costas BA, McManus G, Doherty M, et al. Use of species- specific primers and PCR to measure the distributions of planktonic ciliates in coastal waters. Limnology & Oceanography Methods, 2007, 5(6): 163-173 |
Deiner K, Altermatt F. Transport distance of invertebrate environmental DNA in a natural river. PLoS One, 2014, 9(2): e88786 DOI:10.1371/journal.pone.0088786 |
Deiner K, Walser JC, Mächler E, et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA. Biological Conservation, 2015, 183(9): 53-63 |
Eichmiller JJ, Miller LM, Sorensen PW. Optimizing techniques to capture and extract environmental DNA for detection and quantification of fish. Molecular Ecology Resources, 2016, 16(1): 56-68 DOI:10.1111/1755-0998.12421 |
Evans NT, Lamberti GA. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research, 2017 |
Ficetola GF, Miaud C, Pompanon F, et al. Species detection using environmental DNA from water samples. Biology Letters, 2008, 4(4): 423-425 DOI:10.1098/rsbl.2008.0118 |
Geerts AN, Boets P, Heede SVD, et al. A search for standardized protocols to detect alien invasive crayfish based on environmental DNA (eDNA): A lab and field evaluation. Ecological Indicators, 2018, 84(84): 564-572 |
Goldberg CS, Pilliod DS, Arkle RS, et al. Molecular detection of vertebrates in stream water: A demonstration using rocky mountain tailed frogs and Idaho giant salamanders. PLoS One, 2011, 6(7): e22746 DOI:10.1371/journal.pone.0022746 |
Haile J, Froese DG, Macphee RDE, et al. Ancient DNA reveals late survival of mammoth and horse in interior Alaska. Proceedings of the National Academy of Sciences of USA, 2009, 106(52): 22352-22357 DOI:10.1073/pnas.0912510106 |
Hebert PD, Gregory TR. The promise of DNA barcoding for taxonomy. Systematic Biology, 2005, 54(5): 852-859 DOI:10.1080/10635150500354886 |
Jerde CL, Mahon AR, Chadderton WL, et al. "Sight-unseen" detection of rare aquatic species using environmental DNA. Conservation Letters, 2011, 4(2): 150-157 DOI:10.1111/conl.2011.4.issue-2 |
Jiao MC, Zhao DX, Ouyang S, et al. Application prospect of DNA barcode technology in taxonomy. Hubei Agricultural Sciences, 2011, 50(5): 886-890 [ 焦明超, 赵大显, 欧阳珊, 等. DNA条形码技术在生物分类学中的应用前景. 湖北农业科学, 2011, 50(5): 886-890 DOI:10.3969/j.issn.0439-8114.2011.05.006] |
Klymus KE, Richter CA, Chapman DC, et al. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation, 2015, 183: 77-84 DOI:10.1016/j.biocon.2014.11.020 |
Minamoto T, Yamanaka H, Takahara T, et al. Surveillance of fish species composition using environmental DNA. Limnology, 2012, 13(2): 193-197 DOI:10.1007/s10201-011-0362-4 |
Minamoto T, Naka T, Moji K, et al. Techniques for the practical collection of environmental DNA: Filter selection, preservation, and extraction. Limnology, 2016, 17(1): 23-32 DOI:10.1007/s10201-015-0457-4 |
Moyer GR, Díaz-Ferguson E, Hill JE, et al. Assessing environmental DNA detection in controlled lentic systems. PLoS One, 2014, 9(7): e103767 DOI:10.1371/journal.pone.0103767 |
Rees HC, Maddison BC, Middleditch DJ, et al. The detection of aquatic animal species using environmental DNA - A review of eDNA as a survey tool in ecology//Dynamic 3D Imaging, DAGM 2009 Workshop, Dyn3D 2009, Jena, Germany, September 9, 2009. Proceedings. DBLP, 2014, 110-125
|
Renshaw MA, Olds BP, Jerde CL, et al. The room temperature preservation of filtered environmental DNA samples and assimilation into a phenol-chloroform-isoamyl alcohol DNA extraction. Molecular Ecology Resources, 2015, 15(1): 168-176 DOI:10.1111/men.2014.15.issue-1 |
Shan XJ, Li M, Wang WJ. Application of environmental DNA technology in aquatic ecosystem. Progress in Fishery Sciences, 2018, 39(3): 23-29 [ 单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29] |
Sigsgaard EE, Nielsen IB, Carl H, et al. Seawater environmental DNA reflects seasonality of a coastal fish community. Marine Biology, 2017, 164(6): 128 DOI:10.1007/s00227-017-3147-4 |
Takahara T, Minamoto T, Yamanaka H, et al. Estimation of fish biomass using environmental DNA. PLoS One, 2012, 7(4): e35868 DOI:10.1371/journal.pone.0035868 |
Thomsen PF, Kielgast J, Iversen LL, et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples. PLoS One, 2012, 7(8): e41732 DOI:10.1371/journal.pone.0041732 |
Thomsen PF, Kielgast J, Iversen LL, et al. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 2012, 21(11): 2565 DOI:10.1111/j.1365-294X.2011.05418.x |
Turner CR, Barnes MA, Xu CCY, et al. Particle size distribution and optimal capture of aqueous macrobial eDNA. Methods in Ecology & Evolution, 2014, 5(7): 676-684 |
Yan H, Dong J, Huang J, et al. Study on the DNA extraction from shrimp specimens by different methods. Applied Chemical Industry, 2012, 41(3): 548-551 [ 闫晗, 董君, 黄静, 等. 不同固定剂处理的对虾基因组DNA提取方法研究. 应用化工, 2012, 41(3): 548-551 DOI:10.3969/j.issn.1671-3206.2012.03.054] |
Yuan W, Lin Q, Wang J, et al. Assessment of enhancement effectiveness of Chinese shrimp Fenneropenaeus chinensis stock in the Laoshan Bay. Progress in Fishery Sciences, 2015, 36(4): 27-34 [ 袁伟, 林群, 王俊, 等. 崂山湾中国对虾(Fenneropenaeus chinensis)增殖放流的效果评价. 渔业科学进展, 2015, 36(4): 27-34] |
Vermeulen S, Koziell I. Integrating local and global biodiversity values: A review of biodiversity assessment. 2002-IIED, 2002 |



