新安江源于黄山南麓,流域上游多为山间溪流,地势复杂,水流湍急。下游东入浙江省西部,经淳安至建德与兰江汇合后为钱塘江。温州光唇鱼(Acrossocheilus wenchowensis)隶属鲤形目(Cypriniformes)、鲤科(Cyprinidae)、光唇鱼属(Acrossocheilus)(Nelson et al, 2016),是新安江流域中上游栖息的淡水型鱼类,在浙江、广东、福建境内也有分布。因其成鱼常栖息于石缝间,体侧具有多条鲜艳的黑色横斑而被地方称为“石斑鱼”。由于温州光唇鱼肉质细嫩、体色鲜艳,具有较高的食用和观赏价值,深受消费者的青睐(冀德伟等, 2009)。近年来,受水质污染、栖息地破坏、过度捕捞等人类活动的影响,新安江流域温州光唇鱼的野生资源急剧减少,其分布范围日益减缩,资源数量逐渐下降,个体规格也越来越小(胡玉婷等, 2017)。因此,开展新安江流域温州光唇鱼群体遗传结构研究和保育工作已迫在眉睫。
由于具有高突变率、低重组率及严格的母系遗传等特征,线粒体基因序列一直是群体遗传学和系统发生学研究中的理想标记(Cann et al, 1987)。马春艳等(2009)采用线粒体细胞色素b基因(Cyt b)对鲳属3种鱼类系统进化关系进行分析,提出银鲳(Pampus argenteus)是3种鲳属鱼类中较早分化出的种,而刺鲳(Psenopsis anomala)则是鲳属鱼类的原始类群的观点。李大命等(2017)利用线粒体Cyt b和细胞色素氧化酶亚基Ⅰ(COⅠ)基因作为分子标记,分析洪泽湖大银鱼(Protosalanx hyalocranius) 40个个体的序列多态性,提出该群体可能经历种群扩张事件,且单倍型未出现遗传分化。张争世等(2017)基于线粒体Cyt b基因探讨齐口裂腹鱼(Schizothorax prenanti)野生群体的遗传多样性和遗传结构,长江上游4个齐口裂腹鱼野生群体未经历过种群扩张,且群体间和群体内存在显著的遗传分化。
本研究采用线粒体Cyt b基因,探讨新安江流域的6个不同温州光唇鱼地理群体的遗传结构,并重建其群体进化历史,以期为该类鱼种的保育和开发提供理论依据。
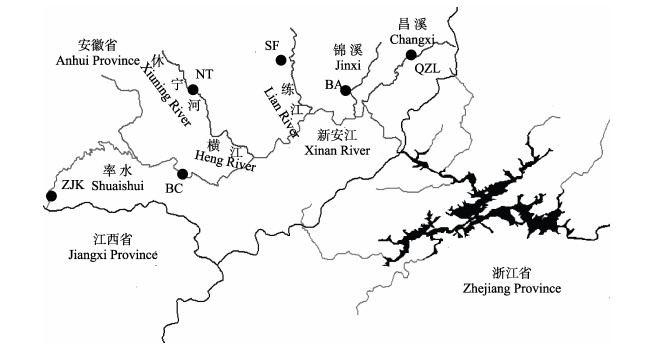
1 材料与方法 1.1 样本采集实验所用的温州光唇鱼样本于2016年10月~ 2017年8月采集自新安江流域。6个自然群体分别采自毕村(BC)、杞梓里(QZL)、南塘(NT)、上丰(SF)、北岸(BA)、朱家坑(ZJK),共170个个体(表 1,图 1)。样本采集后做好标记,保存于95%的酒精中固定。
|
|
表 1 温州光唇鱼样本采集信息 Tab.1 Sampling information of A. wenchowensis |
|
图 1 温州光唇鱼6个群体样本采集点 Fig.1 Sampling sites of 6 populations of A. wenchowensis BC:毕村; QZL:杞梓里; NT:南塘; SF:上丰; BA:北岸; ZJK:朱家坑。下同 BC: Bicun; QZL: Qizili; NT: Nantang; SF: Shangfeng; BA: Bei'an; ZJK: Zhujiakeng. The same as below |
取样本背部肌肉组织,采用Sambrook法提取基因组DNA(Sambrook et al, 1989)。
1.3 PCR扩增及测序Cyt b基因的扩增和测序引物为H15915(5'-CTC CGATCTCCGGATTACAAGAC-3')和L14724(5'-GAC TTGAAAAACCACCGTTG-3')(Xiao et al, 2001)。
PCR反应体系:200 ng的模板DNA,10×Buffer缓冲液(TaKaRa) 5 µl,dNTP(2.5 mmol/L) 3 µl,MgCl2 (2.5 mol/L) 4 µl,上下游引物(5 µmol/L)各2 µl,Taq DNA聚合酶0.75 U,补充无菌超纯水至50 µl。
PCR反应条件:96℃预变性1 min;94℃变性45 s,55℃退火45 s,72℃延伸1 min,共34个循环;72℃延伸10 min。PCR产物经1%的琼脂糖凝胶电泳检测后,委托生工生物工程(上海)有限公司双向测序。
1.4 数据分析所得序列通过Seqman软件(Burland, 2000)辅助人工进行拼接组装,获得完整的mtDNA Cyt b基因。序列集通过MAFFT v.7(Katoh et al, 2013)在线比对,并辅以人工校正。采用DNAsp 5.0(Librado et al, 2009)计算多态位点、单倍型数目、错配分布(Mismatch distribution)、单倍型多样性(Haplotype diversity, Hd)和核苷酸多样性(Nucleotide diversity, Pi)。使用Network 5绘制单倍型网络关系图。通过MrBayes3.1.2 (Ronquist et al, 2003)采用贝叶斯法(Bayesian inference, BI)构建单倍型系统进化树。使用MEGA 6(Tamura et al, 2013)(Kumara2-parameter)计算种群遗传距离,并通过Arlequin 3.11(Excoffier et al, 2005)计算种群遗传分化指数(F-statistics, Fst)并进行分子变异分析(Analysis of molecular variance, AMOVA)。使用Beast1.6软件包(Drummond et al, 2007)进行贝叶斯天际线(Bayesian skyline plot, BSP)分析,检测种群变化的历史动态。基于聚类树的拓扑学结构,使用RASP 3.0软件(Yu et al, 2015)重建温州光唇鱼祖先种群起源地,追溯其原种群。
2 结果与分析 2.1 序列特征和遗传多样性分析测序获得的170条温州光唇鱼mtDNA Cyt b序列已上传至NCBI数据库,登录号为MH141624~ MH141793。经过同源比对后,用于下游分析的基因片段全长1039 bp,共检测到69个突变位点,突变率6.6%,包含简约信息位点(Parsimony-informative sites) 31个,单突变位点(Singleton sites)38个。序列中A、T、G和C的平均含量分别为27.8%、27.3%、14.8%和30.1%,遵循A+T > G+C的规律。编码序列出现了明显的反G偏倚(Anti-G bias)现象,尤其体现在第3位密码子G的含量(7%)上。Saccone等(1999)认为这种偏倚现象与线粒体基因组的进化趋势相关,物种由低等向高等进化过程中,反G偏倚会逐渐弱化,在鸟类和哺乳类动物线粒体中G的含量达到最高。
温州光唇鱼6个群体共170条序列定义了28个单倍型,其中,群体间共享单倍型9个,包含148个个体,占总样本数的87.06%;特有单倍型19个,包含22个个体,占总样本数的12.94%。
各群体的单倍型多样性为0.308~0.876,核苷酸多样性为0.00112~0.00838。其中,BC群体定义13条单倍型,单倍型多样性最高;NT群体检测到38个多态位点,核苷酸多样性最高(表 2)。
|
|
表 2 温州光唇鱼6个群体遗传多样性参数 Tab.2 Population genetic diversity of 6 populations of A. wenchowensis |
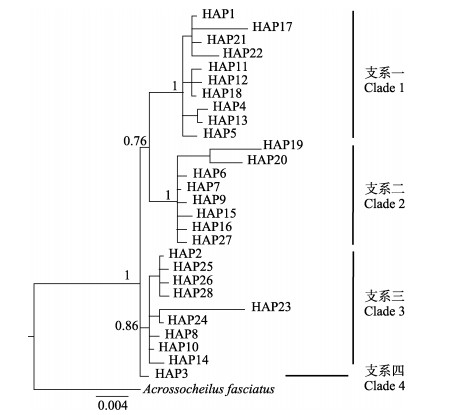
单倍型系统发生分析选择光唇鱼(Acrossocheilus fasciatus)(登录号:KF781289)作为外群,构建进化树。如图 2所示,28个单倍型形成了4个支系,其中HAP1、4、5、11、12、13、17、18、21、22聚为支系一,HAP6、7、9、15、16、19、20、27聚为支系二,HAP2、8、10、14、23、24、25、26、28聚为支系三,HAP3聚为支系四。
|
图 2 基于贝叶斯法构建的28个单倍型系统进化树 Fig.2 Phylogenetic tree for 28 haplotypes constructed based on BI method |
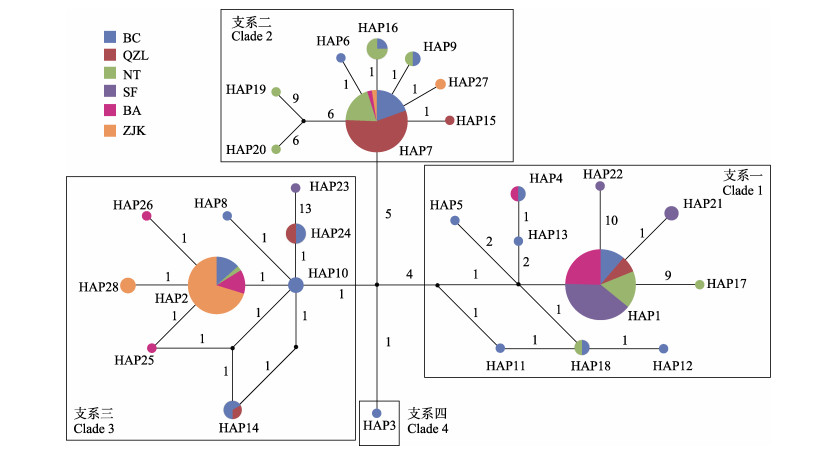
采用中介网络理论(Median joining, MJ)绘制的单倍型网络图中,28个单倍型也形成了4个支系。不同支系内出现了多个主体单倍型(支系一为HAP1,支系二为HAP7,支系三为HAP2),其他单倍型呈辐射状环绕在主体单倍型周围(图 3)。
|
图 3 基于中介网络理论构建的单倍型中介网络图 Fig.3 Haplotypes network based on MJ method 线段上的数字代表单倍型间的突变次数,不同的颜色代表不同的群体 The numbers on the lines represents mutation steps; Different colors represents different populations |
将温州光唇鱼6个不同地理种群设定为同一组群,执行AMOVA分析(表 3),估算种群间和种群内个体间变异率。结果表明,新安江温州光唇鱼整体的变异主要来自于群体内,变异比例为66.85%,而种群间的变异仅占33.15%。
|
|
表 3 温州光唇鱼6个群体分子变异分析 Tab.3 AMOVA of 6 populations of A. wenchowensis |
不同群体间的Fst值见表 4,温州6个地理群体出现了不同程度的分化,其中,SF和ZJK两个群体之间的分化程度最高(Fst=0.73281),而BC和NT群体间未检测到明显分化(Fst=0.02389)。少数群体间存在着不同水平的基因流(Nm > 1),6个群体间的平均基因流Nm=2.04。
|
|
表 4 群体间遗传分化指数(Fst)和群体间有效迁徙个体数(Nm) Tab.4 Pairwise values of Fst (below diagonal) and Nm (above diagonal) between populations |
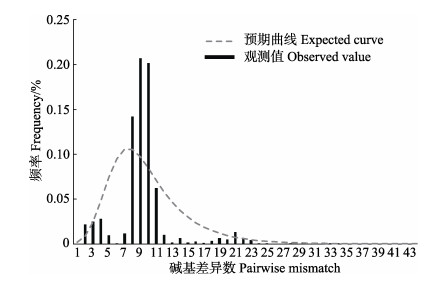
6个群体的中性检验显示Tajima’s D和Fu’s Fs为负值,其中Fu’s Fs结果显著(表 5)。基于期望值持续增长模式下的错配分布分析结果显示,曲线呈现单峰泊松分布,且实际观测结果与期望的假定模型吻合度较高(图 4)。因此,认为新安江温州光唇鱼群体可能经过扩张或者仍处于持续增长状态。
|
|
表 5 温州光唇鱼群体中性检验和错配分布参数 Tab.5 Neutral test and mismatch distribution of nucleotide of A. wenchowensis populations |
|
图 4 温州光唇鱼碱基错配分布 Fig.4 Nucleotide mismatch distribution of A. wenchowensis |
选择GTR+Gamma替代模型,基于宽松分子钟(Relaxed clock)理论构建了贝叶斯天际线图来模拟群体变化动态。BSP结果显示(图 5),新安江温州光唇鱼6个群体发生过扩张事件。由于未见温州光唇鱼mtDNA Cyt b基因分子进化速率的报道,研究基于Gamma替代模型并辅以选择压估算校正,估算了温州光唇鱼mtDNA Cyt b基因位点进化速率u为2.87~3.00/百万年。考虑到光唇鱼属于2龄性成熟鱼类,将表 5中的τ值代入公式τ=2ut,推算出新安江温州光唇鱼群体扩张时间约为335万年前。
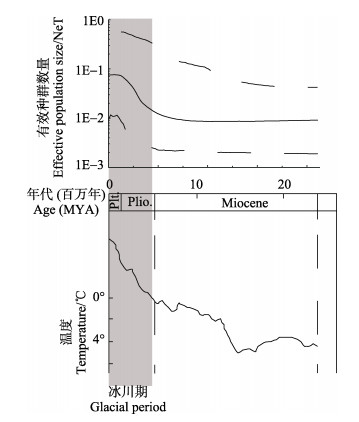
|
图 5 温州光唇鱼群体历史动态 Fig.5 Historical demography of A. wenchowensis 上图为反映温州光唇鱼群体动态的贝叶斯天际线图,下图为Zachos(2001)等研究结果的全球温度变化趋势图。图中阴影部分表示冰川时期 Top plot represents the BSP of 6 populations; bottom plot represents the trend of global temperature change redrawn from Zachos et al's results. The shadow zone represents the glacial period |
为了追溯该流域光唇鱼群体的扩张历史,重建其祖先群体的分布情况,本研究基于贝叶斯二进制法(Bayesian binary method, BBM)和分散–隔绝理论(Statistical dispersal-vicariance method, S-DIVA),重新构建了温州光唇鱼祖先群体分布地(图 6)。图中上半区是新安江的地图,按照采集到的群体分布,将该区域划分成A~F六片区域,分别为A率水上游、B率水下游、C昌溪、D休宁河、E练江、F锦溪。下半区则是基于Fst值构建的邻接树(Neighbor joining)拓扑学结构,进行的祖先起源地重建以及随后的物种区域进化推测结果。结果显示,新安江水系温州光唇鱼可能起源于率水上游和练江上游,随后群体逐渐扩张,形成现在的分布。
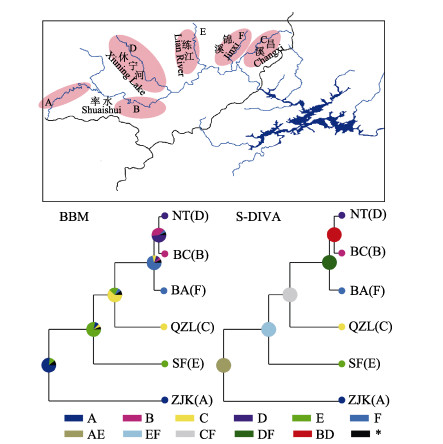
|
图 6 基于BBM和S-DIVA理论重建的祖先起源地 Fig.6 The ancestral area reconstruction inferred from BBM and S-DIVA |
遗传多样性是种群适应外界环境、延续物种生存的遗传基础,与种群的扩张、迁徙、选择和分化息息相关(Crawford et al, 2010; Lacy, 1987)。单倍型多样性和核苷酸多样性是评价种群遗传多样性的重要指标,其数值的大小可直观地反映遗传多样性的高低。
新安江流域温州光唇鱼6个地理群体的单倍型多样性为0.799,核苷酸多样性为0.00631,群体遗传多样性水平较低,这与胡玉婷等(2017)对皖南山区光唇鱼的研究结果相一致。新安江水系较为封闭,与其他水系的光唇鱼物种群体交流较少,加之近年来光唇鱼价格越来越高,当地村民捕鱼热情高涨,温州光唇鱼野生群体数量较少,多种原因造成了该水系温州光唇鱼群体遗传多样性较低的现象。建议该水域的温州光唇鱼资源应以保护为主,科学划定休渔期和禁捕区,对资源量丰富、遗传多样性相对高的群体(BC、NT)可适度开发。
3.2 温州光唇鱼群体遗传分化新安江流域河流呈现出支流多、蜿蜒盘曲、河道窄、河水浅、河床地质为岩石、水质清澈、河道比降大等特点(王文剑, 2014)。采样过程中,发现光唇鱼分布丰度由一级支流至二级、三级支流呈现显著的增长趋势,这与Zhu等(2017)的研究结果相印证。复杂的水系加之季节性的干旱断流和人为建坝的影响,导致了温州光唇鱼出现了栖息地斑块化(Habitat Patch)现象(Erős et al, 2005),不同地理群体间很少有基因交流的机会。因此,在长期遗传漂变的作用下,各个群体分别出现了不同程度的遗传分化。BC群体采集于多条支流交汇后的水域,个体可能来自上游多个群体,因此,未检测到地理分化,群体个体分布于4个进化支系中。
然而,AMOVA分析结果显示,6个地理群体的主要遗传变异仍来自于群体内部,群体间的遗传变异占33.15%。结合估算的群体扩张时间为335万年前,认为新安江温州光唇鱼群体扩张–隔绝–分化事件所处地质年代较新(上新世晚期),群体间隔绝时间较短,较之遗传漂变,基因流在形成群体遗传结构中占主导作用(平均基因流Nm=2.04)。因此,虽然6个群体检测到一定的地理分化,但大多数的分子遗传变异仍来自于群体内个体间。
3.3 新安江温州光唇鱼群体扩张历史研究表明,多数物种,如鱼类、爬行类,在冰川时期都经历过种群收缩(Zhou et al, 2015; 周伟等, 2017)。由于地表温度过低,物种仅能存活在冰期避难所中,以免灭绝。而处于避难所中的物种,由于温度适宜、资源丰富,往往比冰期后扩张的物种有着更长的群体进化历史。对新安江温州光唇鱼群体历史动态分析暗示,温州光唇鱼于335万年前(上新世晚期)发生过群体扩张事件,此时全球处于末次冰川时期的早期(Zachos et al, 2001)。地表温度骤降并未影响到新安江温州光唇鱼群体的扩张。因此,推测新安江流域可能是潜在的冰期避难所。
原种保护一直是保育生物学的重点工作,检测到不同地理群体间的遗传分化,引起了作者对该流域温州光唇鱼原种群鉴定的兴趣。结合温州光唇鱼现有的分布和祖籍地重建分析结果,推测新安江流域温州光唇鱼起源于ZJK和SF两个种群,即原种群,经过一系列的种群扩散–隔绝–分化事件,形成现今的分布。研究通过广泛采样,采用严格母系遗传的mtDNA Cyt b基因序列作为分子标记,追溯该物种的起源,鉴定新安江流域温州光唇鱼的原种群,为该物种种群保护的优先级提供理论依据。
Burland TG. DNASTAR's Lasergene sequence analysis software. Bioinformatics methods and protocols. Humana Press, 2000
|
Cann RL, Stoneking M, Wilson AC. Mitochondrial DNA and human evolution. Nature, 1987, 325(3): 31-36 |
Crawford K, Whitney K. Population genetic diversity influences colonization success. Molecular Ecology, 2010, 19(6): 1253-1263 DOI:10.1111/mec.2010.19.issue-6 |
Drummond AJ, Rambaut A. BEAST: Bayesian evolutionary analysis by sampling trees. BMC Evolutionary Biology, 2007, 7(1): 214 DOI:10.1186/1471-2148-7-214 |
Erős T, Grossman G. Effects of within-patch habitat structure and variation on fish assemblage characteristics in the Bernecei stream, Hungary. Ecology of Freshwater Fish, 2005, 14(3): 256-266 DOI:10.1111/eff.2005.14.issue-3 |
Excoffier L, Laval G, Schneider S. Arlequin (Version 3. 0): An integrated software package for population genetics data analysis. Evolutionary Bioinformatics, 2005, 1(4A): 47-50 |
Hu YT, Jiang H, Duan GQ, et al. Population genetic structure of Acrossocheilus wenchowensis in mountainous area of Southern Anhui Province based on mitochondrial Cyt b gene. Chinese Agricultural Science Bulletin, 2017, 33(32): 121-126 [ 胡玉婷, 江河, 段国庆, 等. 基于线粒体Cyt b基因的皖南山区温州光唇鱼种群遗传结构. 中国农学通报, 2017, 33(32): 121-126] |
Ji DW, Li MY, Shi YH, et al. The nutritional composition and evaluation in muscle of Acrossocheilus fasciatus. Acta Nutrimenta Sinica, 2009, 31(3): 298-300 [ 冀德伟, 李明云, 史雨红, 等. 光唇鱼的肌肉营养组成与评价. 营养学报, 2009, 31(3): 298-300] |
Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability. Molecular Biology and Evolution, 2013, 30(4): 772-780 DOI:10.1093/molbev/mst010 |
Lacy RC. Loss of genetic diversity from managed populations: interacting effects of drift, mutation, immigration, selection, and population subdivision. Conservation Biology, 1987, 1(2): 143-158 DOI:10.1111/cbi.1987.1.issue-2 |
Li DM, Li K, Zhang TQ, et al. Genetic polymorphism analysis of Cytb and COⅠgene sequences of Protosalanx hyalocranius in the Hongze Lake. Progress in Fishery Sciences, 2017, 38(6): 25-31 [ 李大命, 李康, 张彤晴, 等. 洪泽湖大银鱼(Protosalanx hyalocranius) Cytb和COⅠ基因序列多态性分析. 渔业科学进展, 2017, 38(6): 25-31] |
Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 |
Ma CY, Zhao F, Meng YY, et al. Phylogenetic relationships of Pampus inferred from mitochondrial cytochrome b sequences. Progress in Fishery Sciences, 2009, 30(5): 20-26 [ 马春艳, 赵峰, 孟彦羽, 等. 基于线粒体细胞色素b基因片段序列变异探讨3种鲳属鱼类系统进化. 渔业科学进展, 2009, 30(5): 20-26 DOI:10.3969/j.issn.1000-7075.2009.05.004] |
Nelson JS, Grande TC, Wilson MV. Fishes of the world. New York: John Wiley & Sons Press, 2016
|
Ronquist F, Huelsenbeck JP. MrBayes 3: Bayesian phylogenetic inference under mixed models. Bioinformatics, 2003, 19(12): 1572-1574 DOI:10.1093/bioinformatics/btg180 |
Saccone C, De Giorgi C, Gissi C, et al. Evolutionary genomics in Metazoa: The mitochondrial DNA as a model system. Gene, 1999, 238(1): 195-209 DOI:10.1016/S0378-1119(99)00270-X |
Sambrook J, Fritsch EF, Maniatis T. Molecular cloning: A laboratory manual. New York: Cold Spring Harbor Laboratory Press, 1989
|
Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729 DOI:10.1093/molbev/mst197 |
Wang WJ. Spatial pattern in the structure and diversity of the taxonomic and functional organizations of stream fish in the mountains of southern Anhui. Master's Thesis of Anhui Normal University, 2014 [王文剑.皖南山区溪流鱼类分类群与功能群结构及其多样性的空间格局.安徽师范大学硕士研究生学位论文, 2014] http://cdmd.cnki.com.cn/article/cdmd-10370-1015512431.htm
|
Xiao W, Zhang Y, Liu H. Molecular systematics of Xenocyprinae (Teleostei: Cyprinidae): Taxonomy, biogeography, and coevolution of a special group restricted in East Asia. Molecular Phylogenetics and Evolution, 2001, 18(2): 163-173 |
Yu Y, Harris AJ, Blair C, et al. RASP (reconstruct ancestral state in phylogenies): A tool for historical biogeography. Molecular Phylogenetics and Evolution, 2015, 87: 46-49 DOI:10.1016/j.ympev.2015.03.008 |
Zachos J, Pagani M, Sloan L, et al. Trends, rhythms, and aberrations in global climate 65 Ma to present. Science, 2001, 292(5517): 686-693 DOI:10.1126/science.1059412 |
Zhang ZS, Hu BJ, Ye XY, et al. Genetic diversity of the prenant's schizothoracin (Schizothorax prenanti) based on partial mtDNA Cyt b sequences. Acta Hydrobiologica Sinica, 2017, 41(3): 609-616 [ 张争世, 胡冰洁, 叶祥益, 等. 基于mtDNA Cyt b序列分析齐口裂腹鱼群体遗传多样性. 水生生物学报, 2017, 41(3): 609-616] |
Zhou HX, Jiang Y, Nie LW, et al. The historical speciation of Mauremys Sensu Lato: Ancestral area reconstruction and interspecific gene flow level assessment provide new insights. PLoS One, 2015, 10(12): e0144711 DOI:10.1371/journal.pone.0144711 |
Zhou W, Gao TX, Wang J, et al. Genetic diversity and structure analysis based on the mitochondrial DNA control region of the northern snakehead (Channa argus). Journal of Fisheries of China, 2017, 41(10): 1521-1532 [ 周伟, 高天翔, 王俊, 等. 乌鳢群体遗传多样性和遗传结构分析. 水产学报, 2017, 41(10): 1521-1532] |
Zhu R, Li Q, Wang W, et al. Effects of local, river-network and catchment factors on fish assemblages in the headwater streams of the Xin'an Basin, China. Journal of Freshwater Ecology, 2017, 32(1): 309-322 DOI:10.1080/02705060.2016.1278408 |



