2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071
刺参又名仿刺参(Apostichopus japonicus),广泛分布于太平洋西部的中国、俄罗斯、韩国与日本沿海,含有丰富的蛋白质,粘性多糖及多种微量元素,具有很高的药用价值和营养保健价值,是我国海水养殖中重要的经济品种(廖玉麟, 1997)。自20世纪80年代刺参人工繁育技术突破以后,刺参养殖产业快速发展,截止2017年,全国刺参养殖面积达21.9万hm2,养殖产量21.9万t,直接产值逾300亿元(中国渔业统计年鉴, 2018)。然而,随着产业的迅猛发展,刺参养殖产业出现生长速度慢、病害频发、养殖成活率低等种质退化现象。而在韩国和俄罗斯等刺参自然分布海域,由于过度捕捞和环境污染的加剧,野生刺参自然资源趋于枯竭,根据世界自然保护联盟的数据,近30~50年以来,野生刺参的生物资源量,日本下降了30%,韩国下降了40%,俄罗斯下降了80%,中国下降了95%以上。刺参已被世界自然保护联盟收录到濒危物种红色名录的濒危Endangered(EN)等级(Purcell et al, 2013)。因此,开展刺参种质资源评估和遗传结构分析,不仅可以为刺参良种选育提供科学数据,还可为客观评估刺参的种质资源现状提供技术支撑。
目前,用于种质资源评估的分子标记主要包括微卫星DNA和线粒体DNA等。其中,线粒体DNA(mtDNA)是细胞核外具有自主复制、转录和翻译能力的遗传因子,与核DNA相比,具有分子小、结构简单、演化速度快和母系遗传等优点,是用于进行种群遗传结构分析的理想标记。目前,在水产动物中,用于遗传结构解析的mtDNA片段主要包括16S rDNA、D-loop和COⅠ基因。本研究采集中国、韩国和俄罗斯沿海6个海域8个不同地理群体的刺参,分别采用16S rDNA、D-loop和COⅠ这3个片段对所采集的地理群体种质资源和遗传结构进行评估,以期为科学评估目前刺参种质资源现状提供数据支撑,并为种质资源保护及开发利用提供理论依据。
1 材料与方法 1.1 样本来源实验用野生刺参分别采集于中国青岛、烟台、俄罗斯符拉迪沃斯托克、韩国浦项、韩国木浦和韩国群山共6个海域,根据所采集刺参体色的差异,韩国浦项采集的刺参又分为红参、黄参和黑参3个群体,每个群体随机选取10头刺参作为实验刺参,合计采集6个海域8个不同地理群体刺参80头。采集的刺参采用冷冻方式运回实验室,剖取纵肌用于DNA的提取。不同群体的采样地点及信息见图 1和表 1。
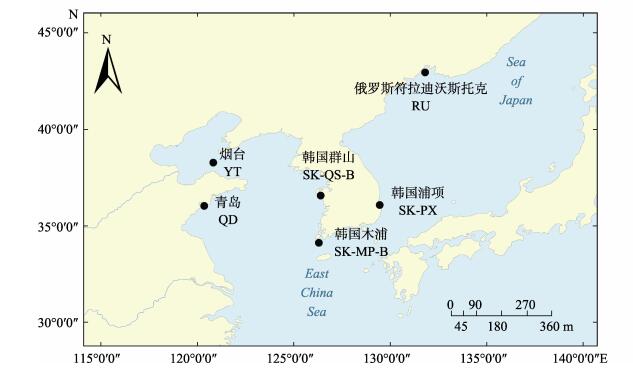
|
图 1 刺参采样地点 Fig.1 Locations of sea cucumber A. japonicus sampling |
|
|
表 1 不同地理种群刺参样本采样信息 Tab.1 Information of A. japonicus sampling |
利用Omega Mollusc DNA kit提取海参基因组DNA,利用NanoDrop 1000及1%琼脂糖凝胶电泳检测DNA纯度及完整性后,置于–20℃冰箱保存。利用刺参16S rDNA、COⅠ和D-loop序列特异引物对3个mtDNA片段进行PCR扩增,对PCR产物测序,3对引物序列见表 2(Kerr et al, 2005; Arndt et al, 1996;姬南京等, 2014),引物由青岛擎科生物公司合成。PCR总体积为50 μl:2×Tsingke Master mix 25 μl,10 μmol/L正反向引物各1 μl,模板DNA 1 μl (50 ng/μl),加ddH2O补足至50 μl。PCR反应扩增程序:95℃预变性60 s、95℃ 30 s、52℃ 30 s,72℃ 60 s,经35个循环后,72℃延伸10 min,4℃保存。PCR产物经1.5%的琼脂糖凝胶电泳检测后,送青岛擎科生物公司进行双向测序。
|
|
表 2 刺参线粒体DNA扩增用引物序列 Tab.2 Primer sequences of mtDNA of A. japonicus |
测序所得序列采用ClustalX1.83软件编辑、校对和排序(Thompson et al, 1997)。采用DnaSP 5.0软件计算多态位点数目(Number of polymorphicsites)、单倍型数目(Number of haplotypes)、单倍型多样性(Haplotype diversity)、核苷酸多样性(Nucleotide diversity)、平均核苷酸差异数(Average number of nucleotide differences)、两两群体间的遗传分化系数(Fst)(Librado et al, 2009)。利用MEGA 6.0软件统计碱基组成、变异位点数,计算群体内及群体间遗传距离,采用Kimura 2- Parameter方法构建N-J(Neighbor-joining)系统发育树(Saitou et al, 1987)。利用Network 5.0软件构建基于Median-Joining的单倍型网络图。
2 结果与分析 2.1 刺参16S rDNA、COⅠ和D-loop序列特征分析经PCR扩增、测序,获得刺参线粒体16S rDNA、COⅠ和D-loop序列,利用ClustalX1.83软件进行序列比对、校正去除序列两端非保守碱基后获得80个个体的相应序列信息。16S rDNA序列长度为543 bp,碱基组成比例相对稳定(表 3),A的平均含量为27.5%,T的平均含量为29.1%,G的平均含量为21.7%,C的平均含量为21.7%,G与C的平均含量及各群体含量均相等,A+T的含量为56.6%,略高于G+C含量。COⅠ的序列长度为656 bp,A、T、G、C的平均含量分别为32.4%、26.5%、21.7%、19.4%,A+T的含量为58.9%,高于G+C含量。D-loop的序列长度为509~527 bp,A、T、G、C的平均含量分别为34.5%、25.3%、27.0%、13.3%,A+T的含量为59.7%,显著高于G+C含量。A+T含量在不同基因片段中差异显著,3个片段中D-loop序列最高,按照不同地理群体进行相应数据统计发现,在同一序列不同群体中差异不显著。
|
|
表 3 刺参不同地理群体16S rDNA、COⅠ和D-loop序列的碱基组成(%) Tab.3 Base compositions of 16S rDNA, COⅠ and D-loop sequences in different populations of A. japonicus(%) |
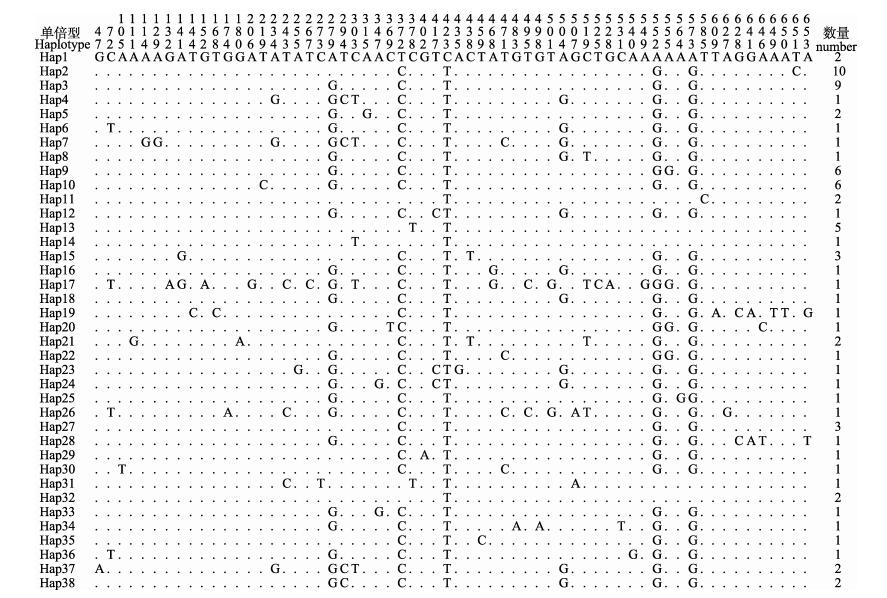
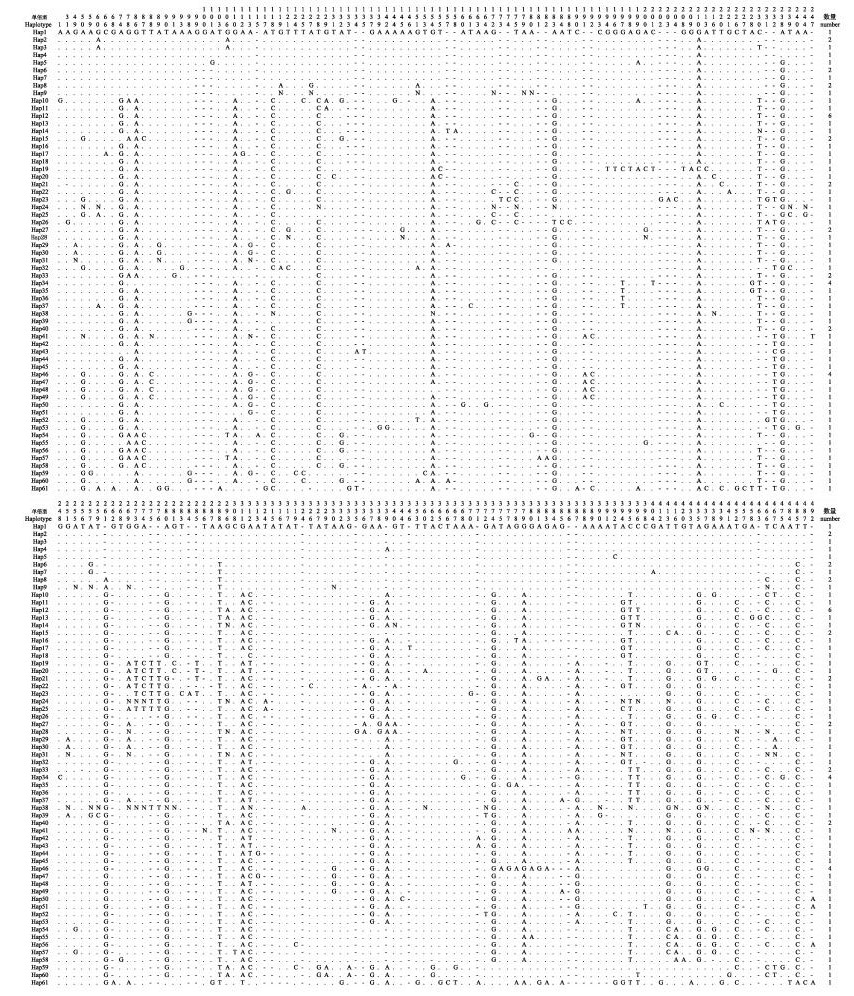
16S rDNA的543个位点中,变异位点有16个(图 2),占总位点数的2.9%,其中没有发现插入/缺失现象,转换多于颠换。在COⅠ基因的656个位点中,变异位点62个(图 3),占总位点数的9.5%,其中,没有发现插入/缺失位点,转换多于颠换。D-loop的总长度为551 bp (包含缺失位点),变异位点200个(图 4),占总位点数的36.3%,其中,68个位点存在碱基的插入/缺失位点。

|
图 2 刺参16S rDNA序列变异位点 Fig.2 Variable site of 16S rDNA in A. japonicus 上方数字表示变异位点在单倍型中对应的位置,左侧表示单倍型名称,右侧表示个体数目 Numbers on the top represent the locations of mutative nucleotide acids, on the left are the names of haplotypes, on the right side represent the number of individuals |
|
图 3 刺参COⅠ基因序列变异位点 Fig.3 Variable site of COⅠ gene in A. japonicus 上方数字表示变异位点在单倍型中对应的位置,左侧表示单倍型名称,右侧表示个体数目,N代表序列简并 Numbers on the top represent the locations of mutative nucleotide acids, on the left are the names of haplotypes, on the right side represent the number of individuals, N represents sequence degeneracy |
|
图 4 刺参D-loop序列变异位点 Fig.4 Variable site of D-loop sequence in A. japonicus 上方数字表示变异位点在单倍型中对应的位置,左侧表示单倍型名称,右侧表示个体数目,N代表序列简并 Numbers on the top represent the locations of mutative nucleotide acids, on the left are the names of haplotypes, on the right side represent the number of individuals, N represents sequence degeneracy |
8个群体间的16S rDNA、COⅠ、D-loop的核苷酸遗传多样性参数见表 4。从8个群体80个个体的16S rDNA序列中共检测到16个多态位点、16种单倍型,单倍型多样性指数为0.629,核苷酸多样性指数为0.0016,平均核苷酸差异数0.880,整体遗传多样性较低。8个群体中80个个体的COⅠ序列共检测到62个多态位点、38种单倍型,单倍型多样性指数为0.958,核苷酸多样性指数为0.0073,平均核苷酸差异数为4.796,说明8个群体的COⅠ序列单倍型及多态位点比较丰富,核苷酸差异数较大,其中,韩国浦项地区3个不同体色的刺参单倍型多样性比其他群体更高。从8个群体中80个个体的D-loop序列共检测到200个多态位点、61种单倍型,单倍型多样性指数为0.933,说明8个群体的D-loop序列的多态位点数及单倍型数最为丰富,单倍型多样性最高,核苷酸多样性指数为0.0157,平均核苷酸差异数为6.834,核苷酸差异数最大。对比3个片段的遗传多样性结果可以看出,COⅠ基因和D-loop的单倍型多样性、多态位点数及核苷酸差异数都显著高于16S rDNA。鉴于NCBI数据库中收录的海参纲COⅠ基因数据量高,而D-loop序列较少,为了保证后期数据分析的准确性和可比性,本研究选取COⅠ基因用于后续不同地理群体刺参遗传结构分析。
|
|
表 4 刺参不同群体16S rDNA、COⅠ和D-loop片段的遗传多样性指数 Tab.4 The genetic diversity indices of partial 16S rDNA, COⅠ and D-loop sequence in different population of A. japonicus |
根据所测定的8个群体的COⅠ序列信息,采用MEGA 6.0软件计算8个群体内和群体间的遗传距离的结果见表 5。从表 5可以看出,群体内遗传距离为0.0023~0.0084,群体内遗传距离最大的是韩国浦项红参群体(0.0084),其次是韩国浦项黑参群体(0.0075),俄罗斯符拉迪沃斯托克群体的群体内遗传距离最小(0.0023)。8个群体的群体间遗传距离为0.0027~ 0.0081,韩国浦项红参群体与韩国浦项黑参群体间的遗传距离最大(0.0081),青岛海参群体、烟台群体与韩国浦项海域的3个海参群体遗传距离较大(0.0054~ 0.0071),俄罗斯符拉迪沃斯托克群体与韩国群山群体的遗传距离最小(0.0027)。
|
|
表 5 基于COⅠ序列的8个刺参群体内(对角线)和群体间的遗传距离(对角线下方)及遗传分化系数(对角线上方) Tab.5 Genetic distances within (diagonal) and between eight different populations (below diagonal), the fixation index (above diagonal) of A. japonicus |
通过DnaSP软件基于COⅠ序列信息分析刺参群体间的遗传分化的结果见表 5。遗传分化系数(Fst)反映了群体间的遗传分化程度,当Fst值在0~0.05时,群体间遗传差异很小,不存在分化;在0.05~0.15时,存在中等程度的遗传分化;0.15~0.25时,遗传分化程度较大;大于0.25时,则遗传分化很大(Wright, 1931)。俄罗斯群体与烟台群体间的遗传分化系数最大(0.1598),遗传分化程度较高,烟台群体与韩国浦项、群山、木浦群体间均存在中等的遗传分化,青岛群体与韩国浦项黄参群体、韩国浦项黑参群体间也存在着中等遗传分化,而其他群体间差异较小,不存在分化。韩国浦项地区3个不同颜色的群体虽然遗传多样性较大,但遗传分化系数却较小,没有形成显著的遗传分化。
2.4 基于COⅠ序列的系统进化基于COⅠ基因构建的进化树(图 5)显示青岛海参群体与烟台海参群体聚为一支,再与韩国群山黑参群体聚为一支,与韩国木浦黑参群体聚在一起,再与俄罗斯群体聚为一支,向外依次为韩国浦项的3个不同体色的群体,这与其地理分布具有一定的相关性。韩国浦项由于其群体内遗传分化较大,在进化树中处于最外侧,可能与受其洋流影响有关。
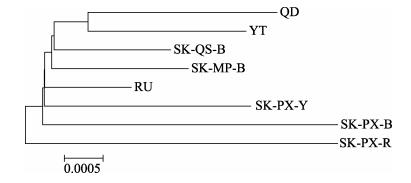
|
图 5 8个群体刺参COⅠ基因的NJ系统发育树 Fig.5 Neighbor-joining tree of eight A. japonicus groups based on COⅠ gene |
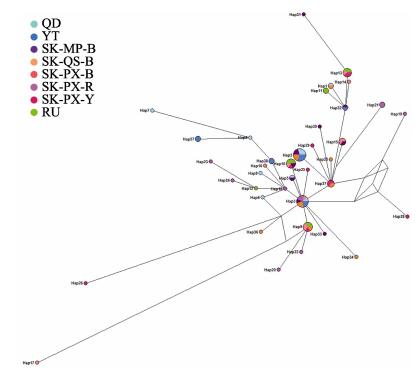
利用Network软件对所获得的COⅠ的38个单倍型构建单倍型网络图发现(图 6),37个单倍型构成以Hap3为中心呈放射状分布的网络状,此外,Hap4、Hap6、Hap9、Hap12、Hap13、Hap18、Hap27、Hap32和Hap38成为外部辐射状网络节点,而其余单倍型则成为辐射图外部端点独立存在。Hap3存在于除俄罗斯群体和韩国浦项黄参群体外的群体中,为6个群体所共有,在37个单倍型中有10个单倍型为共享单倍型,占总单倍型的26.3%,28个单倍型为某个群体所特有。从网络图中难以分析出不同单倍型与地理位置的关系,因为其聚类无显著地理差异性,不同单倍型在不同群体间交叉分布,不同群体的单倍型具有共享性又具有特异性。
|
图 6 刺参COⅠ基因单倍型网络结构 Fig.6 The haplotype network of A. japonicus based on COⅠ gene |
遗传多样性是指地球上所有生物所携带的遗传信息的总和,是评价群体资源状况的一个重要依据。mtDNA作为核外转录因子,具有演化速度快、母系遗传等优点,常被用于种群遗传结构分析(Liu et al, 2007)。其中,常用到的mtDNA标记包括16S rDNA、Cytb、COⅠ、D-loop。这些标记曾应用于不同种类鱼(郝君等, 2013;李大命等, 2017;孙超等, 2018)、甲鱼(Onychostoma rara)(彭珊等, 2009)、海胆属(Temnopleurus)(曾晓起等, 2012;刘晓慧等, 2007)、水母(Aurelia sp.)(张珰妮等, 2015;王建艳等, 2013)等的遗传结构分析。刺参的线粒体全长包含37个基因和3个非编码区(李云峰等, 2012)。前期多位研究者分别用16S rDNA、Cytb、COⅠ、D-loop等mtDNA片段对不同地理群体的遗传多样性进行了分析。如姬南京等(2014)利用D-loop序列的多态性对不同地理群体仿刺参的遗传结构进行了分析;李颖等(2006)利用16S rDNA、COⅠ、IrRNA-COⅠ基因对中国烟台、威海、莱州刺参进行遗传多样性分析及遗传结构分析。为评价不同片段分析刺参遗传多样性的效果,本研究应用16S rDNA、COⅠ、D-loop 3个mtDNA片段对6个地理种群8个群体进行了分析。3个片段对群体的遗传多样性检测结果表明,基于3个序列的各群体的核苷酸多样性指数为0.0007~0.0441,16S rDNA序列最为保守,表现出较低的遗传多样性,而COⅠ和D-loop遗传多样性较高。这与陈丽梅等(2008)研究的海参16S rDNA遗传多样性低于COⅠ基因的遗传多样性一致。相关结果均表明,16S rDNA不适于用于同一物种的种内不同群体的遗传结构分析,COⅠ和D-loop更适于进行种内群体间遗传多样性及遗传结构分析。在海参纲中,相比于D-loop序列,COⅠ基因信息收录于NCBI数据库中的种类及数目较多,更适合于进行比较分析,因而,本研究后续分析选用COⅠ基因。
本研究检测16S rDNA、D-loop和COⅠ序列的核苷酸多样性指数分别为0.0016、0.0157和0.0073,多样性指数较低。单倍型分别是16种、61种和38种,单倍型多样性分别为0.629、0.922和0.958,单倍型多样性较高,这种核苷酸多样性指数较低,单倍型多样性较高的现象,在其他物种海参线粒体基因片段中也广泛存在(Soliman et al, 2016; Uthicke et al, 2010)。对所获得的8个群体的遗传多样性分析结果表明,韩国浦项3个不同体色的野生刺参群体遗传多样性指数最高,相比之下,中国青岛和烟台野生群体刺参的遗传多样性较低,这与王鹤等(2016)的研究结果相一致,即韩国刺参群体的遗传多样性比中国群体的遗传多样性丰富。这种状况出现的原因可能是多方面的,首先地理位置差异导致其聚居区域洋流差异及海水温度差异较大,另一方面可能由于在中国青岛和烟台沿海刺参养殖较多,在排水过程中存在苗种逃逸现象,养殖刺参与野生刺参存在一定程度上的基因交流,近亲繁殖现象严重,从而导致青岛和烟台野生刺参出现遗传多样性较低的现象。
利用COⅠ基因对所采集的8个群体进行遗传距离分析并构建系统进化树,基于COⅠ基因序列的遗传距离为0.0023~0.0084。系统进化分析显示,不同群体的遗传结构与地理位置具有一定的相关性,即青岛、烟台、韩国木浦群体和韩国群山的刺参群体采样点为环渤海、黄海、东海相连通的海域地区,其中,青岛、烟台距离最近,在进化树中,这2个地理群体的刺参聚为一支,然后再与韩国群山、木浦群体聚为一支。韩国浦项群体位于韩国东海,俄罗斯群体采样点位于太平洋沿岸穆拉维约夫–阿穆尔斯基半岛的南端,临近日本海,地理距离与其他群体相对较远,这两地群体位于进化树外侧。本研究结果与潘传燕等(2012)利用微卫星标记进行的相关分析一致,即种群间相似程度与其地理分布具有一定的相关性。
本研究对采自于韩国浦项的3种不同颜色的刺参群体遗传多样性结果分析表明,该海域的刺参具有较高的遗传多样性,这可能与其所处的位置受日本暖流及千岛寒流影响,海水在次表层和中层2个层面中不断流动,运输着来自不同海域的营养物质,使海域中的浮游生物丰富度增加(王燕, 2015),致使此地刺参群体变异比较大,具体的作用机制有待于进一步研究。战爱斌(2007)研究认为,洋流会促使幼虫甚至成体发生交换,会促使不同地域的海洋生物的生殖细胞、幼虫甚至成体发生交换,从而影响不同地理群体的遗传分化(Pante et al, 1992)。韩国浦项3个体色群体的遗传分化较小,还未达到分化成不同种的水平。Zhang等(2016)利用线粒体全基因组分析得出不同颜色的刺参属于同一物种,体色差异是由于海洋气候及洋流变化造成的。Sun等(2010)通过对红色、绿色、黑色刺参的线粒体基因组比较发现,不同体色刺参分化亦未达到不同物种的分化水平,这与本研究的结果是一致的。
本研究基于刺参3种线粒体DNA序列对中、韩、俄沿海野生刺参进行遗传多样性和遗传结构分析结果表明,COⅠ基因更适于进行刺参不同地理群体遗传结构分析;不同地理群体的进化关系跟其地理位置和所处海域的洋流关系密切;处于同一海域不同体色的刺参虽然其体色差异显著,但遗传分化不显著。
Arndt A, Marquez C, Lambert P, et al. Molecular phylogeny of eastern Pacific sea cucumbers (Echinodermata: Holothuroidea) based on mitochondrial DNA sequence. Molecular Phylogenetics and Evolution, 1996, 6(3): 425 |
Chen LM, Li Q, Li Y. Sequence analysis of mitochondrial 16S rRNA and COⅠ gene and molecular phylogeny of four species of sea cucumber. Journal of Fishery Sciences of China, 2008, 15(6): 935-942 [陈丽梅, 李琪, 李赟. 4种海参16S rRNA和COⅠ基因片段序列比较及系统学研究. 中国水产科学, 2008, 15(6): 935-942 DOI:10.3321/j.issn:1005-8737.2008.06.006] |
Fishery Administration Bureau of Ministry of Agriculture. China fishery statistic yearbook. Beijing: China Agriculture Press, 2018 [农业部渔业渔政管理局. 中国渔业统计年鉴. 北京: 中国农业出版社, 2018]
|
Hao J, Yang Q, Bao D, et al. The sequence comparison of mtDNA D-loop and adjacen regions in six fish species. Jounrnal of Dalian Ocean University, 2013, 28(2): 160-165 [郝君, 杨蔷, 鲍迪, 等. 6种鱼mtDNA D-loop及其邻近区段的序列比较分析. 大连海洋大学学报, 2013, 28(2): 160-165 DOI:10.3969/j.issn.2095-1388.2013.02.009] |
Ji NJ, Chang YQ, Zhao C, et al. D-loop sequences variation and phylogenetic analysis in three geographical populations of the sea cucumber Apostichopus japonicas. Journal of Fisheries of China, 2014, 38(4): 476-481 [姬南京, 常亚青, 赵冲, 等. 3个地理群体仿刺参D-loop序列的变异及系统发生分析. 水产学报, 2014, 38(4): 476-481] |
Kerr AM, Janies DA, Clouse RM, et al. Molecular phylogeny of coral-reef sea cucumbers (Holothuriidae: Aspidochirotida) based on 16S mitochondrial ribosomal DNA sequence. Marine Biotechnology, 2005, 7(1): 53-60 DOI:10.1007/s10126-004-0019-y |
Li DM, Li K, Zhang TQ, et al. Genetic polymorphism analysis of Cytb and COⅠgene sequences of Protosalanx hyalocranius in the Hongze Lake. Progress in Fishery Sciences, 2017, 38(6): 25-31 [李大命, 李康, 张彤晴, 等. 洪泽湖大银鱼(Protosalanx hyalocranius) Cytb和COⅠ基因序列多态性分析. 渔业科学进展, 2017, 38(6): 25-31] |
Li Y, Liu P, Sun HL, et al. Sequence analysis of three mitochondrial genes of sea cucumber (Apostichopus japonicas). Oceanologia et Limnologia Sinica, 2006, 37(2): 143-152 [李颖, 刘萍, 孙慧玲, 等. 仿刺参(Apostichopus japonicus) mtDNA三个基因片段的序列分析. 海洋与湖沼, 2006, 37(2): 143-152 DOI:10.3321/j.issn:0029-814X.2006.02.008] |
Li YF, Li MY, Wang J, et al. Comparative structure of complete mitochondrial genome in sea cucumer Apostichopus japonicas. Fisheries Science, 2012, 31(8): 454-461 [李云峰, 李梦瑶, 王建, 等. 仿刺参线粒体全基因组序列结构及比较分析. 水产科学, 2012, 31(8): 454-461 DOI:10.3969/j.issn.1003-1111.2012.08.002] |
Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 |
Liao YL. Fauna Sinica Echinodermata Holothuroidea. Beijing: Science Press, 1997: 148-150 [廖玉麟. 中国动物志棘皮动物门海参纲. 北京: 科学出版社, 1997: 148-150]
|
Liu XH, Huang JQ, Zhou ZC, et al. Sequence analysis of mtDNA 16S rRNA gene fragments among five species of sea urchins. Fisheries Science, 2007, 26(6): 331-334 [刘晓慧, 黄佳琪, 周遵春, 等. 5种经济海胆线粒体16S rRNA基因片段的序列分析. 水产科学, 2007, 26(6): 331-334 DOI:10.3969/j.issn.1003-1111.2007.06.006] |
Liu ZJ, Cordes JF. DNA marker technologies and their applications in aquaculture genetics. Biotechnology Bulletin, 2007, 238(1): 1-37 |
Pan CY, Zang YP, Liao MJ, et al. Development of microsatellite DNA markers of sea cucumber Apostichpus japonicas and their application in population genetic variation and structure analysis. Progress in Fishery Sciences, 2012, 33(4): 72-82 [潘传燕, 臧云鹏, 廖梅杰, 等. 仿刺参微卫星标记的筛选及群体遗传结构分析. 渔业科学进展, 2012, 33(4): 72-82 DOI:10.3969/j.issn.1000-7075.2012.04.010] |
Pante, M, Williams S, Ablan C, et al. Genetic-structure of giant clam (Tridacna derasa) populations from reefs in the Indo-Pacific. Marine Biology, 1992, 113(2): 231-238 |
Peng S, Dai YG. Sequence polymorphism of mtDNA D-loop in the population of the endangered species Onychostoma rara from the Qingshui River. Journal of fisheries of China, 2009, 33(2): 196-200 [彭珊, 代应贵. 濒危鱼类稀有白甲鱼清水江种群mtDNA D-loop序列多态性. 水产学报, 2009, 33(2): 196-200] |
Purcell SW, Mercier A, Conand C, et al. Sea cucumber fisheries: Global analysis of stocks, management measures and drivers of overfishing. Fish and Fisheries, 2013, 14(1): 34-59 DOI:10.1111/j.1467-2979.2011.00443.x |
Saitou N, Nei M. The neighbor-joining method: A new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 1987, 4(4): 406-425 |
Soliman T, Fernandez-Silva I, Reimer JD. Genetic population structure and low genetic diversity in the over-exploited sea cucumber Holothuria edulis, Lesson, 1830 (Echinodermata: Holothuroidea) in Okinawa Island. Conservation Genetics, 2016, 17(4): 1-11 |
Sun C, Liu HB, Jiang T, et al. Comparison of D-loop and cytochrome b sequences among different coilia species and their potential use in analysis for the identification of species at early life stages. Progress in Fishery Sciences, 2018, 39(5): 27-35 [孙超, 刘洪波, 姜涛, 等. 不同鲚属鱼类Cyt b和D-loop序列的比较及其判别早期生活史个体的潜力分析. 渔业科学进展, 2018, 39(5): 27-35] |
Sun XJ, Li Q, Kong LF. Comparative mitochondrial genomics within sea cucumber (Apostichopus japonicus): Provide new insights into relationships among color variants. Aquaculture, 2010, 309: 280-285 DOI:10.1016/j.aquaculture.2010.08.001 |
Thompson JD, Gibson TJ, Plewniak F, et al. The ClustalX windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25: 4876-4882 DOI:10.1093/nar/25.24.4876 |
Uthicke S, Benzie JA. Gene flow and population history in high dispersal marine invertebrates: Mitochondrial DNA analysis of Holothuria nobilis (Echinodermata: Holothuroidea) populations from the Indo-Pacific. Molecular Ecology, 2010, 12(10): 2635-2648 |
Wan H, Wang TT, Hu LP, et al. Microsatellite analysis of genetic characteristics in crosses between Korea population and China population of sea cucumber Apostichopus japonicas. Fisheries Science, 2016, 35(1): 60-66 [王鹤, 王田田, 胡丽萍, 等. 仿刺参中国群体与韩国群体杂交子代微卫星标记分析. 水产科学, 2016, 35(1): 60-66] |
Wang JY, Zhen Y, Wang GS, et al. Molecular identification and detection of moon jellyfish (Aurelia sp.) based on partial sequencing of mitochondrial 16S rDNA and COⅠ. Chinese Journal of Applied Ecology, 2013, 24(3): 847-852 [王建艳, 甄毓, 王国善, 等. 基于mt-16S rDNA和mt-COⅠ基因的海月水母分子生物学鉴定方法和检测技术. 应用生态学报, 2013, 24(3): 847-852] |
Wang Y. Microbial community diversity of the surface and intermediate seawater and a columnar sediment of Maluku Strait in Tropical Western Pacific. Masterxs Thesis of Ocean University of China, 2015 [王燕.热带西太平洋海区中上层海水及马鲁古海峡柱状深海沉积物微生物群落多样性研究.中国海洋大学硕士研究生学位论文, 2015]
|
Wright S. Evolution in Mendelian populations. Genetics, 1931, 16(2): 97-159 |
Zeng XQ, Zhang WF, Gao TX. Molecular phylogenetics of genus Temnopleurus based on 16S rRNA and COⅠ sequences. Periodical of Ocean University of China, 2012, 42(6): 47-51 [曾晓起, 张文峰, 高天翔. 基于线粒体16S rRNA与COⅠ基因序列的刻肋海胆属系统发育研究. 中国海洋大学学报, 2012, 42(6): 47-51] |
Zhan AB. Development, characterization and application of microsatellite markers in Zhikong scallop (Chlamys farreri). Doctoral Dissertation of Ocean University of China, 2007 [战爱斌.栉孔扇贝(Chlamys farreri)微卫星标记的筛选及应用.中国海洋大学博士研究生学位论文, 2007]
|
Zhang DN, Zheng LM, He JR, et al. DNA barcoding of hydromedusae in northern Beibu Gulf for species identification. Biodiversity Science, 2015, 23(1): 50-60 [张珰妮, 郑连明, 何劲儒, 等. 基于线粒体COⅠ和16S片段序列的北部湾北部水螅水母DNA条形码分析. 生物多样性, 2015, 23(1): 50-60] |
Zhang W, Cao ZF, Li YC, et al. Taxonomic status of the three color variants in sea cucumber (Apostichopus japonicus): Evidence from mitochondrial phylogenomic analyses. Mitochondrial DNA, 2016, 27(4): 2330-2333 |



