2. 中国水产科学研究院淡水渔业研究中心 中国水产科学研究院长江中下游渔业生态环境评价与资源养护重点实验室 无锡 214081
2. Key Laboratory of Fishery Eco-Environment Assessment and Resource Conservation in Middle and Lower Reaches of the Yangtze River, Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081
鲚属(Coilia)隶属于鲱形目、鳀科,是我国常见的经济鱼类。一般认为,我国分布的鲚属鱼类有4种,即刀鲚(C. nasus)、凤鲚(C. mystus)、七丝鲚(C. grayii)和短颌鲚(C. brachygnathus) (袁传宓等, 1976、1980)。另外,在太湖、巢湖等长江附属湖泊中分布有能自然繁殖的刀鲚定居型群体,袁传宓等(1976)将此群体定为刀鲚的亚种,即太湖湖鲚(Coilia nasus taihuensis)。目前,湖鲚为太湖鱼类组成的绝对优势种群(刘恩生等, 2005; 毛志刚等, 2011),关于太湖湖鲚的生长特征(毛志刚等, 2011)、生殖特性(孙雪兴等, 1987)、种群数量变动(刘恩生等, 2005)、食物组成(王银平等, 2016)等方面已有研究。然而,实际上太湖的鲚属鱼既有上颌骨长度大于头长的个体,也有上颌骨长度小于头长而符合短颌鲚定义(袁传宓等, 1976、1980)的个体,但多数研究者将太湖内的鲚属鱼类都归为太湖湖鲚(唐文乔等, 2007; 许志强等, 2009; 周晓犊等, 2010),只有少数研究者(Yang et al, 2010)在其研究中将太湖中的短颌鲚与湖鲚分别叙述。
基于生化和形态特征(刘文斌等, 1995),线粒体序列(唐文乔等, 2007; 许志强等, 2009; 周晓犊等, 2010; 张凌, 2012),核基因AFLP分析(Yang et al, 2010)都得出湖鲚、短颌鲚与刀鲚之间未达到亚种、种分化程度的结论,认为湖鲚、短颌鲚都只为刀鲚的淡水生态型。然而,由于湖鲚定义较难界定,在涉及湖鲚的研究中,存在误将太湖中的短颌鲚个体当做湖鲚的可能,进而导致遗传分析结论的客观性存疑。近期,有研究(程方圆等, 2019)开发了可以用来区分短颌鲚与刀鲚、湖鲚与刀鲚的SNP位点。然而,对于湖鲚与短颌鲚之间的分化程度和可区分度,仍需进一步研究。
线粒体DNA(mtDNA)具有母系遗传、结构简单无内含子、缺少重组、进化速率较快的特点(Rosel et al, 2017; DeSalle et al, 2017),已被广泛用于物种鉴定和遗传多样性研究。其中,线粒体细胞色素b (Cyt-b)基因进化速率适中,适合种群水平遗传差异的检测,被广泛应用于水产动物群体遗传多样性和遗传分化研究中(Ma et al, 2011; 李大命等, 2017; 周华兴等, 2019)。本研究室前期的工作中开发了利用鲚属鱼类线粒体序列区分刀鲚、凤鲚、七丝鲚早期生活史个体的方法(孙超等, 2018),证明了线粒体序列用于判别鲚属鱼类的潜力,为了解湖鲚与短颌鲚之间尤其是共同分布在太湖内的湖鲚与短颌鲚之间的遗传分化程度和可区分度提供了可能。因此,本研究拟通过对太湖区域内同水域存在的湖鲚与短颌鲚mtDNA Cyt-b全序列的测定和分析,来尝试评估其遗传多样性,探讨其遗传分化关系,以期为其分化程度的把握、今后资源的区分及相关的保护和开发利用提供理论依据。
1 材料与方法 1.1 样本采集及DNA提取47尾活体鲚采自太湖无锡鼋头渚游船码头附近水域渔船的渔获物,立即取少量肌肉组织固定在95%的乙醇中,其余鱼体保存在放置了冰盒的便携式采样箱中,带回实验室后均保存在-20℃冰箱中备用,解剖前测量全长、体重、上颌骨长、头长等数据,并计算上颌骨长/头长的值以区分湖鲚与短颌鲚,上颌骨长/头长低于1的为短颌鲚,高于1的为湖鲚(袁传宓等, 1976) (表 1)。DNA提取采用康为世纪生物科技有限公司的柱式基因提取试剂盒,按照说明书进行操作。提取后的DNA溶液在1.0%的琼脂糖凝胶上进行电泳,以检测其质量。检测合格后保存在-20℃备用。
|
|
表 1 太湖湖鲚与短颌鲚的上颌骨长/头长、全长、体重 Tab.1 The ratio of the supermaxilla length to head length, total length and body weight in C. nasus taihuensis and C. brachygnathus collected from the Taihu Lake |
参照刀鲚线粒体全基因组序列(Zhao et al, 2016),通过Primer premier 5.0软件(Singh et al, 1998)设计引物(F: CAACCAGACCCAAAATCAAT, R: CT TTGGGAGCTAGTGGTGG)用于扩增Cyt-b序列,该对引物扩增的区域包括刀鲚Cyt-b序列全长及上下游其他基因部分序列,以保证测序结果的准确性。
PCR扩增反应混合物体系包括:100 ng DNA模板,12.5 μl 2×Taq Master Mix(康为世纪生物科技有限公司),引物(10 mmol/L)各0.5 μl,加蒸馏水至终体积25 μl。PCR反应程序:95℃预变性5 min;94℃变性1 min,58.1℃退火45 s,72℃延伸90 s,共30个循环;最后,72℃延伸10 min。PCR扩增产物用1%琼脂糖凝胶电泳检测,然后,用上海生工生物工程技术有限公司的UNIQ-1柱式PCR产物纯化试剂盒进行纯化回收,对产物使用ABI3730XL测序仪(天霖生物技术有限公司)进行双向测序。
1.3 数据分析正反序列经过组装后辅以人工校正,得到一致序列,校对后的序列在GenBank中进行BLAST搜索,确定是目标Cyt-b序列。对获得的序列在BioEdit软件(https://bioedit.software.informer.com)中进行多重比对,检视比对过的结果,手工去除多余的碱基,获得1141 bp的Cyt-b全序列。从文献中下载刀鲚Cyt-b序列(登录号FJ460629-FJ460640, FJ460669-FJ460680)与本实验获得的湖鲚、短颌鲚共同用于分析系统发育关系。另外,从GenBank中下载七丝鲚与凤鲚的Cyt-b序列,与本研究所获得的湖鲚与短颌鲚序列进行遗传距离的计算。
由DnaSP软件(Rozas et al, 2003)输出单倍型文件,由Popatr软件(http://popart.otago.ac.nz)构建median joining network (MJN)以分析单倍型之间的关系。采用Arlequin软件(Excoffier et al, 2005)计算两种群间的Fst值,用以检验种群遗传结构,通过10000次重抽样检验其显著性。MEGA7软件用于计算基于Kimura two-parameter(K2P)模型的种间的平均遗传距离和各自种群内部的平均遗传距离(Kumar et al, 2016)。通过DAMBE软件进行碱基替换的饱和性检验(Xia et al, 2001):基于转换和颠换对K2P遗传距离作图分析和指数ISS检测,以检验Cyt-b序列是否适合用于分析系统发育关系。以凤鲚、七丝鲚作外类群,在MEGA7软件中计算整体序列的平均遗传距离以判断是否适合邻接法(Neighbor-joining method, NJ)建树,平均距离计算过程中误差评估采取Bootstrap法,重复次数为1000次。基于K2P模型采用邻接法构建系统发育树,各分支的支持率采用1000次Bootstrap检验。
2 结果测序共得到47条序列,经BLAST比对,确定是目标序列,比对后使用BioEdit软件截取1141 bp的Cyt-b全序列(序列已提交到GenBank)。经DnaSP软件检测,共有变异位点26个,其中,简约信息位点12个,共得到单倍型22个。湖鲚具有单倍型19个,其中,湖鲚特有单倍型18个;短颌鲚中具有单倍型4个;湖鲚与短颌鲚共享1个单倍型,该单倍型对应的9个个体中,有1个个体为湖鲚,其他都是短颌鲚。
根据上颌骨长度与头长之比将样本分为湖鲚与短颌鲚2个种群(表 1),两种群间基于的Cyt-b序列的固定指数Fst值达0.703(表 2),分化程度较高;两种群间的基因流仅为0.212,显示两种群之间的基因流十分微弱。Fst的值在统计上达到了显著水平(P < 0.001)。
|
|
表 2 不同种类间的Fst值与基因流 Tab.2 Fst value and gene flow between C. nasus taihuensis and C. brachygnathus |
湖鲚和短颌鲚种群内平均Kimura双参数遗传距离分别为0.002和0.001,而湖鲚与短颌鲚种群间平均Kimura双参数遗传距离为0.005,高于湖鲚和短颌鲚种群内部的平均Kimura双参数遗传距离。但湖鲚与七丝鲚的遗传距离为0.034,与凤鲚的遗传距离为0.086;短颌鲚与七丝鲚的遗传距离为0.033,与凤鲚的遗传距离为0.085,远高于湖鲚与短颌鲚之间的遗传距离(表 3)。
|
|
表 3 湖鲚、短颌鲚与凤鲚种类间及种类内平均Kimura双参数遗传距离 Tab.3 Mean value of the Kimura's two-parameter distance between and within C. nasus taihuensis, C. brachygnathus, C. grayii and C. mystus |
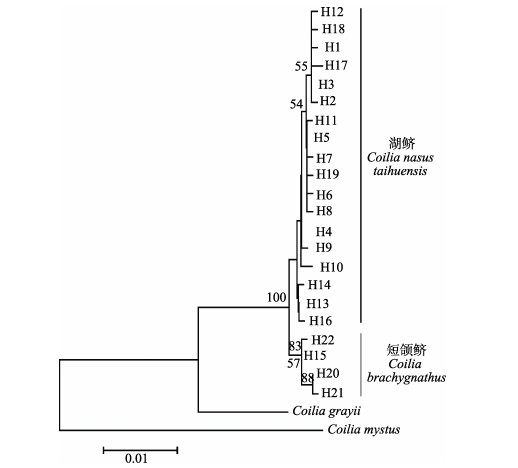
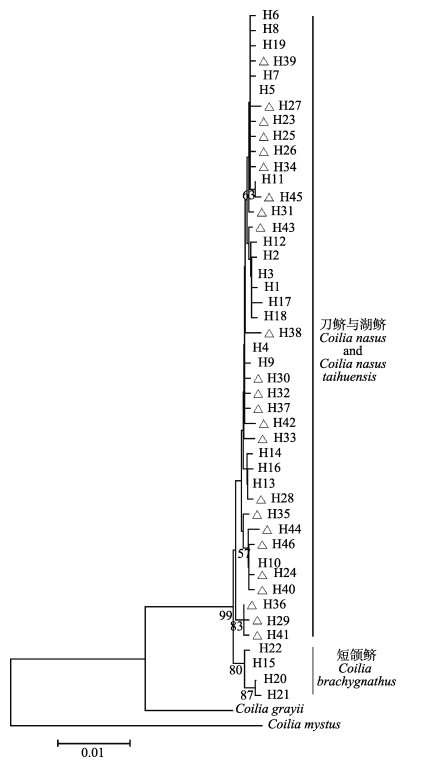
Cyt-b序列的碱基替代饱和度检验结果显示,对称的进化树和非对称的进化树其Iss.c值均大于Iss值,且差异极显著(双尾检验P=0.0000),这表明碱基替换未饱和,适合构建系统进化树。经检验,整体平均距离为0.013(0 < 0.013 < 1),适合构建NJ系统发育树。在NJ系统发育树上,22个单倍型构成了2个大的分支,其中一个分支的4个单倍型中,3个为短颌鲚的单倍型,1个为湖鲚、短颌鲚的共享单倍型,另一个分支的18个单倍型全部为湖鲚的单倍型(图 1),两大主要分支之间的支持度达到100。基于湖鲚与短颌鲚的Cyt-b序列构建的单倍型网络图分为截然不同的两部分(图 2),在A部分与B部分之间的变异步数较大。A部分里,主要是湖鲚的特有单倍型;B部分里,1个湖鲚个体具有该单倍型。从GenBank下载的刀鲚单倍型与湖鲚、短颌鲚单倍型共同构建NJ系统发育树上,46个单倍型仍然构成2个大的分支,其中一个分支上的4个单倍型中,3个为短颌鲚的单倍型,1个为湖鲚、短颌鲚的共享单倍型,另一个分支上的42个单倍型为湖鲚与刀鲚的单倍型,湖鲚与刀鲚单倍型互相交杂,共同构成一个大的分支(图 3),两大主要分支之间的支持度达到99。
|
图 1 基于湖鲚、短颌鲚Cyt-b基因序列构建的邻接法(NJ)系统进化树 Fig.1 Neighbor-joining (NJ) phylogenetic tree of C. nasus taihuensis and C. brachygnathus based on the Cyt-b gene sequences 分支上的数字为自举检验支持率 Numbers on each branch refer to bootstrap value |

|
图 2 基于Cyt-b单倍型构建的单倍型网络关系图 Fig.2 The haplotype network diagram constructed based on Cyt-b haplotypes 圆的大小代表单倍型频率,横线代表突变步数 The size of circles are corresponded to haplotype frequency, the number of vertical bars on the line indicates the steps of substitutions separating two haplotypes |
|
图 3 基于刀鲚、湖鲚、短颌鲚Cyt-b基因序列构建的邻接法(NJ)系统进化树 Fig.3 Neighbor-joining (NJ) phylogenetic tree of C. nasus, C. nasus taihuensis and C. brachygnathus based on the Cyt-b gene sequences 分支上的数字为自举检验支持率,△表示从GenBank上下载的刀鲚Cyt-b序列 Numbers on each branch refer to bootstrap value, △ represents Cyt-b sequences of C. nasus downloaded from GenBank |
在本研究中,湖鲚与短颌鲚间的平均遗传距离为0.005,高于湖鲚、短颌鲚各自内部的平均遗传距离,但远低于湖鲚、短颌鲚分别与凤鲚、七丝鲚之间的遗传距离。这一结果与唐文乔等(2007)、周晓犊等(2010)、张凌(2012)、许志强等(2009)基于线粒体研究得出刀鲚、湖鲚、短颌鲚之间的遗传距离的结果相似。本研究基于Cyt-b序列使用NJ法得出的系统发生树及单倍型网络关系图都显示,除1个湖鲚个体具有共享单倍型外,湖鲚与短颌鲚分为截然不同的2个支系,显示了利用该方法进行湖鲚与短颌鲚早期生活史个体区分的潜力。同时,为了进一步验证该方法的可靠性,作者加入了从GenBank上下载到的刀鲚Cyt-b单倍型序列,发现湖鲚与刀鲚构成一个大的支系,且相互混在一起,而短颌鲚构成另一个大的支系(图 3),表明了该方法的可靠性。袁传宓等(1976)指出,湖鲚为“定居型刀鲚”,并指出刀鲚上颌骨长超过头长,短颌鲚上颌骨较短一般不超过头长。在本研究中,湖鲚(上颌骨长/头长 > 1)与刀鲚的单倍型聚在一起,相互混杂,显示刀鲚与湖鲚之间缺乏遗传分化,为湖鲚是“定居型刀鲚”的观点提供了证据。另外,王倩等(2017)等基于大量单拷贝核基因SNP分析了长江刀鲚与洞庭湖短颌鲚,证明刀鲚、短颌鲚之间具有明显的遗传分化,已经达到了种间分化的程度。那么湖鲚作为定居型的刀鲚,与短颌鲚之间也应该具有遗传分化,而本研究的结果证实了这一点。本研究中,湖鲚与短颌鲚分为截然不同两支系的情况与前人研究有所不同。在唐文乔等(2007)、周晓犊等(2010)、张凌(2012)的研究中,仅指出其研究中湖鲚的采样地点为太湖、巢湖,未对上颌骨长/头长进行描述。许志强等(2009)的研究中报道了湖鲚样本的上颌骨长/头长范围为0.882~ 1.133,显示这批样本中有部分个体的上颌骨长是小于头长的。同样,袁传宓等(1980)研究也发现,太湖中同时存在上颌骨长/头长 > 1、上颌骨长/头长 < 1两种形态的鲚鱼。那么,在上述的遗传分析中,很可能有将湖中的短颌鲚当作湖鲚分析的情况,由此得出的短颌鲚与湖鲚遗传上不能区分的结果显然不够可靠。本研究在区分上颌骨长/头长比率这一形态差异的基础上进行的遗传分析发现形态差异与遗传差异具有良好对应关系,即上颌骨长/头长 > 1的湖鲚与上颌骨长/头长 < 1的短颌鲚之间是具有明显遗传分化的。因此,建议在今后关于湖鲚的研究中明确湖鲚的定义,即湖鲚为定居型刀鲚,避免将同一湖泊内共存的湖鲚与短颌鲚混淆。同时,湖鲚与短颌鲚的形态型与Cyt-b遗传型之间良好的对应关系,显示了Cyt-b序列在区分形态上难以区分的湖鲚与短颌鲚的卵及仔稚鱼等早期生活史个体方面的应用潜力。
本研究结果显示,湖鲚与短颌鲚之间的固定系数Fst达0.703,基因流为0.212。Wright(1965)提出的遗传分化范围(Fst < 0.05,低度分化;0.05 < Fst < 0.15,中度分化;Fst > 0.15,分化程度较高;Fst > 0.25,分化程度极大),在群体遗传研究中得到广泛应用(Bracken et al, 2015; Zhou et al, 2018; 黄镇宇等, 2019)。该参数显示,湖鲚与短颌鲚的分化程度已经达到极大。当不同种群间的基因交流越充分,其遗传分化就越小,当Nm < 1时,意味着基因流不足以维持遗传匀质化,遗传漂变会导致遗传分化(Slatkin et al, 1987)。本研究得到的湖鲚与短颌鲚间的线粒体基因流为0.212,显示其基因流已不足以维持遗传匀质化,遗传漂变等因素会导致湖鲚与短颌鲚进一步分化。湖鲚与短颌鲚之间遗传距离低于湖鲚与凤鲚、短颌鲚与凤鲚之间的种间遗传距离,这种情况在一些其他属中也比较常见。例如,在北美鲱科(Clupeidae)、西鲱属(Alosa)中,Alosa alabamae与A. sapidissima之间的净核苷酸差异(0.0042),远小于它们分别与其他西鲱属物种间的差异(0.0329~0.0936)(Bowen et al, 2008);在蝠鲼科(Mobulidae)中Manta alfredi和M. birostris间的遗传距离(0.00313~0.00681)也远小于它们分别与属内其他物种间的遗传距离(0.02992~0.13198),研究者认为这是由于其分化的时间比起属内其他物种的分化时间短(Kashiwagi et al, 2012)。Nei(2013)提出,在遗传距离较小时,遗传距离与分化时间近似成线性关系。苏思博等(2019)基于Cyt-b序列发现,黑锦蛇(Elaphe schrenckii)和赤峰锦蛇(Elaphe anomala)各自的少部分群体之间存在Fst值显示分化极大而遗传距离极小,并认为这可能是由于种群处于分化早期,单倍型之间的关系较近,因此,种群间遗传距离较低,但各种群间单倍型的频率差异造成了Fst值较高,也可能与所采集样本分布不均匀有关。与之类似,湖鲚与短颌鲚之间遗传距离较低,而Fst值较高的原因可能就是它们之间的分化时间较短,还没有积累足够多的变异位点,但两种群间的单倍型分布已经有了极大变化,从而导致Fst值较高。当然,也不能完全排除采样不均的可能性。
本研究中湖鲚与短颌鲚之间存在的共享单倍型应该是不完全谱系分选或渐渗杂交的结果。Avise等(2000)提出,物种形成初期的物种首先形成一个复系的类群,之后会演化成并系的类群,最终形成互为单系的簇,湖鲚、短颌鲚之间尚未达到单系,表明它们之间的分化可能仍处于较早的阶段,相同的祖先单倍型仍残留在不同的种群中,最终可能会在遗传漂变等因素的作用下,共享单倍型在其中一个种群中固定下来,成为其特有单倍型(Funk et al, 2003; Tang et al, 2012),本研究共有单倍型较低的出现频率也暗示了这一点。除了不完全谱系分选,渐渗杂交也可能是共享单倍型出现的原因(Rapson et al, 2012; Zhang et al, 2018)。因此,对湖鲚与短颌鲚之间的共有单倍型,另一个可能的解释就是湖鲚与短颌鲚之前已经分化为2个不同的支系,但仍存在杂交的潜力,发生过渐渗杂交。湖鲚与短颌鲚都属于淡水定居生态型,具有类似的形态和生态特征,并且在太湖中处于陆封状态,产卵时间、产卵场所都具有较大重叠(袁传宓等, 1976)。然而,湖鲚与短颌鲚之间较高的遗传分化、较低的基因流显示它们之间的基因交流程度很低。这似乎显示了湖鲚与短颌鲚之间存在生殖隔离,正是这种生殖隔离机制阻止了湖鲚与短颌鲚之间自由交配,维持了形态与谱系之间的对应关系。而对于这种生殖隔离的程度与机制,目前的数据还不能精确得出结论,尚需进一步的研究核基因及其生态特征以获得更多的信息。
本研究结果显示,太湖中同域存在的湖鲚与短颌鲚种群之间具有高度的遗传分化,种群间基因流微弱;除少数共享单倍型外,在系统发育树上形成相互独立的支系。这就相应提示研究者,在今后进行相关研究时,宜注意区别对待湖鲚与短颌鲚个体。同时,也为湖鲚与短颌鲚早期生活史个体的判别提供了一种相对可靠的方法。当然,要更准确全面地了解湖鲚与短颌鲚的精细遗传结构和更准确的判别方法,尚需要进一步扩大采样范围,并应用其他线粒体、微卫星等分子标记,进一步开展深入的分析探索。
Avise JC. Phylogeography: The history and formation of species. Cambridge, Massachusetts, London: Harvard University Press, 2000
|
Bowen BR, Kreiser BR, Mickle PF, et al. Phylogenetic relationships among North American Alosa species (Clupeidae). Journal of Fish Biology, 2008, 72(5): 1188-1201 |
Bracken FSA, Hoelzel AR, Hume JB, et al. Contrasting population genetic structure among freshwater-resident and anadromous lampreys: The role of demographic history, differential dispersal and anthropogenic barriers to movement. Molecular Ecology, 2015, 24(6): 1188-1204 |
Cheng FY, Tao ZY, Li CH. Species identification of Coilia brachygnathus, C. nasus and C. nasus taihuensis with SNP markers. Journal of Shanghai Ocean University, 2019, 28(1): 10-18 [程方圆, 陶紫玉, 李晨虹. 应用单核苷酸多态性(SNP)标记鉴定短颌鲚、湖鲚和刀鲚. 上海海洋大学学报, 2019, 28(1): 10-18] |
DeSalle R, Schierwater B, Hadrys H. MtDNA: The small workhorse of evolutionary studies. Frontiers in Bioscience, Landmark, 2017, 22: 873-887 |
Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis. Evolutionary Bioinformatics, 2005, 1(4A): 47-50 |
Funk DJ, Omland KE. Species-level paraphyly and polyphyly: Frequency, causes, and consequences, with insights from animal mitochondrial DNA. Annual Review of Ecology, Evolution, and Systematics, 2003, 34(1): 397-423 |
Huang ZY, Zhang Q, Lu LF, et al. Genetic diversity of Liza affinis in coastal waters of China deduced from mitochondrial COⅠsequences. Progress in Fishery Sciences, 2019, 40(1): 46-52 [黄镇宇, 章群, 卢丽锋, 等. 基于线粒体COⅠ序列的中国前鳞遗传多样性分析. 渔业科学进展, 2019, 40(1): 46-52] |
Kashiwagi T, Marshall AD, Bennett MB, et al. The genetic signature of recent speciation in manta rays (Manta alfredi and M. birostris). Molecular Phylogenetics and Evolution, 2012, 64(1): 212-218 |
Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870 |
Li DM, Li K, Zhang TQ, et al. Genetic polymorphism analysis of Cytb and CO I gene sequences of Protosalanx hyalocranius in the Hongze Lake. Progress in Fishery Sciences, 2017, 38(6): 25-31 [李大命, 李康, 张彤晴, 等. 洪泽湖大银鱼(Protosalanx hyalocranius) Cytb和CO I基因序列多态性分析. 渔业科学进展, 2017, 38(6): 25-31] |
Liu ES, Liu ZW, Chen WM, et al. A study on the change of lake anchovy (Coilia ectenes taihuensis yen et lin) catches and its mutual relationship to the biological environment in Lake Taihu. Journal of Lake Sciences, 2005, 17(4): 340-345 [刘恩生, 刘正文, 陈伟民, 等. 太湖湖鲚渔获量变化与生物环境间相互关系. 湖泊科学, 2005, 17(4): 340-345] |
Liu WB. Biochemical and morphological comparison and interspecific relationships of four species of the genus Coilia in China. Oceanologia et Limnologia Sinica, 1995, 26(5): 558-565 [刘文斌. 中国鲚属4种鱼的生化和形态比较及其系统发育的研究. 海洋与湖沼, 1995, 26(5): 558-565] |
Ma C, Cheng Q, Zhang Q, et al. Genetic variation of Coilia ectenes, (Clupeiformes: Engraulidae) revealed by the complete cytochrome b, sequences of mitochondrial DNA. Journal of Experimental Marine Biology and Ecology, 2010, 385(1): 14-19 |
Mao ZG, Gu XH, Zeng QF, et al. Community structure and diversity of fish in Lake Taihu. Chinese Journal of Ecology, 2011, 30(12): 2836-2842 [毛志刚, 谷孝鸿, 曾庆飞, 等. 太湖鱼类群落结构及多样性. 生态学杂志, 2011, 30(12): 2836-2842] |
Nei M. Genetic distance. In: Encyclopedia of genetics. Amsterdam: Academic Press, 2013, 248-250
|
Rapson SA, Goldizen AW, Seddon JM. Species boundaries and possible hybridization between the black mongoose (Galerella nigrata) and the slender mongoose (Galerella sanguinea). Molecular Phylogenetics and Evolution, 2012, 65(3): 831-839 |
Rosel PE, Taylor BL, Hancock-Hanser BL, et al. A review of molecular genetic markers and analytical approaches that have been used for delimiting marine mammal subspecies and species. Marine Mammal Science, 2017, 33(S1): 56-75 |
Rozas J, Sánchez-DelBarrio JC, Messeguer X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioinformatics, 2003, 19(18): 2496-2497 |
Singh VK, Mangalam AK, Dwivedi S, et al. Primer premier: Program for design of degenerate primers from a protein sequence. Biotechniques, 1998, 24(2): 318-319 |
Slatkin M. Gene flow and the geographic structure of natural populations. Science, 1987, 236(4803): 787-792 |
Su SB, Huang S, Dong BJ. Sequence polymorphism and genetic diversity of Elaphe schrenckii and Elaphe anomala in northeast three provinces based on Cyt b gene. Journal of Changchun Normal University, 2019, 38(4): 76-86 [苏思博, 黄松, 董丙君. 东北三省棕黑锦蛇(Elaphe schrenckii)与赤峰锦蛇(Elaphe anomala)基于Cyt b基因的序列多态性及遗传多样性. 长春师范大学学报, 2019, 38(4): 76-86] |
Sun C, Liu HB, Jiang T, et al. Comparison of D-loop and cytochrome b sequences among different Coilia species and their potential analysis on identification of the species at early life stages. Progress in Fishery Sciences, 2018, 39(5): 27-35 [孙超, 刘洪波, 姜涛, 等. 不同鲚属鱼类Cyt b和D-loop序列的比较及其判别早期生活史个体的潜力分析. 渔业科学进展, 2018, 39(5): 27-35] |
Sun XX. A study of breeding characteristics of the lake anchovy, Coilia ectens taihuensis (yuan et lin) in Taihu Lake. Transactions of Oceanology and Limnology, 1987(2): 91-97 [孙雪兴. 太湖湖鲚生殖特性. 海洋湖沼通报, 1987(2): 91-97] |
Tang QY, Liu SQ, Yu D, et al. Mitochondrial capture and incomplete lineage sorting in the diversification of balitorine loaches (Cypriniformes, Balitoridae) revealed by mitochondrial and nuclear genes. Zoologica Scripta, 2012, 41(3): 233-247 |
Tang WQ, Hu XL, Yang JQ. Species validities of Coilia brachygnathus and C. nasus taihuensis based on sequence variations of complete mtDNA control region. Biodiversity Science, 2007, 15(3): 224-231 [唐文乔, 胡雪莲, 杨金权. 从线粒体控制区全序列变异看短颌鲚和湖鲚的物种有效性. 生物多样性, 2007, 15(3): 224-231] |
Wang Q, Cheng F Y, Li C H. Developing SNP markers for Coilia and its application in species delimitation. Journal of Shanghai Ocean University, 2017, 26(1): 8-16 [王倩, 程方圆, 李晨虹. 鲚属单核苷酸多态性位点(SNPs)标记开发及在物种界定中的应用初探. 上海海洋大学学报, 2017, 26(1): 8-16] |
Wang YP, Gu XH, Zeng QF, et al. Seasonal variations in the diet composition of Coilia ectenes taihuensis in different ecotypes of Lake Taihu. Journal of Lake Sciences, 2016, 28(5): 1078-1085 [王银平, 谷孝鸿, 曾庆飞, 等. 太湖不同生态型湖区湖鲚(Coilia ectenes taihuensis)食物组成及其季节变化. 湖泊科学, 2016, 28(5): 1078-1085] |
Wright S. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution, 1965, 19(3): 395-420 |
Xia X, Xie Z. DAMBE: Software package for data analysis in molecular biology and evolution. Journal of Heredity, 2001, 30(7): 1720-1728 |
Xu ZQ, Ge JC, Huang C, et al. Taxonomy of shortjaw tapertail anchovy Coilia brachygnathus by jaw length and mitochondrial Cytochrome b gene analysis. Journal of Dalian Fisheries University, 2009, 24(3): 242-246 [许志强, 葛家春, 黄成, 等. 基于颌骨长度和线粒体Cytb序列变异探讨短颌鲚的分类地位. 大连海洋大学学报, 2009, 24(3): 242-246] |
Yang QL, Han ZQ, Sun DR, et al. Genetics and phylogeny of genus Coilia in China based on AFLP markers. Chinese Journal of Oceanology and Limnology, 2010, 28(4): 795-801 |
Yuan CM, Lin JB, Qin AL, et al. On the classification history and status quo of genus Coilia in China. Journal of Nanjing University (Natural Sciences), 1976(2): 1-12 [袁传宓, 林金榜, 秦安舲, 等. 关于我国鲚属鱼类分类的历史和现状-兼谈改造旧鱼类分类学的几点体会. 南京大学学报(自然科学版), 1976(2): 1-12] |
Yuan CM, Qin AL, Liu RH, et al. On the classification of the anchovies, Coilia, from the lower Yangtze River and the southeast coast of China. Journal of Nanjing University (Natural Sciences), 1980(3): 67-82 [袁传宓, 秦安舲, 刘仁华, 等. 关于长江中下游及东南沿海各省的鲚属鱼类种下分类的探讨. 南京大学学报(自然科学版), 1980(3): 67-82] |
Zhang L. The species validity analysis of Coilia brachygnathus and Coilia nasus taibuensis based on the mitochondrial 12S rRNA genes sequences data. Journal of Chaohu College, 2012, 14(6): 55-58 [张凌. 基于线粒体12S rRNA基因序列探讨湖鲚、短颌鲚物种有效性. 巢湖学院学报, 2012, 14(6): 55-58] |
Zhang Q, Hu W, Zhou T, et al. Interspecies introgressive hybridization in spiny frogs Quasipaa (Family Dicroglossidae) revealed by analyses on multiple mitochondrial and nuclear genes. Ecology and Evolution, 2018, 8(2): 1260-1270 |
Zhao L, Zhao Y, Zhang N, et al. The complete mitogenome of Coilia nasus (Clupeiformes, Engraulidae) from Poyang Lake. Mitochondrial DNA Part A: DNA Mapping, Sequencing, and Analysis, 2016, 27(3): 1608-1609 |
Zhou HX, Hu YT, Duan GQ, et al. Population genetic analyses of Acrossocheilus wenchowensis in Xin'an Basin based on the mitochondrial DNA cytochrome b gene. Progress in Fishery Sciences, 2019, 40(2): 43-50 [周华兴, 胡玉婷, 段国庆, 等. 基于线粒体细胞色素b基因序列的新安江流域温州光唇鱼群体遗传研究. 渔业科学进展, 2019, 40(2): 43-50] |
Zhou XD, Yang JQ, Tang WQ, et al. Species validities analyses of Chinese Coilia fishes based on mtDNA COI barcoding. Acta Zootaxonomica Sinica, 2010, 35(4): 819-826 [周晓犊, 杨金权, 唐文乔, 等. 基于线粒体COI基因DNA条形码的中国鲚属物种有效性分析. 动物分类学报, 2010, 35(4): 819-826] |
Zhou Y, Lei Y, Lu Y, et al. Population genetics of a Chinese endemic, Gymnocypris potanini, Herzenstein, threatened by population isolation: Conflicting patterns between microsatellites and mitochondrial DNA. Hydrobiologia, 2018, 1-15
|



