线粒体DNA为环状闭合结构,因具有突变率高、重组率低、严格遵循母系遗传等特点,常被作为鱼类分子群体遗传学和分子系统学的重要标记(Xiao et al, 2000)。其中,应用较为广泛的有线粒体控制区序列(D-loop)、细胞色素B基因(Cyt b)和线粒体细胞色素C氧化酶Ⅰ(COⅠ)。孙超等(2018)通过Cyt b和D-loop基因判别不同鲚属鱼类早期生活史个体;李瑶瑶等(2018)通过线粒体COⅠ基因研究魁蚶(Scapharca brouhtonii)中国群体的分类学地位;黄镇宇等(2019)通过线粒体COⅠ基因分析中国前鳞鮻(Liza affinis)的遗传多样性;周华兴等(2019)通过线粒体D-loop序列解析温州光唇鱼(Acrossocheilus wenchowensis)的群体遗传变异规律。诸多应用均表明,线粒体中部分基因在鱼类遗传进化的研究方面具有重要作用。
卷口鱼(Ptychidio jordani)隶属于鲤形目(Cypriniformes)、鲤科(Cyprinidae)、野鲮亚科(Labeoninae)、卷口鱼属(Ptychidio),俗称嘉鱼,其因肉质细嫩、味道鲜美而深受消费者喜爱,经济价值极高,与斑鳠、鲈鱼、鳜鱼并称为“珠江四大淡水名鱼”。近年来,因过度捕捞和江河污染加剧,卷口鱼自然资源急剧衰退,因此,其种质资源保护和人工养殖越来越引起重视。
关于卷口鱼的研究在21世纪初期主要集中在形态分析、年龄与生长、生理生态、人工繁殖等(崔淼等, 2001; 谢刚等, 2001; 廖显平等, 2016; 廖国璋等, 2016; 祁宝崙等, 2001)。随着珠江水系鱼类资源日益下降,特别是梯级电站开发后,鱼类种群规模与种类日趋减少(王崇等, 2015),众多学者的关注点开始转向卷口鱼自然种群的健康发展,即通过分子生物学的手段,检测卷口鱼的遗传多样性以评估该群体未来的遗传发展趋势,但相关研究相对较少(刘毅辉等, 2007)。杜合军等(2006)利用RAPD技术检测了广西桂平至广东肇庆之间的卷口鱼群体,认为其中至少存在2个群体;赵建(2007)采用了传统形态学和框架分析的方法对珠江水系柳江、浔江、西江3个地理群体的卷口鱼的形态变异进行分析,认为3个江段卷口鱼形态差异明显,有一定程度的分化。范凤娟等(2010)开展了广西合山、柳州、桂平和广东郁南4个地理群体卷口鱼线粒体Cyt b基因的遗传变异分析,认为4个地理群体间无显著的遗传分化。众多学者对珠江水系卷口鱼的遗传分化情况得出不同结论,可能是由于分析方法不同造成的差异,亦有可能是样品数量较少造成的误差。但是,现行捕捞强度是否对某些群体造成毁灭性打击,梯级电站的开发是否对不同河段的卷口鱼群体造成地理隔离,种群是否能持续健康的发展仍需进行持续的调查评估。
本研究采用mtDNA D-loop序列测定的方法,对采自西江流域广西境内6条支流的卷口鱼群体进行遗传结构、遗传分化程度和基因交流状况分析,对广西境内的卷口鱼野生群体遗传状况进行评价,掌握其遗传背景资料,为今后西江卷口鱼的种质资源管理、保护和开发利用提供基础数据和科学依据。
1 材料与方法 1.1 材料采集卷口鱼样本于2014年6月~2016年12月采自西江水系广西境内,分别为红水河(HSH)、柳江(LIUJ)、西江(XIJ)、右江(YOUJ)、郁江(YUJ)和左江(ZJ)江段,共139尾。活体每尾取约5 g背部肌肉样本,用95%酒精保存于15 ml离心管备用,每天更换1次酒精(持续2 d)。样本数量、采样点见表 1。
|
|
表 1 卷口鱼D-loop遗传多样性指数 Tab.1 Genetic diversity parameters of P. jordani |
取约50 mg保存肌肉于1.5 ml的EP管中,采用醋酸铵法提取卷口鱼的总DNA (彭敏等, 2011),采用微量核酸蛋白分析仪对所提DNA的浓度和纯度进行检测,DNA质量检测采用1%的琼脂糖凝胶电泳,经检测合格的DNA模板保存于-20℃冰箱备用。采用由生工生物工程(上海)技术服务有限公司合成的D-loop通用引物对卷口鱼的D-loop序列进行扩增,引物序列为D-loop-F (5′-CACCCYTRRCTCCCAAAGCYA-3′)和D-loop-R (5′-GGTGCGGRKACTTGCATGTRTAA-3′) (Xiao et al, 2001)。
PCR反应体系50.0 μl,包括2×Ex Taq Master Mix 25 μl,10 μmol/L的上、下游引物各2 μl,250 ng/μl DNA模板2.0 μl,用ddH2O补至50.0 μl。反应条件:94℃预变性3 min,94℃变性45 s,60℃退火45 s,72℃延伸90 s,共进行35次循环。
经1.5%琼脂糖凝胶电泳检测合格的PCR扩增产物送至深圳华大基因公司进行正反2次重复测序。
1.3 数据处理采用MEGA 4.1软件中的ClustalW程序并辅以人工校对去除序列两端不稳定部分,分析序列碱基含量及变异位点,利用该软件的Kimura 2-parameter模型计算遗传距离,并以大眼卷口鱼(Ptychidio macrops) (GenBank登录号:MF457481.1)作为外类群,构建单倍型的NJ系统树(金逍逍等, 2013)。单倍型及遗传多样性参数使用DnsSP 5.10软件(Librado et al, 2009)统计。采用Arlequin 3.5软件(Excoffier et al, 2010)进行分子方差分析、评估种群的遗传分化,并进行Tajima’s D和Fu′s Fs中性检验以及核苷酸不配对分布;用Network 4.0软件(Bandelt et al, 1999)构建单倍型网络结构图。
2 结果与分析 2.1 卷口鱼线粒体D-loop碱基组成及突变类型 2.1.1 碱基组成6个卷口鱼群体的139条D-loop序列(去除序列两端不稳定部分)忽略插入、缺失时长度均为725 bp,碱基T、C、A和G的平均含量分别为34.6%、20.1%、31.0%和14.2%,其中,A+T (65.7%)远远高于C+G (34.3%),表现出较强的碱基偏倚性,与脊椎动物线粒体DNA序列特征类似。其中,本研究所获得的卷口鱼23个单倍型序列已上传至GenBank (登录号:MN093128-MN093150)。
2.1.2 突变类型139尾卷口鱼D-loop序列共检测出变异位点25个,占总位点数的3.45%,其中,简约信息位点19个,单一变异位点6个,共检测到1个插入或缺失位点。其中,红水河群体变异位点最多,共17个;其次为右江和郁江群体,为11个;柳江和西江群体变异位点最少,均为5个(表 1)。转颠换比R值为11.5,其中,23个为转换,2个为颠换。
2.2 卷口鱼线粒体D-loop的遗传多样性 2.2.1 群体间遗传多样性差异将6个不同地理群体设置为同一组群,单倍型多样性Hd=0.91263,核苷酸多样性Pi=0.00517。群体间的Hd=0.71585~ 0.92063,其中,红水河群体最高(0.92063),其次为西江群体(0.88889),最低为柳江群体(0.71585);群体间Pi=0.00173~0.00668,与单倍型多样性表现相一致。平均核苷酸差异以红水河群体最高,为4.83333,最低为柳江群体,为1.25366。遗传多样性各指数均以红水河最好,柳江最差(表 1)。
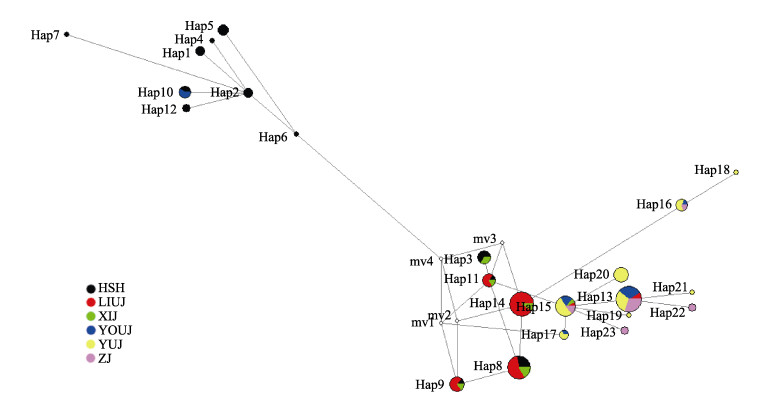
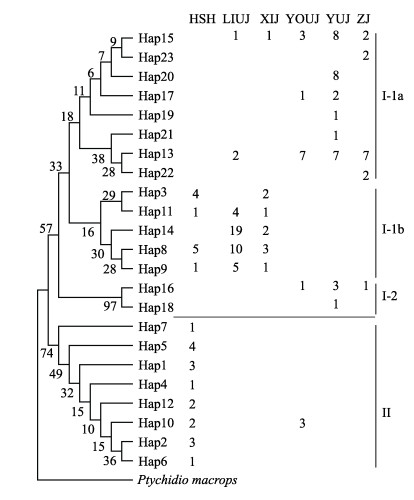
2.2.2 NJ系统树与单倍型网络结构单倍型网络结构(图 1)和NJ系统树(图 2)表现出较高的一致性。139尾个体拥有23个单倍型,主要分为2大支(Ⅰ和Ⅱ),分支Ⅱ仅由红水河群体大半个体和右江群体的3个个体构成,其余个体构成分支Ⅰ。hap13为优势单倍型,由23尾个体共享,占总个体数的16.55%,主要分布于柳江、右江、郁江和左江群体;hap14仅分布于柳江和西江群体,有21尾个体,占总个体数的15.11%。单倍型丰度最多的为红水河群体,拥有12个单倍型;其次为郁江群体,拥有8个单倍型;柳江与西江群体均拥有6个单倍型;左江与右江群体有5个单倍型。共有13个单倍型为不同群体独享,其中,红水河群体独享7个,郁江群体4个,左江群体2个,右江群体1个,柳江与西江群体均无独享单倍型。单倍型网络结构和NJ系统树拓扑结构显示,不同地理群体个体的单倍型分布混杂,未能观察到明显的地理聚群。
|
图 1 卷口鱼单倍型网络结构 Fig.1 Haplotype network structure of P. jordani 圆面积代表单倍型出现的频率,彩色扇形代表各群体在同一单倍型中所占的比例,空圈代表未发现或已经灭绝的单倍型 Circle portion represents the frequencies of haplotype, and the colored portion represents the percentage of the same haplotype in each populations, the empty circles represents the haplotypes which were not found or extinct |
|
图 2 卷口鱼单倍型系统树 Fig.2 Haplotype system tree of P. jordani |
利用MEGA 4.1软件分析结果如表 2所示:红水河群体和左江、郁江群体间的遗传距离最远,均为0.00863;遗传距离最近的是西江群体和柳江群体(0.00210)。群体内遗传距离由大到小依次为为红水河群体(0.00673) > 右江(0.00513) > 郁江(0.00329) > 西江(0.00228) > 左江(0.00233) > 柳江(0.00174)。依据NJ系统树划分的2大分支计算,分支Ⅰ遗传距离为0.003120,分支Ⅱ遗传距离为0.004869,2分支间遗传距离为0.010280。
|
|
表 2 卷口鱼遗传距离与遗传分化系数(Fst) Tab.2 Genetic distance and genetic differentiation coefficient (Fst) of P. jordani |
群体间Fst=0.05533~0.58351,除了左江与郁江、右江群体外,其余群体间均存在显著的遗传分化(P < 0.05),左江与柳江群体的遗传分化程度最大(0.52351),与郁江群体间的遗传分化程度最小(0.05533)。
AMOVA分析(表 3)发现,各地理群体间存在极显著的遗传分化(Fst=0.36737,P < 0.01)。群体间变异占36.74%,群体内变异占63.26%,变异大部分来自群体内部,基因流Nm=0.430513。按图 2将所有个体划分为2个谱系,分支间仍存在极显著的遗传分化(Fst=0.69318,P < 0.01),其中,分支间变异占69.32%,分支内变异占30.68%,基因流Nm=0.221313,表明2分支间基因交流处于较低水平。
|
|
表 3 AMOVA分析 Tab.3 Analysis of molecular variances (AMOVA) |
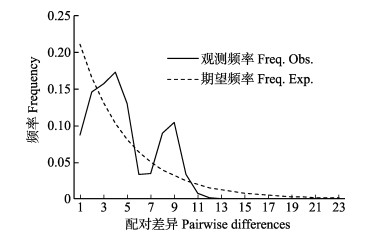
核苷酸错配分布(图 3)及Tajima's D和Fu′s Fs的检验(表 4)结果如下:Fu’s Fs检验(Fu’s Fs=-5.05210, P=0.08800)和Tajima’s D(Tajima’s D=-0.50322, P= 0.34600)检验均为不显著负值(P > 0.05),核苷酸错配分布为双峰,表明西江水系卷口鱼基于D-loop序列的群体动态分析支持西江水系卷口鱼未经历过种群扩张。各江段分析结果发现,除西江群体Fu's Fs检验为显著负值外(P=0.031),其他为不显著负值或正值(P > 0.05),表明除西江群体可能发生过种群扩张外,其他江段并无种群扩张迹象。
|
图 3 卷口鱼核苷酸错配分布 Fig.3 Nucleotide mismatch profile of P. jordani |
|
|
表 4 卷口鱼D-loop序列Tajimaxs D和Tajimaxs D检验 Tab.4 Tajimaxs D and Tajimaxs D test of P. jordani mtRNA D-loop |
遗传多样性通常是物种长期进化的结果,是物种或其群体持续生存并适应不断变化的环境而进化的前提。通常物种的遗传多样性或变异性越丰富,则表明该物种的进化潜力越大,对环境改变响应的进化能力就越强(Laikre et al, 2005; 宋娜等, 2018)。遗传多样性大小决定该生物能否继续在生物圈繁衍和生活,多样性下降将会对种群的健康发展产生负面影响(Ryman et al, 1987; 姜艳艳等, 2003; 杨子拓, 2016)。单倍型多样性指数和核苷酸多样性指数通常被用于评估生物遗传多样性(Vrijenhoek, 1994; 李大命等, 2017)。本研究发现,西江流域广西境内6个地理群体的卷口鱼多样性指数Hd=0.91260,Pi=0.00517。与同样基于D-loop序列为分子标记分析的同一流域的其他鲤科鱼类相比,高于位于可渡河的光唇裂腹鱼(Schizothorax lissolabiatus)种群(Hd=0.733,Pi=0.0027) (韩虎峰等, 2010),低于赤眼鳟(Spualiobarbus curriculus)种群(Hd=0.978,Pi=0.00791)(陈方灿等, 2015; 李潮等, 2018)和大眼华鳊(Sinibrama macrops)群体(Hd=0.91600,Pi=0.01569);与翘嘴鲌(Culter alburnus)群体(Hd= 0.87506, Pi=0.0070),和大眼近红鲌(Ancherythroculter lini)群体(Hd=0.847,Pi=0.00693)相比,具有更高的单倍型多样性与较低的核苷酸多样性(杨子拓等, 2016; 杨子拓, 2016)。本研究中,各群体的遗传多样性差异较大,除了红水河与右江群体,其他各江段的遗传多样性仍相对较低。西江流域广西境内多个江段卷口鱼群体呈现出高单倍型和低核苷酸多样性特征,可能是种群发生瓶颈效应后快速增长,使得单倍型的丰度提高,但时间不足以提高核苷酸多样性所造成(Grant, 1998)。
3.2 卷口鱼的遗传分化群体间的遗传距离是物种分类的一个重要依据。遗传距离越大表明群体间亲缘关系越远(张鹤千等, 2015)。本研究中,各群体间遗传距离为0.00210~ 0.00863,各群体内的遗传距离为0.00228~0.00673,无明显差异,表明该6个群体间具有较近的亲缘关系。单倍型的NJ系统树和网络图显示,本次采样的6个不同地理群体个体来源的单倍型分布混杂,未能观察到明显的地理聚群。但按NJ系统树划分的两分支间存在显著较高的遗传分化(Fst=0.69318,P < 0.01)。
群体遗传学认为,Fst是测量群体间遗传分化的重要参数,Fst值越大表明分化程度较高(Wright, 1979; 沈朕等, 2017)。Wright(1965)提出遗传分化标准(Fst < 0.05,无遗传分化;0.05 < Fst < 0.15,较小遗传分化;0.15 < Fst < 0.25,中度遗传分化;Fst > 0.25,遗传分化较大)。研究表明,柳江与西江、右江与郁江/左江、郁江与左江群体之间的遗传分化值为0.05~0.15,遗传分化较小;红水河与右江间的遗传分化值为0.15~0.25,为中度遗传分化;而红水河与柳江/西江/郁江/左江、柳江与右江/郁江/左江、西江与右江/郁江/左江间Fst > 0.25,遗传分化较大,地理隔离造成群体间基因交流减少可能是造成该结果的原因之一。如左江、右江是郁江、西江的上游,群体间遗传距离均小于0.00500,其主要受水坝阻隔的影响;又如红水河与郁江、右江群体的遗传距离均大于0.00500,因受大坝阻隔与不同支流的双重影响,遗传距离远大于前者,水坝阻隔与不同支流间的地理阻碍使基因交流更加困难,相应分化程度就越高,而红水河与其他群体遗传距离较远,可能与红水河过于密集的梯级开发程度有关。在2010年的调查中,红水河干流11个梯级中有10个已建或在建(薛联芳等, 2010)。匡天旭等(2018)在对珠江鲫(Carassius auratus auratus)遗传种质资源分析中亦发现,大量的水利开发可能是导致遗传多样性差异分布的原因之一。
3.3 西江流域卷口鱼的历史动态Tajima(1989)认为,如果Tajima’s D与Fu’s Fs值呈负值,且在统计学上达到显著标准,则说明序列中含有比中性进化模型更多的核苷酸位点变化,可能预示着被研究种群曾经历过一个扩张的历史。同一组群AMOVA分析及核苷酸错配分布图表明,Fu’s Fs检验(Fu’s Fs=-5.05210,P=0.08800)和Tajima’s D (Tajima's D=-0.50322,P=0.34600)检验均为不显著负值,核苷酸错配分布为双峰。结果支持西江流域卷口鱼未经历过种群扩张。
3.4 西江流域卷口鱼的种质资源保护鱼类作为水域生态系统的消费者,在水域环境中处于食物链的末端,对维护水域生态系统的物质循环与能量流动具有重要作用,任何水环境污染及水利工程开发等都有可能对鱼类群体的健康发展造成不良影响,从而影响水域生态系统的稳定及正常运行。西江流域广西境内的卷口鱼群体仍保持着较好的遗传多样性,但部分群体间遗传分化程度较大,水坝阻隔和捕捞因素可能促进了卷口鱼的遗传分化,其中,以红水河水利梯级开发对卷口鱼的基因交流造成的影响更为明显。建议加强对卷口鱼以及西江流域其他土著鱼类遗传多样性的监控;水电大坝合理设置过鱼设施以促进各江段群体间迁移;适当引入同一流域不同江段的亲鱼进行人工繁育以获得杂合度更高的子代,并通过增殖放流增进各江段群体间基因交流,以此弥补因地理阻隔造成的基因缺失。
Bandelt HJ, Forster P, Rohl A. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16(1): 37-48 DOI:10.1093/oxfordjournals.molbev.a026036 |
Chen FC. Study on the population diversity of Squaliobarbus curriculus in Xijiang. Masterxs Thesis of Shanghai Ocean University, 2015 [陈方灿.西江赤眼鳟群体多样性研究.上海海洋大学硕士研究生学位论文, 2015] http://cdmd.cnki.com.cn/Article/CDMD-10264-1015974713.htm
|
Cui M, Zhao J, Chen XL. Age and growth of Ptychidio jordani. Journal of South China Normal University (Natural Science), 2001(4): 9-15 [崔淼, 赵俊, 陈湘粦. 卷口鱼的年龄和生长. 华南师范大学学报(自然科学版), 2001(4): 9-15] |
Du HJ, Zhu XP, Chen KC, et al. RAPD analysis of genetic diversity of wild Ptychidio jordani in Pearl River. Journal of Fisheries of China, 2006, 30(3): 305-310 [杜合军, 朱新平, 陈昆慈, 等. 珠江野生卷口鱼遗传多样性的RAPD分析. 水产学报, 2006, 30(3): 305-310] |
Excoffier L, Lischer H. Arlequin suite Ver 3.5: A new series of programs to perform population genetics analysis under Linux and Windows. Molecular Ecology Resources, 2010, 10(3): 564-567 DOI:10.1111/j.1755-0998.2010.02847.x |
Fan FJ, Zhang Q, Zhao S, et al. Sequence analysis of mitochondrial Cytb gene in genetic variation of endemic Ptychidio jordani in Pearl River. Agricultural Science in Guangdong Province, 2010, 46(4): 161-164 [范凤娟, 章群, 赵爽, 等. 珠江水系特有卷口鱼遗传变异的线粒体Cytb基因序列分析. 广东农业科学, 2010, 46(4): 161-164] |
Grant W. Shallow population histories in deep evolutionary lineages of marine fishes: Insight from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89(5): 415-426 DOI:10.1093/jhered/89.5.415 |
Han HF, Dai YG. Polymorphism analysis of mtDNA D-loop from Schizothorax lissolabiatus Tsao in Kedu River population of Pearl River. Journal of Fishery Sciences of China, 2010, 17(1): 143-149 [韩虎峰, 代应贵. 珠江水系光唇裂腹鱼可渡河种群mtDNA D-loop序列多态性分析. 中国水产科学, 2010, 17(1): 143-149] |
Huang ZY, Zhang Q, Lu LF, et al. Genetic diversity of Liza affinis in coastal waters of China deduced from mitochondrial COⅠsequence. Progress in Fishery Sciences, 2019, 40(1): 46-52 [黄镇宇, 章群, 卢丽锋, 等. 基于线粒体COⅠ基因序列的中国前鳞鮻遗传多样性分析. 渔业科学进展, 2019, 40(1): 46-52] |
Jiang YY, Kong XY, Yu ZN, et al. Variation analysis of mtDNA D-loop from Scomberomorus niphonius in Yellow Sea. Journal of Fishery Sciences of China, 2003, 10(3): 177-183 [姜艳艳, 孔晓瑜, 喻子牛, 等. 黄海蓝点马鲛mtDNA D- loop序列变异分析. 中国水产科学, 2003, 10(3): 177-183] |
Jin XX, Sun YN, Wang RX, et al. Characteristics and phylogenetic analysis of mitochondrial genome in the gobies. Hereditas, 2013, 35(12): 1391-1402 [金逍逍, 孙悦娜, 王日昕, 等. 虾虎鱼线粒体全基因组序列结构特征分析及系统发育关系探讨. 遗传, 2013, 35(12): 1391-1402] |
Kuang TX, Shuai FM, Chen WT, et al. Genetic diversity and population structure of Carassius auratus auratus from Xijiang. South China Fisheries Science, 2018, 14(5): 29-35 [匡天旭, 帅方敏, 陈蔚涛, 等. 西江鲫的遗传多样性与群体结构. 南方水产科学, 2018, 14(5): 29-35] |
Laikre L, Palm S, Ryman N. Genetic population structure of fishes: Implications for coastal zone management. AMBIO: A Journal of the Human Environment, 2005, 34(2): 111-120 DOI:10.1579/0044-7447-34.2.111 |
Li C, Lan ZJ, Zhao J. Genetic diversity and differentiation of Squaliobarbus curriculus in Pearl River based on mitochondrial control region (D-loop) sequence. Journal of Applied and Environmental Biology, 2018, 24(3): 615-622 [李潮, 蓝昭军, 赵俊. 基于线粒体控制区(D-loop)序列的珠江赤眼鳟遗传多样性与遗传分化. 应用与环境生物学报, 2018, 24(3): 615-622] |
Li DM, Li K, Zhang TQ, et al. Sequence polymorphism of Cytb and COⅠ genes in Protosalanx hyalocranius from Hongze Lake. Progress in Fisheries Science, 2017, 38(6): 25-31 [李大命, 李康, 张彤晴, 等. 洪泽湖大银鱼(Protosalanx hyalocranius)Cytb和COⅠ基因序列多态性分析. 渔业科学进展, 2017, 38(6): 25-31] |
Li YY, Liu YG, Liu XL, et al. The taxonomic status of Chinese population of Scapharca broughtonii based on sequences of COⅠand 12S rRNA genes. Progress in Fishery Sciences, 2018, 39(5): 73-80 [李瑶瑶, 刘运国, 刘晓玲, 等. 基于线粒体基因组COⅠ和12S rRNA序列的魁蚶中国群体分类学地位研究. 渔业科学进展, 2018, 39(5): 73-80] |
Liao GZ, You BZ, Bai YQ, et al. Study on age growth, feeding and reproduction of Ptychidio jordani in Pearl River. Journal of Fisheries of China, 2001, 10(1): 71-86 [廖国璋, 尤炳赞, 白岳强, 等. 珠江卷口鱼年龄生长食性和繁殖的研究. 水产学报, 2001, 10(1): 71-86] |
Liao XP, Lin WQ, Xu ZS, et al. Study on artificial reproduction of Ptychidio jordani. Agricultural Science Bulletin of Anhui, 2016, 22(11): 117-119 [廖显平, 林伟强, 许政生, 等. 卷口鱼人工繁殖技术研究. 安徽农学通报, 2016, 22(11): 117-119] |
Librado P, Rozas J. Dnasp v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 |
Liu YH, Jiao ZY, Chen YL, et al. Morphological characteristics and karyotype of the Pearl River of Ptychidio jordani. Journal of Fisheries of China, 2007, 31(6): 721-725 [刘毅辉, 焦宗垚, 陈永乐, 等. 珠江卷口鱼形态特征与染色体组型. 水产学报, 2007, 31(6): 721-725] |
Peng M, Chen XL, Jiang WM, et al. Extraction of genomic DNA from Trachinotus ovatus by ammonium acetate. Tianjin Journal of Agricultural Science, 2011, 17(1): 114-117 [彭敏, 陈秀荔, 蒋伟明, 等. 醋酸铵法提取卵形鲳鲹基因组DNA. 天津农业科学, 2011, 17(1): 114-117] |
Qi BL, Xie G, Ye X, et al. Preliminary experiment of artificial domestication of Ptychidio jordani. Water Fishery, 2001, 21(3): 26 [祁宝崙, 谢刚, 叶星, 等. 野生卷口鱼的人工驯养初步试验. 水利渔业, 2001, 21(3): 26] |
Ryman N, Utter F. Population genetics and fishery management. Washington: University of Washington Press, 1987
|
Shen Z, Guan HB, Zheng FR, et al. Genetic diversity analysis of four Hexagrammos otakii populations based on Cytb and D-loop. Advances in Marine Science, 2017, 35(4): 524-534 [沈朕, 关洪斌, 郑风荣, 等. 基于cytb和D-loop的4个大泷六线鱼群体遗传多样性分析. 海洋科学进展, 2017, 35(4): 524-534] |
Song N, Zhou W, Jin BS, et al. Population genetics analysis of Pelteobaggrus nitidus in the Yellow River Basin based on mitochondrial DNA control region. Journal of Ocean University of China (Natural Science), 2018, 48(8): 20-26 [宋娜, 周伟, 金斌松, 等. 基于线粒DNA控制区高变区的黄河流域光泽黄颡鱼群体遗传学分析. 中国海洋大学学报(自然科学版), 2018, 48(8): 20-26] |
Sun C, Liu HB, Jiang T, et al. Comparison of D-loop and cytochrome b sequences among different Coilia species and their potential use in analysis for the identification of species at early life stages. Progress in Fishery Sciences, 2018, 39(5): 27-35 [孙超, 刘洪波, 姜涛, 等. 不同鲚属鱼类Cyt b和D-loop序列的比较及其判别早期生活史个体的潜力分析. 渔业科学进展, 2018, 39(5): 27-35] |
Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics, 1989, 123: 585-595 |
Vrijenhoek RC. Genetic diversity and fitness in small populations. Conservation Genetics, 1994, 68: 37-53 |
Wang C, Xie S, Xie WX, et al. Investigation of fish resources in the main stream of Hongshui River after cascade operation. Journal of Freshwater Fisheries, 2015, 45(2): 30-36 [王崇, 谢山, 谢文星, 等. 红水河干流梯级运行后鱼类资源调查. 淡水渔业, 2015, 45(2): 30-36] |
Wright S. Evolution and the genetics of populations, volume 4: Variability within and among natural populations. International Biometric Society, 1979 |
Wright S. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution, 1965, 395-420 |
Xiao WH, Zhang YP. Genetics and evolution of mitochondrial DNA in fish. Acta Hydrobiologica Sinica, 2000, 24(4): 384-391 [肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化. 水生生物学报, 2000, 24(4): 384-391] |
Xiao W, Zhang Y, Liu H. Molecular systematics of Xenocyprinae (Teleostei: Cyprinidae): Taxonomy, biogeography, and coevolution of a special group restricted in East Asia. Molecular Phylogenetics and Evolution, 2001, 18(2): 163-173 DOI:10.1006/mpev.2000.0879 |
Xie G, Qi BL, Xu SY, et al. Critical water temperature and dissolved oxygen content of Ptychidio jordani. Journal of Freshwater Fisheries, 2001, 31(2): 47-48 [谢刚, 祁宝伦, 许淑英, 等. 卷口鱼的临界水温和溶氧量. 淡水渔业, 2001, 31(2): 47-48] |
Xue LF, Gu HB, Cui L, et al. Investigation and study on the impact of cascade development on water temperature accumulation in the main stream of Hongshui River. Journal of Hydropower, 2010, 36(11): 9-12 [薛联芳, 顾洪宾, 崔磊, 等. 红水河干流梯级开发对水温累积影响的调查研究. 水力发电, 2010, 36(11): 9-12] |
Yang ZT. Analysis of genetic structure and diversity of three species of subfamily cockroaches in the Pearl River Basin. Masterxs Thesis of South China Agricultural University, 2016 [杨子拓.珠江流域三种鲌亚科鱼类的遗传结构及遗传多样性分析.华南农业大学硕士研究生学位论文, 2016] http://d.wanfangdata.com.cn/Thesis/D01037482
|
Yang ZT, Sun JJ, Li GF, et al. Study on autogenetic diversity and genetic differentiation of Pearl River Culter alburnus Basilewsky based on mitochondrial D-loop gene. Journal of Sun Yat-Sen University (Natural Science), 2016, 55(3): 89-96 [杨子拓, 孙际佳, 李桂峰, 等. 基于线粒体D-loop基因的珠江翘嘴鱼自遗传多样性与遗传分化研究. 中山大学学报(自然科学版), 2016, 55(3): 89-96] |
Zhao J, Zhu XP, Chen YL, et al. Morphological variation of different geographical populations of Ptychidio jordani in Pearl River. Acta Animal, 2007, 53(5): 921-927 [赵建, 朱新平, 陈永乐, 等. 珠江卷口鱼不同地理种群的形态变异. 动物学报, 2007, 53(5): 921-927] |
Zhang HQ, Yang ZT, Li GF, et al. Sequence analysis of Cytb gene of wild Peheobagrus fulvidraco in Pearl River Basin. Journal of Sun Yat-Sen University (Natural Science), 2015, 54(5): 102-108 [张鹤千, 杨子拓, 李桂峰, 等. 珠江流域野生黄颡鱼Peheobagrus fulvidraco的Cytb基因序列分析. 中山大学学报(自然科学版), 2015, 54(5): 102-108] |
Zhou HX, Hu YT, Duan GQ, et al. Population genetic analyses of Acrossocheilus wenchowensis in Xin'an Basin based on the mitochondrial DNA cytochrome b gene. Progress of Fishery Sciences, 2019, 40(2): 43-50 [周华兴, 胡玉婷, 段国庆, 等. 基于线粒体细胞色素b基因序列的新安江流域温州光唇鱼群体遗传研究. 渔业科学进展, 2019, 40(2): 43-50] |



