2. 中国水产科学研究院黄海水产研究所 农业农村部水产品质量安全检测与评价重点实验室 青岛 266071;
3. 大连工业大学 大连 116000
2. Key Laboratory of Testing and Evaluation for Aquatic Product Safety and Quality, Ministry of Agriculture and Rural Affairs; Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
3. School of Food Science and Technology, Dalian Polytechnic University, Dalian 116000
金枪鱼是栖息在大洋上层的大型鱼类,具有高度洄游的特性,在世界渔业中占有重要的地位(戴小杰等, 2007)。从渔业利用角度讲,金枪鱼一般指经济价值较大的蓝鳍金枪鱼(Thunnus thynnus)、马苏金枪鱼(Thunnus maccoyii)、黄鳍金枪鱼(Thunnus albacares)、大目金枪鱼(Thunnus obesus)、长鳍金枪鱼(Thunnus alalunga)和正鲣(Katsuwonus pelamis)等(徐坤华等, 2014),其中,蓝鳍、马苏、黄鳍和大目金枪鱼等主要用于生鱼片的加工,长鳍金枪鱼和正鲣等小型金枪鱼主要作为金枪鱼罐头和日本“木鱼”(Katsuobushi)原料(方健民等, 2006)。不同品种金枪鱼价格差异很大,尤其是其去头去皮的销售方式,导致金枪鱼市场上标签错乱和以次充好的问题尤为严重,损害了消费者利益,破坏了正常的经营秩序;由于市售金枪鱼及其制品均缺少失主要的形态学鉴定特征,因此,金枪鱼及其制品的分子鉴定方法成为国内外的研究热点。
DNA条形码是物种分子鉴定研究中的重要方法,美国FDA(Food and Drug Administration)也将其作为食品标签认证的方法(王敏等, 2015; Karim et al, 2016),弥补了传统形态学鉴定方法的不足。Barrett等(2005)最早提出DNA条形码的定义,且最先以细胞色素氧化酶亚基Ⅰ基因(COⅠ)为研究对象。COⅠ基因具有足够的遗传变异信息,广泛用于鱼类的鉴定和分类(Hanner et al, 2011; Khedkar et al, 2014)。随着基因条形码技术的发展,除了COⅠ基因序列作为标准条形码外,作为重要条形码的细胞色素b基因(Cyt b)、16S rRNA和线粒体细胞色素C氧化酶亚基Ⅱ基因(COⅡ)等,近年也越来越多地被应用于物种鉴定和遗传多样性分析(Chapela et al, 2005; 崔文涛等, 2013)。Cyt b能在同一物种内表现出稳定的遗传性,却在不同物种间具有明显的遗传差异,因此,被用于物种种属鉴定(Becker et al, 2015);线粒体16S rRNA既含有高度保守的序列区域,又有中度保守和高度变化的序列区域,已成为广泛应用于水生动物群体遗传多样性和系统进化分析研究的分子标记(孙超等, 2014; 陈文炳等, 2017)。
本研究以主要生食金枪鱼及其易混品种(蓝鳍、马苏、黄鳍、大目、长鳍金枪鱼和正鲣)为研究对象,利用COⅠ、Cyt b和16S rRNA基因对其进行DNA条形码研究、遗传差异分析和进化树分析,探讨3种基因在金枪鱼分子鉴定研究中的适用性,为生食金枪鱼的物种鉴定及金枪鱼产业的健康发展提供技术支撑。
1 材料与方法 1.1 材料 1.1.1 样品长鳍金枪鱼(组编号:CQ,下同)、黄鳍金枪鱼(HQ)、蓝鳍金枪鱼(LQ)、马苏金枪鱼(MS)、大目金枪鱼(DM)、正鲣(JY)肌肉组织共63份,每份均来自于捕捞地点、外形特征明确的金枪鱼个体,如表 1所示,由本实验室收集保存。
|
|
表 1 样品信息及编号 Tab.1 Sample information and number |
TIANGEN海洋动物组织DNA提取试剂盒、2×Taq PCR Master Mix和DL2000 DNA marker购自天根生化科技(北京)有限公司;其他试剂为国产分析纯;引物合成及序列测定由生工生物工程(上海)股份有限公司完成;PCR仪为T1常规PCR仪(德国Whatman Biomerra公司),凝胶成像分析系统Infinity 3000(法国Vilber Lourmat公司),核酸蛋白测定仪NanoPhotometer Pearl(德国Implen公司)。
1.2 方法 1.2.1 DNA提取及PCR扩增取30 mg鱼肉,剪碎后置于离心管中,按照试剂盒说明书提取DNA,用核酸蛋白测定仪测定DNA浓度和纯度,–20℃保存。本实验选取引物见表 2,PCR反应体系见表 3。PCR反应程序为:95℃ 10 min;94℃ 1 min,52℃或53℃ 1 min,72℃ 1 min,35个循环;72℃ 10 min。
|
|
表 2 引物信息 Tab.2 Information of primers |
|
|
表 3 PCR扩增体系 Tab.3 PCR amplification system |
将PCR产物进行1.0%琼脂糖凝胶电泳,在凝胶成像系统下观察并记录实验结果;PCR产物由生工生物工程(上海)股份有限公司进行序列测定。
1.2.2 序列比对与建树分析测序结果经人工校对后去除引物区,在NCBI网站上通过BLAST分析将测序结果与GenBank中参考序列比对分析,其中,COⅠ基因采用BOLD(Barcode of Life Data System生命条形码数据系统www.boldsystems.org)进行分析;通过DnaSP 5.10软件计算其二者序列的单倍型数(h)、单倍型多样性指数(Hd)、平均核苷酸差异数(k)、核苷酸多样性指数(π)等遗传多样性参数。运用Mega 7.0软件,统计序列碱基组成,计算序列保守位点(C-conserved sites)、简约信息位点(PI-parsimony- informative sites)和变异位点(V-variable sites),并采用邻接法(Neighbor joining, NJ)法,自展检验1000次,采用Kimura 2-parameter模型,以牛(Bos grunniens, GenBank:KM233416)线粒体DNA(mtDNA,包含COⅠ、Cyt b和16S rRNA基因)作为外类群,同时在NCBI网站选取6种金枪鱼mtDNA(T. alalunga GenBank: AB101291; T. albacares GenBank: JN086153; T. maccoyii GenBank: KF925362; T. obesus GenBank: GU256525; T. thynnus GenBank: KF906720; K. pelamis GenBank: KM605252)作为标准基因,构建系统发育进化树。
2 结果与分析 2.1 PCR扩增长鳍金枪鱼等63个实验样本均扩增出单一的片段,未发现其片段长度多态性,测序得到片段大小分别为COⅠ 652 bp、Cyt b 307 bp、16S rRNA 576 bp(图 1),与预期一致。
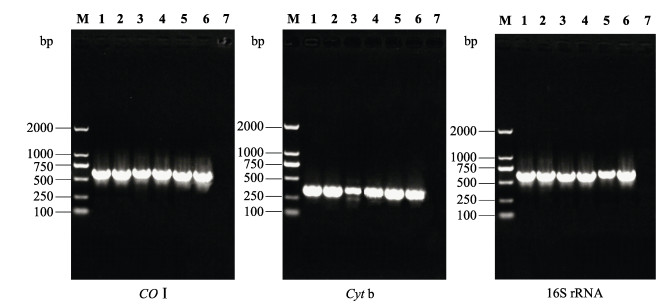
|
图 1 部分样品3种基因PCR产物电泳图
Fig.1 Electrophoresis map of three gene PCR products
M:DNA marker;1:长鳍金枪鱼;2:蓝鳍金枪鱼;3:大目金枪鱼; 4:黄鳍金枪鱼;5:马苏金枪鱼;6:鲣鱼;7:空白对照 M: DNA marker; 1: T. alalunga; 2: T. thynnus; 3: T. obesus; 4: T. albacares; 5: T. maccoyiis; 6: K. pelamis; 7: Blank control |
3种基因扩增产物序列经GenBank数据库比对,确定位于mtDNA 16S rRNA、COⅠ、Cyt b区域,BLAST结果和BOLD比对结果与各样本来源鱼的形态学鉴定结果一致。
经人工校正去除上下游引物,得到COⅠ、Cyt b和16S rRNA基因片段中T、C、A和G平均含量(表 4),其中,16S rRNA基因中A+T平均含量为52.7%,稍高于G+C平均含量(47.3%),且6种金枪鱼的GC含量差异不大;其中,COⅠ基因中A+T平均含量为53.0%,稍高于G+C平均含量(47.0%),且6种金枪鱼的GC含量差异不大;其中,Cyt b基因A+T平均含量为51.9%,稍高于G+C平均含量(48.1%),且6种金枪鱼相互间的GC含量差异也不大。3种基因的GC含量在47.0%~48.1%之间,其中,Cyt b基因GC含量最高(48.1%)。
|
|
表 4 6种金枪鱼COⅠ、Cyt b和16S rRNA基因碱基组成 Tab.4 Base composition of COⅠ, Cyt b and 16S rRNA genes in six tuna species |
各项遗传多样性参数统计如表 5所示。基于16S rRNA序列的金枪鱼各物种变异位点(V)为12,变异率仅为2.08%,远小于COⅠ(11.35%)、Cyt b(15.96%)。16S rRNA序列转换/颠换比(R)为1.49,也小于COⅠ(2.50)和Cyt b(2.48)。从平均核苷酸差异数(k)和核苷酸多样性指数(π)来看,16S rRNA是3种基因中最小的,而保守位点所占比率为97.92%,是3种基因中最保守的,说明了该核苷酸变异程度较低,表明了遗传物质的稳定性。
|
|
表 5 3种基因片段的遗传多样性参数 Tab.5 Genetic diversity parameters of three gene fragments |
基于Cyt b序列的金枪鱼各物种的变异位点(V)为49,小于COⅠ(71),但其变异率为15.96%,高于COⅠ变异率(11.35%)。Cyt b序列的单倍型多样性指数(Hd)低于COⅠ,平均核苷酸差异数(k)低于COⅠ,而核苷酸多样性指数(π)却是3种基因片段中最高的,说明较之16S rRNA和COⅠ序列,Cyt b序列核苷酸变异程度最高,遗传多样性较高,分化程度要比其余二者高,但其遗传多样性没有COⅠ序列高,COⅠ序列遗传资源较丰富。
2.3 系统进化树 2.3.1 16S rRNA基于16S rRNA基因构建的NJ系统进化树见图 2。图 2显示,6种金枪鱼分为4大支,黄鳍金枪鱼(HQ)与大目金枪鱼(DM)聚为一支。蓝鳍金枪鱼(LQ)与马苏金枪鱼(MS)聚为一支,其中3条标准序列T. albacares (JN086153),T. thynnus (KF906720)和T. obesus (GU256525)聚为一小支,与T. maccoyii (KF925362)共同在这一大支下。长鳍金枪鱼(CQ)和T. alalunga (AB101291)的遗传差异与上述4种金枪鱼较大,单独聚为一支。鲣鱼(JY)和K.pelamis (KM605252)单独归为一大支,且和上述5种金枪鱼属遗传距离最远,亲缘关系也最远。这一结果表明,16S rRNA基因只能单独区分出长鳍金枪鱼和鲣鱼,不能很好区分黄鳍、大目、蓝鳍与马苏金枪鱼,而且不能对同一物种不同地理群体进行聚类分析。16S rRNA基因序列相对较为保守,6种金枪鱼的遗传距离较为接近,遗传差异较小。
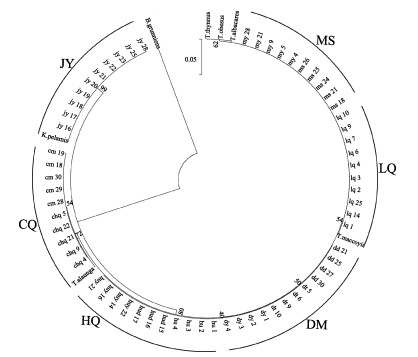
|
图 2 6种金枪鱼基于16S rRNA基因的系统进化树 Fig.2 Phylogenetic tree of 16S rRNA in six species of tuna gene |
基于COⅠ基因构建的NJ系统进化树见图 3。图 3显示,6种金枪鱼分为6大支,大西洋大目(dd)、太平洋大目(dt)和印度洋大目(dy)以及T.obesus (GU256525)归为一大支,虽然可以看出有地理群体聚类的趋势(dd-21、dd-25和dd-27聚为一小支等),但也存在混杂不同地理群体大目金枪鱼的问题(dy和dt相互参杂,dd-30与它们在同一分支下)。虽然蓝鳍金枪鱼(LQ)和T. thynnus (KF906720)与马苏金枪鱼(MS)遗传距离较为接近,但明显单独成为一支。印度洋马苏(my)和养殖马苏(ms)以及T. maccoyii (KF925362)归为马苏金枪鱼(MS)一大支,但无法从进化树当中明显看出二者因地理位置导致的遗传差异。除了huy-16的其他印度洋黄鳍(huy)、大西洋黄鳍(hud)和太平洋黄鳍(hu)以及T. albacares(JN086153)归为黄鳍金枪鱼(HU)一大支,三者同样无法从进化树当中看出明显的因地理位置导致的遗传差异。huy-16进化分支地位与HQ、DM、MS和LQ四者并列,这一个体单独成一支。南太平洋长鳍(chq)和美国长鳍(cm)以及T.alalunga (AB101291)归为长鳍金枪鱼(CQ)一大支,同样无法从进化树明显看出二者因地理位置导致的遗传差异。鲣鱼(JY)和K.pelamis (KM605252)单独归为一大支,且和上述5种金枪鱼遗传距离最远,亲缘关系也最远。结果表明,COⅠ基因能较好区别6种金枪鱼,其进化速率与16S rRNA基因相比较快,遗传信息较为丰富。
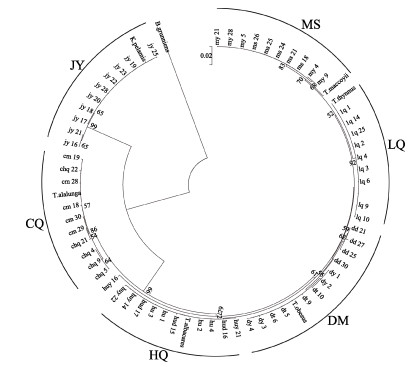
|
图 3 6种金枪鱼基于COⅠ基因的系统进化树 Fig.3 Phylogenetic tree of COⅠ gene in six species of tuna |
基于Cyt b基因构建的NJ系统进化树见图 4,其建树结果与COⅠ建树结果大体一致。不同的是,样品huy-16在Cyt b建树结果中归为黄鳍金枪鱼(HU)一大支,且Cyt b基因建树结果存在一定程度的地理群体聚类的趋势(dd-21、dd-25和dd-27聚为一小支等),但总体而言,无法从进化树中明显看出同种金枪鱼因地理位置导致的遗传差异。结果表明,相比于16S rRNA和COⅠ基因,Cyt b基因对于6种金枪鱼的区分最为准确,基因分化程度较高,区分度较好。
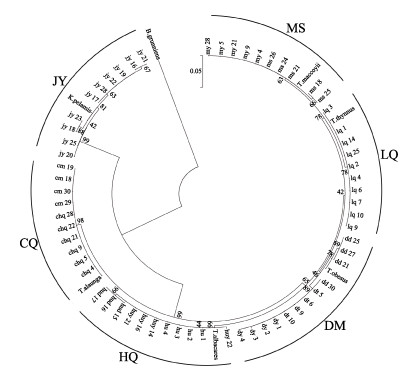
|
图 4 6种金枪鱼基于Cyt b基因的系统进化树 Fig.4 Phylogenetic tree of Cyt b gene in six species of tuna |
随着现代分子生物技术的快速发展,物种鉴定的研究方法已从形态学、生理生化水平发展到分子水平,鉴定的依据从形状、大小、颜色和解剖特征等方面,发展到DNA、RNA和蛋白质等生物大分子层面(蒋守富等, 2014; 柳淑芳等, 2016)。基于DNA开发的多种分子鉴定方法,已广泛应用于金枪鱼及其易混物种的鉴定中。Liu等(2016)采用实时荧光PCR技术成功鉴定了马苏、大目、黄鳍、长鳍和正鲣等金枪鱼;Lin等(2008)建立了多重PCR方法,用来区分鲣(Euthynnus pelamis)、圆舵鲣(Auxis rochei)、扁舵鲣(Auxis thazard)、东方狐鲣(Sarda orientalis)和鲔(Euthynnus affinis)等5种小型金枪鱼;Abdullah等(2016)运用单链构象多态性(Single-strand conformation polymorphism, SSCP)方法将圆舵鲣从金枪鱼属中鉴定出来。近年来,微卫星(Simple sequence repeats, SSRs)分子标记技术越来越多地应用于物种的鉴定和遗传多样性研究,邱凡等(2008)运用RAPD (Random Amplified Polymorphic DNA,随机扩增多态性DNA标记)和微卫星技术对比分析了太平洋蓝鳍金枪鱼野生群体的遗传多样性,发现微卫星与RAPD相比能更好更完善地揭示群体的遗传多样性;Riccioni等(2010)采用微卫星技术分析了大西洋蓝鳍金枪鱼从20世纪初到2007年的遗传差异,发现大西洋蓝鳍金枪鱼在空间和时间上保留了高水平的遗传多样性,为蓝鳍金枪鱼的鉴定研究奠定基础。上述技术手段能对特定范围内的金枪鱼进行鉴定,但也在不同程度上存在操作繁琐、鉴定时间长、成本较高等不足。
DNA条形码是分子鉴定研究中的主要方法,具有很多优点:基本不受样品组织形态的影响;不受样品生命周期的限制;通过基因测序鉴定,准确性高;操作简便,成本低;效率高,可快速鉴定大量样本;可帮助无专业分类学知识的人获得物种信息(律迎春等, 2011)。理想的标准DNA条形码片段并不局限于COⅠ基因序列,能有效鉴定物种的DNA片段都可以应用。此外,多基因遗传信息的组合使用,能获得更理想的物种系统进化分析结果(Aoyama et al, 2001)。针对金枪鱼采用多基因片段组合使用的鉴定方法已成为目前的关注重点,Neils等(2015)利用COⅠ和ITS1(内转录间隔1区)基因成功识别了地中海的3个产卵区的蓝鳍金枪鱼幼体;Lowenstein等(2009)采用多条COⅠ基因引物鉴定68种金枪鱼寿司样本,其中的22个样本商品标签与实际物种信息不符,显示了多基因组合使用的思路在金枪鱼物种分子鉴定领域中的广泛应用。邻接法、最大似然法(Maximum likelihood, ML)和最大简约法(Maximum parsimony, MP)是目前构建系统进化树的主要方法。其中,NJ法是基于最小进化原理常用的一种算法,具有重建的树相对准确、假设少、计算速度快等优点。故NJ法适用于进化距离不大,信息位点少的短序列,广泛用于鳕鱼、鲳鱼和银鱼等遗传进化分析。
本研究以主要渔获海域的6种金枪鱼及其易混种为研究对象,采用COⅠ、Cyt b和16S rRNA基因序列作为DNA条形码进行进化分析,探讨多基因条形码在金枪鱼鉴定研究中的适用性。从实验数据来看,16S rRNA基因序列变异率仅为2.08%,远小于COⅠ(11.35%)、Cyt b(15.96%),且其他遗传多样性参数也是3种序列中最小的;从建树结果来看,16S rRNA基因并不能完全区分出6种金枪鱼,只能鉴定出长鳍金枪鱼和鲣鱼,不能区分黄鳍和大目金枪鱼,也不能区分蓝鳍与马苏金枪鱼,更不能获得同一物种因地理位置导致的遗传差异。结果表明,COⅠ和Cyt b基因的进化速率较16S rRNA基因快,16S rRNA则相对保守,这在狭鳕(Theragra chalcogramma)、太平洋鳕(Gadus macrocephalus)等4种鳕鱼的遗传分析上也有类似结果(毕潇潇等, 2009)。
单倍型多样性指数、核苷酸多样性指数是衡量一个物种群体多样性的非常重要的指标(Vrijenhoek, 1994)。单倍型多样性指数是群体变异程度的重要指标,单倍型多样度高的群体说明其遗传多样性高,遗传资源丰富;而核酸多样性指数小,说明核苷酸变异程度较低,表明了遗传物质的稳定性。Cyt b序列的单倍型多样性指数(Hd)和平均核苷酸差异数(k)均低于COⅠ,而核苷酸多样性指数(π)却高于COⅠ,说明金枪鱼中Cyt b基因较COⅠ基因序列变异速率快,这与鲳属(Pampus)鱼类、波纹唇鱼(Cheilinus undulatus)和大银鱼(Protosalanx hyalocranius)中的规律相似(孙鹏等, 2011; 胡静等, 2014; 李大命等, 2017)。相比之下,COⅠ基因序列进化速率较慢,且比Cyt b拥有更多系统发育信息,更适合解析亲缘关系密切的分类类群(莫帮辉等, 2008)。相比而言,Cyt b进化速度适中,可以用于分析物种种内,甚至种属科间的进化遗传信息,有较强的适用性(辛翠娜等, 2009)。从建树结果来看,针对huy-16样本,Cyt b序列建树结果将huy-16归为黄鳍金枪鱼(HQ)这一大支;而COⅠ序列建树结果显示,该样本单独归为一支,独立于黄鳍、马苏、蓝鳍和大目金枪鱼之外,无法成功鉴定该样品。关于样品huy-16,16S rRNA和Cyt b序列建树结果都显示为黄鳍金枪鱼,而该样本来源经形态学鉴定,确属黄鳍金枪鱼,而从COⅠ序列建树结果并未得出这一结论,所以从该角度来看,Cyt b序列建树结果要比COⅠ序列建树结果要准确。COⅠ、Cyt b和16S rRNA 3种基因都无法从进化树中看出同种金枪鱼因地理位置导致的遗传差异,无法进行同一物种不同地理群体的聚类分析,但COⅠ与Cyt b在一定程度上体现了地理聚类趋势,可作为群体遗传进化研究的辅助参考。
目前,国内外研究多采用COⅠ基因作为鱼类分子鉴定及进化分析的目标基因,也有学者认为,如果仅以一种基因作为参考序列,有可能导致鉴定结果发生偏差,因此,建议同时使用多种基因进行研究(Page et al, 2010; Krück et al, 2013),这一观点已在石斑鱼和河鲀中得到证实(陈双雅等, 2012; 李楠等, 2018)。本研究结果进一步表明,对金枪鱼这种大洋性鱼类,不宜单独使用单一基因作为鉴定依据,建议COⅠ序列与Cyt b序列联合用于金枪鱼及其制品的物种鉴定研究。
Abdullah A, Rehbein H. The differentiation of tuna (family: Scombridae) products through the PCR-based analysis of the Cytochrome b gene and parvalbumin introns. Journal of the Science of Food and Agriculture, 2016, 96(2): 456-464 DOI:10.1002/jsfa.7111 |
Aoyama J, Nishida M, Tsukamoto K. Molecular phylogeny and evolution of the freshwater eel, Genus anguilla. Molecular Phylogenetics and Evolution, 2001, 20(3): 450-459 DOI:10.1006/mpev.2001.0959 |
Barrett RDH, Hebert PDN. Identifying spiders through DNA barcodes. Canadian Journal of Zoology, 2005, 83(3): 481-491 DOI:10.1139/z05-024 |
Becker RA, Sales NG, Santos GM, et al. DNA barcoding and morphological identification of neotropical ichthyoplankton from the Upper Paraná and São Francisco. Journal of Fish Biology, 2015, 87(1): 159-168 DOI:10.1111/jfb.12707 |
Bi XX, Gao TX, Xiao YS, et al. Sequence comparison of mtDNA 16S rRNA, COⅠ and Cyt b gene fragments in four species of Gadidae fish. South China Fisheries Science, 2009, 5(3): 46-52 [毕潇潇, 高天翔, 肖永双, 等. 4种鳕鱼线粒体16S rRNA、COⅠ和Cyt b基因片段序列的比较研究. 南方水产, 2009, 5(3): 46-52] |
Chapela MJ, Sotelo CG, Calo-Mata P, et al. Identification of cephalopod species (Ommastrephidae and Loliginidae) in seafood products by forensically informative nucleotide sequencing (FINS). Food Sciences, 2002, 67(5): 1672-1676 DOI:10.1111/j.1365-2621.2002.tb08703.x |
Chen SY, Wang JH, Chen WL, et al. Application of 16S rRNA gene and COⅠ gene sequence analysis in species identification of grouper. Biotechnology Bulletin, 2012(10): 124-130 [陈双雅, 王嘉鹤, 陈伟玲, 等. 16S rRNA基因和COⅠ基因序列分析在石斑鱼物种鉴定中的应用. 生物技术通报, 2012(10): 124-130] |
Chen WB, Yan TY, Peng J, et al. Identification of Anguilla rostrata, Anguilla anguilla and Anguilla japonica using DNA barcoding based on 16S rRNA gene. Food Science, 2017, 38(4): 283-289 [陈文炳, 缪婷玉, 彭娟, 等. 基于16S rRNA基因DNA条形码鉴定美洲鳗、欧洲鳗、日本鳗. 食品科学, 2017, 38(4): 283-289] |
Cui WT, Liu LQ, Li HM, et al. Phylogenic study of Octopodinae in the China Seas based on COⅡ partial sequences. Progress in Fishery Science, 2013, 34(3): 21-28 [崔文涛, 刘立芹, 李红梅, 等. 基于COⅡ基因序列的中国近海12种蛸亚科动物的分子系统进化研究. 渔业科学进展, 2013, 34(3): 21-28] |
Dai XJ, Xu LX. The primary color map of the world tuna fishery catch species. Beijing: Ocean Press, 2007 [戴小杰, 许柳雄. 世界金枪鱼渔业渔获物物种原色图鉴. 北京: 海洋出版社, 2007]
|
Fang JM, Huang FX, Zheng ZX, et al. Nutritional value and processing of tuna. Fisheries Science and Technology, 2006(2): 8-13 [方健民, 黄富雄, 郑钟新, 等. 金枪鱼的营养价值和加工利用. 水产科技, 2006(2): 8-13] |
Hanner R, Becker S, Ivanova NV, et al. FISH-BOL and seafood identification: Geographically dispersed case studies reveal systemic market substitution across Canada. Mitochondrial DNA, 2011, 22(S1): 106-122 |
Hu J, Hou XY, Yin SW, et al. Genetic diversity and divergence of Cheilinus undulatus of different geographic populations of the South China Sea revealed by COⅠ and Cytb gene analyses. Acta Hydrobiologica Sinica, 2014, 38(6): 1008-1016 [胡静, 侯新远, 尹绍武, 等. 基于mtDNA COⅠ和Cytb基因序列对南中国海不同海域波纹唇鱼群体遗传多样性的研究. 水生生物学报, 2014, 38(6): 1008-1016] |
Inoue J, Miya M, Tsukamoto K, et al. A mitogenomic perspective on the basal teleostean phylogeny: Resolving higher-level relationships with longer DNA sequences. Molecular Phylogenetics and Evolution, 2001, 20(2): 275-285 |
Jiang SF, Zhang XP, He YY. Application progress on rapid detection technology of parasites in food. Chinese Journal of Food Hygiene, 2014, 26(1): 95-100 [蒋守富, 张小萍, 何艳燕. 食品寄生虫快速检测技术的应用进展. 中国食品卫生杂志, 2014, 26(1): 95-100] |
Karim A, Iqbal A, Akhtar R, et al. Barcoding of fresh water fishes from Pakistan. DNA Sequence, 2016, 27(4): 2685-2688 |
Khedkar GD, Jamdade R, Naik S, et al. DNA barcodes for the fishes of the Narmada, one of India's longest rivers. PLoS One, 2014, 9(7): e101460 DOI:10.1371/journal.pone.0101460 |
Li DM, Li K, Zhang TQ, et al. Genetic polymorphism analysis of Cyt b and COⅠ gene sequences of Protosalanx hyalocranius in the Hongze Lake. Progress in Fishery Science, 2017, 38(6): 25-31 [李大命, 李康, 张彤晴, 等. 洪泽湖大银鱼(Protosalanx hyalocranius) Cyt b和COⅠ基因序列多态性分析. 渔业科学进展, 2017, 38(6): 25-31] |
Li N, Wang JH, Han CH, et al. Application of COⅠ and Cyt b gene in species identification of pufferfish. Chinese Journal of Food Hygiene, 2018, 30(1): 6-11 [李楠, 王佳慧, 韩春卉, 等. COⅠ及Cyt b基因对河鲀鱼鱼种鉴定的适用性研究. 中国食品卫生杂志, 2018, 30(1): 6-11] |
Lin WF, Hwang DF. A multiplex PCR assay for species identification of raw and cooked bonito. Food Control, 2008, 19(9): 879-885 DOI:10.1016/j.foodcont.2007.08.015 |
Liu S, Xu K, Wu Z, et al. Identification of five highly priced tuna species by quantitative real-time polymerase chain reaction. DNA Sequence, 2016, 27(5): 1-10 |
Liu SF, Li XR, Du TF, et al. DNA barcoding and electronic microarray for common fish species in Shandong coastal waters. Journal of Fishery Sciences of China, 2016, 23(4): 777-790 [柳淑芳, 李献儒, 杜腾飞, 等. 山东近海习见鱼类DNA条形码及其电子芯片分析. 中国水产科学, 2016, 23(4): 777-790] |
Lowenstein JH, Amato G, Kolokotronis SO. The real maccoyii: Identifying tuna sushi with DNA barcodes - Contrasting characteristic attributes and genetic distances. PLoS One, 2009, 4(11): e7866 DOI:10.1371/journal.pone.0007866 |
Lü YC, Zuo T, Tang QJ, et al. Construction and application of DNA barcoding in sea cucumber Holothurians. Journal of Fishery Sciences of China, 2011, 18(4): 782-789 [律迎春, 左涛, 唐庆娟, 等. 海参DNA条形码的构建及应用. 中国水产科学, 2011, 18(4): 782-789] |
Marko PB, Lee SC, Rice AM. Fisheries: Mislabeling of a depleted reef fish. Nature, 2004, 430(6997): 309-310 |
Mo BH, Qu L, Han S, et al. DNA barcoding identificationⅠ. Research progress and applied perspective of DNA barcoding. Sichuan Journal of Zoology, 2008, 27(2): 303-306 [莫帮辉, 屈莉, 韩松, 等. DNA条形码识别Ⅰ. DNA条形码研究进展及应用前景.四川动物, 2008, 27(2): 303-306] |
Neils PG, Haritz A, Francisco A, et al. Molecular identification of Atlantic bluefin tuna (Thunnus thynnus, Scombridae) larvae and development of a DNA character-based identification key for Mediterranean scombrids. PLoS One, 2015, 10(7): e0130407 DOI:10.1371/journal.pone.0130407 |
Krück NC, Tibbetts IR, Ward RD, et al. Multi-gene barcoding to discriminate sibling species within a morphologically difficult fish genus (Sillago). Fisheries Research, 2013, 143: 39-46 DOI:10.1016/j.fishres.2013.01.007 |
Qiu F, Su YQ, Fu MN, et al. Primary research of genetic diversity of the natural stock of Pacific bluefin tuna (Thunnus thynnus). Journal of Xiamen University (Natural Science), 2008(4): 585-590 [邱凡, 苏永全, 傅蒙娜, 等. 太平洋蓝鳍金枪鱼野生群体遗传多样性的初步研究. 厦门大学学报(自然科学版), 2008(4): 585-590] |
Riccioni G, Landi M, Ferrara G, et al. Spatio-temporal population structuring and genetic diversity retention in depleted Atlantic bluefin tuna of the Mediterranean Sea. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(5): 2100-2107 |
Sun C, Liu ZH, Yang AG, et al. Genetic analysis of mitochondrial COⅠ and 16S rRNA gene fragments in four Potamocorbula species. Progress in Fishery Science, 2014, 35(1): 82-90 [孙超, 刘志鸿, 杨爱国, 等. 4种河蓝蛤线粒体COⅠ和16S rRNA基因序列的种间遗传分析. 渔业科学进展, 2014, 35(1): 82-90] |
Sun P, Peng SM, Yin F, et al. Population genetic structure of three Pampus species from East China Sea inferred by COⅠ and Cyt b sequences. Marine Fisheries, 2011, 33(4): 398-404 [孙鹏, 彭士明, 尹飞, 等. 利用COⅠ和Cyt b序列探讨东海区3种鲳属鱼类的种群遗传结构. 海洋渔业, 2011, 33(4): 398-404] |
Page TJ, Hughes JM. Comparing the performance of multiple mitochondrial genes in the analysis of Australian freshwater fishes. Journal of Fish Biology, 2010, 77(9): 2093-2122 DOI:10.1111/j.1095-8649.2010.02821.x |
Vrijenhoek RC. Genetic diversity and fitness in small populations. Conservation Genetics, 1994, 68: 37-53 |
Wang M, Liu W, Huang H, et al. Identifying fish products in Shenzhen through DNA barcoding. Food Science, 2015, 36(20): 247-251 [王敏, 刘荭, 黄海, 等. DNA条形码技术在深圳鱼肉制品鉴定中的应用. 食品科学, 2015, 36(20): 247-251] |
Ward RD, Hanner R, Hebert PDN. The campaign to DNA barcode all fishes, FISH-BOL. Journal of Fish Biology, 2009, 74(2): 329-356 DOI:10.1111/j.1095-8649.2008.02080.x |
Xin CN, Peng JJ, Wang Y, et al. Application of Cyt b gene as a molecular marker in species identification. Chinese Journal of Wildlife, 2009, 30(4): 217-221 [辛翠娜, 彭建军, 王莹, 等. Cyt b分子标记技术在物种鉴定中的应用. 野生动物杂志, 2009, 30(4): 217-221] |
Xu KH, Zhao QL, Liao MT, et al. Research of relationship between texture and sensory evaluation of tuna. Journal of Chinese Institute of Food Science and Technology, 2014, 14(12): 190-197 [徐坤华, 赵巧灵, 廖明涛, 等. 金枪鱼质构特性与感官评价相关性研究. 中国食品学报, 2014, 14(12): 190-197] |



