2. 四川省畜牧科学研究院 成都 610041
2. Animal Science Academy of Sichuan Province, Chengdu 610041
生活污水排放量的剧增、部分工厂未正确处理工业废水等原因使水体中含氮有害物质积累,导致水体氨氮污染,这对水生生物的生存造成威胁(Liu et al, 2004)。因此,水体净化已是养殖业的关注焦点,当前主要提倡微生物脱氮,以微生物作为生态调节剂来维持水生态平衡,不仅效率高、成本低,而且不易造成二次污染(康传磊等, 2018)。
枯草芽孢杆菌(Bacillus subtilis)为革兰氏阳性菌,其抗逆性强、环境兼容性好,优异的蛋白质分泌功能使其成为生产抗生素、药用蛋白和工业酶的重要宿主(Kewcharoen et al, 2019; 王成强等, 2019)。枯草芽孢杆菌因具有多种优良特性而在水产养殖中得到广泛应用。枯草芽孢杆菌HAINUP40可降低模拟废水及养殖废水水体中氨氮含量,是净化水质的一种良好生物制剂(刘树彬等, 2018)。枯草芽孢杆菌对氨的利用途径为谷氨酰胺合成酶/谷氨酸合成酶(Glutamine synthetase/glutamate synthase, GS/GOGAT)途径(Magasanik, 1982)。本研究以枯草芽孢杆菌R47为研究对象,以(NH4)2SO4作为水体氨氮污染来源,模拟富含氨氮污水,采用高通量测序技术建立富含氨氮条件下枯草芽孢杆菌转录组数据库,初步筛选参与枯草芽孢杆菌响应富含氨氮环境的相关基因及通路,为进一步探究其对氨氮应答的分子机制提供数据支撑。
1 材料与方法 1.1 实验菌株枯草芽孢杆菌R47分离于鲫鱼(Carassius auratus)粪便,保存于西南民族大学青藏高原动物遗传资源保护与利用教育部和四川省重点实验室。
1.2 枯草芽孢杆菌R47对氨氮的应答实验将枯草芽孢杆菌R47接种于LB液体培养基,37℃培养,待菌液浓度达1.4×108 CFU/ml时,取10 ml菌液加入100 ml水样中,分别于6、18和24 h取样,根据水质检测试剂盒(杭州陆恒生物科技有限公司)操作指南测定氨氮含量(mg/L)。
1.3 转录组测序分析 1.3.1 测序样品制备及测序以(NH4)2SO4为氨氮污染来源,模拟富含氨氮污水。将枯草芽孢杆菌R47接种于LB液体培养基,37℃培养,待菌液浓度达1.4×108 CFU/ml时,向LB液体培养基中添加(NH4)2SO4,对照组(C)和处理组(T)的(NH4)2SO4浓度分别为0和40 mmol/L,37℃ 180 r/min培养24 h后,4℃ 12000 r/min离心2 min,收集菌体沉淀,–80℃中保存。每个样品3个生物学重复。参照细菌总RNA提取试剂盒(北京天根生化科技有限公司)提取2组细菌总RNA,经NanoDrop ND-1000核酸检测仪(LabTech, 美国)检测RNA的浓度与纯度。基于Illumina二代高通量测序平台(HiSeq 4000)测序,由上海美吉生物医药科技有限公司完成。
1.3.2 转录组数据处理与分析将质控后的原始数据,即Clean data,与参考基因组使用软件Bowtie进行比对,获得用于后续分析的Mapped data,同时,对本次转录组测序的比对结果进行质量评估,Reads在参考基因组不同区域分布以及Reads在不同染色体分布分析,以此获得各样品Reads的比对效率和Reads在基因组上的位置信息。比对参考基因组进行基础功能注释,基于蛋白序列与NR库(Non-Redundant Protein Database)、Swiss-prot库(Swissprotein sequence database)、Pfam数据库、COG数据库(Cluster of Orthologous Groups of proteins)和KEGG数据库(Kyoto Encyclopedia of Genes and Genomes) 6大数据库进行比对,得到相应的功能注释信息,综合NR、Swiss-Prot、KEGG、COG和Pfam数据库的注释结果,选择最佳的Unigene进行分析。利用软件RSEM(RNA-Seq expression estimation by Expectation-Maximization)以TPM为定量指标对基因的表达水平进行定量分析。基于负二项分布的DESeq2软件对Raw counts进行组间表达差异的基因统计分析,标准为对比组样品间表达倍数(Fold Change)≥2和错误发现率(False Discover Rate,FDR) < 0.05 & |log2FC| > 1。采用软件Goatools对差异表达基因进行GO和KEGG Pathway富集分析,预测其可能参与的生物学过程和功能。
1.3.3 small RNA(sRNA)分析采用软件Rockhopper获得sRNA预测结果后,使用Blast及公共数据sRNAMap、sRNATarBase、SIPHT及Rfam资源对鉴定到的sRNA进行注释。采用RNAplex和IntaRNA分别对sRNA靶基因预测,然后对潜在靶基因的功能进行分析。
1.4 实时荧光定量PCR分析使用PrimeScriptTM 1st strand cDNA Sythesis Kit试剂盒(TaKaRa公司)制备cDNA。选择了5个差异表达基因,以高水平、恒定表达的16S rDNA基因为内参基因,设计特异性引物(表 1)。以合成的cDNA为模板,使用TB GreenTM Premix Ex TaqTM Ⅱ试剂盒(TaKaRa公司)进行实时荧光定量分析,检测其mRNA水平的相对表达量,采用2–∆∆Ct法分析结果。Real-Time PCR结果与转录组测序数据结果均以|Log2FC|表示。
|
|
表 1 Real-Time PCR引物 Tab.1 Primers of Real-Time PCR |
枯草芽孢杆菌菌株R47对氨氮的应答实验结果显示,在6 h后水样中氨氮含量从0.8降至0.01 mg/L,18和24 h后,水样中氨氮含量也均维持在0.01 mg/L,这表明枯草芽孢杆菌R47可明显降低氨氮含量,可能存在同化吸收作用。
2.2 转录组测序数据拼接与组装平均每个测序样本得到约1.40×107条reads。质控后碱基错误率(Clean Error Rate)为0.01%,碱基质量值大于20的占99.24%,大于30的占97.55%,GC含量为43.51%。以上结果显示,转录组测序结果质量较高。将各样本比对到参考基因组上的Reads (Mapped Reads)占Clean Reads的百分比均大于80%,表明所选参考基因组组装可以满足信息分析的需求。
2.3 Unigene的功能注释分别基于NR、Swiss-prot、Pfam、COG、GO和KEGG数据库进行总Unigene注释,注释到NR库的基因占比最高,高达96.95%,注释到KEGG库的基因占总基因数最少,为42.51%。6个数据库总计能注释的Unigene数目为4420。
2.4 转录组测序差异表达基因分析转录组测序数据相关性检查结果显示,每组样品间基因表达水平皮尔逊相关系数的平方(R2)均大于0.65,即各组样品之间表达模式的相似度较高,增加了实验的可靠性。将2个组基因表达水平分析中得到的数据采用DESeq2进行分析,结果显示,与对照组相比,处理组共筛选出3918条差异表达基因(Differentially expressed genes, DEGs),其中,包括1887个DEGs上调表达,2031个DEGs下调表达。利用KEGG数据库,可将DEGs按照参与的Pathway通路或行使的功能进行分类,结果发现,表达下调基因主要参与氨基酸代谢、碳水化合物代谢、膜运输过程,上调基因则主要参与碳水化合物代谢、氨基酸代谢与辅助因子和维生素的代谢等过程。
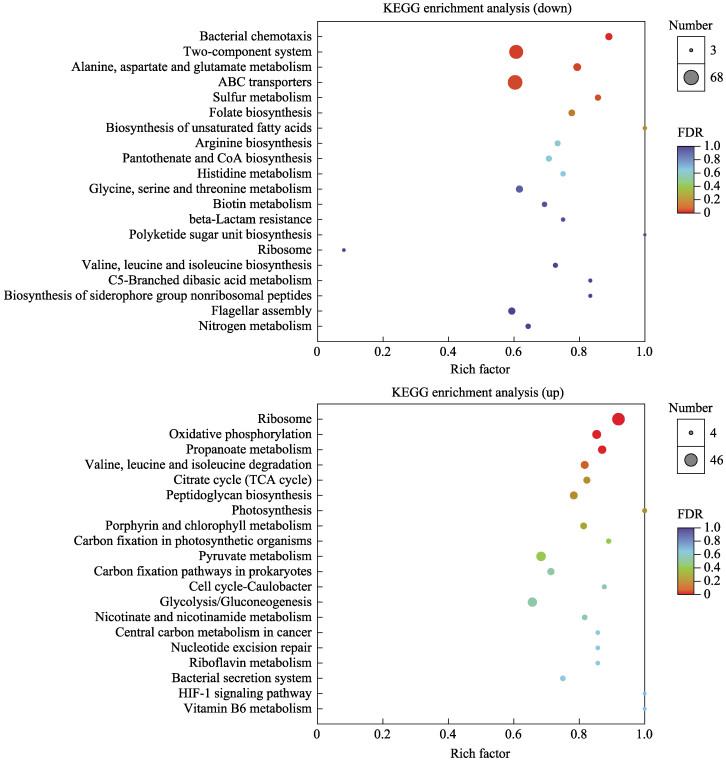
2.5 差异表达基因功能富集分析采用软件Goatools对差异表达基因进行GO富集分析,获得差异表达基因主要具有的GO功能(图 1)。上调基因中对与氨氮代谢功能有关的GO条目及候选基因进行筛选,共得到1个GO条目,包含37个DEGs。下调基因中GO条目及候选基因筛选共得到13个GO条目,包含574个DEGs。将DEGs标注到KEGG数据库中,对基因进行KEGG Pathway富集分析,结果显示,共富集到176个信号通路,上调基因与下调基因共同信号通路有130个。在差异基因KEGG富集结果中,挑选了富集最显著的20条通路在图形中展示(图 2)。在所有KEGG通路中,对参与枯草芽孢杆菌氨氮代谢通路及候选基因筛选,共得到8个信号通路,包含260个DEGs (表 2)。根据对差异表达基因GO与KEGG富集分析中筛选出9个与枯草芽孢杆菌R47对水中氨氮应答的相关基因(表 3)。

|
图 1 差异表达基因GO富集散点图 Fig.1 Scattered plot of GO enrichment of DEGs |
|
图 2 差异表达基因KEGG富集散点图 Fig.2 Scattered plot of KEGG enrichment of DEGs |
|
|
表 2 氨氮代谢相关差异表达基因KEGG富集通路 Tab.2 The enrichment pathway of ammonia nitrogen metabolism related DEGs |
|
|
表 3 氨氮代谢相关差异表达基因 Tab.3 DEGs related ammonia nitrogen metabolism |
去除接头并滤去低质量数据后,获得3348条长度为50~500 nt的sRNA序列,其中,以50~100 nt的序列居多。用Blast及公共数据库sRNA Map、sRNA TarBase、SIPHT及Rfam资源对鉴定到的sRNA进行注释,被注释到的sRNA总数为62。
2.7 sRNA的潜在靶位点及功能分类对sRNA靶基因的分析结果显示,其有3960个对应的潜在靶基因。KEGG数据库将潜在的靶基因分为代谢、环境信息处理等6大类(表 4)。这些潜在靶基因涉及较广的生物学过程及功能,其中主要参与碳水化合物运输和新陈代谢、氨基酸转运和代谢、转录过程等,但由于数据有限,有1285个靶基因功能不明确。根据富集分析所得9个相关基因中,其中,sinR和tnrA分别是sRNA2073和sRNA2182的靶基因。
|
|
表 4 sRNA潜在靶基因的KEGG代谢通路分类统计 Tab.4 Classification of KEGG metabolic pathways of sRNA potential target genes |
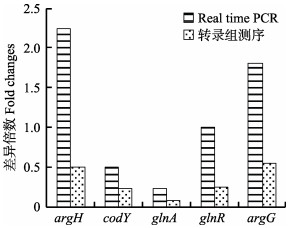
为评估转录组测序结果的可靠性,随机挑选argH、codY、argG、glnA和glnR共5个基因进行Real-time PCR分析。相关性分析结果显示,皮尔逊相关系数为0.947,P < 0.05,Real-time PCR与转录组测序结果具有一致性,表明基于转录组分析差异表达基因的表达结果较为可靠(图 3)。
|
图 3 转录组测序结果的实时定量PCR验证 Fig.3 Verification of RNA-sequence by Real-time PCR |
微生物可参与水环境中的物质代谢,将有机和无机污染物转化为无毒化合物,从而达到改善水质的作用(Hu et al, 2012)。比较来自海水分离的枯草芽孢杆菌、粪产碱杆菌(Alcaligenes faecalis)和绿脓杆菌(Pseudomonas aeruginosa),枯草芽孢杆菌所分泌的酶具有最高的活性,去除污渍能力最强(Marathe et al, 2018)。分离于水产养殖水体及底泥中枯草芽孢杆菌B7具有良好的水质调控作用,对水体中氨氮的去除率大于80%(陈静, 2008)。鲫鱼粪便中分离的枯草芽孢杆菌R47对氨氮具有相似响应,在一定时间内可降低氨氮含量,从而改善水质。
生物体对某一刺激作出反应时,阐明其生物功能涉及的信号通路是至关重要的。本研究在GO富集分析的基础上,进一步开展了KEGG信号通路分析,发现DEGs富集最多的候选信号通路为细菌双组分系统(Two component system, TCS)。TCS在细菌、古细菌中均有发现,是其对环境刺激作出反应的多种信号转导过程。枯草芽孢杆菌通过TCS来应对高浓度氨氮环境,得以在逆境中生存(Galperin et al, 2018; Krell et al, 2010)。epsA、tasA和sinR参与枯草芽孢杆菌对高浓度氨氮的应激反应。TCS可影响细菌的生物膜形成(Plate et al, 2012)。枯草芽孢杆菌生物被膜的胞外基质主要由胞外多糖和胞外蛋白质TasA 2个主要组分组成,分别由epsA-O操纵子和tapA-sipW-tasA操纵子诱导合成(Kolodkin et al, 2010; Nagorska et al, 2010)。SinR是枯草芽孢杆菌生物被膜形成过程中重要的调控蛋白,通过抑制胞外多糖和胞外蛋白TasA的合成,进而抑制生物被膜的形成(Kearns et al, 2005)。差异表达分析结果显示,epsA和tasA均表现出下调趋势,而sinR表现出上调趋势。这可能是因为枯草芽孢杆菌R47正处于高浓度氨氮水体的初级适应阶段。
枯草芽孢杆菌没有谷氨酸脱氢酶活性,通过GS/GOGAT途径同化氨,glnR、glnA、ureABC和tnrA参与此过程。glnR基因编码的蛋白是氮代谢全局调控因子(Global nitrogen regulator, GlnR),一种转录调控因子。枯草芽孢杆菌氮代谢途径的谷氨酰胺合成酶基因glnA受GlnR的调节(杨帆等, 2019),这一观点在本研究也得以证实。glnR位于glnA上游,参与双顺反子glnRA操纵子的组成。具有一个α-螺旋–转角–螺旋结构域的GlnR形成二聚体后,结合到glnRA操纵子上,在氮源丰富时抑制glnA转录,降低谷氨酸合成酶的活性(Brown et al, 1996; Wray et al, 2008),同时,GlnR也能抑制脲酶ureABC操纵子的转录,作为阻遏物阻碍tnrA基因表达(Brandenburg et al, 2002; Randazzo et al, 2017)。差异表达基因分析结果显示,glnA、ureABC和tnrA呈下调趋势,与其保持一致。枯草芽孢杆菌对氨氮的应答是一个复杂反应,多个信号通路相互协调完成。本研究仅对枯草芽孢杆菌R47的双组分系统与部分基因进行了探讨,还需深入开展研究。
sRNA介导的转录后基因调控是生物体的一种新型基因调控机制,它在有机体适应环境、调节生命活动等生物过程中有极为重要的作用。氮胁迫诱导RNA1和RNA4可调节念珠藻(Nostocales)和蓝细菌(Cyanobacteria)对氮的可利用性(Álvarez-Escribano et al, 2018; Klähn et al, 2018)。Gaimster等(2019)发现,副球菌(Paracoccus denitrificans) sRNA29的过表达下调亚硝酸盐还原酶并限制细胞产生NO和N2O。本研究对样本sRNA的靶基因进行预测分析,sRNA2073和sRNA2182所介导的调控通路可能是枯草芽孢杆菌R47进行氮代谢的重要辅助。
本研究利用Illumina二代高通量测序技术对在富含氨氮环境中枯草芽孢杆菌R47进行测序,并初步分析发现细菌双组分系统和epsA、tasA、sinR、glnR、glnA、tnrA、ureABC等基因可能参与枯草芽孢杆菌R47对氨氮应答过程,同时进行了sRNA分析,为后续深入探讨枯草芽孢杆菌的脱氮分子机理提供数据支撑。
Álvarez-Escribano I, Vioque A, Muro-Pastor AM. NsrR1, a nitrogen stress-repressed sRNA, contributes to the regulation of nblA in Nostoc sp. PCC 7120. Frontiers in Microbiology, 2018, 9(1): 2267 |
Brandenburg JL, Wray LV, Beier L, et al. Roles of PucR, GlnR, and TnrA in regulating expression of the Bacillus subtilis ure P3 promoter. Journal of Bacteriology, 2002, 184(21): 6060-6064 DOI:10.1128/JB.184.21.6060-6064.2002 |
Brown SW, Sonenshein AL. Autogenous regulation of the Bacillus subtilis glnRA operon. Journal of Bacteriology, 1996, 178(8): 2450-2454 DOI:10.1128/JB.178.8.2450-2454.1996 |
Chen J, Xu HY, Gu W. Bacillus subtilis B7: Isolation and function in water purification. Hebei Fisheries, 2008, 11061106(11): 10-11, 29 [陈静, 徐海燕, 谷巍. 枯草芽孢杆菌B7的分离和净化水质的初步研究. 河北渔业, 2008, 11061106(11): 10-11, 29 DOI:10.3969/j.issn.1004-6755.2008.11.004] |
Commichau FM, Gunka K, Landmann JJ, et al. Glutamate metabolism in Bacillus subtilis: Gene expression and enzyme activities evolved to avoid futile cycles and to allow rapid responses to perturbations of the system. Journal of Bacteriology, 2008, 190(10): 3557-3564 DOI:10.1128/JB.00099-08 |
Gaimster H, Hews CL, Griffiths R, et al. A central small RNA regulatory circuit controlling bacterial denitrification and N2O emissions. mBio, 2019, 10(4): e01165-19 |
Galperin MY, Makarova KS, Wolf YI, et al. Phyletic distribution and lineage-specific domain architectures of archaeal two-component signal transduction systems. Journal of Bacteriology, 2018, 200(7): e00681-17 |
Hu M, Wang X, Wen X, et al. Microbial community structures in different wastewater treatment plants as revealed by 454. pyrosequencing analysis. Bioresource Technology, 2012, 117: 72-79 DOI:10.1016/j.biortech.2012.04.061 |
Kang CL, Li QF, Zhang Y, et al. Purifying effect of three heterotrophic nitrification-aerobic denitrification bacteria strains on the farming water of Verasper variegates. Progress in Fishery Sciences, 2018, 39(2): 42-48 [康传磊, 李秋芬, 张艳, 等. 三株异养硝化-好氧反硝化细菌对圆斑星鲽养殖水质的净化效果. 渔业科学进展, 2018, 39(2): 42-48] |
Kearns DB, Chu F, Branda SS, et al. A master regulator for biofilm formation by Bacillus subtilis. Molecular Microbiology, 2005, 55(3): 739-749 |
Kewcharoen W, Srisapoome P. Probiotic effects of Bacillus spp. from Pacific white shrimp (Litopenaeus vannamei) on water quality and shrimp growth, immune responses, and resistance to Vibrio parahaemolyticus (AHPND strains). Fish and Shellfish Immunology, 2019, 94: 175-189 |
Klähn S, Bolay P, Wright PR, et al. A glutamine riboswitch is a key element for the regulation of glutamine synthetase in cyanobacteria. Nucleic Acids Research, 2018, 46(19): 10082-10094 |
Kolodkin-Gal I, Romero D, Cao S, et al. D-amino acids trigger biofilm disassembly. Science, 2010, 328(5978): 627-629 DOI:10.1126/science.1188628 |
Krell T, Lacal J, Busch A, et al. Bacterial sensor kinases: Diversity in the recognition of environmental signals. Annual Review of Microbiology, 2010, 64(1): 539-5593 DOI:10.1146/annurev.micro.112408.134054 |
Liu CH, Chen JC. Effect of ammonia on the immune response of white shrimp Litopenaeus vannamei and its susceptibility to Virio alginolyticus. Fish and Shellfish Immunology, 2004, 16(3): 321-334 DOI:10.1016/S1050-4648(03)00113-X |
Liu SB, Wang XR, Lin ZQ, et al. Purification of aquaculture effluent by Bacillus subtilis HAINUP40. Journal of Fisheries Science, 2018, 37(2): 159-166 [刘树彬, 王新锐, 林壮其, 等. 枯草芽孢杆菌HAINUP40水质净化作用的研究. 水产科学, 2018, 37(2): 17-24] |
Magasanik B. Genetic control of nitrogen assimilation in bacteria. Annual Review of Genetics, 1982, 16: 135-168 DOI:10.1146/annurev.ge.16.120182.001031 |
Marathe SK, Vashistht MA, Prashanth A, et al. Isolation, partial purification, biochemical characterization and detergent compatibility of alkaline protease produced by Bacillus subtilis, Alcaligenes faecalis and Pseudomonas aeruginosa obtained from sea water samples. Journal of Genetic Engineering and Biotechnology, 2018, 16(1): 39-46 DOI:10.1016/j.jgeb.2017.10.001 |
Nagorska K, Ostrowski A, Hinc K, et al. Importance of EPS genes from Bacillus subtilis in biofilm formation and swarming. Journal of Applied Genetics, 2010, 51(3): 369-381 DOI:10.1007/BF03208867 |
Plate L, Marletta MA. Nitric oxide modulates bacterial biofilm formation through a multicomponent cyclic-di-GMP signaling network. Molecular Cell, 2012, 46(4): 449-460 DOI:10.1016/j.molcel.2012.03.023 |
Randazzo P, Aucouturier A, Delumeau O, et al. Revisiting the in vivo GlnR-binding sites at the genome scale in Bacillus subtilis. BMC Research Notes, 2017, 10(1): 422 DOI:10.1186/s13104-017-2703-9 |
Wang CQ, Li BS, Wang JY, et al. Effects of dietary Bacillus subtilis and yeast culture on growth, serum biochemical indices and antioxidant capacity of juvenile hybrid grouper (Epinephelus fuscoguttatus♀×E.lanceolatus♂).. Progress in Fishery Sciences, 2019, 40(4): 47-56 [王成强, 李宝山, 王际英, 等. 饲料中添加枯草芽孢杆菌和酵母培养物对珍珠龙胆石斑鱼幼鱼生长、血清生化指标及抗氧化能力的影响. 渔业科学进展, 2019, 40(4): 47-56] |
Wray LV, Fisher SH. Bacillus subtilis GlnR contains an autoinhibitory C-terminal domain required for the interaction with glutamine synthetase. Molecular Microbiology, 2008, 68(2): 277-285 DOI:10.1111/j.1365-2958.2008.06162.x |
Yang F, Qin JX, Peng Q, et al. Transcriptional regulation and overexpression of glnA gene in Bacillus thuringiensis. Chinese Journal of Biological Control, 2019, 35(4): 563-569 [杨帆, 秦嘉欣, 彭琦, 等. 苏云金芽胞杆菌谷氨酰胺合成酶基因glnA的转录调控和过表达. 中国生物防治学报, 2019, 35(4): 563-569] |



