 生长性状全基因组关联分析
生长性状全基因组关联分析
2. 上海海洋大学水产与生命学院 上海 201306
 (Seriola lalandi)个体进行测序,共获得黄条
(Seriola lalandi)个体进行测序,共获得黄条 SNP分子标记26665个,对黄条
SNP分子标记26665个,对黄条 个体的体质量和全长这2个重要生长性状进行全基因组关联分析,筛选与体质量和全长性状相关的SNP位点和候选基因。结果显示,黄条
个体的体质量和全长这2个重要生长性状进行全基因组关联分析,筛选与体质量和全长性状相关的SNP位点和候选基因。结果显示,黄条 体质量性状中共筛选到17个体质量显著关联的SNP位点,找到17个可能的候选基因,全长性状共筛选到12个潜在显著关联位点,找到12个可能的候选基因。利用KEGG数据库对可能的候选基因进行Pathway分析,得知候选基因主要参与了细胞或组织生长发育相关的代谢通路调控过程,可能是影响黄条
体质量性状中共筛选到17个体质量显著关联的SNP位点,找到17个可能的候选基因,全长性状共筛选到12个潜在显著关联位点,找到12个可能的候选基因。利用KEGG数据库对可能的候选基因进行Pathway分析,得知候选基因主要参与了细胞或组织生长发育相关的代谢通路调控过程,可能是影响黄条 生长性状密切相关的重要候选SNP位点和功能基因,结果可为今后黄条
生长性状密切相关的重要候选SNP位点和功能基因,结果可为今后黄条 种质资源可持续利用和育种提供遗传信息资料积累。
种质资源可持续利用和育种提供遗传信息资料积累。 生长性状 全基因组关联分析 2b-RAD 简化基因组
生长性状 全基因组关联分析 2b-RAD 简化基因组 2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
全基因组关联分析(Genome-wide association study, GWAS)是应用全基因组范围内的大量分子标记(一般为SNP),将标记基因型结合性状表型进行联合分析,统计每个标记与目标性状之间的关联性大小(一般用P值表示),鉴定出与目标性状密切相关且具有特定功能和育种潜力的基因位点或分子标记,主要用于物种经济性状相关SNP分子标记以及功能基因的鉴定,从而达到缩短育种周期和提高育种效率的目的,目前已在畜禽等脊椎动物育种中广泛应用(Tavares et al, 2020; Müller et al, 2019; Cui et al, 2016; Zhang et al, 2019)。近年来,随着基因组高通量测序技术的发展及测序成本的降低,GWAS开始应用于水产养殖动物的育种研究,如在大黄鱼(Larimichthys crocea)、鲶鱼(Silurus asotus)、凡纳滨对虾(Litopeneaus vannamei)、龙胆石斑鱼(Epinephelus lanceolatus)、虾夷扇贝(Patinopecten yessoensis)等物种的生长性状关联SNP位点、候选基因的挖掘和鉴定(Zhou et al, 2019; Li et al, 2017; Yu et al, 2019; Wu et al, 2019; Ning et al, 2019)方面应用并取得了一定进展。但是,与陆生脊椎动物相比,GWAS在水产动物育种中的应用尚处于起步阶段。
黄条












选择来自我国黄海种群养殖黄条
使用QIAGEN公司生产的动物基因组DNA提取试剂盒(DP121221),参照试剂盒使用说明,提取鳍条基因组DNA。用NanoDrop 2000分光光度计(Thermo, 美国)测定基因组DNA浓度,通过1%琼脂糖凝胶电泳检测DNA的完整性,通过A260 nm/A280 nm的比值来判断DNA的质量。将质检合格的DNA浓度稀释至100 ng/µl,于–20℃条件保存备用。
1.3 文库构建与测序将≥200 ng的各样品基因组DNA采用IIB型限制性内切酶BsaXI进行酶切,酶切产物分别加入5组不同的接头,使用T4 DNA Ligase连接,然后PCR扩增连接产物,最后根据5组接头信息,将5个标签按顺序串联,连接产物添加barcode序列,混库,使用Illumina Hiseq测序平台对混合好的文库进行Paired-end测序。
1.4 数据分析 1.4.1 表型数据分析使用R语言中fivenum()函数,对黄条
Illumina HiSeq测序平台得到的原始图像数据文件经碱基识别转化为Raw Reads,过滤删除含有接头序列的Reads,得到Clean Reads,过滤删除含有N碱基比例大于8%的Reads,过滤删除低质量Reads(质量值低于Q30的碱基超过15%);利用Pear (Zhang et al, 2014)软件(V0.9.6)将成对的Clean Reads拼接,提取出各样品对应的Reads,过滤删除不含酶切识别位点的Reads后,得到各样品的Enzyme Reads;利用电子酶切从参考基因组中提取含有酶切识别位点的标签,作为参考序列,利用SOAP软件将各样品的Enzyme Reads比对到参考序列上,主要参数为-r0–M4–v2 (-r0指唯一比对;–M4指最优比对;–v2指比对允许2个错配),对比对到相同标签的reads聚类,得到unique标签深度,选择样品深度 > 3×且深度 < 500、标签长度为27 bp的标签,利用SOAP软件(V 2.21) (Li et al, 2008)将测序数据比对到参考序列,利用最大似然法(ML)进行位点的分型(Fu et al, 2013),过程中使用的RAD分型软件包(RAD typing),包含10余个软件组分,覆盖了从数据预处理至最终分型结果输出的全过程。
1.4.3 全基因组关联分析使用EMMA eXpedited (EMMAX)高效混合模型(Kang et al, 2010),通过方差分量方法进行SNP分子标记和表型性状的全基因组关联分析,所用模型:
| $y = Xb + Ga + e$ |
式中,y为表型值;X为固定效应关联矩阵,b为固定效应向量,G为通过SNP标记计算得到的关系矩阵,a为随机加性遗传方差的参数,e为剩余效应的向量。
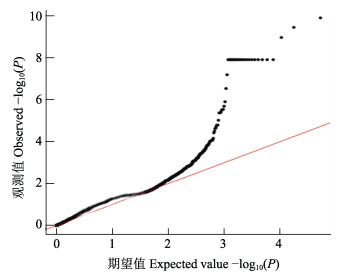
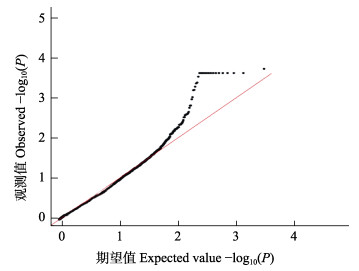
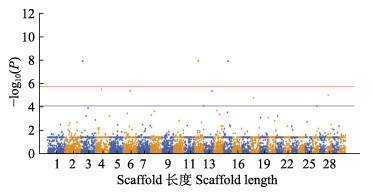
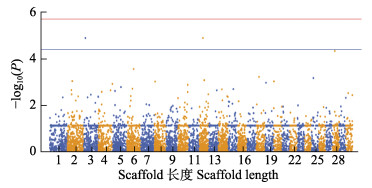
每个SNP位点能得到1个关联值P。对GWAS给出的P值划定2条显著性水平线,其中1条经Bonferroni校正P=0.05/N来确定全基因组显著性阈值(Bonferroni, 1936),N为SNP标记的个数,2个性状经Bonferroni校正后显著关联阈值–lgP=5.726;另一条使用R软件包中的p.adjust()函数计算得到经FDR校正后的阈值,体质量性状潜在显著关联阈值–lgP= 4.091,全长性状潜在显著关联阈值–lgP=4.413,挑选Scaffold长度的前30使用R软件包的qqman绘制曼哈顿图,绘制QQ图对关联分析进行评价,判断关联分析结果是否可靠。
1.5 候选基因鉴定及功能分析将筛选到的具有关联性的SNP位点上下游1 Mb范围内的碱基序列与GenBank数据库中已有的黄条 参考基因组(https://www.ncbi.nlm.nih.gov/genome/? term=Seriola+lalandi+dorsalis)进行序列比对,使用SnpEff软件(Version 4.3T) (Cingolani et al, 2012)对得到的SNP位点进行注释,以确定SNP位点在基因元件的位置、对氨基酸的变化影响等找到距离SNP位点最近的基因。将所有关联基因与KEGG数据库比对,进行Pathway分析,并用超几何分布检验的方法计算每个Pathway条目中基因富集的显著性,公式如下:
参考基因组(https://www.ncbi.nlm.nih.gov/genome/? term=Seriola+lalandi+dorsalis)进行序列比对,使用SnpEff软件(Version 4.3T) (Cingolani et al, 2012)对得到的SNP位点进行注释,以确定SNP位点在基因元件的位置、对氨基酸的变化影响等找到距离SNP位点最近的基因。将所有关联基因与KEGG数据库比对,进行Pathway分析,并用超几何分布检验的方法计算每个Pathway条目中基因富集的显著性,公式如下:
| $p = 1 - \mathop \sum \limits_{i = 0}^{m - 1} \frac{{\left({\begin{array}{*{20}{c}} M \\ i \end{array}} \right)\left({\begin{array}{*{20}{c}} {N - M} \\ {n - i} \end{array}} \right)}}{{\left({\begin{array}{*{20}{c}} N \\ n \end{array}} \right)}}$ |
式中,N为所有基因中具有KEGG注释的基因数目,n为N中差异表达基因中具有的KEGG注释的基因数目,M为所有基因中注释为某特定KEGG的基因数目,m为注释某特定KEGG的差异表达基因的数目。计算的结果会返回一个富集显著性的P值,小的P值表示基因在该Pathway中出现富集,当P≤0.05表示显著富集。
2 结果 2.1 表型性状描述性统计根据本研究所用黄条

|
|
表 1 黄条 |
对2b-RAD简化基因组测序数据按照以下指标进一步过滤。剔除所有样品中低于80%个体可以分型的位点;剔除MAF低于0.05的位点,剔除等位基因大于2的位点。最终,测序获得26665个SNP位点进行GWAS分析。
2.3 全基因组关联分析利用R软件包分别绘制黄条

|
图 1 黄条 |
|
图 2 黄条 |
|
图 3 黄条 蓝色实线代表潜在显著关联阈值: –log10P=4.091 The red solid line indicates the genome wide significant threshold: –log10P=5.726. The blue solid line indicates the threshold for the significance of "suggestive association": –log10P=4.091 |
|
图 4 黄条 蓝色实线代表潜在显著关联阈值–log10P=4.413 The red solid line indicates the genome wide significant threshold: –log10P=5.726. The blue solid line indicates the threshold for the significance of "suggestive association": –log10P=4.413 |
|
|
表 2 黄条 |
|
|
表 3 黄条 |
本研究共鉴定黄条
|
|
表 4 体质量性状和全长性状KEGG注释结果 Tab.4 KEGG annotation results of body weight and total length trait |
黄条











GWAS依赖于连锁不平衡(Linkage Disequilibrium, LD)检测目标物种群体的遗传变异与性状之间的关联,然后通过统计基因型和表型的关联性大小筛选出影响显著的遗传变异,定位影响表型性状的重要数量性状位点(QTL)和候选基因,确定其遗传机制(陶林等, 2019; Naha et al, 2016)。本研究根据鉴定出与体质量和全长性状关联的SNP位点,在每个SNP上、下游1 Mb序列范围内扫描,共挖掘到17个体质量性状显著关联基因、12个全长性状潜在显著关联基因,但这些基因尚未在黄条


本研究发现了29个与黄条

Bonferroni CE. Teoria statistical delle classi e calcolo delle probabilita. Pubblicazioni del R Istituto Superiore di Scienze Economiche e Commericiali di Firenze, 1936, 8: 3-62 |
Chen SL, Xu WT, Liu Y. Fish genomic research: Decade review and prospect. Journal of Fisheries of China, 2019, 43(1): 1-14 [陈松林, 徐文腾, 刘洋. 鱼类基因组研究十年回顾与展望. 水产学报, 2019, 43(1): 1-14] |
Chen ZD, Wang WH. Genome-wide association study on feet weight in chicken (Gallus gallus). Journal of Agricultural Biotechnology, 2016, 24(10): 1569-1577 [陈则东, 王文浩. 鸡脚重性状的全基因组关联分析. 农业生物技术学报, 2016, 24(10): 1569-1577] |
Cingolani P, Platts A, Wang LL, et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly, 2012, 6(2): 80-92 DOI:10.4161/fly.19695 |
Cui ZH, Luo JD, Qi CY, et al. Genome-wide association study (GWAS) reveals the genetic architecture of four husk traits in maize. BMC Genomics, 2016, 17(1): 946 DOI:10.1186/s12864-016-3229-6 |
Fu X, Dou J, Mao J, et al. RAD typing: An integrated package for accurate de novo codominant and dominant RAD genotyping in mapping populations. PLoS One, 2013, 8(11): e79960 DOI:10.1371/journal.pone.0079960 |
Johannessen M, Moller S, Hansen T, et al. The multifunctional roles of the four-and-a-half-LIM only protein FHL2. Cellular and Molecular Life Sciences, 2006, 63(3): 268-284 DOI:10.1007/s00018-005-5438-z |
Kang HM, Sul JH, Service SK, et al. Variance component model to account for sample structure in genome-wide association studies. Nature Genetics, 2010, 42(4): 348-354 DOI:10.1038/ng.548 |
Li N, Zhou T, Geng X, et al. Identification of novel genes significantly affecting growth in catfish through GWAS analysis. Molecular Genetics and Genomics, 2017, 293(3): 1-13 |
Li R, Li Y, Kristiansen K, et al. SOAP: Short oligonucleotide alignment program. Bioinformatics, 2008, 24(5): 713-714 DOI:10.1093/bioinformatics/btn025 |
Li R, Xu YJ, Liu XZ, et al. Morphometric analysis and internal anatomy of yellowtail kingfish (Seriola aureovittata). Progress in Fishery Sciences, 2017, 38(1): 142-149 [李荣, 徐永江, 柳学周, 等. 黄条  (Seriola aureovittata)形态度量与内部结构特征. 渔业科学进展, 2017, 38(1): 142-149] (Seriola aureovittata)形态度量与内部结构特征. 渔业科学进展, 2017, 38(1): 142-149] |
Liu YS, Liu XZ, Shi B, et al. Analysis of the banding patterns of Seriola aureovittata. Journal of Fisheries of China, 2018, 42(9): 138-147 [刘永山, 柳学周, 史宝, 等. 黄条  染色体多种显带的形态特征分析. 水产学报, 2018, 42(9): 138-147] 染色体多种显带的形态特征分析. 水产学报, 2018, 42(9): 138-147] |
Matthias C, Edwige T, Bernadette B, et al. FHL2 interacts with both ADAM-17 and the cytoskeleton and regulates ADAM- 17 localization and activity. Journal of Cellular Physiology, 2006, 208: 363-372 DOI:10.1002/jcp.20671 |
Müller BSF, de Almeida Filho JE, Lima BM, et al. Independent and joint-GWAS for growth traits in Eucalyptus by assembling genome-wide data for 3373 individuals across four breeding populations. New Phytologist, 2019, 221(2): 818-833 DOI:10.1111/nph.15449 |
Naha BC, Prasad A, Sailo L, et al. Concept of genome wide association studies and its progress in livestock. International Journal of Science and Nature, 2016, 7(1): 39-42 |
Nguyen NH, Premachandra HKA, Kilian A, et al. Genomic prediction using DArT-Seq technology for yellowtail kingfish Seriola lalandi. BMC Genomics, 2018a, 19(1): 107 DOI:10.1186/s12864-018-4493-4 |
Nguyen NH, Rastas PMA, Premachandra HKA, et al. First high- density linkage map and single nucleotide polymorphisms significantly associated with traits of economic importance in yellowtail kingfish Seriola lalandi. Frontiers in Genetics, 2018b, 9: 127 DOI:10.3389/fgene.2018.00127 |
Ning XH, Li X, Wang J, et al. Genome-wide association study reveals E2F3 as a candidate gene for scallop growth. Aquaculture, 2019, 73(4): 734216 |
Ohara E, Nishimura T, Nagakura Y, et al. Genetic linkage maps of two yellowtails (Seriola quinqueradiata and Seriola lalandi). Aquaculture, 2005, 244: 41-48 DOI:10.1016/j.aquaculture.2004.10.022 |
Pi X, Ren R, Kelley R, et al. Sequential roles for myosin-X in BMP6 dependent filopodial extension, migration, and activation of BMP receptors. Journal of Cell Biology, 2008, 179(7): 1569-1582 |
Premachandra HKA, De la Cruz FL, Takeuchi Y, et al. Genomic DNA variation confirmed Seriola lalandi comprises three different populations in the Pacific, but with recent divergence. Scientific Reports, 2017, 7(1): 9386 DOI:10.1038/s41598-017-07419-x |
Raise A, Stefanie W, Ralf J, et al. Hunting for the function of orphan GPCRs-beyond the search for the endogenous ligand. British Journal of Pharmacology, 2015, 172(13): 3218-3228 |
Sepulveda FA, Gonzalez M. Spatio-temporal patterns of genetic variations in populations of yellowtail kingfish Seriola lalandi from the southeastern Pacific Ocean and potential implications for its fishery management. Journal of Fish Biology,, 2017, 90(1): 249-264 DOI:10.1111/jfb.13179 |
Shi B, Liu YS, Liu XZ, et al. Study on the karyotype of yellowtail kingfish (Seriola aureovittata). Progress in Fishery Sciences, 2017, 38(1): 139-144 [史宝, 刘永山, 柳学周, 等. 黄条  (Seriola aureovittata)染色体核型分析. 渔业科学进展, 2017, 38(1): 136-144] (Seriola aureovittata)染色体核型分析. 渔业科学进展, 2017, 38(1): 136-144] |
Sicuro B, Luzzana U. The state of Seriola spp. other than yellowtail (S. quinqueradiata) farming in the world. Reviews in Fisheries Science and Aquaculture, 2016, 24(4): 314-325 DOI:10.1080/23308249.2016.1187583 |
Swart BL, Merwe BVD, Kerwath SE, et al. Phylogeography of the pelagic fish Seriola lalandi at different scales: Confirmation of inter-ocean population structure and evaluation of southern African genetic diversity. South African Journal of Marine Science, 2016, 38(4): 513-524 DOI:10.2989/1814232X.2016.1238410 |
Symonds JE, Walker SP, Pether S, et al. Developing yellowtail kingfish (Seriola lalandi) and hāpuku (Polyprion oxygeneios) for New Zealand aquaculture. New Zealand Journal of Marine and Freshwater Research, 2014, 48(3): 371-384 DOI:10.1080/00288330.2014.930050 |
Tao L, He XY, Di R, et al. Research progress on genome-wide association study for growth-related traits in livestock and poultry. Chinese Journal of Animal Science, 2019, 55(11): 34-41 [陶林, 贺小云, 荻冉, 等. 畜禽生长发育相关性状的全基因组关联分析研究进展. 中国畜牧杂志, 2019, 55(11): 34-41] |
Tavares V, Pinto R, Assis J, et al. Venous thromboembolism GWAS reported genetic makeup and the hallmarks of cancer: Linkage to ovarian tumour behavior. Biochimica et Biophysica Acta - Reviews on Cancer, 2020, 1873(1): 188331 DOI:10.1016/j.bbcan.2019.188331 |
Wang B, Xu Y, Liu X, et al. Molecular characterization and expression profiles of insulin-like growth factors in yellowtail kingfish (Seriola lalandi) during embryonic development. Fish Physiology and Biochemistry, 2019, 45(1): 375-390 DOI:10.1007/s10695-018-0570-5 |
Whatmore P, Nguyen NH, Miller A, et al. Genetic parameters for economically important traits in yellowtail kingfish Seriola lalandi. Aquaculture, 2013, 400(25): 77-84 |
Woolner S, O'Brien LL, Wiese C, et al. Myosin-10 and actin filaments are essential for mitotic spindle function. Journal of Cell Biology, 2008, 182(1): 77-88 DOI:10.1083/jcb.200804062 |
Wu LN, Yang Y, Li BJ, et al. First genome-wide association analysis for growth traits in the largest coral reef-dwelling bony fishes, the giant grouper (Epinephelus lanceolatus). Marine Biotechnology, 2019, 21(5): 707-717 DOI:10.1007/s10126-019-09916-8 |
Yu Y, Wang QC, Zhang Q. Genome scan for genomic regions and genes associated with growth trait in pacific white shrimp Litopeneaus vannamei. Marine Biotechnology, 2019, 21(3): 374-383 DOI:10.1007/s10126-019-09887-w |
Zhang J, Kobert K, Flouri T, et al. PEAR: A fast and accurate Illumina Paired-End reAd mergeR. Bioinformatics, 2014, 30(5): 614-620 DOI:10.1093/bioinformatics/btt593 |
Zhang YF, Zhang JJ, Gong HF, et al. Genetic correlation of fatty acid composition with growth, carcass, fat deposition and meat quality traits based on GWAS data in six pig populations. Meat Science, 2019, 150: 47-55 DOI:10.1016/j.meatsci.2018.12.008 |
Zhou Z, Han K, Wu Y, et al. Genome-wide association study of growth and body-shape-related traits in large yellow croaker (Larimichthys crocea) using ddRAD sequencing. Marine Biotechnology, 2019, 21(5): 655-670 DOI:10.1007/s10126-019-09910-0 |



