2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室 山东省渔业资源与生态环境重点实验室 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Shandong Provincial Key Laboratory of Fishery Resources and Eco-Environment, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071)
准确地掌握目标物种的分布信息是生物多样性研究的必要前提之一,且对生物地理学、保护生物学和生态学等学科领域的研究也具有重要意义(Lodge et al, 2012, Ficetola et al, 2008)。与陆地环境不同,受到水深、温度、光照、压力等诸多因素限制,对水生生物进行直接观察和识别难度很大(Bogich et al, 2008),特别是某些低密度或具有独特生活史的种群(Dejean et al, 2011)。目前,传统调查方法,例如拖网、影像和声学调查等,通常成本高昂且会对珍稀物种造成巨大威胁(Murphy et al, 2010)。随着分子生物学研究的不断发展,环境DNA (environmental DNA, eDNA)技术的出现使得“非侵入”式资源调查和物种跟踪成为可能(Taberlet et al, 2012)。该技术是通过采集并检测生物体向水环境中释放的DNA片段来判断生物是否存在于该水域,并进行生物量评估的一种新兴技术(Ficetola et al, 2008; Takahara et al, 2012)。eDNA的来源广泛,可以是生物的表皮、黏液、粪便、尿液和生殖细胞等(Turner et al, 2015; Klymus et al, 2015; Wilcox et al, 2015),并且只需通过少量水样的采集分析即可满足调查目的(Thomsen et al, 2012a、b),从而降低采样成本,减少采样工作对环境和生物体的干扰和破坏。eDNA技术很大程度地弥补了传统调查方法的不足,在物种保护和生物量评估等方面发挥重要作用。
已有学者对eDNA在河流中的扩散或降解进行研究(Deiner et al, 2014; Jane et al, 2015; Sansom et al, 2017),发现水体中的eDNA在自然条件下易降解,且会在环境中扩散,以短片段的形式移动到远离释放源的位置。此外,仅依靠分析eDNA本身无法就其源头及释放时间进一步溯源。然而,对于在面积、水流及深度相比河流都更为复杂的海洋环境中,eDNA的分布规律及变化研究仍旧十分有限(Yamamoto et al, 2016; O’Donnell et al, 2017)。了解eDNA在水体中扩散和滞留的规律对于采样设计和空间推断(即样本所代表的可能出现的目标生物的位置)具有重要意义(Shogren et al, 2017)。对水体中eDNA变化规律的阐释是利用其进行目标物种追踪及生物量评估的前提。一般而言,利用eDNA技术对目标物种进行追踪是为获取其准确的分布位置和种群信息。而eDNA在水体中的分布受多种因素影响,包括物种分布、水流、沉降和沉积物再悬浮等(Deiner et al, 2014; Robison et al, 1981; Saba et al, 2012; Wotton et al, 2001; Maggi, 2013)。这些除物种分布以外的其他因素的影响所导致eDNA的杂乱分布是研究者不想看到的。如何避免这些因素影响,从而通过eDNA获取有效的目标物种信息是研究者一直以来想要解决的问题。而目前已有研究对eDNA的水平分布讨论较多(Thalinger et al, 2019),很少涉及垂直分布的特点,或只涉及了单一介质(Turner et al, 2015)。本研究选用中国对虾( Fenneropenaeus chinensis)作为目标物种,选择平均水深较大的黄海作为研究水域,以eDNA垂直分布作为主要考察要素。鉴于沉积物也会对水体中eDNA浓度造成一定程度的影响(Turner et al, 2015; 魏楠等, 2020)。将水体与沉积物的相互效应也考虑在内,希望能更加科学合理地解释eDNA的分布特点。本研究分为两部分内容:(1) 在黄海中南部海域8个站位的不同水深(表、中、底)采水,研究水体中中国对虾eDNA的分布规律;(2) 采集表层沉积物并进行室内实验,分析沉积物对水体的影响方式。通过本研究希望能够了解中国对虾eDNA在水中的分布规律和影响因素,更合理地设计采样工作以及更有效地将eDNA技术推广到其他物种的研究中。
1 材料与方法 1.1 采样站位样品来源于中国水产科学研究院黄海水产研究所蓝海101号科研调查船于2019年12月21日在黄海的调查。根据邓景耀等(1990)对中国对虾的研究方法,在中国对虾越冬洄游路线上,选取8个站位,利用分层采水方法在各站位分别采集表、中、底3个水层水样。其中,表层水在距海面3 m深的位置取水,中层水在20 m左右水深采水,底层水在该站位水深于海底以上5 m左右位置取水(具体采样深度根据海况及作业难度有所变动),具体采样位置见图 1,采样水深信息见表 1。

|
图 1 2019年12月黄海采样站位 Fig.1 Sampling stations in the Yellow Sea in Dec. 2019 |
|
|
表 1 各站位采样水深 Tab.1 Station location and sampling depth |
选择中国对虾的线粒体细胞色素C氧化亚基Ⅰ基因(mtDNA COⅠ)进行研究。引物参照李苗等(2019),其中,COI PF与COI PR作为普通PCR引物用来制备质粒标准品DNA。COI DF与COI DR作为定量PCR引物,扩增的目的片段为普通PCR引物所扩增片段的一部分。引物与探针由生工生物工程(上海)股份有限公司合成。
1.3 中国对虾eDNA富集使用滤膜法对海水中eDNA进行富集。选取直径为47 mm、孔径为0.45 μm的玻璃纤维滤膜对1 L所采水样过滤(李苗等, 2019),每个水样取3个平行样本及3个阴性对照,共计75个样本。为避免样品相互污染,过滤完后每张滤膜用锡纸单独包裹再放入封口袋中并做好标记,–20℃保存直到进行DNA提取。
1.4 eDNA提取使用试剂盒进行eDNA提取,具体提取方法参照Renshaw等(2015)并加以改进,其具体提取步骤为:
(1) 将滤膜从冰箱取出后,用剪刀去除滤膜边缘多余部分,再剪成条状,置于2 ml无菌离心管中;向离心管内加入570 μl的Buffer ATL和60 μl的蛋白酶K溶液(20 mg/ml),涡旋震荡,将混合液混合均匀,在恒温水浴锅内65℃水浴3 h,水浴期间,每隔15 min轻轻颠倒混匀离心管,使其充分裂解,裂解完成后取出并挤干剩余滤膜;
(2) 向离心管内加入630 μl的Buffer AL和630 μl的无水乙醇,涡旋震荡15 s,使其混合均匀;将2 ml离心管中的混合液转移到DNeasy离心柱中,25℃ 8000 g离心1 min;
|
|
表 2 本研究用到的引物与探针 Tab.2 Primers and probes used in this study |
(3) 向离心柱内加入500 μl Buffer AW1,25℃ 8000 g离心1 min,再向离心柱内加入500 μl Buffer AW2,25℃ 8000 g离心3 min;
(4) 将离心柱放入1.5 ml的无菌离心管中,在离心柱中央加入100 μl Buffer TE,25℃孵育1 min。25℃ 8000 g离心1 min;
eDNA提取完成后,立即使用超微量紫外分光光度计检测其浓度及质量,若eDNA样品的浓度高于250 ng/μl (实时荧光定量PCR试剂盒要求待测样品的DNA浓度低于250 ng/μl),则将其进行稀释。在每个eDNA样品内吸取10 μl DNA溶液,用作琼脂糖凝胶电泳检测及PCR定量分析,其余90 μl eDNA溶液–80 ℃保存备用。
1.5 中国对虾eDNA定量分析所有提取的eDNA样品采用BBI生命科学有限公司的2×TaqMan Fast qPCR Master Mix (Low Rox)实时荧光定量PCR试剂盒进行定量分析。定量PCR (Quantitative PCR, qPCR)扩增的反应体系按照试剂盒说明书要求采用20 μl体系(表 3)。qPCR扩增反应程序:94℃ 3 min;94℃ 5 s,60℃ 35 s,40个循环。qPCR扩增仪器为ABI 7500型定量PCR仪,质粒标准品DNA与未知浓度eDNA样品在每个96孔板均设计3个qPCR重复,每个96孔板设置3个阴性对照(无模板)与3个阳性对照(中国对虾基因组DNA),质粒标准品DNA依次从107 copies/μl以10倍的浓度梯度稀释到10 copies/μl。实验获得的所有数据采用绝对定量法分析,每个eDNA样品最终的拷贝数以阳性扩增样品的平均值为准,应用系统软件SDS 1.4.0.25自动计算Ct值,并生成标准曲线与扩增曲线。
|
|
表 3 实时荧光定量PCR反应体系及用量 Tab.3 Real-time fluorescence quantitative PCR reaction system and dosage |
选择黄海中南部的3个站位(8694、10594和13994)采集表层沉积物(采样站位根据该海域底质类型进行选择)。表层沉积物的采集工作在同一航次中完成,采样时,将采泥器垂直下放至海底抓取沉积物,待采泥器闭合后提出海面移至夹板,排出采泥器中残留海水以避免沉积物悬浮进入水体。采样前后,采泥设备用双蒸水冲洗2遍以避免样品交叉污染。样品放入无菌封口袋,于–20℃冷库中保存,运输过程用冰块保持温度在–10℃左右,避免样品反复冻融。2020年1月3日样品送达实验室后立即进行实验。3个站位的表层沉积物分为3个实验组。每个实验组为1个15 L的塑料整理箱(长×宽×高:35 cm × 24 cm × 20 cm),内装1000 g沉积物样品和10 L人工海水。实验过程中,所用人工海水经实验室qPCR检测,无中国对虾eDNA存在。实验启动时,分别混匀3个实验组,待沉积物重新沉淀后,采集1 L上层水样(3个平行样品)进行中国对虾eDNA富集,滤膜用锡纸单独包装,–20℃保存待用。取样后向整理箱中重新加入3 L人工海水以保证下次取样浓度一致。每日15:00取样,连续进行1周。所有实验组均在室内进行,室温保持在10℃,水温保持在6℃并通过空气泵持续曝气。所有实验设备均在实验前用高锰酸钾消毒,并用双蒸水冲洗。实验结束后,对所有样品统一进行eDNA的提取和qPCR检测。室内实验中国对虾eDNA的富集、提取及qPCR分析方法同上(1.3、1.4和1.5)。
|
|
表 4 实验条件设置 Tab.4 Setting of experimental condition |
所有数据经标准化处理后使用Excel 2016、R 3.6.3及其集成开发环境R Studio软件处理。
2 结果与分析 2.1 引物特异性验证2%的琼脂糖凝胶电泳检测表明,引物COI DF/ DR成功扩增了中国对虾mtDNA COⅠ长度为106 bp的目的片段,电泳条带单一明亮、无杂带(图 2),与预期结果一致,说明所选引物特异性良好。

|
图 2 qPCR产物的琼脂糖凝胶凝胶电泳检测结果 Fig.2 Detection of qPCR production by agarose gel electrophoresis M: DNA Marker DL 2000;–:负极;+:正极;连续3列泳道为同一采样站位 M: DNA Marker DL 2000; –: Negative electrode; +: Positive electrode; Three rows of lanes are for the same sampling station |
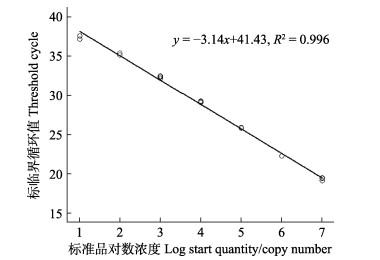
从图 3可以看出,本实验稀释的质粒标准品DNA浓度在10~107 copies/μl范围内具有良好的线性关系,说明本研究建立的标准曲线能够准确地反应中国对虾mtDNA COⅠ基因的扩增。
|
图 3 中国对虾mtDNA COⅠ基因qPCR标准曲线 Fig.3 The standard curve of qPCR of F.chinensis mtDNA COⅠ gene |
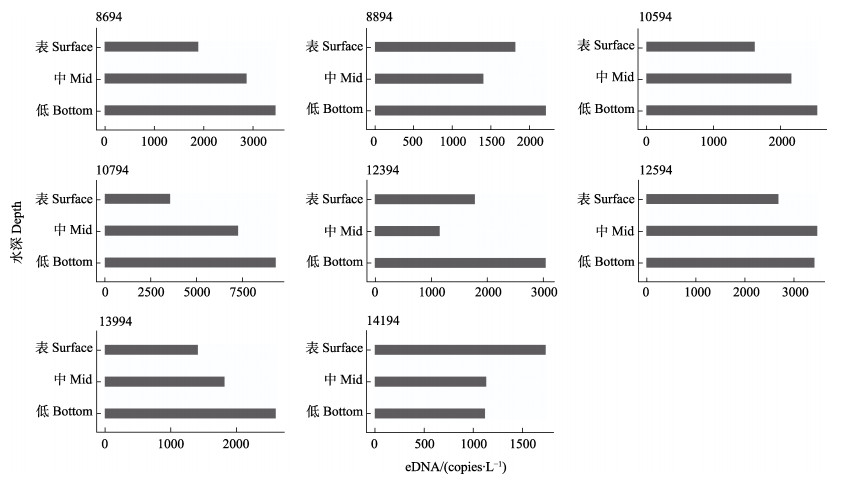
检测结果显示,在黄海8个站位的表、中、底水样中,底层eDNA拷贝数与表层和中层具有显著差异(P=0.003, P=0.0009)。除个别站位外,底层水体eDNA拷贝数均大于表、中层水体(表 5)。8894、12394和14194站位表层eDNA浓度大于中层,其中,14194站位表层eDNA浓度最高,呈现了中国对虾eDNA分布规律为表层少、底层多(图 4)。
|
|
表 5 黄海8个站位不同水深中国对虾eDNA浓度 Tab.5 Concentrations of F.chinensis eDNA in different water depths at eight stations in Yellow Sea (copies/L) |
|
图 4 黄海8个站位不同水深中国对虾eDNA浓度 Fig.4 Concentrations of F.chinensis eDNA in different water depths at eight stations in Yellow Sea |
室内沉积物释放实验结果显示,表层沉积物在外力作用下会向水体中释放大量中国对虾eDNA,其中,8694与10594站位在实验第1天释放量最高,分别为1624.06和3453.34 copies/L,而13994站位在第2天释放量达到最高,为1143.24 copies/L。3个实验组沉积物释放的eDNA拷贝数与时间呈负相关关系。均在第7天(2020年1月9日)趋于稳定且保持在较低水平(表 6和图 5)。
|
|
表 6 表层沉积物释放中国对虾eDNA浓度 Tab.6 Variations of environmental DNA concentration of F.chinensis released from the surface substrate (copies/L) |

|
图 5 室内实验中国对虾eDNA的浓度变化 Fig.5 Variation of eDNA concentration of F.chinensis in laboratory experiments |
本研究发现,中国对虾在垂直方向上具有显著的分布规律。结合目标物种在不同发育阶段的生活习性和实际环境条件,对eDNA浓度进行分析推测,能够分析得到更为准确的物种分布位置和生物量情况。以往研究表明,中国对虾在越冬场主要以底栖的甲壳类、瓣鳃类、多毛类、蛇尾类(Ophiuroidea)及小型鱼类为食,还会摄食寄居蟹皮海绵(Suberites domumeula) (邓景耀等, 1990)。根据中国对虾这一摄食习性,可以推测中国对虾在黄海越冬洄游过程中应该集中分布于底层水体。而检测结果与这一推测相符:越冬期中国对虾eDNA底层浓度相对较大。同时,粪便和外壳作为越冬期中国对虾的主要eDNA来源,由于涌浪和沉降作用,这些物质会变成细小碎屑沉至底层水体(Turner et al, 2015),也会导致底层水体中国对虾eDNA浓度升高。此外,不同物种或同一物种在不同发育阶段,其eDNA的释放速率也存在一定差异(Minamoto et al, 2017; Maruyama et al, 2014)。将种群构成因素纳入到考量范围,也是提高eDNA技术准确性的有效途径。
3.2 水体浑浊度对eDNA分布的影响结果显示,中国对虾的eDNA在自然水体中总体呈现底层浓度高、表层浓度低的特点,但8894、12394和14194的检测结果与其他站位存在差异。在采样过程中发现,这3站的水体较为浑浊,泥沙量大,可能是导致eDNA浓度反常变化的原因之一,怀疑该海域存在垂直流,导致底部泥沙悬浮,从而对eDNA浓度存在一定程度影响。具体影响方式需要开展更多实验来验证。
3.3 沉积物对eDNA分布的影响本研究发现,eDNA会在水体与沉积物之间双向移动,粪便和外壳作为中国对虾越冬期的主要eDNA来源,不仅会在沉降作用下进入沉积物,并且沉积物会在外力(如垂直流、底栖生物扰动和人为干扰等)作用下,导致沉积物再悬浮(Turner et al, 2015),向周围释放eDNA,使得底层水体eDNA浓度升高,从而使测量值高于真实值。研究表明,eDNA会在水体与沉积物之间转移,因此,将沉积物与水体结合进行分析更有意义(Leff et al, 1992)。相比水体,沉积物中含有更多的eDNA(Corinaldesi et al, 2005; Dell’Anno et al, 2004)。其原因是:(1) 生物的粪便、外壳和脱落的组织会在沉降作用下进入沉积物中,使沉积物中eDNA浓度升高(Turner et al, 2015);(2) eDNA可被沉积物中的表面活性颗粒吸附,受保护而不被降解,并且这些颗粒还会吸附核酸酶,导致酶活性降低,从而延长eDNA降解时间(Pietramellara et al, 2009),使沉积物中eDNA不断积累,浓度上升。以上两点说明了沉积物在eDNA研究中的重要性。但大多数研究只是单独对沉积物中的eDNA进行研究,鲜有研究者将沉积物与周围环境结合看待(魏楠等, 2020),因而沉积物对周围环境的影响极易被忽视。本研究揭示了沉积物通过再悬浮释放eDNA的方式对周围水体造成巨大影响,如何屏蔽这一影响,减少测量误差,是接下来研究的重点。
3.4 再悬浮eDNA残留时间的区别室内实验发现,表层沉积物中的中国对虾eDNA溶解进入水体7 d后近乎完全降解。而这与李苗等(2018)对中国对虾eDNA水中残留时间的研究结果存在差异,其研究发现,新鲜的eDNA可以在水体中残留27 d。同时还发现,eDNA降解时间受eDNA浓度影响。eDNA浓度较高,其降解时间较长(李苗等, 2020)。考虑到本研究的样本是在野外采集到的,其浓度远比养殖场中的样品浓度小。因此,本研究中沉积物释放的eDNA残留时间相对较短。另外,新鲜的eDNA被生物体释放进入水体时发生降解,致使片段长度缩短,并在沉降作用下保留在沉积物中,沉积物又在外力作用下再悬浮,导致所释放短片段eDNA环境改变,加速了降解过程,因此,沉积物释放的eDNA仅能残留较短时间。已有研究表明,不同片段长度的eDNA在残留时间上具有显著差异(Dejean et al, 2011)。在以后的研究中,可以根据eDNA片段长度的不同,对eDNA进行区分,选择合适的引物长度进行qPCR检测,以此降低沉积物释放的干扰。
在了解目标生物的生活习性的前提下,研究人员进行有针对性的采样设计,可以使检测结果更加可信。但需要对再悬浮的eDNA进行有效区分,以避免测量结果的误差,从而将eDNA技术更好地推广到其他水生物种的生态系统研究中。
Bogich TL, Liebhold AM, Shea K. To sample or eradicate? A cost minimization model for monitoring and managing an invasive species. Journal of Applied Ecology, 2008, 45: 1134-1142 DOI:10.1111/j.1365-2664.2008.01494.x |
Corinaldesi C, Danovaro R, Dell'Anno A. Simultaneous recovery of extracellular and intracellular DNA suitable for molecular studies from marine sediments. Journal of Applied Environmental Microbiology, 2005, 71: 46-50 DOI:10.1128/AEM.71.1.46-50.2005 |
Deiner K, Altermatt F. Transport distance of invertebrate environmental DNA in a natural river. PLoS One, 2014, 9: e88786 DOI:10.1371/journal.pone.0088786 |
Dejean T, Valentini A, Duparc A, et al. Persistence of environmental DNA in freshwater ecosystems. PLoS One, 2011, 6(8): e23398 DOI:10.1371/journal.pone.0023398 |
Dell'Anno A, Corinaldesi C. Degradation and turnover of extracellular DNA in marine sediments: Ecological and methodological considerations. Journal of Applied Environmental1 Microbiology, 2004, 70(7): 4384-4386 DOI:10.1128/AEM.70.7.4384-4386.2004 |
Deng JY, Ye CC, Liu YC. Shrimp and its resource management in Bo Sea and Yellow Se. Beijing: China Ocean Press, 1990 [邓景耀, 叶昌臣, 刘永昌. 渤黄海的对虾及其资源管理. 北京: 海洋出版社, 1990]
|
Ficetola GF, Miaud C, Pompanon F, et al. Species detection using environmental DNA from water samples. Journal of Biology Letters, 2008, 4(4): 423-425 DOI:10.1098/rsbl.2008.0118 |
Jane SF, Wilcox TM, McKelvey KS, et al. Distance, flow and PCR inhibition: eDNA dynamics in two headwater streams. Journal of Molecular Ecology Resources, 2015, 15(1): 216-227 DOI:10.1111/1755-0998.12285 |
Klymus KE, Richter CA, Chapman DC, et al. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Journal of Biological Conservation, 2015, 183: 77-84 DOI:10.1016/j.biocon.2014.11.020 |
Leff L, McArthur J, Shimkets L. Information spiraling: Movement of bacteria and their genes in streams. Journal of Microbial Ecology, 1992, 24: 11-24 DOI:10.1007/BF00171967 |
Li M, Shan XJ, Wang WJ, et al. Establishment and optimization of environmental DNA detection techniques for assessment of Fenneropenaeus chinensis biomass. Progress in Fishery Sciences, 2019, 40(1): 12-19 [李苗, 单秀娟, 王伟继, 等. 中国对虾生物量评估的环境DNA检测技术的建立及优化. 渔业科学进展, 2019, 40(1): 12-19] |
Li M, Shan XJ, Wang WJ, et al. Studying the retention time of Fenneropenaeus chinensis eDNA in water. Progress in Fishery Sciences, 2020, 41(1): 51-57 [李苗, 单秀娟, 王伟继, 等. 环境DNA在水体中存留时间的检测研究——以中国对虾为例. 渔业科学进展, 2020, 41(1): 51-57] |
Lodge DM, Turner CR, Jerde CL, et al. Conservation in a cup of water: Estimating biodiversity and population abundance from environmental DNA. Journal of Molecular Ecology, 2012, 21(11): 2555-2558 DOI:10.1111/j.1365-294X.2012.05600.x |
Maggi F. The settling velocity of mineral, biomineral, and biological particles and aggregates in water. Journal of Geophysical Research: Oceans, 2013, 118(4): 2118-2132 DOI:10.1002/jgrc.20086 |
Maruyama A, Nakamura K, Yamanaka H, et al. The release rate of environmental DNA from juvenile and adult fish. PLoS One, 2014, 9(12): e114639 DOI:10.1371/journal.pone.0114639 |
Minamoto T, Fukuda M, Katsuhara KR, et al. Environmental DNA reflects spatial and temporal jellyfish distribution. PLoS One, 2017, 12(2): e0173073 DOI:10.1371/journal.pone.0173073 |
Murphy HM, Jenkins GP. Observational methods used in marine spatial monitoring of fshes and associated habitats: A review. Journal of Marine and Freshwater Research, 2010, 61: 236-252 DOI:10.1071/MF09068 |
O'Donnell JL, Kelly RP, Shelton AO, et al. Spatial distribution of environmental DNA in a nearshore marine habitat. Journal of Peer, 2017, 5: e3044 DOI:10.7717/peerj.3044 |
Pietramellara G, Ascher J, Borgogni F, et al. Extracellular DNA in soil and sediment: Fate and ecological relevance. Journal of Biology and Fertility Soils, 2009, 45(3): 219-235 DOI:10.1007/s00374-008-0345-8 |
Renshaw MA, Olds BP, Jerde CL, et al. The room temperature preservation of filtered environmental DNA samples and assimilation into a phenol-chloroform-isoamyl alcohol DNA extraction. Journal of Molecular Ecology Resources, 2015, 15(1): 168-176 DOI:10.1111/1755-0998.12281 |
Robison BH, Bailey TG. Sinking rates and dissolution of midwater fish fecal matter. Journal of Marine Biology, 1981, 65(2): 135-142 DOI:10.1007/BF00397077 |
Saba GK, Steinberg DK. Abundance, composition, and sinking rates of fish fecal pellets in the Santa Barbara Channel. Journal of Scientific Reports, 2012, 2: 716 DOI:10.1038/srep00716 |
Sansom BJ, Sassoubre LM. Environmental DNA (eDNA) shedding and decay rates to model freshwater mussel eDNA transport in a river. Journal of Environmental Science and Technology, 2017, 51: 14244-14253 DOI:10.1021/acs.est.7b05199 |
Shogren AJ, Jennifer l, Andruszkiewicz TE, et al. Controls on eDNA movement in streams: Transport, retention, and resuspension. Journal of Scientific Reports, 2017, 7(1): 5065 DOI:10.1038/s41598-017-05223-1 |
Taberlet P, Prud'Homme, S M, Campione E, et al. Soil sampling and isolation of extracellular DNA from large amount of starting material suitable for metabarcoding studies. Journal of Molecular Ecology, 2012, 21(8): 1816-1820 DOI:10.1111/j.1365-294X.2011.05317.x |
Takahara T, Minamoto T, Yamanaka H, et al. Estimation of fish biomass using environmental DNA. PLoS One, 2012, 7(4): e35868 DOI:10.1371/journal.pone.0035868 |
Thalinger B, Wolf E, Traugott M, et al. Monitoring spawning migrations of potamodromous fish species via eDNA. Journal of Scientific Reports, 2019, 9(1): 1033-1047 DOI:10.1038/s41598-018-36760-y |
Thomsen PF, Kielgast J, Iversen LL, et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples. PLoS One, 2012a, 7: e41732 DOI:10.1371/journal.pone.0041732 |
Thomsen PF, Kielgast J, Iversen LL, et al. Monitoring endangered freshwater biodiversity using environmental DNA. Journal of Molecular Ecology, 2012b, 21(11): 2565-2573 DOI:10.1111/j.1365-294X.2011.05418.x |
Turner CR, Uy KL, Everhart RC. Fish environmental DNA is more concentrated in aquatic sediments than surface water. Journal of Biology Conservation, 2015, 183: 93-102 DOI:10.1016/j.biocon.2014.11.017 |
Wei N, Wang XH, Zhang CP. Application of environmental DNA on monitoring surface sediment and the relationship to environment variables. Journal of Environmental Engineering, 2020, 14(8): 2262-2269 [魏楠, 王夏晖, 张春鹏. 环境DNA在监测表层沉积物中的运用及其与环境变量的关系. 环境工程学报, 2020, 14(8): 2262-2269] |
Wilcox TM, McKelvey KS, Young MK, et al. Environmental DNA particle size distribution from brook trout (Salvelinus fontinalis). Journal of Conservation Genetics Resources, 2015, 7(3): 639-641 DOI:10.1007/s12686-015-0465-z |
Wotton RS, Malmqvist B. Feces in aquatic ecosystems. Journal of Bioscience, 2001, 51: 537-544 DOI:10.1641/0006-3568(2001)051[0537:FIAE]2.0.CO;2 |
Yamamoto S, Masuda R, Sato Y, et al. Environmental DNA metabarcoding reveals local fish communities in a species- rich coastal sea. Journal of Scientific Reports, 2017, 7: 40368 DOI:10.1038/srep40368 |
Yamamoto S, Minami K, Fukaya K, et al. Environmental DNA as a 'snapshot' of fish distribution: A case study of Japanese jack mackerel in Maizuru Bay, Sea of Japan. PLoS One, 2016, 11(3): e0149786 DOI:10.1371/journal.pone.0149786 |



