2. 中国水产科学研究院东海水产研究所 农业农村部低洼盐碱地 水产养殖重点实验室 盐碱水域渔业工程技术研究中心(上海) 上海 200090;
3. 通威股份有限公司 成都 610093
2. Key Laboratory of Aquaculture on Saline-Alkaline Land, Ministry of Agriculture and Rural Affairs, East China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Fishery Engineering Technology Research Center for Saline-Alkaline Waters (Shanghai), Shanghai 200090, China;
3. Tongwei Co, LTD, Chengdu, Sichuan 610093, China
我国拥有丰富的盐碱水资源,其高pH、高碳酸盐碱度水质特征制约了水生动物的生存、生长和繁殖。凡纳滨对虾(Litopenaeus vannamei)具有较强的抗逆性,在盐碱水中有一定的耐受性。短期盐碱胁迫下,凡纳滨对虾碳酸酐酶(CA)、Na+-K+-ATPase等离子调控类基因差异表达,以增强离子调节的方式进行酸碱和渗透平衡的调控(么宗利等, 2010、2012),但针对长期高碱胁迫下凡纳滨对虾的基因调控研究相对较少。目前,虽然盐碱水凡纳滨对虾养殖取得一定成功,但养殖存活率不稳定,凡纳滨对虾耐盐碱选育鲜有报道。国内通过引进、消化、吸收和自主创新,建立了基于规模化家系的“水产动物多性状复合育种技术”(张天时, 2010),选育的优良性状包括抗病(黄永春等, 2013)、耐低温(景福涛等, 2006)、耐盐碱(李明栋等, 2021)、耐低盐度(郝登春等, 2018)、养殖存活率(孔杰等, 2017)等,为实现凡纳滨对虾良种本土化打下了良好的基础。为有效利用盐碱水资源,迫切需要开展凡纳滨对虾耐盐碱选育工作,从而促进凡纳滨对虾盐碱水养殖业的健康发展。
水生生物耐盐碱性状由多基因互相作用协同控制(么宗利等, 2010),其分子响应机制是一个复杂的过程。研究发现,耐盐碱性状具有一定的遗传潜力,在尼罗罗非鱼(Oreochromis niloticus)、瓦氏雅罗鱼(Leuciscus waleckii)、脊尾白虾(Exopalaemon carinicauda)等水产动物中获得了若干耐盐碱功能基因(唐首杰等, 2018; Chang et al, 2021; 李明栋等, 2021),但功能位点尚不清晰。随着基因组学和生物信息学的高速发展,高通量测序技术逐渐成熟,并广泛应用于功能基因挖掘、分子标记筛选及信号转导等研究中,为耐盐碱响应机制的研究提供了有效的技术手段。其中,转录组测序可以针对各种环境条件下的物种细胞或组织进行高通量测序,并对测序结果进行基因结构分析和功能注释,分析特定条件下相关基因的表达水平,以揭示其代谢网络及调控响应机理(Sims et al, 2014)。本研究采用高通量测序技术对2种处理下的凡纳滨对虾肠道和鳃组织进行转录组测序,筛选长期高碱胁迫下差异表达基因以及与盐碱胁迫相关代谢通路,为解析凡纳滨对虾耐盐碱调控机制提供基础数据,同时为盐碱水环境下凡纳滨对虾分子辅助育种提供理论依据。
1 材料与方法 1.1 实验材料实验所用凡纳滨对虾取自通威股份有限公司2020年5月培育的G10代育种群体。对现有20个凡纳滨对虾家系进行驯化、筛选、荧光标记后,进行96 h高碱急性胁迫实验,依据实验结果,开展42 d的高碱胁迫实验。挑选活力相对较好、个体规格比较一致的凡纳滨对虾作为实验用虾,起始平均体长为(7.2±0.5) cm,体重为(4.7±0.6) g。
1.2 碳酸盐碱度胁迫实验碳酸盐碱度胁迫实验于2020年8月开始,每个家系挑选规格相对一致的凡纳滨对虾96尾,设置低碱对照组(LSW)和高碱胁迫组(AW),每组设置3个平行(每个平行16尾),每个平行单独放置在50 cm× 50 cm×50 cm网箱中养殖。AW组养殖用水通过添加相应的Na2CO3和NaHCO3配制而成(碳酸盐碱度为10 mmol/L,盐度为6,pH=8.8),LSW组为对照组(碳酸盐碱度为3 mmol/L,盐度为6,pH=8.1)。为保证实验组盐碱水碳酸盐碱度和pH稳定,每隔24 h换水50%。经过42 d的养殖,选择一个高碱敏感家系[养殖存活率为(42.5±8.33)%]取样,LSW和AW每组每个平行各取6尾虾[体长为(9.8±0.8) cm,体重为(9.6±0.9) g],活体解剖,取鳃和肠道组织放入RNA保存液保存备用,其中,3尾用于转录组测序,3尾用于后期qRT-PCR验证。
1.3 RNA提取及转录组测序按照常规Trizol法提取每尾虾的鳃和肠道组织总RNA。通过1%琼脂糖凝胶电泳和NanoDrop ND-2000分光光度计(Thermo, 美国)检测RNA质量及浓度。总RNA浓度 > 250 ng/μL,OD260 nm/OD280 nm介于1.8~2.2之间,确保RNA无降解、无污染,然后,进行文库构建和高通量测序(Hiseq 2500, Illumina,美国)。
1.4 转录组序列组装及注释测序原始数据经过质量分析,去除带接头、低质量和N (无法确定碱基信息)比例大于10%的序列,质控后的序列用Trinity软件进行拼接,每个基因以拼接得到的最长序列为该基因序列(unigene) (Grabherr et al, 2011)。转录组测序数据用CASAVA Base Calling软件、RSEM软件分析,并利用HISAt2将Clean Reads与指定参考基因组(ASM378908v1-NCBI)进行序列比对,获取在参考基因组或基因上的位置信息以及测序样本特有的序列特征信息。
1.5 差异表达分析及差异基因富集分析采用FPKM方法计算基因表达量(Trapnell et al, 2010)。利用R语言DEGseq软件包筛选差异表达基因(DEGs),筛选阀值为fold change > 2和Q < 0.05 (Anders et al, 2010)。对于DEGs,依据测序结果中的基因GO (gene ontology)功能注释(Young et al, 2010),并结合KEGG数据库分析差异显著基因参与的代谢通路(Kanehisa et al, 2008)。
1.6 qRT-PCR验证为了进一步验证转录组数据,选取10个差异倍数较大的DEGs进行鳃组织qRT-PCR验证分析,其中包含5个上调和5个下调表达的基因。利用Primer 5.0软件设计特异性引物(表 1),送交生工生物工程(上海)股份有限公司合成。qRT-PCR实验采用TaKaRa相对荧光定量试剂盒,以18S rRNA为内参基因,每个样品3次重复,验证所用样品为前期实验经过相同处理的平行样品。利用2–∆∆CT法分析基因的相对表达量(Schmittgen et al, 2008),与转录组表达数据进行比较分析。
|
|
表 1 用于转录组表达验证的引物序列 Tab.1 Genes and primers used for validation of RNA-seq data |
转录组测序分析共完成12个样本的有参转录组测序,获得75.62 Gb的Clean data,各样本有效数据量在5.72~7.01 Gb,碱基Q30在92.83%~93.47%,平均GC含量为47.76%,说明测序质量符合生物信息学分析要求,测序数据质量如表 2所示。
|
|
表 2 样品测序数据与组装结果统计 Tab.2 Summary of sequencing and transcriptome assembly |
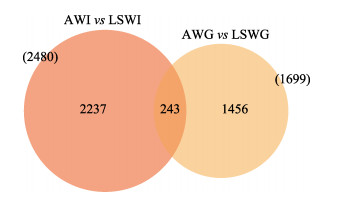
通过将Clean reads比对到参考基因组,得到各个样本的基因组比对情况,比对率为80.93%~91.2%。基于比对结果,进行蛋白编码基因表达量分析。根据蛋白编码基因在不同样本中的表达量进行差异筛选,设有肠道和鳃2个差异分组,其检测到的差异基因数量分别为2480和1699,共同差异表达基因为243个(图 1)。
|
图 1 高碱胁迫下凡纳滨对虾肠道和鳃组织差异表达基因维恩图 Fig.1 Numbers of differentially expressed genes in intestine and gills of L. vannamei under high alkaline stress |
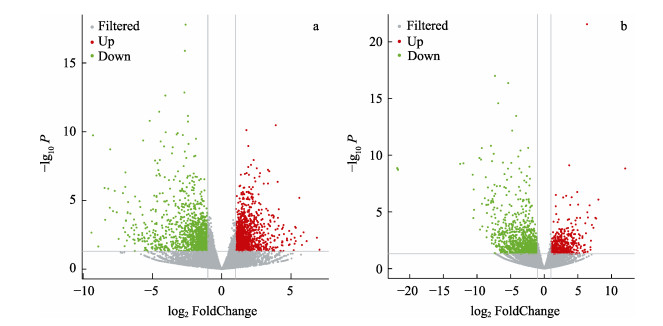
对AW和LSW的差异表达基因分析结果绘制火山图(图 2)。结果显示,在高碱胁迫条件下,肠道组织有1224个基因表达上调,有1256个基因表达下调;鳃组织有613个基因表达上调,1086个基因表达下调。将2个组织在高碱胁迫下的差异表达基因进行比较,2个组织有243个共同的差异表达基因,其中,98个基因上调表达,145个基因下调表达,这243个共同的差异表达基因可能与高碱胁迫下凡纳滨对虾组织间协同调控分子机制有关。
|
图 2 高碱胁迫下凡纳滨对虾差异表达基因火山图 Fig.2 The volcano plot of differentially expressed genes of L. vannamei under high alkaline stress a:肠道组织差异基因火山图;b:鳃组织差异基因火山图灰色为非显著性差异的基因,红色为显著上调表达基因,绿色为显著下调表达基因。 a: The volcanic plot of the differentially expressed genes in intestine; b: The volcanic plot of the differentially expressed genes in gills; The gray means non-significant different genes, the red and green means a significant different genes, red means the up-regulated genes, and green means down-regulated genes. |
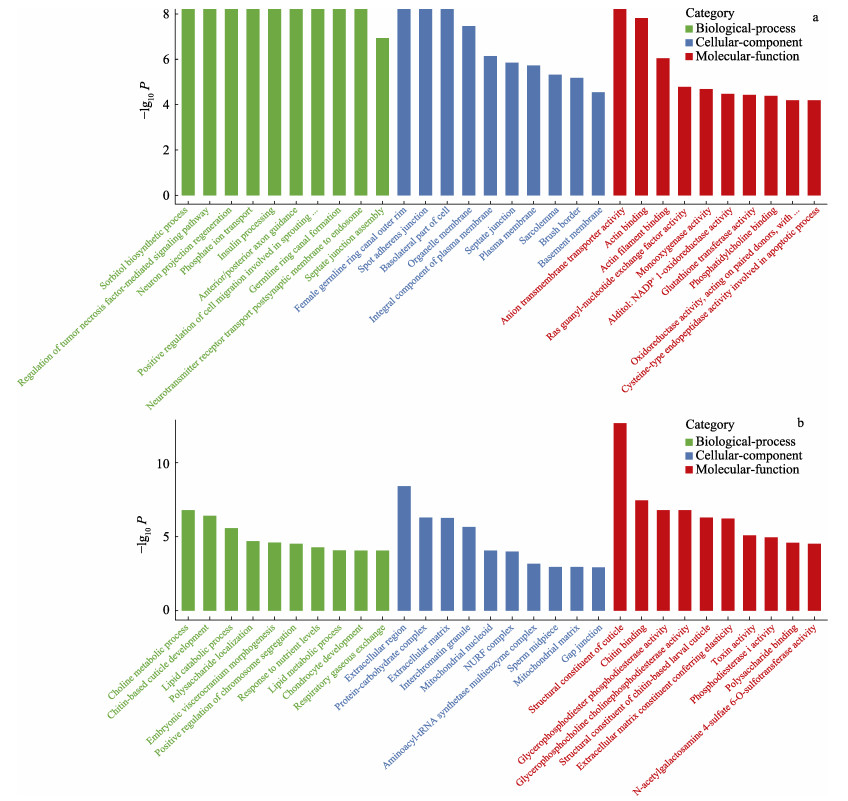
GO富集分析显示,差异基因在生物过程(biological process, BP)、细胞成分(cellular component, CC)和分子功能(molecular function, MF)中均有分布,其中,以生物过程居多。肠道中,差异表达基因共富集到5253个terms,其中,生物过程3223个,细胞组分910个,分子功能1120个。鳃组织中,差异表达基因共富集到3647个terms,其中,生物过程1244个,细胞组分1185个,分子功能1218个。在生物过程中,生物调节、代谢途径、细胞途径、离子转运以及单有机体过程是包含差异基因最多的几类;在细胞成分类中,差异基因最多的是细胞组分和细胞这两类;而在分子功能类中,结合和催化活性这两类包含了最多的差异基因,挑选每个分类中富集最显著的前10个GO terms进行展示(图 3)。
|
图 3 高碱胁迫下凡纳滨对虾肠道、鳃组织差异基因前30 GO富集分析 Fig.3 Top 30 GO enrichment of differentially expressed genes of intestine and gills of L. vannamei under high alkaline stress a:肠道组织差异基因GO富集;b:鳃组织差异基因GO富集 a: GO enrichment terms of differentially expressed genes in intestine tissue; b: GO enrichment terms of differentially expressed genes in gills tissue |
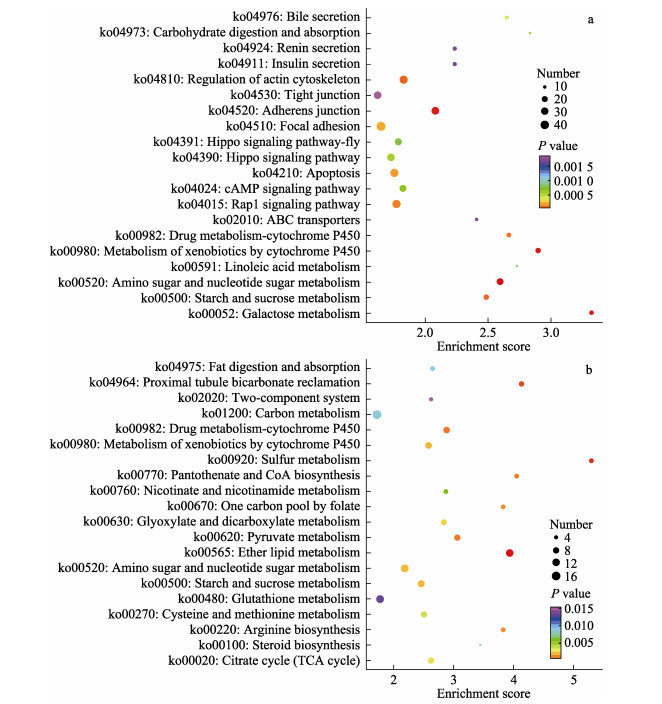
KEGG富集结果显示,2种处理在肠道组织的DEGs涉及238个通路,在鳃组织的DEGs涉及225个通路,从KEGG富集分析中选取富集最显著的20个代谢途径,绘制KEGG富集气泡图(图 4)。其中,肠道中差异表达基因主要集中在糖代谢、碳水化合物消化吸收、胆汁分泌、ABC跨膜转运、紧密连接、免疫调节等途径。鳃中差异表达基因主要集中在谷胱甘肽代谢、碳酸氢盐转运、精氨酸合成、糖代谢以及离子转运等相关途径。
|
图 4 高碱胁迫下凡纳滨对虾肠道和鳃差异表达基因KEGG富集分析气泡图 Fig.4 KEGG enrichment results of differentially expressed genes in intestine and gills of L. vannamei under high alkalinity stress a:肠道差异表达基因KEGG富集分析;b:鳃差异表达基因KEGG富集分析P值大小用点的颜色来表示,P-value越小则颜色越接近红色,每个通路下包含差异基因的多少用点的大小来表示。 a: KEGG enrichment results of differentially expressed genes in intestine; b: KEGG enrichment results of differentially expressed genes in gills. The P-value is represented by the color of dots. Red color indicates small P-value. The number of differential genes contained in each pathway is represented by the size of dots. |
从2种不同处理下凡纳滨对虾的肠道和鳃中筛选的243个共同DEGs中,根据GO和KEGG富集结果,选取10个差异倍数较大DEGs进行验证(图 5)。各基因qRT-PCR验证表达趋势与转录组表达趋势一致。其中,NKA-α、NKAIN、MDH、Rhbg和Nmdar1基因在高碱胁迫下呈上调表达,差异倍数在1.6~4.1之间;Eip74EF、CA1、CA10、UGT8和Hex-t2基因呈下调表达,差异倍数在1.9~4.4之间。

|
图 5 高碱胁迫下凡纳滨对虾鳃组织差异基因qRT-PCR验证 Fig.5 Validation of differentially expressed genes in gills of L. vannamei using real-time qRT-PCR |
长期高碱胁迫下,凡纳滨对虾肠道中差异基因主要富集在糖代谢、碳水化合物、胆汁分泌等消化吸收相关通路以及细胞凋亡等免疫相关通路,鳃中差异基因主要富集在谷胱甘肽代谢等氮代谢相关通路以及碳酸氢根转运等离子转运相关通路。鳃和肠道是凡纳滨对虾酸碱调节、渗透调节和生长调控的重要器官。鳃是水生动物特有的呼吸器官,在气体交换、渗透压平衡和氨氮排泄等方面发挥重要作用(龚仕玲等, 2019)。肠道在机体的生长调节、营养代谢以及免疫防御等方面起到重要作用。研究表明,高pH胁迫会影响肠道组织的消化酶活性和抗氧化酶活性(Duan et al, 2019)。因此,本研究以选育凡纳滨对虾的鳃和肠道组织作为实验材料,通过高通量测序技术分析其在高碱胁迫和正常碱度条件下转录水平的差异。高碳酸盐碱度会影响对虾的生长和存活(么宗利等, 2010、2012; 柳飞等, 2016),而青海湖裸鲤(Gymnocypris przewalskii)、瓦氏雅罗鱼(Leuciscus waleckii)、尼罗罗非鱼(Oreochromis niloticus)等鱼类在高碱环境下,其体内会积累氨氮(衣晓飞等, 2017; 徐悦等, 2021; 吴俊伟等, 2016)。本研究中,凡纳滨对虾肠道消化吸收、鳃氮代谢等通路富集了大量差异表达基因,推测其在应对长期高碱胁迫时,生长和氮废物排泄受到较大影响。同时,凡纳滨对虾细胞凋亡相关通路的变化提示,胁迫环境可能会破坏其免疫防御系统,导致免疫抑制(Xiao et al, 2019)。
3.2 高碱环境下凡纳滨对虾相关基因表达本研究发现,在应对长期高碱胁迫时,鳃组织中NKAIN、NKA-a等离子转运基因和Rhbg氨转运基因表现为上调模式,而CA1、CA10、Eip74EF等基因表现为下调模式。凡纳滨对虾应对高碱胁迫的响应机制较为复杂,当其暴露在高碱环境中时,碱性水环境中HCO3–、CO32–等离子可直接腐蚀对虾鳃和其他表面暴露部位,造成器质性损伤,破坏其离子交换体系,进而影响对虾的存活。高碱度水体中较高浓度的CO32–、HCO3–导致水生动物体内摄入过多的HCO3–,同时,外界环境的高pH会抑制一些离子交换(如Na+/H+、Cl–/HCO3–等),HCO3–大量累积,造成水生动物体内酸碱平衡遭到破坏,最终导致持续性碱中毒(Yao et al, 2015)。本研究中,鳃中NKA相关基因表达上调,有利于高碱环境下凡纳滨对虾维持体内离子平衡,而低表达的碳酸酐酶则有助于降低体内HCO3–含量,从而维持酸碱平衡。脊尾白虾中3种碳酸酐酶(CAs)在盐碱水中响应高碱胁迫分子机制的研究也发现,CAc和CAg在高碱环境中具有重要调节能力,对高碱环境下对虾的存活发挥重要作用,为对虾的存活争取了时间(Ge et al, 2019)。研究表明,高碱环境会影响水生动物氨氮的排泄,鱼类可以通过上调Rh、Ut基因增加氨的排泄(衣晓飞等, 2017)。本研究发现,凡纳滨对虾鳃中Rhbg表达上调,推测其可能参与了长期高碱胁迫下氨排泄。甲壳类动物外壳由大量Ca2+化合物构成,甲壳类动物在蜕皮前期会将外壳中的部分Ca重新吸收,但蜕皮后仍需从食物或者通过鳃组织从水体中获取Ca (董少帅等, 2005)。在长期高碱环境下,由于Ca2+的流失,蜕壳周期延长,AW组在养殖过程中的蜕壳率较低。本研究发现,Eip74EF在鳃中表达下调,而这一基因在调控果蝇(Drosophila)蜕皮变态发育上有着重要的协同作用(Fletcher et al, 1995),推测其在高碱胁迫下参与对虾蜕壳调控过程。此外,本研究发现,鳃部Hex-t2、UGT8等能量代谢基因表达下调,推测在高碱胁迫下,对虾能量消耗较大,通过降低能量消耗维持其基本生命活动。对虾耐碱胁迫是一个复杂的过程,涉及生理、生化及遗传多个方面。因此,虾类对高碳酸盐碱度的抵抗能力不仅是单个耐受基因的启动和识别,更重要的是在整个生理过程中多个基因的相互作用。
综合转录组和定量PCR实验结果显示,凡纳滨对虾表现出较强的高碱适应性,可能是通过下调鳃中CA的表达补偿体内碱中毒,上调Rh氨转运蛋白防止氨在体内积累,上调NKA相关基因维持体内渗透平衡而得到的结果;蜕皮激素诱导蛋白(Eip74EF)显著下调,推测其蜕皮功能受到影响。本研究通过对凡纳滨对虾鳃和肠道进行转录组分析,寻找高碱环境下差异表达基因,为后续高碱环境下凡纳滨对虾的选育及良种培育提供了理论基础。
ANDERS S, HUBER W. Differential expression analysis for sequence count data. Genome Biology, 2010, 11: R106 DOI:10.1186/gb-2010-11-10-r106 |
CHANG Y M, ZHAO X F, LIEW H J. Effects of bicarbonate stress on serum ions and gill transporters in alkali and freshwater forms of Amur ide (Leuciscus waleckii). Aquatic Physiology, 2021, 12(5): 676-696 |
DONG S S, DONG S L, WANG F, et al. The effect of Ca2+ concentration on the growth of juvenile Litopenaeus vannamei. Journal of Fisheries of China, 2005, 29(2): 211-215 [董少帅, 董双林, 王芳, 等. Ca2+浓度对凡纳滨对虾稚虾生长的影响. 水产学报, 2005, 29(2): 211-215] |
DUAN Y F, WANG Y, LIU Q S. Changes in the intestine barrier function of Litopenaeus vannamei in response to pH stress. Fish and Shellfish Immunology, 2019, 88(3): 142-149 |
FLETCHER C, THUMMEL C S. The Drosophila E74 gene is required for the proper stage- and tissue-specific transcription of ecdysone-regulated genes at the onset of metamorphosis. Development, 1995, 121(5): 1411-1421 DOI:10.1242/dev.121.5.1411 |
GE Q Q, LI J, WANG J J, et al. Characterization, functional analysis, and expression levels of three carbonic anhydrases in response to pH and saline-alkaline stresses in the ridgetail white prawn Exopalaemon carinicauda. Cell Stress and Chaperones, 2019, 2(3): 212-224 |
GONG S L, XIE D M, LI Y W, et al. Cadmium exposure induces histological damage, oxidative stress and immune response in yellow catfish. Acta Hydrobiologica Sinica, 2019, 43(2): 340-347 [龚仕玲, 谢冬梅, 李英文, 等. 镉暴露诱导黄颡鱼鳃的组织学损伤、氧化应激和免疫反应. 水生生物学报, 2019, 43(2): 340-347] |
GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nature Biotechnology, 2011, 29(7): 644-652 DOI:10.1038/nbt.1883 |
HAO D C, LUAN S, CAO B X, et al. Genetic parameters of survival of juvenile families of Litopenaeus vannamei during desalination and culture stages. Progress in Fishery Sciences, 2018, 39(1): 90-96 [郝登春, 栾生, 曹宝祥, 等. 凡纳滨对虾家系幼虾淡化和养殖阶段存活性状遗传参数估计. 渔业科学进展, 2018, 39(1): 90-96] |
HUANG Y C, AI H S, PAN Z C, et al. Establishment and WSSV resistant characteristics of selective breeding families for resistance to the white spot syndrome virus of Litopenaeus vannamei. Journal of Fisheries of China, 2013, 37(3): 359-366 [黄永春, 艾华水, 潘忠诚, 等. 凡纳滨对虾抗WSSV选育家系的建立及其抗病特性. 水产学报, 2013, 37(3): 359-366] |
JING F T, PAN L Q, HU F W. The immune response of white shrimp Litopenaeus vannamei to the change of temperature. Periodical of Ocean University of China (Natural Science), 2006, 36(S1): 40-44 [景福涛, 潘鲁青, 胡发文. 凡纳滨对虾对温度变化的免疫响应. 中国海洋大学学报(自然科学版), 2006, 36(S1): 40-44] |
KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment. Nucleic Acids Research, 2008, 36(3): 480-484 |
KIM D, LANGMEAD B, SALZBERG S L. HISAT: A fast spliced aligner with low memory requirements. Nature Methods, 2015, 12(4): 357-362 DOI:10.1038/nmeth.3317 |
KONG J, LUAN S, LUO K, et al. Genetic evaluation for body weight and survival of Pacific white shrimp (Litopenaeus vannamei) at different salinity. Journal of Fisheries of China, 2017, 41(4): 573-578 [孔杰, 栾生, 罗坤, 等. 不同盐度下凡纳滨对虾生长和存活性状遗传评估. 水产学报, 2017, 41(4): 573-578] |
LI M D, LI J T, SHI K P, et al. Estimation of heritability and genetic correlation of saline-alkali tolerance in Exopalaemon carinicauda. Progress in Fishery Sciences, 2021, 42(1): 117-123 [李明栋, 李吉涛, 史鲲鹏, 等. 脊尾白虾耐盐碱性状遗传力和遗传相关的估计. 渔业科学进展, 2021, 42(1): 117-123] |
LIU F, LI J, LI J T, et al. Effect of carbonate alkalinity stress on the survival, growth, reproduction and immune enzyme activities of Exopalaemon carinicauda. Journal of Fishery Sciences of China, 2016, 23(5): 1137-1147 [柳飞, 李健, 李吉涛, 等. 碳酸盐碱度对脊尾白虾生存、生长、繁殖及免疫酶活性的影响. 中国水产科学, 2016, 23(5): 1137-1147] |
SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols, 2008, 3(6): 1101-1108 DOI:10.1038/nprot.2008.73 |
SIMS D, SUDBERY I, ILOTT N E, et al. Sequencing depth and coverage: Key considerations in genomic analyses. Nature Reviews Genetics, 2014, 15(2): 121-132 DOI:10.1038/nrg3642 |
TANG S J, LI S F, ZHAO J L, et al. Heritability analysis of the late selection generations of Nile Tilapia (Oreochromis niloticus) new gift strain using microsatellites. Transactions of Oceanology and Limnology, 2018, 48(5): 171-174 [唐首杰, 李思发, 赵金良, 等. "新吉富"罗非鱼选育后期世代遗传潜力的微卫星分析. 海洋湖沼通报, 2018, 48(5): 171-174] |
TRAPNELL C, WILLIAMS B A, PERTEA G. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation. Nature Biotechnology, 2010, 28(5): 511-515 DOI:10.1038/nbt.1621 |
WU J W, ZHAO J L, ZHAO Y, et al. Effects of high carbonate alkalinity stress on changes in ammonia metabolism expression of Oreochromis niloticus. Journal of Fishery Sciences of China, 2016, 23(6): 1290-1299 [吴俊伟, 赵金良, 赵岩, 等. 高碳酸盐碱胁迫对尼罗罗非鱼氨代谢基因表达变化的影响. 中国水产科学, 2016, 23(6): 1290-1299] |
XIAO J, LI Q Y, TU J P, et al. Stress response and tolerance mechanisms of ammonia exposure based on transcriptomics and metabolomics in Litopenaeus vannamei. Ecotoxicology and Environmental Safety, 2019, 180(5): 491-450 |
XU Y, MI B H, ZHAO X F, et al. Effects on alkalinity stress on blood biochemical indices Leuciscus waleckii. Chinese Journal of Fisheries, 2021, 34(3): 17-21 [徐悦, 米博瀚, 赵雪飞, 等. 碳酸盐胁迫对瓦氏雅罗鱼血液中部分生理生化指标的影响. 水产学杂志, 2021, 34(3): 17-21 DOI:10.3969/j.issn.1005-3832.2021.03.004] |
YAO Z L, LAI Q F, HAO Z R, et al. Carbonic anhydrase 2-like and Na+-K+-ATPase a gene expression in medaka (Oryzias latipes) under carbonate alkalinity stress. Fish Physiology and Biochemistry, 2015, 41(6): 1491-1500 DOI:10.1007/s10695-015-0101-6 |
YAO Z L, WANG H, ZHOU K, et al. Effects of water carbonate alkalinity and pH on survival rate of post larva Litopenaeus vannamei. Chinese Journal of Ecology, 2010, 29(5): 945-950 [么宗利, 王慧, 周凯, 等. 碳酸盐碱度和pH值对凡纳滨对虾仔虾存活率的影响. 生态学杂志, 2010, 29(5): 945-950] |
YAO Z L, YING C Q, ZHOU K, et al. Gene expression profiles of Litopenaeus vannamei in response to carbonate alkalinity stress. Journal of Fishery Sciences of China, 2012, 19(1): 1-12 [么宗利, 应成琦, 周凯, 等. 碳酸盐碱度胁迫下凡纳滨对虾基因的差异表达. 中国水产科学, 2012, 19(1): 1-12] |
YI X F, LAI Q F, SHI J Q, et al. Nitrogenous waste excretion and gene expression of nitrogen transporter in Gymnocypris przewalskii in high alkaline environment. Journal of Fishery Sciences of China, 2017, 24(4): 681-689 [衣晓飞, 来琦芳, 史建全, 等. 高碱环境下青海湖裸鲤氮废物排泄及相关基因的表达规律. 中国水产科学, 2017, 24(4): 681-689] |
YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene ontology analysis for RNA-seq: Accounting for selection bias. Genome Biology, 2010, 11: R14 DOI:10.1186/gb-2010-11-2-r14 |
ZHANG T S. Analysis of animal models and estimation of genetic parameters in Fenneropenaeus chinensis breeding. Doctoral Dissertation of Ocean University of China, 2010, 13-17 [张天时. 中国对虾(Fenneropenaeus chinensis)育种的模型分析与遗传参数评估. 中国海洋大学博士研究生学位论文, 2010, 13-17] |
ZOU H, SUN J, YU F, et al. Effect of cadmium on lysosomes in BRL 3A cells. Chinese Veterinary Science, 2019, 49(12): 1602-1608 [邹辉, 孙建, 于凡, 等. 镉暴露对BRL3A细胞溶酶体的影响. 中国兽医科学, 2019, 49(12): 1602-1608] |



