2. 海南热带海洋学院 热带海洋生物资源利用与保护教育部重点实验室 海南 三亚 572022
2. Key Laboratory of Utilization and Conservation for Tropical Marine Bioresources, Ministry of Education, Hainan Tropical Ocean University, Sanya 572022, China
淡水鱼类是水域生态系统的重要组成部分,对保持生态平衡、维系人与自然和谐发展起着至关重要的作用(程馨雨等, 2019)。海南省是中国唯一的热带岛屿省份,气候条件独特,岛内存在丰富的淡水鱼类特有种(李高俊等, 2020)。然而,由于过度捕捞、环境污染等原因,海南岛淡水鱼类生境遭到严重破坏,淡水鱼类资源量急剧衰退。根据2015—2017年对海南省淡水鱼类的全面调查数据(申志新等, 2018),与1986年相比,海南省已有14.15%的淡水鱼类处于野外缺失状态,大鳞鲢(Hypophthalmichthys harmandi)、大鳞光唇鱼(Acrossocheilus ikedai)、海南鳅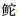
通用引物对eDNA宏条形码技术的应用具有直接影响。已知的鱼类eDNA宏条形码通用引物主要位于线粒体(mtDNA) 12S rRNA (以下简称12S)、16S rRNA (以下简称16S)基因区(Zhang et al, 2020),这些核糖体基因区引物普遍存在部分近缘鱼类难以识别的问题。如蒋佩文等(2022)基于CO Ⅰ和12S基因构建珠江河口鱼类eDNA宏条形码数据库发现,172种鱼类的COⅠ条形码可形成明显的条形码间隙,全部鱼类均可有效鉴定;而12S条形码则有11种鱼类(占总种类的6.4%)存在区分困难的情况。Schenekar等(2020)、陈锦孝(2022)在各自的鱼类eDNA研究中也发现了此问题。此外,相比于COⅠ条形码,目前公共数据库内的12S和16S条形码序列数量不足。陈治等(2022)统计了123种(基于COⅠ)、117种(基于12S)、115种(基于16S)海南岛淡水鱼类在NCBI数据库内的参考序列情况,发现平均每种鱼类有21.98条(COⅠ)、5.54条(12S)、6.40条(12S)参考序列。这表明12S和16S条形码覆盖的地理群体更少,不能有效反映不同地理群体的条形码变异情况。针对该问题,Sato等(2018)基于标本馆样品和Mitofish数据库构建专门的鱼类12S eDNA宏条形码数据库,但该数据库的建设时间较短,目前仅完成了数千种鱼类的参考序列构建,且以海洋鱼类为主。因此,短期内该数据库无法为淡水鱼类eDNA研究提供广泛支持。从近缘鱼类鉴定准确性及可用参考序列两方面考虑,有必要针对海南岛淡水鱼类开发eDNA宏条形码COⅠ通用引物。本研究将在此方面进行尝试,以期为海南岛淡水鱼类多样性研究提供一定的技术支撑。
1 材料与方法本研究将基于数据库(自建+公共)内已有序列设计多组COⅠ条形码引物;基于课题组积累的海南岛淡水鱼类样品使用PCR技术初步筛选所设计引物。野外采集水样,通过高通量测序技术比较所设计引物与已有引物的鱼类多样性调查差异。实验室饲养不同密度组的鲤鱼(Cyprinus carpio carpio),考察所设计引物的定量准确性。
1.1 目标序列筛选或扩增根据《海南淡水及河口鱼类图鉴》(申志新等, 2021)、《海南岛淡水及河口鱼类原色图鉴》(李新辉等, 2020)、《海南岛淡水及河口鱼类志》(珠江水产研究所等, 1986)等资料统计海南岛淡水鱼类名录(含对应拉丁学名)。登录NCBI数据库(https://ncbi.nlm.nih.gov/),下载对应物种的线粒体COⅠ条形码seq文件(优先下载线粒体全序列、含有标本凭证信息和来自海南岛及临近水域的序列)。同时,采用Ward等(2005)设计的引物组合扩增实验室积累的部分海南岛淡水鱼类COⅠ条形码片段。PCR引物(Fish-F1/F2和Fish-R1/R2)见Ward等(2005),反应条件和步骤参考蒋佩文等(2022)。使用DNAstar软件包中的SeqMan程序核对序列长度、方向及所在链,删除overlap区短、含有简并碱基或插入/缺失位点的序列。
1.2 侧翼保守区筛选及引物设计使用MegAlign程序中的“ClustalW method”多重比对功能生成“.pau”文件,基于DnaSP 5.01程序将该文件转化为“.meg”文件;使用MEGA7软件将保守位点用绿色背景标注并生成“.xlsx”文件。采用目测法筛选侧翼保守区:保守区长度≥18 bp,密码子第1、2位点不发生任何变异,密码子第3位点如有变异则必须仅为转换。将引物序列输入Primer Premier 6.0软件,查看退火温度(annealing temperature, Tm)、GC含量等基本信息,并基于默认参数记录引物Premier评分等级:理论扩增效率≥80%的评分等级为Best,80% > 扩增效率≥50%的评分等级为Good,扩增效率 < 50%的评分等级为Poor。
1.3 PCR筛选共有72种海南岛淡水鱼类(见表 1中用*标注的物种)用于PCR筛选。为了减少工作量,首轮PCR统一使用“Fish-R1+Fish-R2”(1∶1混合,作为反向引物)或“Fish-F1+Fish-F2”(1∶1混合,作为正向引物)作为本研究所设计引物的配套引物进行单引物筛选;次轮PCR则对首轮PCR筛选出的单引物进行排列组合,从中筛选出最优引物组合。PCR体系总体积为25 μL,包括PCR混合液(天根生化科技有限公司) 12.5 μL、灭菌蒸馏水10 μL、上下游引物(10 μmol/L)各1 μL、DNA样品0.5 μL。PCR条件为94 ℃预变性5 min;94 ℃变性20 s,Tm温度下退火20 s (Tm= “正向引物Tm+反向引物Tm”的平均值),72 ℃延伸20 s,共39个循环;最后72 ℃再延伸5 min。扩增后对PCR产物进行凝胶电泳检测,各取2.5 μL的PCR产物于1%的琼脂糖凝胶中,电压220 V,电泳5 min,最后在凝胶成像系统上拍照。将jpg格式胶图导入Photoshop 2022软件,调节图片亮度,分别统计原图状态及胶图上100、200、500、1 000和2 000 bp marker恰好肉眼不可见(以下简称marker不可见)时扩增条带情况。
|
|
表 1 本研究用于引物设计的鱼类名录及序列号 Tab.1 Species and accession number of fishes used for primer design in this study |
为了方便运输、降低eDNA降解风险,在G98海南环岛高速与南渡江(编号ND: 110°25ʹ33ʺE, 19°56ʹ54ʺN)、昌化江(编号CH: 108°54ʹ31ʺE, 19°13ʹ56ʺN)、万泉河(编号WQ: 110°26ʹ30ʺE, 19°15ʹ12ʺN)交汇处共设置3个水样采集点,每个取样点取10个平行样。每个平行样用直径47 mm、孔径0.45 μm的WCN硝酸纤维滤膜抽滤5 L。抽滤后的滤膜用液氮冷冻保存后送至华大基因青岛研究院进行高通量测序。所用引物为本研究PCR筛选出的COⅠ引物与MiFish-U (12S)引物(Miya et al, 2015)。建库类型为全基因组测序(whole genome sequencing, WGS),测序平台为BGISEQ-500RS。对测序后的序列进行质控等生物信息学操作后得到可操作分类管理单元(operational taxonomic units, OTUs),将OTUs与DNA条形码参考数据库(自建数据库及GenBank库、MitoFish库等)进行BLAST注释分析。对于相似性≥99%的鱼类,记录其所在的种;相似性在97%~99%的鱼类,记录其所在的属;相似性在95%~97%的鱼类,记录其所在的科(Miya et al, 2015)。同时,基于Kimura双参数模型(Kimura-2-parameter, K2P),采用最大似然法(maximum likelihood, ML)计算OTUs间的两两遗传距离(pairwise distance),评估不同引物的物种鉴别差异。
使用非度量多维尺度(non-metric multidimensional scaling, NMDS)方法对鱼类出现结果[0, 1]矩阵进行排序,该分析在Canoco 5.0软件中完成(魏亚男等, 2017)。选用Bray-Curtis距离,因其在大范围和小范围的坐标轴上都具有稳健性(Kruskal et al, 1964)。分析结果以胁强系数(stress)作为评判标准:当stress < 0.2时,认为可以用NMDS的二维点图表示,该图形有一定的解释意义;当stress < 0.1时,认为该排序是一个好的排序;当stress < 0.05时,则认为该排序结果具有很好的代表性(Newmaster et al, 2008; 魏亚男等, 2017)。NMDS运算的步骤如下:(1)将鱼类出现情况(仅分析确定种,未定种、属及属以上阶元不分析)处理为[0, 1]矩阵,鱼类出现为1,不出现为0;以“水系+引物名”为首行储存xlsx格式的[0, 1]矩阵;(2)打开Canoco 5.0软件,加载xlsx文档,给定table name,选择“import all species as factors”,其余选择默认参数,完成矩阵导入;(3)“analyses”对话框内点击“new”选项,选择“Canoco Adviser”,调出NMDS分析程序;(4)“stress formula”选择“type 1”,“treatment of ties in distance”选择“secondary”,其余选择默认参数。
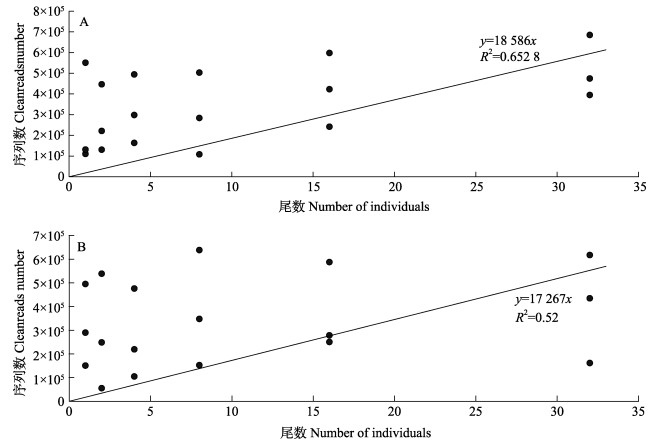
1.5 “生物量-拷贝数”定量拟合于热带海洋生物资源利用与保护教育部重点实验室内设置6个容积为1 m3、具有相同换水装置的圆形水桶,用于比较所用引物的eDNA定量差异。每个水桶用10%次氯酸钠充分消毒后,在水桶中加入0.5 m3淡水(淡水已进行多级沉淀、过滤和消毒)。水桶内的水体源头独立、无相互交汇。水体更新速度恒定保持为0.5 L/min,多余水体从溢流管流出,使体积保持不变。分别在这6个水桶中加入1、2、4、8、16、32尾均重为23.68 g的鲤鱼,在充分供氧条件下,统一喂食一定数量的鱼饵。在加入鲤鱼15 d后,对每个水桶采集500 mL水样,共取3个平行样,同时取等体积纯净水作为空白对照组。取样方式及滤膜处理方式与野外式样相同。使用R软件(version 4.0.3)中的“basicTrendline”包对剔除阴性对照后的高通量测序分子拷贝数及不同尾数组进行散点图拟合,计算“生物量-拷贝数”定量拟合线性回归方程和相关系数。
2 结果与分析 2.1 序列筛选结果经物种名核对、碱基比对等过程筛选,本研究用于引物设计的鱼类共计8目、26科、101属、150种(表 1)。
2.2 侧翼保守区简介基于150种鱼类COⅠ序列,共筛选出6个侧翼保守区(图 1,侧翼保守区A~F)。其中,侧翼保守区F与E距离较远(> 300 bp),其扩增子长度超出常见eDNA分子长度及高通量测序读长范围(2×150 bp) (Turner et al, 2014)。侧翼保守区A与B、C与D距离不足100 bp,扩增子长度较短不利于近缘鱼类区分。因此,本研究在2×150 bp测序读长范围内选取扩增子最长的前3个侧翼保守区组合——A+D、A+E、B+E进行正、反向引物设计。为了比较,本研究同时标注了标准的鱼类DNA条形码(650 bp或652 bp)及其他研究者设计的2对通用性较高的eDNA宏条形码COⅠ通用引物所在位置(图 1,Fish-F1/F2、Fish-R1/R2、PS1、Uni-Minibar) (Zhang et al, 2020)。
|
图 1 侧翼保守区在COⅠ基因前半段(776 bp/1 551 bp)内的相对位置 Fig.1 Relative positions of flanking conserved regions within the first half of COⅠ gene (776 bp/1 551 bp) 以鲤鱼(NCBI序列号:JN105352)为标注模板。 Cyprinus carpio carpio (accession number: JN105352) was used as the labeling template. |
除侧翼保守区A存在2个完全保守密码子外,其余密码子第3位点均发生转换变异(表 2),需要使用适量简并碱基提高“引物序列-DNA模板”匹配度。根据简并碱基数的不同,本研究对每个侧翼保守区分别设计了5~8种引物,其碱基组成、GC含量和Tm值见表 3。引物Premier评分结果显示,26条引物中,3条引物评分处于“Best”等级,表明引物在退火温度、GC含量、二级结构等方面更符合理想引物的标准;17条引物评分处于“Good”等级,理论上仍能够进行DNA模板有效扩增;6条引物评分处于“Poor”等级,不满足常用引物有关标准。
|
|
表 2 本研究中150种鱼类在侧翼保守区位置的碱基分布及已有引物的碱基组成 Tab.2 Base distribution of the 150 fish species in the flanking conserved regions and base composition of existing primers |
|
|
表 3 本研究中简并引物基本信息 Tab.3 Basic information of degenerate primers in this study |
首轮PCR筛选结果显示,20条引物中,侧翼保守区E内的5条引物扩增成功率较低,引物的通用性差。HN-A-F1/F2/F3/F4、HN-B-F1/F2/F3/F4、HN-D-R2/R3/R4扩增出的单一条带物种数较多(表 3);逐步调低图片亮度,当胶图上的marker不可见时,HN-A-F4、HN-B-F3/F4、HN-D-R3/R4存在条带痕迹的物种数较多(亮度大于marker的单一条带物种数≥70种,表 3)。因此,这5条引物的通用性更高。
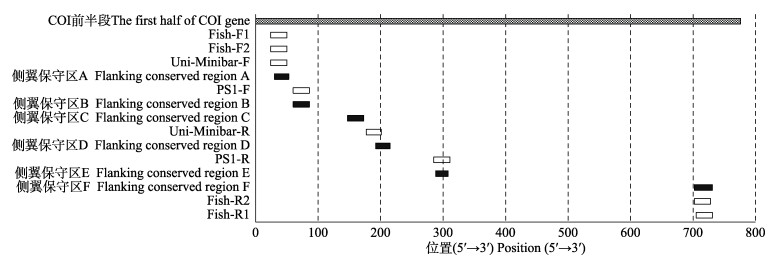
对上述5条引物形成的3 (正向) × 2 (反向)套引物组合进行次轮PCR筛选,结果显示,全部72种鱼类扩增成功率均为100% (PCR胶图未展示)。HN-A-F4、HN-B-F3/F4作为HN-D-R3/R4的正向引物产生的扩增子长度分别为139 bp、106 bp/106 bp。扩增子长度越长,物种鉴定准确性越高,因此,HN-A-F4比HN-B-F3/F4更有优势。虽然HN-D-R3/R4作为HN-A-F4的反向引物产生的扩增子长度相同,且二者的扩增成功率均为100% (图 2A: HN-D-R3; 图 2B: HN-D-R4),但逐步调低图片亮度,当胶图上的marker不可见时HN-D-R3产生的扩增子均存在痕迹(图 2C),而HN-D-R3扩增的23、24、43号样品条带彻底消失(图 2D)。HN-D-R3产生的条带更亮、扩增效率高于HN-D-R4 (见图 2C、图 2D),因此更适合作为反向引物。综上所述,次轮PCR筛选中表现最优的引物组合为“HN-A-F4、HN-D-R3”(以下简称HN-COⅠ)。

|
图 2 HN-D-R3与HN-D-R4的扩增效果比较 Fig.2 Comparison of amplification effect of HN-D-R3 and HN-D-R4 A:HN-D-R3扩增原图;B:HN-D-R4扩增原图;C、D:原图亮度同步调低至只剩750 bp marker可见时的扩增子痕迹(C: HN-D-R3; D: HN-D-R4);1~72:物种编号;M:Marker。 A: Original image of HN-D-R3 amplification; B: Original image of HN-D-R4 amplification; C, D: Amplicon trace when the brightness of the original image was synchronously reduced to only 750 bp marker visible (C: HN-D-R3; D: HN-D-R4); 1~72: Numbers of species; M: Marker. |
全部30个水样稀释曲线(rarefaction curves)末端趋于平滑,样品测序数据量充足,可用于后续分析。虽然生物信息学过程已剔除阴性对照中出现的序列,但水样中仍检测出了一定比例的河口及海洋鱼类OTUs。由于这部分序列仍是引物灵敏性和通用性的反映,因此,本研究未进一步对其进行剔除。MiFish-U的扩增效率高于HN-COⅠ,前者产生的待分析序列(clean reads)总量是后者的1.29倍(表 4);然而,HN-COⅠ对鱼类eDNA靶向性更强,待分析序列内鱼类序列总数和序列平均占比分别是MiFish-U的1.22倍和1.99倍(表 4)。OTUs也表现出同样的现象:MiFish-U共扩增出477种OTUs,其中鱼类OTUs共65种(占比为13.63%);HN-COⅠ共扩增出406种OTUs,其中鱼类OTUs共86种(占比为21.18%) (表 4)。
|
|
表 4 水样高通量测序结果简介 Tab.4 Brief introduction to water sample results after high-throughput sequencing |
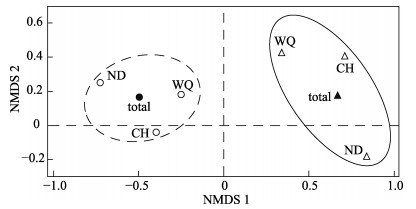
OTUs注释结果显示,HN-COⅠ产生的86种鱼类OTUs注释到种、属、科及科以上水平的数目分别为70 (81.40%)、10 (11.63%)、6 (6.98%);MiFish-U产生的65种鱼类OTUs中注释到种、属、科及科以上水平的数目分别为53(81.54%)、3(4.62%)、9 (13.85%)。种水平的OTUs共注释81种鱼类,其中共享鱼类42种,独享鱼类28种(HN-COⅠ)和11种(MiFish-U)。基于81种鱼类的出现情况进行“引物+水样”的NMDS排序结果显示,样品聚类成边界明显的2组。表明HN-COⅠ、MiFish-U的鱼类多样性调查结果存在较大不同。此外,HN-COⅠ的NMDS结果离散性高于MiFish-U,暗示基于该引物组合的海南岛鱼类多样性调查差异大。NMDS分析结果的胁强系数为0.15,说明将2个引物调查结果划归不同组具有一定的解释意义。
|
图 3 不同引物的NMDS分析 Fig.3 NMDS analysis of different primers 圆形:MiFish-U扩增结果;三角形:HN-COⅠ扩增结果 Round: Amplification results of MiFish-U; Triangle: Amplification results of HN-COⅠ |
42种共享鱼类的两两遗传距离分别为0.007~ 0.377 (0.234±0.087) (HN-COI)和0.006~0.462 (0.298± 0.135) (MiFish-U);对42种鱼类所在阶元进行分组,属内物种间两两遗传距离分别为0.007~0.251 (0.156±0.050) (COⅠ)和0.006~0.202 (0.099 4±0.035) (MiFish-U);HN-COⅠ引物组合的属内物种遗传距离最小值及平均值均高于MiFish-U,分别为后者的1.23倍和1.57倍,暗示HN-COⅠ的近缘鱼类的区分能力高于MiFish-U。此外,全部42种共享鱼类的两两遗传距离的变异系数分别为37.28% (HN-COⅠ)和45.42% (MiFish-U);属内物种间两两遗传距离的变异系数分别为31.88% (HN-COⅠ)和35.51% (MiFish-U)。与MiFish-U相比,HN-COⅠ的变异系数更小,表明其物种鉴定稳定性高于MiFish-U。
2.6 “生物量-拷贝数”定量拟合结果将不同密度的鲤鱼与高通量测序后的分子拷贝数进行拟合:HN-COⅠ、MiFish-U的线性回归方程分别为y=18 586x (R2=0.65)和y=17 267x (R2=0.52) (图 4)。2种引物拟合方程的相关系数均较低,表明尚不能通过eDNA序列反映水槽中鲤鱼的生物量。拟合结果还表明,饲养15 d后低密度组的eDNA浓度极高,6个密度组内的鲤鱼eDNA可能均处于饱和状态;同一密度组内却又存在平行样间分子拷贝数差异较大的现象(图, 4),表明水体内eDNA分布并不均匀。此外,本研究尝试了二项式、指数、对数、幂函数的拟合效果,其相关性均较低(R2≤0.565) (图表略)。
|
图 4 室内条件下鲤鱼eDNA分子拷贝数与其生物量之间的相关关系 Fig.4 Correlation between eDNA reads and biomass of Cyprinus carpio carpio under indoor conditions A: HN-COⅠ; B: MiFish-U |
本研究中,有5条引物的通用性较高(表 3),其中以HN-COⅠ效果最佳,该引物组合的扩增子长度达到139 bp,PCR成功率为100%且扩增条带亮度均大于marker。表明以海南岛淡水鱼类为调查对象时,能够筛选出理想的鱼类eDNA宏条形码COⅠ通用引物。然而,PS1及Uni-Minibar在本研究中表现较差——72种鱼类PCR成功数分别为46种和25种,扩增条带亮度大于marker的物种数更是仅有22种和9种(表 3)。Zhang等(2020)基于北京市水样进行eDNA宏条形码引物比较亦表明:PS1及Uni-Minibar在全部22对测评引物中处于中等偏下水平。然而,在Balasingham等(2018)和Meusnier等(2008)的原始研究中,PS1、Uni-Minibar却展现了极高的通用性。PS1、Uni-Minibar及HN-COⅠ在不同研究中的表现说明,eDNA宏条形码COⅠ通用引物具有明显的地域性,可能无法适用于原始调查水域/类群之外的鱼类多样性研究。造成引物地域性的主要原因,是COⅠ基因变异速率远高于12S、16S,因而找不到严格的侧翼保守区(Riaz et al, 2011; Freeland, 2017; Evans et al, 2018)。综合已有研究结果,本研究认为,鱼类eDNA宏条形码COⅠ通用引物的具体应用需要密切考虑其设计背景和原始调查水域/类群,直接使用他人筛选出的COⅠ通用引物存在一定的物种调查假阴性风险。
3.2 COⅠ通用引物筛选参考及建议虽然鱼类eDNA宏条形码COⅠ通用引物具有一定的地域性,但本研究为其他类似研究提供了一个引物筛选参考。本研究筛选出的6个侧翼保守区与Fish-F、Fish-R、PS1、Uni-Minibar等已知的鱼类COⅠ条形码引物位置重叠(图 1),并且侧翼保守区涉及的鱼类总数在数百种以上(本研究:150种;Fish-F、Fish-R:207种;PS1:114种;Uni-Minibar:187种) (Ward et al, 2005; Meusnier et al, 2008; Balasingham et al, 2018)。因此,图 1标注的侧翼保守区位置具有充分借鉴意义。尽管不同类群在具体碱基构成上可能存在一定差异,如150种海南岛淡水鱼类在HN-A-F4引物3´端存在5个连续保守碱基,Fish-F1/F2、Uni-Minibar-F在相同位置却只存在2个连续保守碱基(表 2),但其他水域的鱼类多样性调查仍然可以以此为导引,通过调整引物起始位置和修改碱基组成,筛选所需的COⅠ通用引物。
侧翼保守区长度会影响引物通用性。本研究中HN-E系列的6条引物PCR成功率明显偏低(表 3)。虽然侧翼保守区E的长度达到20 bp。有研究认为,当引物长度在18 bp以上即可保证引物自身有极高的扩增效率(Kitano et al, 2007; Riaz et al, 2011; Sultana et al, 2018)。但由于密码子第3位点变异碱基的存在,侧翼保守区E与eDNA模板的有效结合长度最小值只有14 bp,导致引物筛选过程中有10~26种鱼类扩增失败。随机扩增片段多态性(random amplification polymorphic DNA, RAPD)技术的广泛使用(所用引物在10 bp左右),也从侧面印证了引物长度的降低会导致PCR出现多条带或者无条带的结果(何舜平等, 2000; 尹绍武等, 2006; 鹿志创等, 2007)。相比之下,Fish-F1/F2及Fish-R1/R2长度均为26 bp,即使密码子第3位点完全不匹配,其有效结合长度也至少为18 bp。这4条引物在未使用任何简并碱基的情况下,PCR条带亮度大于marker的物种数仍高达62~68种;HN-B-F3/F4的PCR成功率更是高达100%。Menning等(2020)和Sultana等(2018)针对阿拉斯加、马来西亚本土鱼类进行的COⅠ通用引物筛选,结果也呈现出类似规律:长度短的引物更容易被率先淘汰。本研究不推荐在短侧翼保守区内进行引物设计,如有必要则应该对引物5´端进行碱基加长修饰。
COⅠ通用引物设计过程中,应使用适量简并碱基。Miya等(2015)在引物筛选过程中发现,即使只添加1~2个简并碱基也会导致PCR扩增中出现不同程度的弥散带,因此,采取将MiFish-U (硬骨鱼类)与MiFish-E (软骨鱼类)引物分开设计的原则。Ward等(2005)在设计标准的鱼类DNA条形码COⅠ引物(Fish-F1/F2及Fish-R1/R2)过程中,也采取了“分别设计多对正、反向引物,不同引物组合应用”的策略。然而,由于没有简并碱基,MiFish-U、Fish-F1/F2及Fish-R1/R2始终存在部分鱼类无法扩增的问题(Ward et al, 2005; Miya et al, 2015);并且多对引物搭配使用会显著提高鱼类eDNA研究的工作量和PCR测序成本。针对此问题,Ivanova等(2007)和Minamoto等(2012)均通过添加简并碱基来提高“引物序列-DNA模板”的匹配度。本研究支持使用适量简并碱基的做法:用于首轮PCR筛选的20条引物中,使用1~2、3~4、5个及以上简并碱基的引物,其PCR条带亮度大于marker的物种数分别为25~66 (54.38±14.13)、49~71 (65.13±9.67)和19~59 (47.25±19.02)种。次轮PCR筛选结果也显示,HN-COⅠ等存在3~4个简并碱基的引物PCR成功率均为100%。然而,当简并碱基在5个及以上时,72种海南岛淡水鱼类的PCR成功数开始明显下降;特别是HN-B-F6/F7/F8、HN-D-R7、HN-E-R5/R6引物,由于自身含有过多的简并碱基,已经无法满足Premier软件的基本要求。PS1对海南岛淡水鱼类的扩增效果较差,可能也受到了反向引物简并碱基过多的影响(Minamoto et al, 2012; Miya et al, 2015)。
3.3 HN-COⅠ与MiFish-U的比较通用性和物种识别能力是鱼类eDNA宏条形码引物的2个核心指标。Collins等(2019)和Milan等(2020)认为:12S和16S引物的通用性一般高于COⅠ和Cytb。本研究支持此结论。30个水样高通量测序结果显示,MiFish-U产生的待分析序列总数和OTUs总数分别是HN-COⅠ的1.29倍和1.18倍(表 4)。然而,MiFish-U可能存在通用性过高的风险——虽然该引物是Miya等(2015)基于880种鱼类线粒体全序列筛选所得,但引物序列对微生物、鸟类、哺乳类等众多类群却普遍适用。过高的通用性会引起PCR竞争性扩增,导致量少的鱼类eDNA模板在扩增过程中因受到其他量多的eDNA模板(如微生物)的竞争而逐步丢失(Kobayashi et al, 2000; 华育平, 2002; 李朋祥, 2012)。本研究中,HN-COⅠ产生的鱼类序列总数及其占比、鱼类OTUs总数及其占比分别为MiFish-U的1.22倍(2 264 965/1 863 905)和1.57倍(25.39%/16.16%)、1.32倍(86/65)和1.57倍(25.39%/16.16%) (表 4)。HN-COⅠ引物对海南岛淡水鱼类具有更高的靶向性,能够在降低微生物等非目标物种有效扩增的同时产生更多的鱼类序列或OTUs,进而满足海南岛淡水鱼类多样性调查需求。
HN-COⅠ的物种识别能力高于MiFish-U。42种共享鱼类遗传距离分析结果显示,HN-COⅠ属内物种遗传距离最小值及平均值是MiFish-U的1.23倍和1.57倍;HN-COⅠ在物种注释过程中停滞在科及科以上水平的OTUs占比则仅为MiFish-U的0.50倍(6.98%/13.85%)。陈治等(2022)通过比较数十种海南岛淡水鱼类的eDNA宏条形码参考数据库发现,HN-COⅠ和MiFish-U分别有5.56% (4种)和16.67% (12种)鱼类存在条形码序列完全相同的现象。因此,MiFish-U扩增出的部分OTUs可能是多个种的复合OTUs (Schenekar et al, 2020; 蒋佩文等, 2022; 王梦等, 2022)。这可能也是该引物的鱼类OTUs比HN-COⅠ少的原因之一。
“引物+水样”的NMDS聚类形成边界明显的两组(胁强系数=0.15),表明HN-COⅠ、MiFish-U的鱼类多样性调查结果存在较大不同。该结果符合Taberlet等(2012)和Pawlowski等(2020)提出的鱼类eDNA调查应多基因片段并用的观点。NMDS图中南渡江、昌化江、万泉河的位点距离较远,该结果也与部分已有研究一致(李高俊等, 2020),表明eDNA能够在中尺度上有效反映鱼类的组成差异(Kelly et al, 2014; Deiner et al, 2017; Yamamoto et al, 2017)。并且,基于HN-COⅠ的南渡江、昌化江、万泉河三大水系NMDS结果离散性高于MiFish-U。海南岛高山地形复杂、环境异质化明显,众多河流互不干扰且呈放射状向海岛四周奔流,因此,物种的隔离效应极其明显(陈辈乐等, 2008)。HN-COⅠ结果可能更符合海南岛淡水鱼类的这一时空分布特点。然而,由于没有同步采用传统调查方法采集鱼类样品,上述推测还需要进一步验证。
虽然有观点认为,eDNA能够反映鱼类的生物量(Doi et al, 2017; Takahara et al, 2019),但本研究2种引物的“生物量-拷贝数”定量拟合方程相关系数较低。这可能是由于本研究为室内实验,水体中的eDNA浓度较高,导致全部水样经过少数几个PCR循环后均达到峰值。Pilliod等(2013)和Anais等(2016)的研究也未能通过eDNA浓度准确反映物种的生物量和养殖密度。本研究同一密度组内却存在平行样间分子拷贝数差异较大的现象(图 4),表明无论鲤鱼密度高低都会出现检出生物量不稳定的风险。eDNA在水体的分布可能并不均匀,而是呈斑片状分布,这与Eichmiller等(2014)和吴昀晟等(2019)的研究报道相同。鱼类eDNA的产生、降解动力学过程极为复杂,目前没有研究能够准确阐明eDNA的微观存在和变化规律。后续还需要进行大量实验厘清HN-COⅠ、MiFsh-U等引物的eDNA定量结果准确性。
4 结论本研究基于8目、26科、101属、150种鱼类COⅠ序列筛选出6个侧翼保守区,从中设计了26条引物,经72种鱼类、30个水样验证后表现最优的引物组合为HN-COⅠ (HN-A-F4、HN-D-R3)——该引物组合扩增成功率为100%且扩增条带单一明亮,扩增子长度为139 bp;高通量测序获得的鱼类OTUs总数是MiFish-U的1.32倍;属内物种扩增子两两遗传距离最小值及平均值分别是MiFish-U的1.23倍和1.57倍。HN-COⅠ的OTUs注释结果及NMDS聚类图形同样与MiFish-U存在较大不同。HN-COⅠ虽然无法准确反映鲤鱼的生物量,但对海南岛淡水鱼类具有更高的靶向性,该引物组合更有利于海南岛淡水鱼类的检出和准确鉴定。
ANAIS L R, ROSABAL M, BERNATCHEZ L. Estimating fish abundance and biomass from eDNA concentrations: Variability among capture methods and environmental conditions. Molecular Ecology Resources, 2016, 16(6): 1401-1414 DOI:10.1111/1755-0998.12522 |
BALASINGHAM K D, WALTER R P, MANDRAK N E, et al. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries. Molecular Ecology, 2018, 27(1): 112-127 DOI:10.1111/mec.14395 |
CHEN B L, CHEN X L. Species diversity and distribution of freshwater fishes at Mt. Yinggeling, Hainan Island, China. Biodiversity Science, 2008, 16(1): 44 [陈辈乐, 陈湘粦. 海南鹦哥岭地区的鱼类物种多样性与分布特点. 生物多样性, 2008, 16(1): 44-52] |
CHEN J X. Fish species composition and diversity in the northern and southern foothills of the Qinling Mountains based on environmental DNA high-throughput barcoding technology. Masterxs Thesis of Northwest A & F University, 2022 [陈锦孝. 基于环境DNA高通量条形码技术的秦岭南北两麓鱼类物种组成及多样性研究. 西北农林科技大学硕士研究生学位论文, 2022]
|
CHEN Z, CAI X W, ZHANG Q F, et al. Preliminary construction and comparative analysis of environmental DNA metabarcoding reference database of freshwater fishes in Hainan Island. South China Fisheries Science, 2022, 18(3): 1-12 [陈治, 蔡杏伟, 张清凤, 等. 海南岛淡水鱼类环境DNA宏条形码参考数据库的初步构建及比较分析. 南方水产科学, 2022, 18(3): 1-12] |
CHENG X Y, TAO J, WU R D, et al. Functional ecology of freshwater fish: Research progress and prospects. Acta Ecologica Sinica, 2019, 39(3): 810-822 [程馨雨, 陶捐, 武瑞东, 等. 淡水鱼类功能生态学研究进展. 生态学报, 2019, 39(3): 810-822] |
COLLINS R A, BAKKER J, WANGENSTEEN O S, et al. Non-specific amplification compromises environmental DNA metabarcoding with COⅠ. Methods in Ecology and Evolution, 2019, 10(11): 1985-2001 DOI:10.1111/2041-210X.13276 |
DEINER K, BIK H M, ELVIRA MACHLER, et al. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities. Molecular Ecology, 2017, 26(21): 5872-5880 DOI:10.1111/mec.14350 |
DOI H, INUI R, AKAMATSU Y, et al. Environmental DNA analysis for estimating the abundance and biomass of stream fish. Freshwater Biology, 2017, 62(1): 30-39 DOI:10.1111/fwb.12846 |
EICHMILLER J J, BAJER P G, SORENSEN P W. The relationship between the distribution of common carp and their environmental DNA in a small lake. PLoS One, 2014, 9(11): e112611 DOI:10.1371/journal.pone.0112611 |
EVANS N T, LAMBERTI G A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research, 2018, 197(4): 60-66 |
FICETOLA G, MIAUD C, POMPANON F, et al. Species detection using environmental DNA from water samples. Biology Letters, 2008, 4(4): 423-425 DOI:10.1098/rsbl.2008.0118 |
FREELAND J R. The importance of molecular markers and primer design when characterizing biodiversity from environmental DNA. Genome, 2017, 60(4): 358-374 DOI:10.1139/gen-2016-0100 |
HE S P, WANG W, CHEN Y Y. The RAPD analysis and the phylogenetic concerning for primitive Cyprinidae. Acta Hydrobiologica Sinica, 2000, 24(2): 101-106 [何舜平, 王伟, 陈宜瑜. 低等鲤科鱼类RAPD分析及系统发育研究. 水生生物学报, 2000, 24(2): 101-106] |
HUA Y P. Development of a competitive PCR assay for quantifying sheep-associated malignant catarrhal fever viral DNA. Chinese Journal of Veterinary Medicine, 2002, 38(9): 10-13 [华育平. 定量测定羊关联性恶性卡他热病毒DNA的竞争性PCR方法的建立. 中国兽医杂志, 2002, 38(9): 10-13] |
IVANOVA N V, ZEMLAK T S, HANNER R H, et al. Universal primer cocktails for fish DNA barcoding. Molecular Ecology Notes, 2007, 7(4): 544-548 DOI:10.1111/j.1471-8286.2007.01748.x |
JIANG P W, LI M, ZHANG S, et al. Construction of DNA meta-barcode database of fish in Pearl River estuary based on mitochondrial cytochrome COⅠ and 12S rDNA gene. South China Fisheries Science, 2022, 18(3): 13-21 [蒋佩文, 李敏, 张帅, 等. 基于线粒体COⅠ和12S rDNA基因构建珠江河口鱼类DNA宏条形码数据库. 南方水产科学, 2022, 18(3): 13-21] |
KELLY R P, PORT J A, YAMAHARA K M, et al. Using Environmental DNA to census marine fishes in a large mesocosm. PLoS One, 2014, 9(1): e86175 DOI:10.1371/journal.pone.0086175 |
KITANO T, UMETSU K, TIAN W, et al. Two universal primer sets for species identification among vertebrates. International Journal of Legal Medicine, 2007, 121(5): 423 DOI:10.1007/s00414-006-0113-y |
KOBAYASHI Y, FORSTER R J, TEATHER R M. Development of a competitive polymerase chain reaction assay for the ruminal bacterium Butyrivibrio fibrisolvens OB156 and its use for tracking an OB156-derived recombinant. FEMS Microbiology Letters, 2000, 188(2): 185-190 DOI:10.1111/j.1574-6968.2000.tb09191.x |
KRUSKAL J B. Nonmetric multidimensional scaling: A numerical method. Psychometrika, 1964, 29(2): 115-129 DOI:10.1007/BF02289694 |
LI G J, GU D E, CAI X W, et al. The species composition and distribution of indigenous freshwater fishes of three main rivers in Hainan Island. Freshwater Fisheries, 2020, 50(6): 15-22 [李高俊, 顾党恩, 蔡杏伟, 等. 海南岛"两江一河"淡水土著鱼类的种类组成与分布现状. 淡水渔业, 2020, 50(6): 15-22] |
LI P X. Analytical methods of gene copy number variations based on multiplex competitive PCR using universal fluorescent primers. Masterxs Thesis of Donghua University, 2012 [李朋祥. 基于荧光通用引物竞争性PCR的基因拷贝数分析方法. 东华大学硕士研究生学位论文, 2012]
|
LI X H, LI J, LI Y F. Primary color Atlas of freshwater and estuarine fishes in Hainan Island. Beijing: Science Press, 2020 [李新辉, 李捷, 李跃飞. 海南岛淡水及河口鱼类原色图鉴. 北京: 科学出版社, 2020]
|
LU Z C, GAO T X, LI Y H, et al. Optimization for the RAPD reaction system in Gadus macrocephalus Tilesius and Cynoglossus semilaevis Gunther. Transactions of Oceanology and Limnology, 2007, 113(3): 96-103 [鹿志创, 高天翔, 李玉晖, 等. 大头鳕和半滑舌鳎RAPD分析条件的优化. 海洋湖沼通报, 2007, 113(3): 96-103] |
MENNING D, SIMMONS T, TALBOT S. Using redundant primer sets to detect multiple native Alaskan fish species from environmental DNA. Conservation Genetics Resources, 2020, 12(1): 109-123 DOI:10.1007/s12686-018-1071-7 |
MEUSNIER I, SINGER G A C, LANDRY J F, et al. A universal DNA mini-barcode for biodiversity analysis. BMC Genomics, 2008, 9(1): 214 DOI:10.1186/1471-2164-9-214 |
MILAN D T, MENDES I S, DAMASCENO J S, et al. New 12S metabarcoding primers for enhanced Neotropical freshwater fish biodiversity assessment. Scientific Reports, 2020, 10(1): 17966 DOI:10.1038/s41598-020-74902-3 |
MINAMOTO T, YAMANAKA H, TAKAHARA T, et al. Surveillance of fish species composition using environmental DNA. Limnology, 2012, 13(1): 193-197 |
MIYA M, SATO Y, FUKUNAGA T, et al. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: Detection of more than 230 subtropical marine species. Royal Society Open Science, 2015, 2(7): 150088 DOI:10.1098/rsos.150088 |
NEWMASTER S G, FAZEKAS A J, STEEVES R A D, et al. Testing candidate plant barcode regions in the Myristicaceae. Molecular Ecology Resources, 2008, 8(3): 480-490 DOI:10.1111/j.1471-8286.2007.02002.x |
PAWLOWSKI J, APOTHELOZ P G L, ALTERMATT F. Environmental DNA: What's behind the term? Clarifying the terminology and recommendations for its future use in biomonitoring. Molecular Ecology, 2020, 29(22): 4258 DOI:10.1111/mec.15643 |
Pearl River Fisheries Research Institute, Shanghai Fisheries University, East China Sea Fishery Research Institute, et al. The freshwater and estuaries fishes of Hainan Island. Guangzhou: Guangdong Science and Technology Press, 1986 [珠江水产研究所, 上海水产大学, 东海水产研究所, 等. 海南岛淡水及河口鱼类志. 广东: 广东科技出版社, 1986]
|
PILLIOD D S, GOLDBERG C S, ARKLE R S, et al. Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples. Canadian Journal of Fisheries and Aquatic Sciences, 2013, 70(8): 1123-1130 DOI:10.1139/cjfas-2013-0047 |
QIAN T Y, WANG W J, LI M, et al. A preliminary study on the vertical distribution of Fenneropenaeus chinensis environmental DNA in the Yellow Sea and its influencing factors. Progress in Fishery Sciences, 2021, 42(2): 1-9 [钱瑭毅, 王伟继, 李苗, 等. 黄海中国对虾环境DNA(eDNA)的垂直分布规律及其影响因素初探. 渔业科学进展, 2021, 42(2): 1-9] |
RIAZ T, SHEHZAD W, VIARI A, et al. ecoPrimers: Inference of new DNA barcode markers from whole genome sequence analysis. Nucleic Acids Research, 2011, 39(21): e145 DOI:10.1093/nar/gkr732 |
SATO Y, MIYA M, FUKUNAGA T, et al. MitoFish and MiFish pipeline: A mitochondrial genome database of fish with an analysis pipeline for environmental DNA metabarcoding. Molecular Biology and Evolution, 2018, 35(6): 1553-1555 DOI:10.1093/molbev/msy074 |
SCHENEKAR T, SCHLETTERER M, LECAUDEY L A, et al. Reference databases, primer choice, and assay sensitivity for environmental metabarcoding: Lessons learnt from a re-evaluation of an eDNA fish assessment in the Volga headwaters. River Research and Applications, 2020, 36(7): 1004-1013 DOI:10.1002/rra.3610 |
SHAN X J, LI M, WANG W J. Application of environmental DNA technology in aquatic ecosystem. Progress in Fishery Sciences, 2018, 39(3): 23-29 [单秀娟, 李苗, 王伟继. 环境DNA (eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29] |
SHEN Z X, LI G J, CAI X W, et al. The evolution and protection of freshwater fish species in Hainan Province. China Fisheries, 2018, 516(11): 56-60 [申志新, 李高俊, 蔡杏伟, 等. 海南省淡水野生鱼类多样性演变及保护建议. 中国水产, 2018, 516(11): 56-60] |
SHEN Z X, WANG D Q, LI G J, et al. Atlas guide to Hainan freshwater and estuarine fish. Beijing: Chinese Agricultural Press, 2021 [申志新, 王德强, 李高俊, 等. 海南淡水及河口鱼类图鉴. 北京: 中国农业出版社, 2021]
|
SULTANA S, ALI M E, HOSSAIN M A M, et al. Universal mini COⅠ barcode for the identification of fish species in processed products. Food Research International, 2018, 105(5): 19-28 |
TABERLET P, COISSAC E, HAJIBABAEI M, et al. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793 DOI:10.1111/j.1365-294X.2012.05542.x |
TAKAHARA T, IKEBUCHI T, DOI H, et al. Using environmental DNA to estimate the seasonal distribution and habitat preferences of a Japanese basket clam in Lake Shinji, Japan. Estuarine, Coastal and Shelf Science, 2019, 221(5): 15-20 |
TAO J, CAO Y, ZUO Q T. Research progress on the application of environmental DNA technology on river ecosystem. Water Resources Protection, 2021, 37(6): 150-156 [陶洁, 曹阳, 左其亭. 环境DNA技术在河流生态系统中的应用研究进展. 水资源保护, 2021, 37(6): 150-156] |
THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 2012, 21(11): 2565-2573 |
THOMSEN P F, WILLERSLEV E. Environmental DNA: An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183(1): 4-18 |
TURNER C R, BARNES M A, XU C C Y, et al. Particle size distribution and optimal capture of aqueous macrobial eDNA. Methods in Ecology and Evolution, 2014, 5(7): 676-684 |
VALENTINI A, TABERLET P, MIAUD C, et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942 |
WANG M, YANG X, WANG W, et al. Fish diversity in Chongqing section of the national nature reserve for rare and endemic fish in the upper Yangtze River based on eDNA technology. Acta Hydrobiologica Sinica, 2022, 46(1): 2-16 [王梦, 杨鑫, 王维, 等. 基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究. 水生生物学报, 2022, 46(1): 2-16] |
WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia's fish species. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360(1462): 1847-1857 |
WEI Y N, WANG X M, YAO P C, et al. Comparison of species resolution rates of DNA barcoding for Chinese coastal halotolerant plants. Biodiversity Science, 2017, 25(10): 1095-1104 [魏亚男, 王晓梅, 姚鹏程, 等. 比较不同DNA条形码对中国海岸带耐盐植物的识别率. 生物多样性, 2017, 25(10): 1095-1104] |
WU Y S, TANG Y K, LI J L, et al. The application of environmental DNA in the monitoring of the Yangtze finless porpoise, Neophocaena phocaenoides asaeorientalis. Journal of Fishery Sciences of China, 2019, 26(1): 124-132 [吴昀晟, 唐永凯, 李建林, 等. 环境DNA在长江江豚监测中的应用. 中国水产科学, 2019, 26(1): 124-132] |
YAMAMOTO S, MASUDA R, SATO Y, et al. Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea. Scientific Reports, 2017, 7(1): 40368 |
YIN S W, HUANG H, LIAO J Q, et al. RAPD analysis of molecular genetic diversity and genetic relationship of four grouper species. Acta Oceanologica Sinica, 2006, 28(6): 119-126 [尹绍武, 黄海, 廖经球, 等. 4种石斑鱼的分子遗传多样性和亲缘关系的RAPD分析. 海洋学报(中文版), 2006, 28(6): 119-126] |
YOCCOZ N G. The future of environmental DNA in ecology. Molecular Ecology, 2012, 21(8): 2031 |
ZHANG H, XIAN W W. Application of environmental DNA technology in ecological conservation and monitoring. Marine Sciences, 2020, 44(7): 96-102 [张辉, 线薇薇. 环境DNA技术在生态保护和监测中的应用. 海洋科学, 2020, 44(7): 96-102] |
ZHANG S, ZHAO J, YAO M. A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish. Methods in Ecology and Evolution, 2020, 11(12): 1609-1625 |




 科Sisoridae
科Sisoridae 科Ictaluridae
科Ictaluridae 属Zacco
属Zacco 属Ochetobius
属Ochetobius 属Toxabramis
属Toxabramis 属Rhodeus
属Rhodeus 属Acheilognathus
属Acheilognathus 属Hemibarbus
属Hemibarbus 属Squalidus
属Squalidus