2. 上海市水生野生动植物保护研究中心 上海 200092;
3. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室 上海 201306;
4. 上海海洋大学 上海水产养殖工程技术研究中心 上海 201306
2. Shanghai Aquatic Wildlife Conservation Research Center, Shanghai 200092, China;
3. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
4. Shanghai Engineering Research Center of Aquaculture, Shanghai Ocean University, Shanghai 201306, China
中华鲟(Acipenser sinensis)是中国特有的江海性洄游性鱼类,是国家Ⅰ级重点保护野生动物,被列为世界自然保护联盟濒危物种红色名录(IUCN red list of threatened species)中的极危物种(CR)和濒危野生动植物种国际贸易公约(CITES)附录Ⅱ的保护物种。在2015年农业部印发《中华鲟拯救行动计划(2015—2023年)》的通知中,通过修复保护栖息地、建立监测和救护体系、人工增殖和放流、保护遗传资源等措施来恢复中华鲟种群数量,而濒危物种的日常健康监测也是预防疾病和恢复自然种群资源的重要一环。
无损的采样方法可大幅降低濒危物种因采样导致的机体损伤而造成的健康风险(Fernández-Alacid et al, 2018)。鱼类体表黏液是保护机体的第一道防线,由上皮黏液细胞分泌,是抵御致病微生物的重要屏障。此外,黏液含有抗菌肽、蛋白酶、溶菌酶和凝集素等化学成分,对鱼体具有重要的保护作用(Dash et al, 2018)。鱼类皮肤中的黏液不断地吸附和清除微生物,微生物通过竞争空间或营养物质(Balcazar et al, 2006)、产生拮抗化合物(Boutin et al, 2012; Lowrey et al, 2015)与宿主免疫系统相互作用来抵御病原体的入侵,避免病原性微生物的定植。
皮肤微生物群落结构受多种因素影响,如环境条件、健康状况和宿主遗传等(van Cise et al, 2020; Xavier et al, 2020)。环境压力会导致体表菌群的变化,促进潜在致病菌的繁殖(Sylvain et al, 2016)。环境条件影响鱼类生理健康已有诸多研究报道,这种外源性压力可能会使得体表黏液中保持共生菌和机会性病原菌之间的平衡状态发生严重失衡(Gomez et al, 2008)。目前,全球变暖的趋势严重,因此,有学者对西伯利亚鲟(Acipenser baerii)进行了热应激实验,发现热应激可使皮肤黏液细胞数量明显减少,并引起表皮损伤,显著破坏了皮肤微生物的多样性和丰度,潜在病原菌增加(Vibrionimonas、Mesorhizobium和Phyllobacterium),益生菌减少(Bradyrhizobium和Methylovirgula) (Yang et al, 2022)。宿主微生物群的失调与感染性疾病之间存在相关性,影响宿主免疫功能。此外,感染鲤春病毒血症病毒(spring viremia of carp, SVC)的鲤(Cyprinus carpio)皮肤黏膜层中,变形菌门(Proteobacteria)的丰度明显升高(Meng et al, 2021)。感染哈维氏弧菌(Vibrio harveyi)的欧洲鲈(Dicentrarchus labrax)体表黏液菌群失调,未感染组中Rubritalea是黏液菌群的优势属,是该组的生物标志物(Cámara-Ruiz et al, 2021)。诊断生物标志物可以作为疾病发展的指标,并且可以在水产养殖场疾病预防和优化治疗中发挥关键作用。
本研究利用高通量测序技术,结合非侵入性的无损采样方式,分析水体、健康与亚健康中华鲟皮肤黏液的微生物组成、结构和多样性,以期了解不同生理状态下中华鲟体表黏液微生物的变化情况,同时与生境水体微生物进行比对并分析其中的差异,为中华鲟的健康养殖管理提供科学依据。
1 材料与方法 1.1 实验对象实验鱼为上海市水生野生动植物保护研究中心饲养的≥10龄的子一代中华鲟。养殖池长为15 m、宽为15 m、深为2 m,采用循环水养殖系统,投喂鲟鱼配合饲料(粗蛋白≥40%;粗脂肪≥12%)。采样时,养殖水温为23 ℃,溶解氧为8.58 mg/L,氨氮为0.13 mg/L,亚硝酸盐为0.07 mg/L,pH为8.3。健康与亚健康实验鱼样本采集于同一养殖环境中,健康鱼平均体长为(183.08±15.43) cm,平均体质量为(68.63±11.91) kg;亚健康鱼平均体长为(172.25±9.43) cm,平均体质量为(61.90±8.42) kg。健康中华鲟体表无畸变,腹腔饱满,活动有力,亚健康中华鲟存在一定程度的身体畸形和腹腔凹陷。
1.2 实验设计 1.2.1 环境样本的采集在养殖池中选取3个点位,利用采水器在每个点位从水面下1 m处采得1 L的养殖水体,然后转移到容量为1 L的聚乙烯瓶中,共采集3 L水体。每升水体用一张直径为47 mm、孔径为0.22 µm的混合醋酸硝化纤维素滤膜过滤,共使用3个滤膜,分别记为S1、S2和S3。过滤完成后,滤膜用无菌锡箔纸包好保存,放于−80 ℃冰箱,用于后续微生物组的测定。
1.2.2 黏液样本的采集采集黏液时,保持中华鲟腹部朝下,头部浸没在水中保证正常呼吸,背部适当抬离水面,使用无菌生理盐水清洗取样区域皮肤,清洗完成后,使用载玻片从皮肤表面收集黏液。在收集过程中避免血液和粪便。选取12条健康中华鲟进行样本采集,每3条健康中华鲟的黏液汇集至一个冻存管中,即健康组共4个重复,记为JN1、JN2、JN3和JN4。亚健康中华鲟样品数量较少,选取4条亚健康中华鲟进行亚健康组的样本采集,该组每条鱼为1个重复,共4个重复,记为YN1、YN2、YN3和YN4。在收集后及时将黏液样品储存在−80 ℃冰箱中,用于微生物组的测定。
1.2.3 水体理化参数的测定使用WTW Multi 3510多参数水质测定仪(Xylem,德国)测量水温、溶解氧和pH值。使用Nova 60和配套试剂包(Merck, 德国)测量氨氮、亚硝酸。
1.2.4 DNA提取与PCR扩增使用E.Z.N.A™ Mag-Bind Soil DNA Kit (OMEGA,中国)对11个样本进行DNA提取,按照制造商的说明书进行。使用Qubit 4.0 (Thermo,美国)测量DNA浓度,以确保提取了足够数量的高质量基因组DNA。采用2种通用细菌16S rRNA基因扩增子PCR引物(PAGE纯化):扩增子PCR上游引物(CCTACGGGNGGCWGCAG)和下游引物(GACTACHVGGGTATCTAATCC),进行2轮扩增。使用Hieff NGSTM DNA Selection Beads (Yeasen, 10105ES03,中国)对扩增子产物中的游离引物和引物二聚体物种进行纯化,通用Illumina适配器和索引构建文库。在测序之前,通过2%琼脂糖凝胶电泳(电泳仪DYY-6C,北京市六一仪器厂)检测文库大小,使用Qubit® 4.0 Green双链DNA分析仪(Q33288,ThermoFisher)测定每个PCR产物的DNA浓度,并使用生物分析仪(Agilent 2100,美国)进行质量控制。每个反应混合物的扩增子根据其浓度以等摩尔比混合(漩涡混合器GL-88B,海门市其林贝尔仪器制造有限公司)。使用Illumina Mi Seq系统(Illumina Mi Seq,美国)进行测序。
1.2.5 数据分析测序完成后,用PEAR软件(version 0.9.8)将2个短Illumina读数按照overlap进行拼接,并对fastq文件进行处理,生成单独的fasta和qual文件,然后用标准方法进行分析。使用Usearch软件(version 11.0.667)将有效标签聚类成≥97%相似性的操作分类单元(operational taxonomic unit, OTU)。去除嵌合体序列和单一OTUs (只读一个),剩余序列根据OTUs排序到每个样品中。在每个簇内选择丰度最高的标签序列作为代表序列。将细菌和真菌OTU代表序列分别与RDP数据库和UNITE真菌ITS数据库进行比对,进行分类统计。
1.2.6 统计分析运用单因素方差分析(one-way ANOVA)检验不同组间α多样性指数(ACE、Chao 1、Shannon和Simpson指数)差异的显著性。测序后,统计各组之间独有和共有OTU数量和各组的OTU总数。对样本微生物门水平的相对丰度进行统计与绘制。对各组进行相似性分析(ANOSIM)以确认分组的合理性。β多样性评估样品间微生物组的差异,包括主坐标分析(PCoA)、主成分分析(PCA)等降维方法,根据样本间距离绘制距离热图并聚类。测序数据与已知代谢功能的微生物参考基因组数据库进行比对,实现对细菌代谢功能的预测。使用生信云平台(https://ngs.sangon.com/)进行可视化分析。
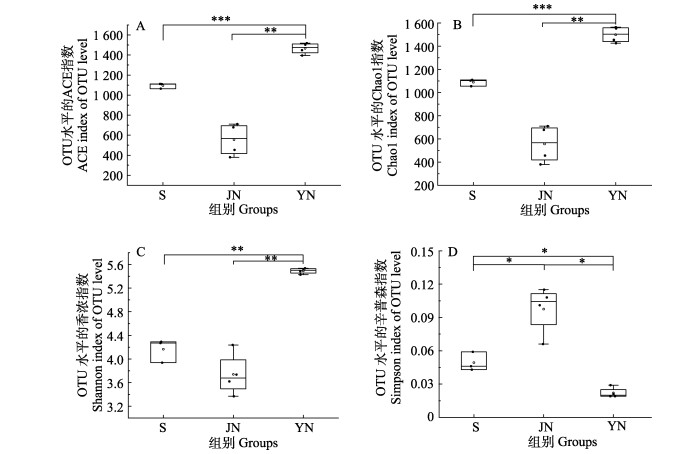
2 结果 2.1 序列的统计分析使用Illumina Mi Seq平台对所有样品的16S rRNA基因进行测序,分析健康和亚健康中华鲟体表黏液及其生活水体的菌群组成。经初始数据质量和嵌合体过滤,所有样本共获得383 963条高质量序列,基于表中所有样本的覆盖值,测出序列可涵盖每组中的大部分微生物,统计了各样本的OTU数目,以及α多样性指数(表 1)。ACE指数和Chao 1指数确定样品的物种丰富度,Shannon和Simpson指数量化物种多样性。以每个样品在3%的遗传距离上的显著差异(P < 0.05)进行汇总。亚健康中华鲟黏液微生物组中α多样性指数相较于其养殖水体以及健康中华鲟的黏液样本存在显著差异(P < 0.05)。Simpson指数在健康中华鲟的黏液样本中与养殖水体存在显著差异(P < 0.05);ACE、Chao1和Shannon指数在健康中华鲟的黏液样本中与养殖水体无显著差异(P > 0.05) (图 1)。
|
|
表 1 丰富度和多样性指数 Tab.1 Richness and diversity indices |
|
图 1 各组样本微生物的丰富度和多样性
Fig.1 Abundance and diversity of microorganisms in each group of samples
箱体上中下线分别为75、50 (中位数)和25分位数,轴须线最长不超过1.5×箱体范围。S代表中华鲟养殖水体,JN代表健康中华鲟体表黏液,YN代表亚健康中华鲟体表黏液,下同。
The upper and lower boxes are 75, 50 (median) and 25th quartiles, respectively, and the axial whisker lines are not longer than 1.5× box range at the longest. S represents sturgeon culture water samples, JN represents healthy sturgeon mucus, and YN represents sub-healthy sturgeon mucus, the same below. *: 0.01 < P≤0.05; **: 0.001 < P≤0.01; ***: P≤0.001. |
图 2展示了各组之间共有和独有OTU数量的韦恩图以及各组所有测序得出的OTU总数。水体共统计到808个OTU,生物样本中OTU总数基本一致,分别为1 321和1 319。水体与健康黏液微生物的共有OTU最少,而与亚健康组的共有OTU最多。相较于水体,生物样本中独有OTU数目可达水体的2倍左右。结果表明,生物与其所在生境菌群的交融与独特性会因生理健康状况而发生明显变化。

|
图 2 所有组的OTU韦恩图 Fig.2 Venn diagram of OTUs for all groups A~C为各组之间的两两对比,D为所有组的OTU总数统计。 A~C shows the comparison between groups, and D shows the total number of OTUs counted for all groups. |
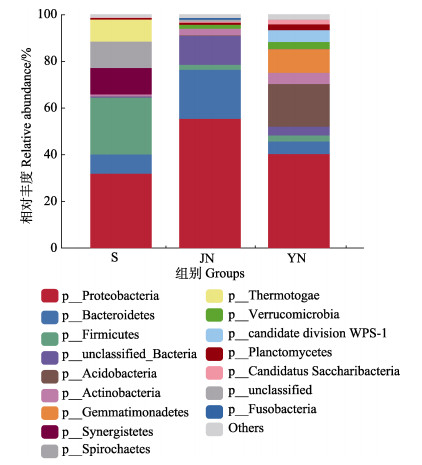
样品经过数据库比对物种注释后,相对丰度较高的门类有变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)、互养菌门(Synergistetes)、螺旋体门(Spirochaetes)、热袍菌门(Thermotogae)、疣微菌门(Verrucomicrobia)、candidate_division_WPS-1、浮霉菌门(Planctomycetes)、Candidatus_Saccharibacteria、梭杆菌门(Fusobacteria) (图 3)。在水体中,丰度较高的为变形菌门、厚壁菌门和互养菌门,占比分别为31.84%、24.37%和11.36%。健康组则为变形菌门、拟杆菌门和放线菌门,占比为55.23%、21.00%和2.85%。亚健康组为变形菌门、酸杆菌门和芽单胞菌门,占比为40.23%、18.29%和10.08%。利用单因素方差分析对各组的优势菌群进行差异分析,结果如图 4所示。水体中厚壁菌门、互养菌门、热袍菌门和螺旋体门的占比显著高于黏液组(P < 0.05),健康组具优势的菌群为变形菌门和拟杆菌门,而亚健康组含多种具优势的菌群,包括芽单胞菌门、candidate_division_WPS-1、酸杆菌门、疣微菌门、Candidatus_Saccharibacteria、放线菌门和浮霉菌门等。
|
图 3 所有组门水平的相对丰度柱状图 Fig.3 Histogram of relative abundance at the phylum level for all groups 相对丰度小于1%的分类合并至“others”。 Classifications with a relative abundance of less than 1% were merged into "others". |

|
图 4 所有组门水平物种差异分析 Fig.4 Analysis of species differences at the phylum level for all groups |
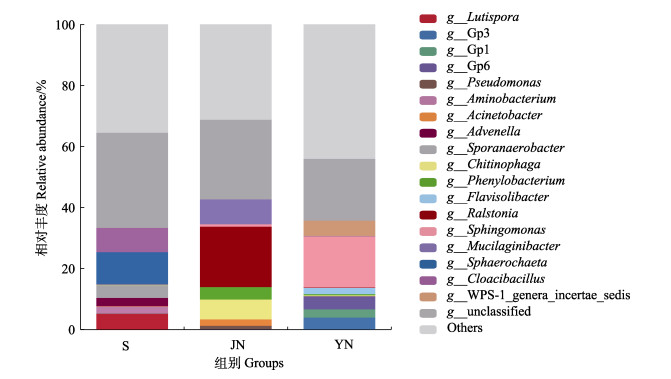
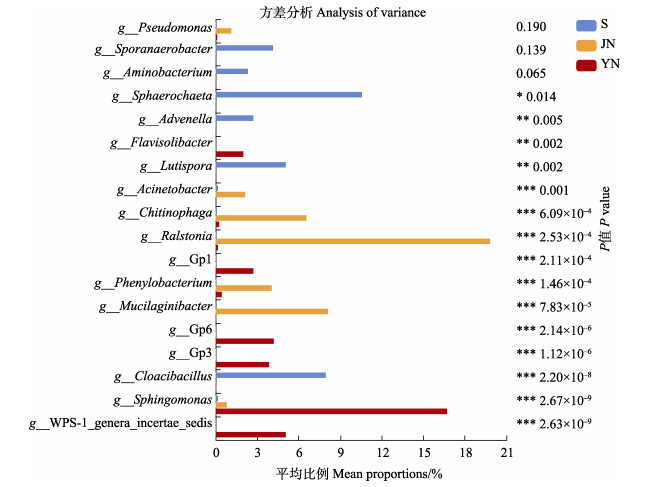
基于属水平的统计结果,水体中排名靠前的优势菌群包括球形发丝菌属(Sphaerochaeta)、下水道菌属(Cloacibacillus)、淤泥孢菌属(Lutispora)、成孢厌氧杆菌属(Sporanaerobacter)等,占比分别为10.56%、7.95%、5.06%和4.13%。健康组中的优势菌群包括罗尔斯通氏菌属(Ralstonia)、黏液杆菌属(Mucilaginibacter)、噬几丁质菌属(Chitinophaga)和苯基小杆菌属(Phenylobacterium)等,占比为19.83%、8.11%、6.55%和4.05%。亚健康组的优势菌群则相较于健康组发生了变化,主要由鞘氨醇单胞菌属(Sphingomonas)、WPS-1_genera_incertae_sedis、Gp6、Gp3等组成,占比为16.71%、5.05%、4.19%和3.86%等(图 5)。利用单因素方差分析对各组属水平具有显著差异的生物标志物进行差异分析(图 6)。水体组中下水道菌属、淤泥孢菌属、小陌生菌属(Advenella)、球形发丝菌属丰度显著高于其他组(P < 0.05),而在健康组中,黏液杆菌属、苯基小杆菌属、罗尔斯通氏菌属、噬几丁质菌属、不动杆菌属(Acinetobacter)的丰度具有显著优势(P < 0.05)。在亚健康组中,出现多种酸杆菌门的优势亚群,如Gp1、Gp3、Gp6等,此外黄色土壤杆菌属(Flavisolibacter)和WPS-1_genera_incertae_sedis也显著高于其他组(P < 0.05)。
|
图 5 所有组属水平的相对丰度柱状图 Fig.5 Histogram of relative abundance at genus level for all groups 相对丰度小于3%的分类合并至“others”,合并未分类的菌群。 Classifications with relative abundance less than 3% were merged to "others", and unclassified classifications were merged. |
|
图 6 所有组属水平物种差异分析 Fig.6 Analysis of species differences at the genus level for all groups |
基于各组样本间OTU水平上的Bray-Curtis距离进行样本距离分析,两两样本间距离由小到大排序后计算得出R≈1.000 0,证明组间差异远大于组内差异。将样本进行999次置换,得出P=0.001 0,证明组间具有极显著的差异。2种指标的计算得出组间差异大于组内差异,分组具有合理性(图 7A)。
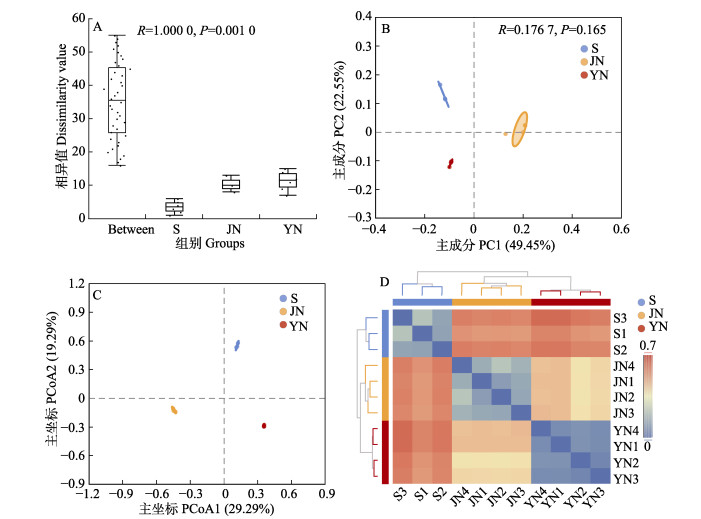
|
图 7 各组样本的相似性与β多样性分析图 Fig.7 The similarity and beta diversity analysis of each group and sample A为多组之间的相似性分析(ANOSIM),B与C分别为多组样本的主成分分析(PCA)与主坐标分析图(PCoA),D为各样本间的距离热图。 A represents the similarity analysis between multiple groups (ANOSIM), B and C represent the principal component analysis (PCA) and principal co-ordinate analysis (PCoA) plots of multiple samples, and D represents the distance heatmap between each sample. |
为进一步检测不同组之间的相似程度,计算β多样性并进行主成分分析(PCA)和主坐标分析(PCoA)。方差分解并进行二维可视化后得到主成分分析图(PCA)(图 7B),主成分1与主成分2的贡献度分别为49.45%和22.55%。基于OTU水平上的Unweighted Unifrac距离进行主坐标分析(PCoA) (图 7C),两主坐标差异解释度的百分比分别为29.29%和19.29%。虽然P值表明各组样本在OTU分类水平方面无显著差异,但无论是PCA还是PCoA图,各组样本都显示出明显的组别分离以及同组样本的集中,一定程度上表明组别之间的差异,且组内样本相似性较高,因此,在二维可视化时各样本点的相对距离较小。根据上述算法得到样本距离后,根据样本之间的距离统计绘制距离热图,并根据样本平均距离进行聚类分析(图 7D)。热图显示,各组中亚健康组内各样本间距离相较于其他组更小,虽然水体与健康组内样本不具有很强的离散性,但组内距离略大于亚健康组。在组间,亚健康组与水体样本的距离相较于健康组更远,生物样本组间的距离较接近。
2.4 PICRUSt功能预测基于扩增子测序结果构建OTU,以KO类别进行KEGG功能注释(图 8)。统计丰度排名前25的功能,多数功能在健康组样本中具有丰度优势。在水体中,肽/镍转运系统、渗透酶蛋白与底物结合蛋白等具有明显的丰度优势。在健康组样本中,具有明显丰度优势的功能有RNA聚合酶sigma-70因子、铁复合外膜接收器蛋白、甲基接受趋化蛋白、LacI家族转录调控因子、谷胱甘肽S-转移酶等,而这些功能在亚健康组中丰度均有下降。亚健康组较高丰度的功能为RNA聚合酶sigma-70。

|
图 8 所有样本的KEGG功能预测热图(右侧为功能对应注释) Fig.8 Heatmap of KEGG functional prediction for all samples, with functional correspondence annotated on the right |
亚健康是一种处于健康与疾病之间的一种临界状态,症状不易察觉,如不及时对其生理状况进行检测,则有可能引发病害问题。皮肤、鳃等部位的共生微生物也可通过持续和频繁的物质交换、能量流动和信号传递来维持黏膜层微环境的稳态,进而调节宿主健康状态(Beklioglu et al, 2006)。亚健康中华鲟存在身体畸形和腹腔凹陷可能是由于先天、环境、人为和饲料等因素的影响,从而导致与健康中华鲟皮肤表面的微生物定植存在差异。鱼类微生物的群落和稳定性由内部和外部因素共同驱动,当鱼体健康状态发生改变时,微生物会表现出“失调”现象,早期发现微生物的变化对于疾病预防和治疗至关重要。为更准确地反映亚健康中华鲟黏液微生物变化情况,借助高通量测序技术对菌群的群落组成与丰度进行表征(Ingham et al, 2007)。
皮肤微生物的群落结构特征可直接影响鱼体健康。研究结果表明,亚健康组具有最多的OTU数目,结合α多样性指标,亚健康组的微生物多样性和丰富度相较于水体和健康组显著升高,而健康组与水体之间的α多样性整体较为相似。亚健康的中华鲟体表微生物多样性显著升高,可能是因为菌群出现了“失调”现象。有研究发现,物理损伤致使金头鲷(Sparus aurata)实验性溃疡后,溃疡和非溃疡区域的微生物存在明显差异,皮肤溃疡提供了微环境,扰乱了体表黏液成分和微生物多样性(Tapia-Paniagua et al, 2018)。另外,昼夜节律变化同样会改变鱼类黏膜微生物群的多样性和组成,持续的光照会使病毒感染的鱼类体表黏液微生物多样性显著增加(Ellison et al, 2021),这与本文亚健康组所呈现的微生物多样性研究结果一致。也有其他研究表明,感染哈维氏弧菌的欧洲海鲈的皮肤微生物群丰度较低(Cámara-Ruiz et al, 2021)。多方面研究表明,非健康状态下鱼类体表黏液微生物群的多样性与组成存在物种特异性,且与其所生活的环境、病原体等因素高度相关。同样有观点认为,即使是与其生长环境高度相关的水环境,鱼类皮肤微生物群在正常情况下仍可存在较大的菌群组成差异,并且在个体之间存在较大的可变性(Berggren et al, 2022)。本研究表明,体表黏液菌群组成与水体存在一定相似性,但多数门类与水体呈现出较大差异,无论是从相对丰度,还是从各组独有和共有OTU来说,体表黏液样本呈现出与水体的独立性,且样本间距离也佐证了这一结论。此外,随着鱼类生理状态的改变,亚健康个体与水体的共有OTU增多。结果表明,生物与其所在生境菌群的交融与独特性,会因生理健康状况而发生明显变化。
在本研究中,热袍菌门和互养菌门在中华鲟养殖水体中被检测到。热袍菌门和互养菌门等有助于在生境中增强菌群之间的协同作用,进而形成环境中具有独立性的群落构型(Li et al, 2022)。变形菌门在鱼类体表微生物中常具有最高的占比,其他菌门则会因物种和环境等因素发生变化(Larsen et al, 2013)。在本研究中,变形菌门作为核心菌群存在于黏液和水体中,黏液中变形菌门的丰度高于养殖水体,其他研究中也发现类似结果:肠道中变形菌门的丰度高于养殖水体(宫晗等, 2023)。其中,健康组黏液变形菌门的丰度高于亚健康组,变形菌门作为细菌中最大和最具多样性的一个族群,具有较强的适应性和丰富的代谢多样性,在生物化学物质循环过程中起重要作用(刘艳霞等, 2023)。本研究的健康组中拟杆菌门的相对丰度高于亚健康组。Ye等(2020)研究表明,拟杆菌门通过抵御致病菌来改善鱼体的消化和免疫功能。亚健康组中鞘氨醇单胞菌是黏液菌群的优势属,该属可作为健康检测的标志物。鞘氨醇单胞菌的主要次级代谢产物为类胡萝卜素和威兰胶。胡萝卜素可以作为抗氧化剂保护细胞免受活性氧的伤害。威兰胶具有黏性,能在细胞周围充当物理屏障,为细胞提供保护,并具有抗炎症和抗氧化等功能(刘辉等, 2023)。鞘氨醇单胞菌属可能在中华鲟抵御外部不良环境和病原微生物中发挥重要作用。健康组中的优势菌群包括罗尔斯通氏菌属(19.83%)和黏液杆菌属(8.11%)。罗尔斯通氏菌属是革兰氏阴性细菌,在环境中普遍存在。有研究表明,罗尔斯通氏菌属与蛋白质消化和吸收、苯丙氨酸代谢、酮体合成和降解以及赖氨酸降解密切相关,上述通路对于鱼体维持健康状态具有重要意义(Huang et al, 2020)。通过PICRUSt功能预测表明,健康组丰度较高的功能主要与信号转导、响应环境和生物代谢有关,如sigma-70因子和铁相关蛋白等(胡莉等, 2018; Jatuponwiphat et al, 2019),甲基接受趋化蛋白等也具有感知化学效应物与调控信号转导的作用(Huang et al, 2016);在亚健康组中,相关功能的丰度均有不同程度的下降,结合功能注释与其相关的研究,亚健康组黏液微生物对环境变化的响应与各种微观调控能力相较于健康组都有所下降。一方面鱼类的生理状态会影响体表微生物的定植,另外,定植的微生物会对鱼类的生理状态产生一定的影响。共生微生物通过持续频繁的物质交换、能量流动和信号传递,维持粘膜层微环境的平衡,调节宿主的健康(张艳敏等, 2022)。体表黏液菌群可作为鱼体健康状态的微生物标记物,有利于评估鱼体的健康状态。
4 结论本研究利用高通量测序技术,对中华鲟体表黏液及其生活水体进行微生物组测定。结果表明,与水体和健康组黏液样本相比,亚健康组样本的微生物多样性和丰富度显著升高。致病菌在亚健康组中占比上升,并影响鱼类代谢、环境适应、微观分子调控等相关基因功能,从而影响鱼类的正常生命活动。这一现象对于理解水产养殖物种中常驻微生物与病原菌之间的协同和拮抗作用具有重要意义。濒危物种的日常健康监测是预防疾病和恢复自然种群资源的重要一环,通过对健康和亚健康中华鲟体表黏液微生物群落的比较,发现了不同健康状态存在不同的微生物群落结构。此外,鞘氨醇单胞菌属作为亚健康中华鲟黏液菌群的优势属,或许可以通过检测它的改变达到一定的诊断目的,有助于未来在实践养殖过程中采取相应的预防措施,为中华鲟的保育工作提供新的视野和途径。
BALCAZAR J L, DE BLAS I, RUIZ-ZARZUELA I, et al. The role of probiotics in aquaculture. Veterinary Microbiology, 2006, 114(3/4): 173-186 |
BEKLIOGLU M, TELLI M, GOZEN A G. Fish and mucus-dwelling bacteria interact to produce a kairomone that induces diel vertical migration in Daphnia. Freshwater Biology, 2006, 51: 2200-2206 DOI:10.1111/j.1365-2427.2006.01642.x |
BERGGREN H, TIBBLIN P, YILDIRIM Y, et al. Fish skin microbiomes are highly variable among individuals and populations but not within individuals. Frontiers in Microbiology, 2022, 12: 67770 |
BOUTIN S, BERNATCHEZ L, AUDET C, et al. Antagonistic effect of indigenous skin bacteria of brook charr (Salvelinus fontinalis) against Flavobacterium columnare and F. psychrophilum. Veterinary Microbiology, 2012, 155(2/3/4): 355-361 |
CÁMARA-RUIZ M, CEREZO I M, GUARDIOLA F A, et al. Alteration of the immune response and the microbiota of the skin during a natural infection by Vibrio harveyi in european seabass (Dicentrarchus labrax). Microorganisms, 2021, 9(5): 964 DOI:10.3390/microorganisms9050964 |
DASH S, DAS S K, SAMAL J, et al. Epidermal mucus, a major determinant in fish health: A review. Iranian Journal of Veterinary Research, 2018, 19(2): 72-81 |
ELLISON A R, WILCOCKSON D, CABLE J. Circadian dynamics of the teleost skin immune-microbiome interface. Microbiome, 2021, 9(1): 222 DOI:10.1186/s40168-021-01160-4 |
FERNÁNDEZ-ALACID L, SANAHUJA I, ORDÓÑEZ-GRANDE B, et al. Skin mucus metabolites in response to physiological challenges: A valuable non-invasive method to study teleost marine species. Science of the Total Environment, 2018, 644: 1323-1335 DOI:10.1016/j.scitotenv.2018.07.083 |
GOMEZ G D, BALCAZAR J L. A review on the interactions between gut microbiota and innate immunity of fish. FEMS Immunology and Medical Microbiology, 2008, 52(2): 145-154 DOI:10.1111/j.1574-695X.2007.00343.x |
GONG H, CHEN P, QIN Z, et al. Analysis of water quality and bacterial community characterization in an industrialized recirculating aquaculture system of Litopenaeus vannamei. Progress in Fishery Sciences, 2023, 44(1): 125-136 [宫晗, 陈萍, 秦桢, 等. 凡纳滨对虾工厂化循环水养殖系统水质指标及微生物菌群结构的分析. 渔业科学进展, 2023, 44(1): 125-136] |
HU L, TAN Z W, GAO C, et al. Advances in the high efficient regulation of microbiological functions by sigma factors. Chinese Bulletin of Life Sciences, 2018, 30(3): 285-292 [胡莉, 谭泽文, 郜晨, 等. Sigma因子高效调控微生物多功能研究进展. 生命科学, 2018, 30(3): 285-292] |
HUANG Q, SHAM R C, DENG Y, et al. Diversity of gut microbiomes in marine fishes is shaped by host-related factors. Molecular Ecology, 2020, 29(24): 5019-5034 DOI:10.1111/mec.15699 |
HUANG Z, NI B, JIANG C Y, et al. Direct sensing and signal transduction during bacterial chemotaxis toward aromatic compounds in Comamonas testosteroni: Sensing aromatics during microbial chemotaxis. Molecular Microbiology, 2016, 101(2): 224-237 DOI:10.1111/mmi.13385 |
INGHAM C J, SPRENKELS A, BOMER J, et al. The micro-Petri dish, a million-well growth chip for the culture and high-throughput screening of microorganisms. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(46): 18217-18222 |
JATUPONWIPHAT T, CHUMNANPUEN P, OTHMAN S, et al. Iron-associated protein interaction networks reveal the key functional modules related to survival and virulence of Pasteurella multocida. Microbial Pathogenesis, 2019, 127: 257-266 DOI:10.1016/j.micpath.2018.12.013 |
LARSEN A, TAO Z, BULLARD S A, et al. Diversity of the skin microbiota of fishes: Evidence for host species specificity. FEMS Microbiology Ecology, 2013, 85(3): 483-494 DOI:10.1111/1574-6941.12136 |
LI Y Z, CHEN Z, PENG Y Y, et al. Deeper insights into the effects of substrate to inoculum ratio selection on the relationship of kinetic parameters, microbial communities, and key metabolic pathways during the anaerobic digestion of food waste. Water Research, 2022, 217: 118440 DOI:10.1016/j.watres.2022.118440 |
LIU H, WEI L L, ZHU L F, et al. Research progress of Sphingomonas. Microbiology China, 2023, 50(6): 2738-2752 [刘辉, 韦璐璐, 朱龙发, 等. 鞘氨醇单胞菌的研究进展. 微生物学通报, 2023, 50(6): 2738-2752] |
LIU Y X, ZHANG J J, ZHOU Y Q, et al. Characteristics and correlation analysis of bacterial community structure in the gut of Apostichopus japonicus and culture water in suspension cages from north China. Progress in Fishery Sciences, 2023, 44(6): 190-202 [刘艳霞, 张靖婧, 周叶青, 等. 北方吊笼养殖刺参肠道及其养殖环境菌群结构特征及其相关性分析. 渔业科学进展, 2023, 44(6): 190-202] |
LOWREY L, WOODHAMS D C, TACCHI L, et al. Topographical mapping of the rainbow trout (Oncorhynchus mykiss) microbiome reveals a diverse bacterial community with antifungal properties in the skin. Applied and Environmental Microbiology, 2015, 81(19): 6915-6925 DOI:10.1128/AEM.01826-15 |
MENG K F, DING L G, WU S, et al. Interactions between commensal microbiota and mucosal immunity in teleost fish during viral infection with SVCV. Frontiers in Immunology, 2021, 12: 654758 DOI:10.3389/fimmu.2021.654758 |
SYLVAIN F É, CHEAIB B, LLEWELLYN M, et al. pH drop impacts differentially skin and gut microbiota of the Amazonian fish tambaqui (Colossoma macropomum). Scientific Reports, 2016, 6: 32032 DOI:10.1038/srep32032 |
TAPIA-PANIAGUA S T, CEBALLOS-FRANCISCO D, BALEBONA M C, et al. Mucus glycosylation, immunity and bacterial microbiota associated to the skin of experimentally ulcered gilthead seabream (Sparus aurata). Fish and Shellfish Immunology, 2018, 75: 381-390 DOI:10.1016/j.fsi.2018.02.006 |
VAN CISE A M, WADE P R, GOERTZ C E C, et al. Skin microbiome of beluga whales: Spatial, temporal, and health-related dynamics. Animal Microbiome, 2020, 2(1): 39 DOI:10.1186/s42523-020-00057-1 |
XAVIER R, PEREIRA A, PAGAN A, et al. The effects of environment and ontogeny on the skin microbiome of two Stegastes damselfishes (Pomacentridae) from the eastern Caribbean Sea. Marine Biology, 2020, 167(7): 102 DOI:10.1007/s00227-020-03717-7 |
YANG S Y, XU W Q, TAN C L, et al. Heat stress weakens the skin barrier function in sturgeon by decreasing mucus secretion and disrupting the mucosal microbiota. Frontiers in Microbiology, 2022, 13: 860079 DOI:10.3389/fmicb.2022.860079 |
YE G L, DONG X H, YANG Q H, et al. A formulated diet improved digestive capacity, immune function and intestinal microbiota structure of juvenile hybrid grouper (Epinephelus fuscoguttatus ♀×Epinephelus lanceolatus ♂) when compared with chilled trash fish. Aquaculture, 2020, 523: 735230 DOI:10.1016/j.aquaculture.2020.735230 |
ZHANG Y M, YANG G K, LI K K, et al. Research progress of mucosal microorganisms of fish. Journal of Fisheries of China, 2022, 46(6): 1117-1127 [张艳敏, 杨国坤, 李克克, 等. 鱼类黏膜层微生物研究进展. 水产学报, 2022, 46(6): 1117-1127] |



