2. 海水养殖生物育种与可持续产出全国重点实验室 中国水产 科学研究院黄海水产研究所 山东 青岛 266071;
3. 青岛海洋科技中心海洋渔业科学与食物产出过程功能实验室 山东 青岛 266237
2. State Key Laboratory of Mariculture Biobreeding and Sustainable Goods, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao, 266071, China;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao Marine Science and Technology Center, Qingdao 266237, China;
4. BLUP Aquabreed Co., Ltd., Weifang 261311, China
凡纳对虾(Penaeus vannamei)因具有生长迅速、抗病性强、经济效益好等特点,已成为世界范围内养殖对虾的主要品种(Chen et al, 2019; Hou et al, 2022)。随着水产养殖规模的不断扩大,细菌、真菌和病毒性疾病的暴发对全球虾类养殖业构成了重大威胁。其中,白斑综合征病毒(white spot syndrome virus, WSSV)对凡纳对虾、日本对虾(Penaeus japonicus)和斑节对虾(Penaeus monodon)等多种商业重要对虾品种具有高致死率。感染WSSV后对虾死亡率可达100% (Chen et al, 2007; 董丽君等, 2019; 冯亚萍等, 2017)。因此,开展对虾抗WSSV的相关机制研究对于对虾养殖业发展具有重要意义。
本实验室前期研究基于全基因组关联分析(genome-wide association study, GWAS)方法筛选到抗WSSV候选基因:parkin共调基因(parkin co-regulated gene, PACRG)。PACRG以头对头的方式与DNA反义链上的parkin连接,共用一个5′核心双向启动子,二者共同参与细胞自噬过程,从而在细胞保护方面发挥作用(West et al, 2003)。parkin编码的蛋白是一种与帕金森病相关的RBR (RING-between-RING) E3泛素连接酶,通过泛素–蛋白酶体系统(ubiquitin-proteasome system, UPS)来降解异常折叠蛋白,同时参与氧化应激及线粒体功能调控等多种途径(Scott et al, 2017; Winklhofer, 2014)。UPS是一种蛋白质降解途径,在DNA损伤修复、细胞周期检查点控制和免疫反应调节等多个细胞过程中发挥重要作用(Dagar et al, 2023)。研究表明,泛素化修饰在先天免疫和炎症反应中发挥重要作用(Di Napoli et al, 2003)。在高等动物体内,parkin能够泛素化病原菌或机体的某些蛋白,作为信号招募自噬膜的延伸然后形成包含病原菌的自噬体,进而介导自噬体与溶酶体融合降解病原菌。PACRG蛋白高度保守,主要参与微管蛋白的调节,对鞭毛、纤毛的运动和精子的发育具有重要作用(Mizuno et al, 2016)。PACRG蛋白还能够与分子伴侣热休克蛋白90、70 (Hsp90、Hsp70)和T-复合物1 (TCP1α、TCP1β和TCP1θ)相互结合,起到保护细胞的作用,同时,PACRG还在肿瘤坏死因子(tumor necrosis factor, TNF)信号传导中发挥作用(Meschede et al, 2020)。Trinklein等(2004)研究发现,共用一个双向启动子的基因表达具有显著的相关性和功能关联性。在果蝇(Drosophila)和人类(Homo sapiens)基因组中发现同一区域的基因呈现共同表达的特性(Caron et al, 2001; Spellman et al, 2002)。PACRG与parkin共用一个双向启动子,它们在功能上可能存在相关性。研究证实,PACRG与parkin蛋白的相互结合能够减弱parkin底物类内皮素受体(parkin associated endothelin receptor-like, Pael-R)的毒性作用,从而增加细胞成活率(Imai et al, 2003)。PACRG和parkin共享调控区域中的单核苷酸多态性位点(single nucleotide polymorphism, SNP)分析结果显示,PACRG与麻风病、伤寒副伤寒、癌症、肺结核等疾病的发生具有相关性(赵海洲等, 2018)。
本研究旨在通过模拟自然环境中凡纳对虾进食感染WSSV,探究凡纳对虾感染WSSV后PACRG和parkin基因是否参与抵御WSSV的免疫应答反应。为深入解析凡纳对虾PACRG和parkin基因在病毒感染中发挥的功能,本研究在转录、mRNA空间定位、SNP等水平进一步验证PACRG和parkin基因与抗WSSV的相关性,旨在为揭示凡纳对虾抗WSSV分子机制提供理论基础。
1 材料与方法 1.1 实验动物凡纳对虾取自潍坊邦普种业科技有限公司,平均体长为(8.52±0.87) cm,平均体重为(4.7±1.5) g。实验地点为山东省青岛市即墨区中国水产科学研究院黄海水产研究所水产遗传育种中心。所有的凡纳对虾均在实验平台(3 m2×1.2 m)饲养,并按照相同的管理模式进行日常管理。实验开始前将凡纳对虾在盐度为29±1、水温为(26±1) ℃的无菌海水中暂养3 d。每日分别于08:00、14:00和20:00投喂饲料,换水量为40%~50%,提供充足的氧气,使凡纳对虾的生存状态保持稳定。
1.2 WSSV胁迫处理利用单尾、等量、口饲法对150尾健康凡纳对虾进行WSSV感染实验,参考孟宪红等(2013)研究中的WSSV毒饵制备及口饲的方式对每尾虾投喂10 mg病毒载量为1×106 copies/mg的对虾碎肌肉组织,使用红色食用色素将WSSV毒饵进行染色,以便观察对虾的食用情况。随机采集健康和感染WSSV后24、48、72、96、144、192和228 h各5尾凡纳对虾的肝胰腺、鳃、肌肉和眼柄,保存在RNAstore reagent (TIANGEN)中,–80 ℃冷冻用于基因表达水平分析。
1.3 总RNA的提取和cDNA的合成使用RNA-easy isolation reagent (Vazyme)提取肝胰腺、鳃、肌肉和眼柄中的总RNA。使用1%的琼脂糖凝胶电泳检测RNA的质量及完整性。使用Nano- Photometer N50 Touch分光光度计(Thermo Fisher Scientific)在260 nm和280 nm下定量总RNA。使用HiScript® Ⅲ RT SuperMix for qPCR (+gDNA wiper) (Vazyme)试剂盒反转录获得cDNA,于–80 ℃保存备用。
1.4 基因序列生物信息学分析基于NCBI数据库中PACRG基因的参考序列(XM_027379415.1)和parkin基因的参考序列(XM_027357483.1),使用EditSeq软件预测氨基酸序列。使用ExPASy-ProtParam tool (https://web.expasy.org/protparam/)预测蛋白质分子质量、理论等电点、不稳定系数等。使用TMHMM软件(https://dtu.biolib.com/DeepTMHMM)分析蛋白质的跨膜区。使用SignalP 4.1 (https://services.healthtech.dtu.dk/services/SignalP-4.1/)软件进行信号肽结构预测。使用NetNGlyc 1.0 Server (http://www.cbs.dtu.dk/services/NetNGlyc/)进行糖基化位点分析。使用NetPhos 3.1 Server (https://services.healthtech.dtu.dk/services/NetPhos-3.1/)进行磷酸化位点分析。运用SMART (https://smart.embl.de/)在线工具进行蛋白质功能结构域预测分析。使用SOPMA (https://npsa-prabi.ibcp.fr/)软件进行蛋白质二级结构预测。使用DNAMAN 6.0进行同源氨基酸多序列比对分析。使用MEGA 7.0软件通过邻接法(neighbour-joining method, NJ)进行系统进化树构建。
1.5 PACRG和parkin基因的表达定量分析使用SYBR Green Real-time PCR Master Mix (TOYOBO)试剂,在Applied BiosystemsTM QuantStudio 1 Real-Time PCR定量仪中进行real-time PCR,检测PACRG和parkin在凡纳对虾肝胰腺、鳃、肌肉和眼柄中的表达水平。以18S rRNA基因作为内参基因。引物序列信息(qPACRG-F、qPACRG-R;qparkin-F、qparkin-R;18S-F、18S-R)见表 1。Real-time PCR体系总体积为20 μL:SYBR Green Real-time PCR Master Mix (TOYOBO) 10 μL,上下游引物(0.2 μmol/L)各0.8 μL,cDNA模板(100 ng/μL) 2 μL,RNase Free ddH2O 6.4 μL。反应程序:95 ℃预变性60 s;95 ℃变性15 s,60 ℃退火15 s,72 ℃延伸45 s,共40个循环。每个样品设置3个平行重复实验。采用2–ΔΔCt方法分析PACRG和parkin在感染WSSV后不同时间凡纳对虾肝胰腺、鳃、肌肉和眼柄中的相对表达水平。同时,利用IBM SPSS Statistics 25.0软件对计算结果进行独立样本t检验,P < 0.05表示差异显著,P < 0.01表示差异极显著。
|
|
表 1 引物和探针信息 Tab.1 Information of primers and probes |
PACRG特异性探针用异硫氰酸荧光素(fluorescein isothiocyanate, FITC)标记。WSSV特异性探针用花青素荧光染料3 (cyanine, Cy3)标记。特异性探针序列(PACRG-probe、WSSV-probe)信息见表 1。细胞核用DAPI染色。荧光原位杂交(fluorescence in situ hybridization, FISH)检测所用特异性探针由武汉塞维尔生物科技有限公司合成。选取健康和感染WSSV 96 h后PACRG表达水平最高的凡纳对虾的肌肉组织在Servicebio®原位杂交固定液(动物) (Servicebio)中固定并于4 ℃保存过夜。经过切片、探针杂交和DAPI染色后,在荧光显微镜下观察并采集图像。
1.7 SNP位点的筛选及抗WSSV关联分析将32个家系中的960尾凡纳对虾进行单尾、定量投喂WSSV毒饵(方法同1.2),测试WSSV抗性。测试至530 h结束,死亡855尾对虾,存活105尾对虾。死亡对虾的平均存活时间为(392.00±209.57) h。取所有测试死亡和存活个体的肌肉组织于–80 ℃酒精保存。筛选前期死亡的30尾对虾[存活时间(123.8± 19.42) h]作为敏感组,后期存活530 h的30尾对虾作为抗性组,进行SNP位点的筛选和抗WSSV关联分析。使用TIANamp Marine Animals DNA Kit (TIANGEN)提取敏感组和抗性组中对虾肌肉组织的DNA。利用1%的琼脂糖凝胶电泳检测DNA的质量。使用NanoPhotometer N50 Touch分光光度计(Thermo Fisher Scientific)在260 nm和280 nm下检测DNA的浓度和纯度。
基于NCBI数据库中的参考序列(PACRG, NW_ 020871716.1; parkin, NW_020869090.1),利用primer 3在线引物设计工具(https://primer3.ut.ee/)设计PACRG和parkin基因的DNA序列扩增引物,引物序列信息(PACRG-F、PACRG-R;parkin-F、parkin-R)见表 1。引物由生工生物工程(上海)股份有限公司合成,于–20 ℃保存备用。以提取的DNA为模板,使用特异性引物扩增PACRG和parkin基因的DNA序列。使用Quick Taq HS DyeMix (TOYOBO)进行PCR,反应体系为50 μL:2×Quick Taq HS DyeMix (TOYOBO) 25 μL,上下游引物(0.2 μmol/L)各1 μL,DNA模板(100 ng/μL) 4 μL,RNase Free ddH2O 19 μL。PCR程序:94 ℃预变性2 min;94 ℃变性30 s,55 ℃退火30 s,68 ℃延伸1 min,35个循环;68 ℃延伸5 min。扩增反应结束后,利用1%的琼脂糖凝胶电泳确定扩增产物特异性和扩增片段大小。PCR产物委托生工生物过程(上海)股份有限公司进行Sanger测序。使用SeqMan软件将测序样本的PACRG和parkin的DNA序列与NCBI数据库参考序列进行比对,同时结合Sanger测序峰图筛选出所有的SNP位点。
根据SNP位点的分型结果,统计各基因型频率,使用PLINK1.9软件(Purcell et al, 2007)计算观测杂合度(Ho)、期望杂合度(He)、最小等位基因频率(MAF)、多态信息含量(PIC)和哈迪–温伯格平衡(Hardy- Weinberg equilibrium, HWE)等遗传多样性参数。通过质控,去除群体中MAF < 0.05的SNP位点,不做进一步的关联分析。PIC < 0.25为低度多态性,0.25≤PIC ≤0.50为中度多态性,PIC > 0.50为高度多态性。利用IBM SPSS 25.0软件对分型结果进行卡方检验(χ2),P < 0.05为差异显著,P < 0.01为差异极显著,分析SNP位点与凡纳对虾抗WSSV性状的相关性。
2 结果与分析 2.1 PACRG基因及氨基酸序列分析PACRG的ORF序列全长600 bp,编码199个氨基酸(图 1)。预测带负电荷的氨基酸残基(Asp+Glu)有29个,带正电荷氨基酸残基(Arg+Lys)有27个。分子量为23.26 kDa。理论等电点为6.44。不稳定系数为56.68,归类为不稳定蛋白。脂肪族指数为89.55,亲水性总平均值为–0.566。无跨膜蛋白,无信号肽结构,无糖基化位点。磷酸化位点分析预测有8个丝氨酸、7个苏氨酸和3个酪氨酸。在27~195氨基酸位置处包含一个ParcG基因结构域(图 1)。预测的二级结构中,α-螺旋(105个氨基酸)占52.76%,延伸链(21个氨基酸)占10.55%,β-转角(12个氨基酸)占6.03%,无规则卷曲(61个氨基酸)占30.65%。

|
图 1 PACRG基因cDNA序列及预测的氨基酸序列 Fig.1 The cDNA sequence and deduced amino acid sequence of the PACRG gene 起始密码子和终止密码子用方框标出;预测的结构域用下划线标出;预测的磷酸化位点用加粗字体标出;SNP位点用红色字体标出。 The start and stop codons are indicated by boxes. The predicted structural domain is indicated by an underline. The predicted phosphorylation sites are indicated in bold font. The SNP sites are indicated in red font. |
parkin mRNA长2 329 bp,包括1 653 bp的ORF,100 bp的5′非翻译区(5′-untranslated region, 5′-UTR),576 bp的3′非翻译区(3′-untranslated region, 3′-UTR),编码550个氨基酸(图 2)。预测带负电荷的氨基酸残基(Asp+Glu)有69个,带正电荷氨基酸残基(Arg+Lys)有49个。分子量为60.15 kDa。理论等电点为5.15。不稳定系数为46.96,归类为不稳定蛋白。脂肪族指数为75.29。亲水性总平均值为–0.254。无跨膜蛋白。1~27氨基酸位置为信号肽结构。糖基化位点分析预测,在95、191和279氨基酸位置有NHSI、NRSC和NATC基序的N-糖基化位点。预测有8个丝氨酸、7个苏氨酸和3个酪氨酸磷酸化位点。在33~104氨基酸位置处包含一个UBQ基因结构域,400~460氨基酸位置包含一个IBR结构域,487~543氨基酸位置包含一个IBR结构域(图 2)。预测的二级结构中,α-螺旋(116个氨基酸)占21.09%,延伸链(130个氨基酸)占23.64%,β-转角(14个氨基酸)占2.55%,无规则卷曲(290个氨基酸)占52.73%。

|
图 2 parkin基因cDNA序列及预测的氨基酸序列 Fig.2 The cDNA sequence and deduced amino acid sequence of the parkin gene 起始密码子和终止密码子用方框标出;预测的结构域用下划线标出;预测的磷酸化位点用加粗字体表示;预测的糖基化位点用“[ ]”标出;SNP位点用红色字体标出。 The start and stop codons are indicated by boxes. The predicted structural domain is indicated by an underline. The predicted phosphorylation sites are indicated in bold font. The predicted glycosylation sites are indicated by square brackets. The SNP sites are indicated in red font. |
将凡纳对虾与多个物种PACRG氨基酸序列进行同源比对(图 3),结果显示,凡纳对虾PACRG氨基酸序列与日本对虾的同源性最高,为89.70%,与其他物种同源性:中华绒螯蟹(Eriocheir sinensis) 87.27%,三疣梭子蟹(Portunus trituberculatus) 86.67%,中国对虾(Penaeus chinensis) 86.67%,克氏原螯虾(Procambarus clarkii) 85.45%,露斯塔野鲮(Labeo rohita) 68.48%,白氏文昌鱼(Branchiostoma belcheri) 64.53%,菲律宾蛤仔(Ruditapes philippinarum) 64.53%,非洲爪蟾(Xenopus laevis) 63.95%,小鼠(Mus musculus) 63.80%,人(Homo sapiens) 63.80%,家蝇(Musca domestica) 46.45%。

|
图 3 PACRG的氨基酸序列比对 Fig.3 Multiple comparison of amino acid sequences for PACRG PACRG序列GenBank登录号:凡纳对虾(XP_027235216.1)、日本对虾(XP_042860455.1)、中华绒螯蟹(XP_050695651.1)、三疣梭子蟹(XP_045128156.1)、中国对虾(XP_047498046.1)、克氏原螯虾(XP_045582115.1)、露斯塔野鲮(XP_050988658.1)、白氏文昌鱼(XP_019623365.1)、菲律宾蛤仔(XP_060575230.1)、非洲爪蟾(XP_041418715.1)、小鼠(NP_081308.1)、人(NP_001073847.1)、家蝇(XP_005176069.2)。 The GenBank accession numbers of PACRG amino acid sequences are as follows: P. vannamei (XP_027235216.1), P. japonicus (XP_042860455.1), E. sinensis (XP_050695651.1), P. trituberculatus (XP_045128156.1), P. chinensis (XP_047498046.1), P. clarkii (XP_045582115.1), L. rohita (XP_050988658.1), B. belcheri (XP_019623365.1), R. philippinarum (XP_060575230.1), X. laevis (XP_041418715.1), M. musculus (NP_081308.1), H. sapiens (NP_001073847.1), M. domestica (XP_005176069.2). |
对PACRG进行系统进化树分析,进一步研究PACRG与所选脊椎动物和无脊椎动物的进化关系(图 4)。系统进化树与该物种的分类地位一致,凡纳对虾与中国对虾和日本对虾聚为一支,他们的亲缘关系最近,形成一个独立的分支。接着与中华绒鳌蟹和三疣梭子蟹聚为一支,与克氏原螯虾、菲律宾蛤仔、白氏文昌鱼、非洲爪蟾、露斯塔野鲮、小鼠、人和家蝇的亲缘关系较远。

|
图 4 PACRG系统进化树分析 Fig.4 The analysis of phylogenetic tree for PACRG |
将凡纳对虾与多个物种parkin氨基酸序列进行同源比对发现(图 5),凡纳对虾的parkin氨基酸序列与中国对虾和斑节对虾的同源性最高,为93.45%。与其他物种同源性:克氏原螯虾72.48%,三疣梭子蟹69.04%,内华达古白蚁(Zootermopsis nevadensis) 58.76%,穴兔(Oryctolagus cuniculus) 51.08%,虎斑响尾蛇(Crotalus tigris) 50.29%,米兰达果蝇(Drosophila mirand) 45.96%,斑马鱼(Danio rerio) 44.03%,蚤状溞(Daphnia pulex) 41.23%,人40.49%,菲律宾蛤仔39.93%,银鳕鱼(Anoplopoma fimbria) 31.86%。

|
图 5 Parkin的氨基酸序列比对 Fig.5 Multiple comparison of amino acid sequences for Parkin Parkin序列GenBank登录号:凡纳对虾(XP_027213284.1)、中国对虾(XP_047495255.1)、斑节对虾(XP_037789710.1)、克氏原螯虾(XP_045609795.1)、三疣梭子蟹(MPC14914.1)、内华达古白蚁(XP_021912942.1)、穴兔(XP_051711121.1)、虎斑响尾蛇(XP_039174700.1)、米兰达果蝇(XP_033242207.1)、斑马鱼(NP_001017635.1)、蚤状溞(XP_046458662.1)、人(KAI4020493.1)、菲律宾蛤仔(XP_060589880.1)、银鳕鱼(XP_054483181.1)。 The GenBank Accession No. of Parkin amino acid sequences: P. vannamei (XP_027213284.1), P. chinensis (XP_047495255.1), P. monodon (XP_037789710.1), P. clarkii (XP_045609795.1), P. trituberculatus (MPC14914.1), Z. nevadensis (XP_021912942.1), O. cuniculus (XP_051711121.1), C. tigris (XP_039174700.1), D. mirand (XP_033242207.1), D. rerio (NP_001017635.1), D. pulex (XP_046458662.1), H. sapiens (KAI4020493.1), R. philippinarum (XP_060589880.1), A. fimbria (XP_054483181.1). |
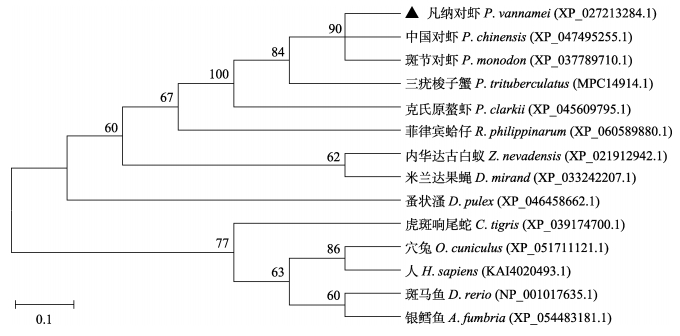
对parkin进行系统进化树分析,进一步研究parkin与所选脊椎动物和无脊椎动物的进化关系(图 6)。系统进化树与该物种的分类地位一致,凡纳对虾与中国对虾和斑节对虾最先聚为一支,它们的亲缘关系最近,形成一个独立的分支。接着与三疣梭子蟹和克氏原螯虾聚为一支,与内华达古白蚁、穴兔、虎斑响尾蛇、米兰达果蝇、斑马鱼、蚤状溞、人、菲律宾蛤仔和银鳕鱼的亲缘关系较远。
|
图 6 parkin系统进化树分析 Fig.6 The analysis of phylogenetic tree for parkin |
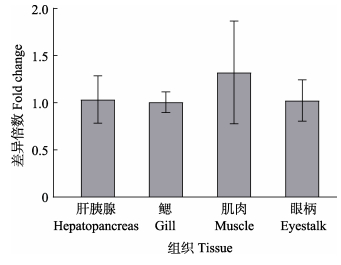
Real-time PCR结果显示,PACRG在健康的凡纳对虾肝胰腺、鳃、肌肉和眼柄中均有表达,且各组织的表达水平基本相同,无显著差异。肌肉中PACRG的表达水平高于肝胰腺、鳃和眼柄。PACRG在鳃中的表达水平约为肝胰腺的0.97倍,在肌肉中的表达水平约为肝胰腺的1.28倍,在眼柄中的表达水平约为肝胰腺的0.99倍(图 7)。
|
图 7 健康凡纳对虾PACRG在肝胰腺、鳃、肌肉和眼柄中的表达 Fig.7 Expression profile of PACRG in hepatopancreas, gill, muscle, and eyestalk of healthy P. vannamei |
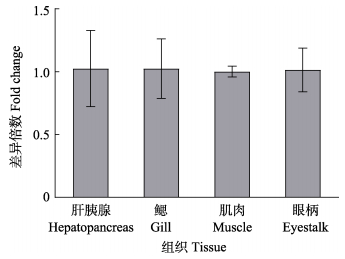
parkin在健康凡纳对虾肝胰腺、鳃、肌肉和眼柄中均有表达,且各组织的表达水平基本相同,无显著差异。parkin在鳃中的表达水平与肝胰腺相同。parkin在肌肉中的表达水平约为肝胰腺的0.98倍。parkin在眼柄中的表达水平约为肝胰腺的0.99倍(图 8)。
|
图 8 健康凡纳对虾parkin在肝胰腺、鳃、肌肉和眼柄中的表达 Fig.8 Expression profiles of parkin in hepatopancreas, gill, muscle, and eyestalk of healthy P. vannamei |
凡纳对虾感染WSSV后48、96、144、192和228 h,PACRG在肝胰腺中的表达水平分别为对照组的(0.67±0.09)、(0.66±0.16)、(1.84±0.38)、(0.45±0.13)和(0.48±0.13)倍,其中48 h和96 h差异显著(P < 0.05),144、192和228 h差异极显著(P < 0.01)(图 9A)。凡纳对虾感染WSSV后48、72、96、144、192和228 h,PACRG在鳃中的表达水平分别为对照组的(0.46±0.11)、(0.72±0.06)、(0.28±0.10)、(0.23±0.06)、(0.24±0.03)和(0.24±0.02)倍,差异极显著(P < 0.01) (图 9B)。凡纳对虾感染WSSV后96、192和228 h,PACRG在肌肉中的表达水平分别为对照组的(18.12±9.89)、(12.80±4.06)和(4.98±1.32)倍,差异极显著(P < 0.01) (图 9C)。凡纳对虾感染WSSV后72、144、192和228 h,PACRG在眼柄中的表达水平分别为对照组的(0.31±0.06)、(0.50±0.18)、(0.31±0.10)和(0.10±0.03)倍,差异极显著(P < 0.01) (图 9D)。

|
图 9 感染WSSV后PACRG在凡纳对虾肝胰腺、鳃、肌肉和眼柄中的表达 Fig.9 Expression profiles of PACRG in hepatopancreas, gill, muscle, and eyestalk of P. vannamei post-WSSV infection A:肝胰腺;B:鳃;C:肌肉;D:眼柄。*:P < 0.05;**:P < 0.01,下同。 A: Hepatopancreas; B: Gill; C: Muscle; D: Eyestalk. *: P < 0.05, **: P < 0.01. The same below. |
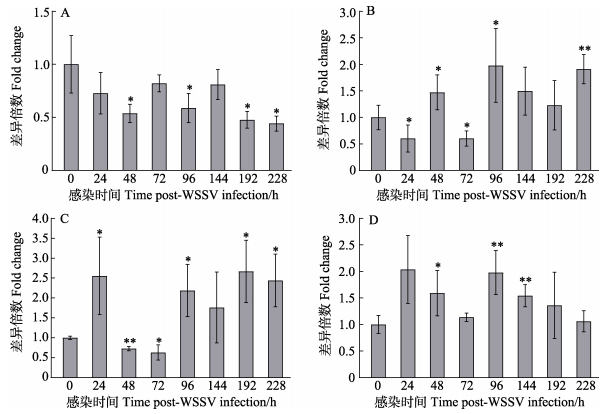
凡纳对虾感染WSSV后48、96、192和228 h,parkin在肝胰腺中的表达水平分别为对照组的(0.54±0.09)、(0.59±0.14)、(0.47±0.08)和(0.44±0.07)倍,差异显著(P < 0.05) (图 10A)。凡纳对虾感染WSSV后24、48、72、96和228 h,parkin在鳃中的表达水平分别为对照组的(0.60±0.26)、(1.47±0.33)、(0.60± 0.14)、(1.98±0.70)和(1.91±0.27)倍,其中228 h差异极显著(P < 0.01),其他时间点差异显著(P < 0.05) (图 10B)。凡纳对虾感染WSSV后24、48、72、96、192和228 h,parkin在肌肉中的表达水平分别为对照组的(2.56±0.97)、(0.73±0.05)、(0.63±0.19)、(2.19±0.66)、(2.67±0.78)和(2.44±0.66)倍,其中48 h和96 h差异极显著(P < 0.01),其他时间点差异显著(P < 0.05) (图 10C)。凡纳对虾感染WSSV后48、96和144 h,parkin在眼柄中的表达水平分别为对照组的(1.59±0.43)、(1.98±0.41)和(1.54±0.21)倍,其中48 h差异显著(P < 0.05),其他时间点差异极显著(P < 0.01) (图 10D)。
|
图 10 感染WSSV后parkin在凡纳对虾肝胰腺、鳃、肌肉和眼柄中的表达 Fig.10 Expression profiles of parkin in hepatopancreas, gill, muscle, and eyestalk of P. vannamei post-WSSV infection A:肝胰腺;B:鳃;C:肌肉;D:眼柄。 A: Hepatopancreas; B: Gill; C: Muscle; D: Eyestalk. |
选取感染WSSV 96 h后PACRG表达水平最高的凡纳对虾肌肉组织进行PACRG mRNA和WSSV DNA空间定位,结果如图 11所示。与对照组相比,感染WSSV 96 h后凡纳对虾肌肉中PACRG的表达量明显增加,并且PACRG mRNA的表达位置大多与WSSV的位置呈现重合的状态(图 11)。
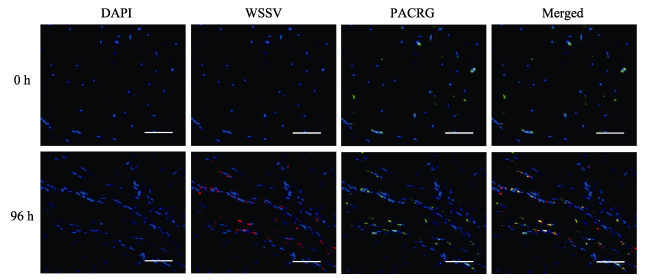
|
图 11 凡纳对虾肌肉中PACRG mRNA和WSSV DNA的位置(比例尺为50 μm) Fig.11 The location of PACRG mRNA and WSSV DNA in the muscle of P. vannamei (The scale bar is 50 μm) |
测序结果显示,在凡纳对虾PACRG ORF中共筛选到2个SNP位点,分别在ORF的369 bp和411 bp处,命名为SNP1和SNP2 (表 2),编码甘氨酸和亮氨酸,2个SNP位点均属于同义突变。
|
|
表 2 PACRG基因敏感组和抗性组SNP位点突变类型及多态性分析 Tab.2 Analysis of SNP mutation types and polymorphism in PACRG between susceptible group (SG) and resistant group (RG) |
通过PLINK1.9软件对PACRG SNP位点的多态性进行分析。结果表明,敏感组群体的Ho为0.400~ 0.500,He为0.375~0.491,MAF为0.250~0.433。PIC为0.305~0.371,表现为中度多态性。无MAF < 0.05的SNP位点。HWE结果表明,SNP1和SNP2位点均符合HWE (表 2)。抗性组群体的Ho为0.300~0.467,He为0.480~0.499,MAF为0.400~0.483。PIC为0.365~0.375,表现为中度多态性。无MAF < 0.05的SNP位点。HWE结果表明,SNP1位点符合HWE,SNP2位点偏离HWE(表 2)。
2.10 PACRG基因SNP位点与抗WSSV性状的关联利用卡方检验(χ2)分析PACRG基因2个SNP位点的基因分型结果与抗WSSV性状之间的关联性,结果见表 3。SNP1和SNP2位点与抗WSSV性状无显著关联。
|
|
表 3 PACRG基因SNP位点与抗WSSV性状的关联分析 Tab.3 Correlation analysis between SNP sites and WSSV resistance traits in the PACRG gene |
测序结果显示,在凡纳对虾parkin中共筛选到15个SNP位点,每个SNP的位置从parkin mRNA序列(XM_027357483.1)开始,命名为SNP1~15 (表 4)。SNP1位点为同义突变,位于parkin的ORF区。其余SNP位点均位于3′-UTR区。
|
|
表 4 parkin基因敏感组和抗性组SNP位点突变类型及多态性分析 Tab.4 Analysis of SNP mutation types and polymorphism in parkin between susceptible group (SG) and resistant group (RG) |
对SNP位点的多态性进行分析结果显示,敏感组群体的Ho为0.100~0.600,He为0.095~0.491,MAF为0.050~0.433。PIC为0.090~0.371,SNP5、SNP6、SNP7、SNP8、SNP11、SNP12、SNP14和SNP15位点表现为低度多态性,SNP1、SNP2、SNP3、SNP4、SNP9、SNP10和SNP13位点表现为中度多态性。无MAF < 0.05的SNP位点。HWE结果表明,15个SNP位点均符合HWE (表 4)。抗性组群体的Ho为0.167~ 0.700,He为0.153~0.495,MAF为0.083~0.450,PIC为0.141~0.372,SNP6、SNP8、SNP11、SNP12、SNP14和SNP15位点表现为低度多态性,SNP1、SNP2、SNP3、SNP4、SNP5、SNP7、SNP9、SNP10和SNP13位点表现为中度多态性。无MAF < 0.05的SNP位点。HWE结果表明,15个SNP位点均符合HWE(表 4)。
2.12 parkin基因SNP位点与抗WSSV性状的关联分析利用卡方检验(χ2)分析parkin基因15个SNP位点的基因分型结果与抗WSSV性状之间的关联性,结果见表 5。其中,SNP3、SNP4、SNP5、SNP7和SNP9位点与抗WSSV性状显著相关。
|
|
表 5 parkin基因SNP位点与抗WSSV性状的关联分析 Tab.5 Correlation analysis between SNP sites and WSSV resistance traits in the parkin gene |
PACRG与parkin基因共用一个双向启动子,二者均受UPS的调控,将泛素与受损或错误折叠的蛋白质结合,随后被蛋白酶体降解,发挥维持细胞稳态的作用(Taylor et al, 2007; West et al, 2003)。parkin是一种具有泛素蛋白E3连接酶活性的多结构域蛋白,可通过类似线粒体的自噬途径对病原菌的某些蛋白进行泛素化标记并清除。许玲(2016)在日本对虾(Penaeus japonicus)中鉴定出泛素连接酶parkin,表明进入细胞内的WSSV病毒的囊膜蛋白被parkin泛素化修饰,然后通过蛋白酶体和异体自噬的方式被清除。PACRG在甲壳类动物中的功能研究较少,在哺乳动物中的研究较为广泛。Taylor等(2007)通过探究PACRG与帕金森病发病机制的相关性,表明PACRG受UPS的调节,可能在帕金森病的发病机制中发挥作用(Taylor et al, 2007)。Liu等(2021)通过重组表达来探究PACRG的生物学功能,发现PACRG参与泛素依赖的蛋白质代谢过程,同时可能在基因转录调控中发挥重要作用。
本研究对凡纳对虾中PACRG和parkin基因进行mRNA和预测氨基酸序列的特征分析,其中,PACRG ORF序列全长600 bp,编码199个氨基酸,包含一个ParcG结构域。对凡纳对虾PACRG蛋白与不同物种间的氨基酸序列进行同源性分析发现,凡纳对虾的PACRG与日本对虾的氨基酸序列同源性最高,为89.70%。凡纳对虾与中国对虾和日本对虾的亲缘关系最近。推测PACRG具有较高的进化保守性。这与Dawe等(2005)证实PACRG是一种保守的轴丝蛋白的结果相一致。parkin ORF序列全长1 653 bp,编码550个氨基酸,包含一个信号肽结构,在33~104氨基酸位置处包含一个UBQ基因结构域,400~460氨基酸位置包含一个IBR结构域,487~543氨基酸位置包含一个IBR结构域(图 2)。对parkin蛋白在不同物种间的氨基酸序列进行同源性分析,发现凡纳对虾的parkin与中国对虾和斑节对虾的同源性最高,为93.45%。凡纳对虾与中国对虾和斑节对虾的亲缘关系最近。推测parkin同样具有较高的进化保守性。
为了证实PACRG和parkin基因在防御WSSV感染过程中发挥作用,采用real-time PCR技术对健康和感染WSSV后不同时间凡纳对虾肝胰腺、鳃、肌肉和眼柄中PACRG和parkin基因的表达水平进行检测。结果显示,健康凡纳对虾中的PACRG和parkin在肝胰腺、鳃、肌肉和眼柄中均有表达,且无显著差异。West等(2003)分析表明,PACRG和parkin在人体许多组织中共表达,如脑、心脏和肌肉。感染WSSV后48、96、192和228 h,对虾肝胰腺中PACRG和parkin的表达水平均呈现显著的下调趋势。感染WSSV后48、72、96、144、192和228 h,对虾鳃中PACRG的表达水平呈极显著的下调趋势;而感染WSSV后48、96和228 h,对虾鳃中parkin的表达水平呈现显著的上调趋势。感染WSSV后96、192和228 h,对虾肌肉中PACRG和parkin的表达水平均呈现显著的上调趋势。感染WSSV后眼柄中PACRG和parkin表现出相似的组织表达模式。研究显示,双向启动子基因通常表现出相似的组织表达模式和生物功能,或者基因产物通常在共同的途径中发挥作用(Li et al, 2006)。本研究中,凡纳对虾在感染WSSV后不同时间、不同组织中的PACRG和parkin表达水平有显著变化,推测其可能参与凡纳对虾抗WSSV的过程。此外,FISH结果表明,感染WSSV 96 h后,凡纳对虾肌肉中PACRG mRNA和WSSV的位置呈现重合状态,推测PACRG可能在凡纳对虾与WSSV的相互作用中发挥功能。
SNP突变引起的碱基改变可以使其编码的蛋白质结构和功能发生改变,从而直接影响生物体的性状(杜玮南等, 2000)。有研究表明,麻风病、结核病、伤寒病、白血病和星形胶质细胞肿瘤这些疾病的易感性与PACRG和parkin共有的双向启动子区域的变异有关(Mira et al, 2000; Ali et al, 2006; Ichimura et al, 2006)。parkin基因的突变或缺失与常染色体隐性遗传的青少年帕金森病有关(Kitada et al, 1998)。本研究对960尾凡纳对虾进行WSSV感染并按照死亡时间筛选出敏感组和抗性组进行SNP分析,其中,在PACRG的ORF区共筛选到2个SNP位点,在parkin的ORF区筛选到1个SNP位点,在parkin的UTR区筛选到14个SNP位点。将这些SNP位点进行抗WSSV性状的关联分析后发现,位于parkin UTR区的SNP3、SNP4、SNP5、SNP7和SNP9位点与抗WSSV性状显著相关。尽管非翻译区的SNP不能直接改变蛋白质的氨基酸序列,但对mRNA的稳定性和翻译调控具有重要作用(余光创等, 2007)。
综上所述,共用一个双向启动子的PACRG和parkin具有较高的进化保守性。凡纳对虾感染WSSV后,PACRG和parkin在对虾肝胰腺、鳃、肌肉和眼柄中的表达水平发生显著变化,其中,眼柄中的PACRG和parkin表现出极为相似的组织表达模式。FISH结果表明,在对虾肌肉中PACRG mRNA和WSSV DNA的位置呈重合状态,进一步提示PACRG在凡纳对虾与WSSV的相互作用中发挥功能。通过SNP位点与抗WSSV性状关联分析,parkin UTR区发现有抗WSSV性状相关的SNP。上述结果提示PACRG和parkin可能参与凡纳对虾的抗WSSV过程,但具体功能需要进一步研究。
ALI S, VOLLAARD A M, WIDJAJA S, et al. PARK2/PACRG polymorphisms and susceptibility to typhoid and paratyphoid fever. Clinical and Experimental Immunology, 2006, 144(3): 425-431 DOI:10.1111/j.1365-2249.2006.03087.x |
CARON H, VAN SCHAIK B, VAN DER MEE M, et al. The human transcriptome map: Clustering of highly expressed genes in chromosomal domains. Science, 2001, 291(5507): 1289-1292 DOI:10.1126/science.1056794 |
CHEN J, LI Z J, HEW C L. Characterization of a novel envelope protein WSV010 of shrimp white spot syndrome virus and its interaction with a major viral structural protein VP24. Virology, 2007, 364(1): 208-213 DOI:10.1016/j.virol.2007.02.030 |
CHEN K, LI E, XU C, et al. Growth and metabolomic responses of Pacific white shrimp (Litopenaeus vannamei) to different dietary fatty acid sources and salinity levels. Aquaculture, 2019, 499: 329-340 DOI:10.1016/j.aquaculture.2018.09.056 |
DAGAR G, KUMAR R, YADAV K K, et al. Ubiquitination and deubiquitination: Implications on cancer therapy. Biochimica et Biophysica Acta-Gene Regulatory Mechanisms, 2023, 1866(4): 194979 DOI:10.1016/j.bbagrm.2023.194979 |
DAWE H R, FARR H, PORTMAN N, et al. The Parkin co-regulated gene product, PACRG, is an evolutionarily conserved axonemal protein that functions in outer-doublet microtubule morphogenesis. Journal of Cell Science, 2005, 118(23): 5421-5430 DOI:10.1242/jcs.02659 |
DI NAPOLI M, PAPA F. The proteasome system and proteasome inhibitors in stroke: controlling the inflammatory response. Current Opinion in Investigational Drugs, 2003, 4(11): 1333-1342 |
DONG L J, LUO K, CAO J W, et al. Comparison of WSSV-resistant traits of different commercial larvae of Pacific white shrimp Litopenaeus vannamei. Progress in Fishery Sciences, 2019, 40(1): 69-75 [董丽君, 罗坤, 曹家旺, 等. 凡纳滨对虾商业苗种抗WSSV性能比较. 渔业科学进展, 2019, 40(1): 69-75] |
DU W N, SUN H X, FANG F D. The research development of single nucleotide polymorphism. Acta Academiae Medicinae Sinicae, 2000, 22(4): 392-394 [杜玮南, 孙红霞, 方福德. 单核苷酸多态性的研究进展. 中国医学科学院学报, 2000, 22(4): 392-394] |
FENG Y P, KONG J, LUO K, et al. The comparison of the sensitivity to the white spot syndrome virus between Fenneropenaeus chinensis and Litopenaeus vannamei. Progress in Fishery Sciences, 2017, 38(6): 78-84 [冯亚萍, 孔杰, 罗坤, 等. 中国对虾(Fenneropenaeus chinensis)和凡纳滨对虾(Litopenaeus vannamei)对白斑综合征病毒的敏感性比较. 渔业科学进展, 2017, 38(6): 78-84] |
HOU C H, SONG W L, YUAN H, et al. Comparative transcriptome analysis revealed that dietary zymosan-A improved the immunity of Penaeus vannamei by regulating the TLR signaling pathway. Aquaculture, 2022, 561: 738603 DOI:10.1016/j.aquaculture.2022.738603 |
ICHIMURA K, MUNGALL A J, FIEGLER H, et al. Small regions of overlapping deletions on 6q26 in human astrocytic tumours identified using chromosome 6 tile path array-CGH. Oncogene, 2006, 25(8): 1261-1271 DOI:10.1038/sj.onc.1209156 |
IMAI Y, SODA M, MURAKAMI T, et al. A product of the human gene adjacent to parkin is a component of Lewy bodies and suppresses Pael receptor-induced cell death. Journal of Biological Chemistry, 2003, 278(51): 51901-51910 DOI:10.1074/jbc.M309655200 |
KITADA T, ASAKAWA S, HATTORI N, et al. Mutations in the parkin gene cause autosomal recessive juvenile parkinsonism. Nature, 1998, 392(6676): 605-608 DOI:10.1038/33416 |
LI Y Y, YU H, GUO Z M, et al. Systematic analysis of head-to-head gene organization: Evolutionary conservation and potential biological relevance. PLoS Computational Biology, 2006, 2(7): e74-112 DOI:10.1371/journal.pcbi.0020074 |
LIU T K, ZHAO H Z, JIAN S F, et al. Functional expression, purification and identification of interaction partners of PACRG. Molecules, 2021, 26(8): 2308-2322 DOI:10.3390/molecules26082308 |
MENG X H, ZHANG T S, KONG J, et al. Equivalent and quantitative test method of white spot syndrome virus resistance of prawns. ZL201210107377.8. 2013-12-25 [孟宪红, 张天时, 孔杰, 等. 对虾抗白斑综合症病毒能力的等量、定量测试方法. ZL201210107377.8. 2013-12-25]
|
MESCHEDE J, ŠADIć M, FURTHMANN N, et al. The parkin-coregulated gene product PACRG promotes TNF signaling by stabilizing LUBAC. Science Signaling, 2020, 13(617): eaav1256-1269 DOI:10.1126/scisignal.aav1256 |
MIRA M T, ALCAÏS A, NGUYEN V T, et al. Susceptibility to leprosy is associated with PARK2 and PACRG. Nature, 2004, 427(6975): 636-640 DOI:10.1038/nature02326 |
MIZUNO K, DYMEK E E, SMITH E F, et al. Microtubule binding protein PACRG plays a role in regulating specific ciliary dyneins during microtubule sliding. Cytoskeleton, 2016, 73(12): 703-711 DOI:10.1002/cm.21340 |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: A tool set for whole-genome association and population-based linkage analyses. American Journal of Human Genetics, 2007, 81(3): 559-575 DOI:10.1086/519795 |
SCOTT L, DAWSON V L, DAWSON T M. Trumping neurodegeneration: Targeting common pathways regulated by autosomal recessive Parkinson's disease genes. Experimental Neurology, 2017, 298(B): 191-201 |
SPELLMAN P T, RUBIN G M. Evidence for large domains of similarly expressed genes in the Drosophila genome. Journal of Biology, 2002, 1(1): 5 DOI:10.1186/1475-4924-1-5 |
TAYLOR J M, SONG Y J C, HUANG Y, et al. Parkin co-regulated gene (PACRG) is regulated by the ubiquitin- proteasomal system and is present in the pathological features of parkinsonian diseases. Neurobiology of Disease, 2007, 27(2): 238-247 DOI:10.1016/j.nbd.2007.04.014 |
TRINKLEIN N D, ALDRED S F, HARTMAN S J, et al. An abundance of bidirectional promoters in the human genome. Genome Research, 2004, 14(1): 62-66 DOI:10.1101/gr.1982804 |
WEST A B, LOCKHART P J, O′ FARELL C, et al. Identification of a novel gene linked to parkin via a bi-directional promoter. Journal of Molecular Biology, 2003, 326(1): 11-19 DOI:10.1016/S0022-2836(02)01376-1 |
WINKLHOFER K F. Parkin and mitochondrial quality control: Toward assembling the puzzle. Trends in Cell Biology, 2014, 24(6): 332-341 DOI:10.1016/j.tcb.2014.01.001 |
XU L. E3 Ligase Parkin mediates the immune response against white spot syndrome virus by ubiquitin-proteasome and xenophage pathways. Master′s Thesis of Shandong University, 2016 [许玲. 泛素连接酶Parkin通过蛋白酶体与异体自噬途径介导对虾的抗病毒免疫反应. 山东大学硕士研究生学位论文, 2016]
|
YU G C, QIN Y D, BO X C, et al. Translational control of eukaryotic gene depending on higher structures in 5′ untranslated regions. Chinese Journal of Biochemistry and Molecular Biology, 2007, 23(11): 881-887 [余光创, 秦宜德, 伯晓晨, 等. 依赖于5′端非编码区高级结构的真核生物mRNA翻译调控. 中国生物化学与分子生物学报, 2007, 23(11): 881-887] |
ZHAO H Z, LI J, MA Y H, et al. Research progress of Parkin co-regulated gene. Chinese Pharmacological Bulletin, 2018, 34(10): 1333-1336 [赵海洲, 李军, 马延红, 等. Parkin共调基因调控功能与疾病研究进展. 中国药理学通报, 2018, 34(10): 1333-1336] |



