2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 上海海洋大学海洋科学学院 上海 201306
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071;
3. College of Marine Sciences, Shanghai Ocean University, Shanghai 201306
全球渔业衰退是21世纪面临的重大挑战之一(Dudgeon, 2010; 金显仕等, 2015; Evans et al, 2018)。然而,对研究人员与政策制定者而言,收集水生生物种群分布与种群动态变化的精确数据是其面临的最大挑战(Thomsen et al, 2015),特别是对于某些密度极小与具有特殊生活史的种群,检测其时空分布与调查其种群动态变化将变得更加困难(Mackenzie et al, 2005; Dejean et al, 2011)。为了克服以上困难,DNA条形码技术被成功地应用到水生生态系统中,检测来自生物体细胞外的DNA(即eDNA)(Bohmann et al, 2014; 单秀娟等, 2018),进而通过eDNA去掌握物种的分布与种群动态变化。该方法不需要采集生物样本,对生态系统无伤害且经济高效。目前,该方法已成功应用到生物检测(Goldberg et al, 2015; Davison et al, 2017)、生物多样性评价(Drummond et al, 2015; Majaneva et al, 2018)、生物量评估(Pillioddavid et al, 2013; Tillotson et al, 2018)、鱼类洄游(Yamanaka et al, 2016)以及其他方面(Sigsgaard et al, 2016、2017)的研究中。
由于eDNA极易降解,且在环境中含量极低,因而,其在环境中的存留时间将会直接影响后期的定性与定量分析。为了能够将eDNA技术准确地应用到水生生态系统的研究领域中,探究eDNA在水体中的存留时间显得尤为重要。eDNA在环境中的存留时间是指切断eDNA的来源之后,eDNA在环境中的持续存在时间(Dejean et al, 2011)。虽然,已有相关研究表明,eDNA在水体中呈指数式降解(Dejean et al, 2011; Strickler et al, 2015),但不同的物种具有不同的生活史,其释放eDNA的速率各不相同(Minamoto et al, 2017),从而导致不同物种释放的eDNA在水体中的存留时间不同。本研究以中国对虾(Fenneropenaeus chinensis)为研究对象,结合实时荧光定量PCR,检测eDNA在水体中的存留时间,同时,探究eDNA降解速率与时间之间的关系。本研究旨在为水生生态系统eDNA的取样、操作流程优化、整体实验方案设计及应用提供理论基础。
1 材料与方法 1.1 特异性引物设计针对中国对虾的线粒体细胞色素酶氧化亚基Ⅰ(COⅠ)基因设计特异性引物。在GenBank数据库中检索中国对虾线粒体DNA (mtDNA) COⅠ基因序列(GenBank登录号: gbHQ700930.1),利用BioEdit和MEGA7软件进行序列比对,使用Primer Premier 6与Beacon Designer 8软件设计引物与探针,并在NCBI网站上进行引物特异性测试。
筛选确定1对特异性扩增中国对虾COⅠ基因的目的片段为597 bp的普通PCR引物(COⅠPF:TTGT AGTTACAGCCCACGCT,COⅠPR:AAATTATCCCGAAGGCGGGT)与1对目的片段为106 bp的荧光定量PCR引物(COⅠDF:AGGGGTAGGAACAGGATGAAC,COⅠDR:GACACCAGCTAGATGCAGCG,Probe:5'FAM-TCAGCTAGAATTGCTCATGCCGGAGCTTCAGT-3'BHQ1),其中,普通PCR引物用来制备质粒标准品DNA,定量PCR引物所扩增的目的片段为普通PCR引物所扩增目的片段的一部分。引物由生工生物工程(上海)股份有限公司合成。
1.2 中国对虾肌肉组织DNA提取及PCR扩增实验所用中国对虾肌肉组织取自2016年渤海渔业资源调查捕获的中国对虾,–20℃保存。
中国对虾肌肉组织DNA的提取采取传统的酚–氯仿–异戊醇方法,提取完成后,使用紫外分光光度计检测DNA浓度并稀释到50 ng/μl,用1.5 ml无菌离心管分装,–20℃保存。25 μl PCR反应体系:10×Taq Buffer 2.5 μl,dNTPs (各2.5 mmol/L) 0.5 μl,正反向引物(COⅠPF/COⅠPR)(10 mmol/L)各0.5 μl,Taq DNA Polymerase(5 U/μl) 0.5 μl,模板DNA (50 ng/μl) 1 μl, MgCl2 (25 mmol/L) 1.5 μl,ddH2O 18 μl。PCR扩增反应条件:94℃预变性3 min;94℃ 30 s,60℃ 30 s,72℃ 1 min,扩增35个循环;72℃复延伸10 min。PCR产物用2%的琼脂糖凝胶电泳进行检测。
1.3 中国对虾mtDNA COⅠ基因克隆及质粒标准品制备将1.2中PCR产物用Gel Extraction Kit (OMEGA)试剂盒进行纯化。将纯化的PCR产物连接到pMD-18-T质粒载体(TaKaRa)上,转化入DH5α感受态细胞(TaKaRa)中,最后在含Amp、X-gal及IPTG的LB固体平板培养基上过夜培养,经过蓝白斑筛选后,挑取8个白色单菌落,分别置于1.5 ml离心管中培养8 h,将菌液送至生工生物工程(上海)股份有限公司测序。
根据测序结果,选取连接转化成功的菌液扩大培养。培养24 h后,使用Plasmid Mini Kit I (OMEGA)进行质粒标准品DNA提取。DNA提取完成后,使用紫外分光光度计检测并计算其拷贝数,最后将其稀释到108 copies/μl,–80℃保存备用。
以上所有实验步骤均按照说明书进行操作。
1.4 样品采集为了检测eDNA在水体中的存留时间,必须保证水体中有一定量的eDNA,且在开始检测时切断其eDNA的释放源头,以免生物体不断地向水体中释放DNA而影响实验结果。因此,通常是在固定的水体中饲养研究对象一段时间,待水体中eDNA有一定量的积累后将生物体从水体中移除。
本实验所用水样取自中国水产科学研究院黄海水产研究所水产遗传育种中心中国对虾养殖池(养殖池内只有中国对虾这单一物种,且养殖池内有足够量的eDNA),养殖池长5.5 m、宽3.6 m、高1.2 m,池内水体体积为14 m3,池内有中国对虾180只,虾体平均体重为27 g。从池内取25 L水样用提前消毒的无菌白色塑料桶带回实验室,室温保存,桶内用充气泵保持充气直至取样完毕(有关养殖池信息及中国对虾体长、体重数据均由遗传育种中心工作人员测量)。
取样工作参照Dejean等(2008)的方法并加以改进,每隔24 h从水桶内取15 ml水样,装进50 ml无菌离心管内,同时,在离心管内加3 mol/L的醋酸钠溶液1.5 ml与无水乙醇33 ml,每次取3个平行样本,–20℃保存直至进行eDNA提取。水样采集时间为2018年1月12日~2月7日,取样时间为每天08:00。
1.5 eDNA提取选用DNeasy Blood and Tissue kit (Qiagen, Hilden, 德国)试剂盒进行eDNA的提取,提取方法参照Ficetola等(2008)、Dejean等(2008)与Renshaw等(2015),并进行相应改进,其具体提取步骤为:
将水样从冰箱拿出,4℃ 9000 g离心1 h。将上一步离心管底部的沉淀物全部刮出,放入2 ml的无菌离心管中,加入570 μl的Buffer ATL与60 μl的蛋白酶K,涡旋震荡混匀,恒温水浴3 h直至沉淀物完全裂解,水浴期间,每隔15 min轻轻颠倒混匀离心管以加速裂解,裂解完成后涡旋震荡15 s。其余步骤按照试剂盒说明书进行,其中,最后一步中DNA的洗脱用60 μl Buffer TE,而非试剂盒中Buffer AE。
eDNA提取完成后,立即使用紫外分光光度计检测其浓度,若eDNA样品浓度高于250 ng/μl,则将其进行稀释。每个eDNA样品吸取10 μl用作琼脂糖凝胶电泳检测及PCR定量分析,其余50 μl eDNA溶液于–80℃保存。
1.6 eDNA的定量分析所有提取的eDNA样品采用BBI生命科学有限公司2×TaqMan Fast qPCR Master Mix(Low Rox)实时荧光定量PCR试剂盒进行定量分析。PCR采用20 μl体系:10 μl 2×TaqMan Fast qPCR Master Mix,正反向引物(10 μmol/L)各0.4 μl,0.4 μl探针(10 μmol/L),2 μl模板DNA,6.8 μl PCR级水。扩增反应程序采用两步法:94℃预变性3 min;94℃变性5 s,60℃退火延伸34 s,40个循环。
PCR扩增使用ABI 7500型定量PCR仪和96孔板(Thermo Fisher),标准品DNA与未知浓度eDNA样品均进行3个重复,每个96孔板进行3个阴性对照(无模板)与3个阳性对照(中国对虾基因组DNA),标准品以10倍浓度梯度从107 copies/μl稀释至101 copies/μl。实验数据采用绝对定量法分析,eDNA拷贝数取阳性扩增样品的平均值,应用系统软件SDS1.4.0.25自动计算Ct值及生成标准曲线与扩增曲线。
1.7 数据分析基于AIC选择了最适于eDNA随时间降解的模型(Burnham, 2002; Wickham, 2009),所有数据使用R3.5.0进行处理,误差控制在95%的置信区间以内。
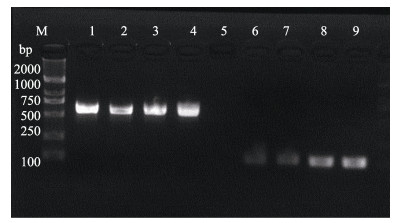
2 结果与分析 2.1 引物特异性验证PCR产物经2%的琼脂糖凝胶电泳检测,结果显示,引物对COⅠPF/PR与COⅠDF/DR成功扩增出597 bp与106 bp的目的片段,与预期结果完全一致,电泳条带单一且明亮,无杂带(图 1),证明所设计的引物特异性很好。
|
图 1 引物COⅠPF/PR与COⅠDF/DR的扩增结果 Fig.1 Amplification of primer COⅠPF/PR and COⅠDF/DR M: DNA Marker DL 2000;泳道1~4为引物COⅠPF/PR的PCR产物;泳道6~9为引物COⅠDF/DR的PCR产物 M: DNA Marker DL 2000; Lane 1~4: PCR products of COⅠPF/PR; Lane 6~9: PCR products of COⅠDF/DR |
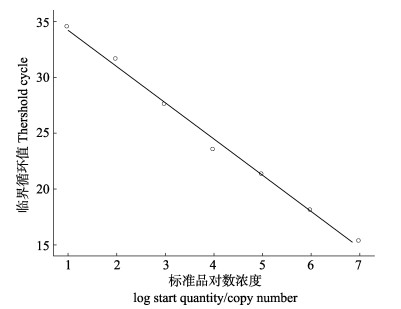
通过实时荧光定量PCR扩增,PCR检测系统根据荧光值的变化规律,自动生成中国对虾COⅠ基因的标准曲线(图 2)。曲线的斜率为K=-3.15,相关系数R2=0.994,回归方程为y=-3.24x+37.47,说明在稀释的质粒标准品DNA浓度范围内具有良好的线性关系,表明本研究建立的标准曲线能够准确地反映中国对虾COⅠ基因的扩增。
|
图 2 中国对虾COⅠ基因AQ-PCR标准曲线 Fig.2 Standard curve of AQ-PCR of Fenneropenaeus chinensis COⅠ gene |
使用紫外分光光度计检测未知浓度eDNA样品发现,大部分样品的A260 nm/A280 nm值均低于1.8,只有极少数样品的A260 nm/A280 nm值在1.8~2.0之间,且eDNA样品浓度普遍较低,说明水体中eDNA含量较少且水体中杂质较多,因而导致提取的DNA纯度较低。
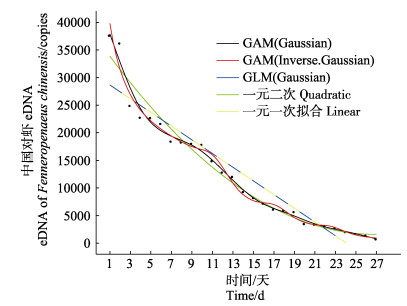
2.4 eDNA降解结果检测结果显示,在eDNA的释放源头去除后,随着时间的推移,水体中eDNA的拷贝数与时间呈负相关关系。第1天(2018年1月12日)检测时每15 ml水样中含有的DNA拷贝数为3.76×104,而在第27天(2018年2月7日)检测时,每15 ml水体中的eDNA拷贝数则降解为711。此外,基于AIC比较了GAM (Gaussian)、GAM(Inverse.Gaussian)、GLM、一元一次回归及一元二次回归5种模型对eDNA降解与时间之间的关系的适用性,发现用GAM(Gaussian)模型拟合eDNA降解与时间之间的关系曲线AIC值最小(AIC=472.0694)(表 1),同时,其相关系数R2也最高(R2=0.984) (图 3),说明GAM(Gaussian)模型能更好反映eDNA的降解与时间之间的关系。
|
|
表 1 基于AIC值对模型的选择 Tab.1 The choice of model based on AIC value |
|
图 3 中国对虾eDNA降解与时间之间的关系拟合 Fig.3 Relationship fitting between degradation of eDNA and time in Fenneropenaeus chinensis |
eDNA的检出率主要取决于释放速率与降解速率,同时,目标种种群数量的大小对eDNA的检出率也有一定的影响。此外,除了不可控的自然因素外,一些人为因素也对eDNA检出率的高低有不可忽视的影响。eDNA从生物体释放进入水环境后,可能会持续存在,吸附在有机或者无机颗粒上成为沉积物,或者在水环境中逐渐被降解(Levy-Booth et al, 2007),影响其降解的因素主要有核酸酶、温度、pH、紫外光照、水化学及微生物的活动等一系列生物与非生物因素(Barnes et al, 2014; Strickler et al, 2015; Tsuji et al, 2016)。
3.1 长片段eDNA与短片段eDNA在研究中的区别eDNA由释放源进入水环境后,随着时间的变化,一般经过由大片段逐渐分解为小片段直至降解的过程,在这个过程中,根据其环境条件的不同,eDNA在不同环境因子的影响下被加速降解。片段大小为300~400 bp的eDNA在人为实验控制的条件下能够在水体中持续存在7 d (Zhu, 2006; Dejean et al, 2011),7 d后仍能检测到的eDNA则为≤100 bp左右的短片段DNA。本研究采用实时荧光定量PCR (TaqMan法)扩增了一段目的片段大小为106 bp的短片段eDNA,检测了短片段eDNA在水体中滞留时间的长短,结果发现,较长片段eDNA而言,短片段eDNA在水体存留的时间要长很多,其完全降解至少需要30 d,但是,长片段eDNA能够更加准确地反映最新的生物信息(Hänfling et al, 2016; Bista et al, 2017)。同时,这也反映出研究者应该针对自己的最终目的设计实验,扩增符合需求的eDNA片段,以达到最佳的研究效果。Jo等(2017)设计了1对能够扩增719 bp的长片段eDNA的引物与1对能够扩增127 bp的短片段eDNA的引物,分别应用实时荧光定量PCR对竹筴鱼(Trachurus japonicus)进行了生物量估测及分布监测,表明扩增长片段的eDNA能够更加精确地评估生物量以及对物种分布做出准确判断。根据长短片段eDNA在水体中滞留时间的不同,建议在利用eDNA技术进行生物量评估与物种时空动态分布监测时,设计能够扩增长片段eDNA的引物以扩增来自于生物体最新释放进入水体的DNA,而不是长期滞留于水体中的DNA,以避免2个资源调查周期之间短片段eDNA的累积效果所导致的实验误差。
3.2 不同物种释放的eDNA在水体中存留时间的区别一般而言,水生动物释放的eDNA在水体的存留时间为7~30 d,但不同的生物种类的生活史特征不同,释放eDNA的速率不同(Minamoto et al, 2017),释放eDNA量的多少与eDNA片段的大小也各不相同(Geerts et al, 2018)。因此,对不同的物种而言,其释放的DNA在环境中的存留时间也不同。Thomsen等(2012)研究发现,在饲养锄足蟾(Pelobates fuscus)和冠北螈(Triturus cristatus)时,养殖水体中eDNA的量随时间不断增加,但将其移除后,水体中eDNA的量快速下降,7~14 d后,eDNA完全降解;而Goldberg等(2013)以淡水螺(Potamopyrgus antipodarum)为研究对象,在15℃恒温条件下饲养淡水螺一段时间后将其移除,发现其释放到水体中的DNA能够在环境中存留14~42 d。本研究与前人的研究相比,中国对虾所释放的eDNA在水体中的存留时间较长,主要是因为甲壳类的整个生活史过程中存在蜕皮现象,因而其释放到水中的eDNA的量较大。此外,本研究所取水样的养殖池中的中国对虾密度较大,是eDNA在水体中存留时间较长的原因之一。
3.3 环境因子对eDNA存留时间的影响除了物种本身生活史差异性造成的存留时间不同外,环境因子的变动也会导致eDNA在水体中存留时间的差异。已有研究表明,温度与eDNA的降解速率呈正相关关系(Strickler et al, 2015; Lacoursière-Roussel et al, 2016)。Strickler等(2015)研究了美国牛蛙(Lithobates catesbeianus)释放到水体中的eDNA分别在5℃、25℃和30℃的条件下的降解情况,发现当环境中eDNA的量相同时,5℃时eDNA的降解最缓慢,主要因为低温条件下微生物的生长过程较为缓慢,降低了有关生命活动导致的eDNA的降解。本研究所用水体是在冬天室温(15℃)的情况下保存的,与夏季相比,eDNA的降解速率较为缓慢。因此,对于自然水体而言,随着季节的交替变化,水温发生变化,其水环境中的eDNA的降解速率也发生变化。此外,研究表明,pH、紫外辐射以及水化学对eDNA的降解也存在一定的影响(Pilliod et al, 2013; Barnes et al, 2014; Eichmiller et al, 2014、2016a、b)。但是,以上环境因子共同作用时如何影响eDNA的降解尚不清楚。未来在应用eDNA技术准确评估目标种生物量之前,解决上述环境因子对eDNA降解的影响机制至关重要。
4 小结通过检测eDNA在水体中的存留时间发现,短片段eDNA在水环境中能够存留长达30 d左右,可以据此来合理设计实验方案、确定合适的采样周期以及寻求妥善的eDNA存储方法,从而为eDNA技术更有力地应用到水生生态系统的研究领域奠定基础。
致谢: 衷心感谢中国水产科学研究院黄海水产研究所水产动物遗传育种中心陈宝龙老师与课题组项目聘用人员王惠宾在取样方面给予的建议与帮助!
Barnes MA, Turner CR, Jerde CL, et al. Environmental conditions influence eDNA persistence in aquatic systems. Environmental Science and Technology, 2014, 48(3): 1819 DOI:10.1021/es404734p |
Bista I, Carvalho GR, Walsh K, et al. Annual time-series analysis of aqueous eDNA reveals ecologically relevant dynamics of lake ecosystem biodiversity. Nature Communications, 2017, 8: 14087 DOI:10.1038/ncomms14087 |
Bohmann K, Evans A, Gilbert MT, et al. Environmental DNA for wildlife biology and biodiversity monitoring. Trends in Ecology and Evolution, 2014, 29(6): 358-367 DOI:10.1016/j.tree.2014.04.003 |
Burnham KP. Information and likelihood theory: A basis for model selection and inference//Model selection and multimodel inference. Springer New York, 2002, 49-97
|
Davison PI, Copp GH, Créach V, et al. Application of environmental DNA analysis to inform invasive fish eradication operations. Science of Nature, 2017, 104(3-4): 35 DOI:10.1007/s00114-017-1453-9 |
Dejean T, Valentini A, Duparc A, et al. Persistence of environmental DNA in freshwater ecosystems. PLoS One, 2011, 6(8): e23398 DOI:10.1371/journal.pone.0023398 |
Drummond AJ, Newcomb RD, Buckley TR, et al. Evaluating a multigene environmental DNA approach for biodiversity assessment. GigaScience, 2015, 4(1): 46 DOI:10.1186/s13742-015-0086-1 |
Dudgeon D. Prospects for sustaining freshwater biodiversity in the 21st century:Linking ecosystem structure and function. Current Opinion in Environmental Sustainability, 2010, 2(5-6): 422-430 DOI:10.1016/j.cosust.2010.09.001 |
Eichmiller JJ, Bajer PG, Sorensen PW. The relationship between the distribution of common carp and their environmental DNA in a small lake. PLoS One, 2014, 9(11): e112611 DOI:10.1371/journal.pone.0112611 |
Eichmiller JJ, Best SE, Sorensen PW. Effects of temperature and trophic state on degradation of environmental DNA in lake water. Environmental Science and Technology, 2016a, 50(4): 1859-1867 DOI:10.1021/acs.est.5b05672 |
Eichmiller JJ, Miller LM, Sorensen PW. Optimizing techniques to capture and extract environmental DNA for detection and quantification of fish. Molecular Ecology Resources, 2016b, 16(1): 56-68 DOI:10.1111/1755-0998.12421 |
Evans NT, Lamberti GA. Freshwater fisheries assessment using environmental DNA:A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research, 2018, 197: 60-66 DOI:10.1016/j.fishres.2017.09.013 |
Ficetola GF, Miaud C, Pompanon F, et al. Species detection using environmental DNA from water samples. Biology Letters, 2008, 4(4): 423-425 DOI:10.1098/rsbl.2008.0118 |
Geerts AN, Boets P, Heede SVD, et al. A search for standardized protocols to detect alien invasive crayfish based on environmental DNA (eDNA):A lab and field evaluation. Ecological Indicators, 2018, 84: 564-572 DOI:10.1016/j.ecolind.2017.08.068 |
Goldberg CS, Sepulveda A, Ray A, et al. Environmental DNA as a new method for early detection of New Zealand mudsnails (Potamopyrgus antipodarum). Freshwater Science, 2013, 32(3): 9 |
Goldberg CS, Strickler KM, Pilliod DS. Moving environmental DNA methods from concept to practice for monitoring aquatic macroorganisms. Biological Conservation, 2015, 183: 1-3 DOI:10.1016/j.biocon.2014.11.040 |
Hänfling B, Lawson HL, Read DS, et al. Environmental DNA metabarcoding of lake fish communities reflects long-term data from established survey methods. Molecular Ecology, 2016, 25(13): 3101-3119 DOI:10.1111/mec.13660 |
Jin XS, Dou SZ, Shan XJ, et al. Hot spots of frontiers in the research of sustainable yield of Chinese inshore fishery. Progress in Fishery Sciences, 2015, 36(1): 124-131 [金显仕, 窦硕增, 单秀娟, 等. 我国近海渔业资源可持续产出基础研究的热点问题. 渔业科学进展, 2015, 36(1): 124-131] |
Jo T, Murakami H, Masuda R, et al. Rapid degradation of longer DNA fragments enables the improved estimation of distribution and biomass using environmental DNA. Molecular Ecology Resources, 2017, 17(6): 1-9 DOI:10.1111/1755-0998.12678 |
Lacoursière-Roussel A, Rosabal M, Bernatchez L. Estimating fish abundance and biomass from eDNA concentrations:Variability among capture methods and environmental conditions. Molecular Ecology Resources, 2016, 16(6): 1401-1414 DOI:10.1111/1755-0998.12522 |
Levy-Booth DJ, Campbell RG, Gulden RH, et al. Cycling of extracellular DNA in the soil environment. Soil Biology and Biochemistry, 2007, 39(12): 2977-2991 DOI:10.1016/j.soilbio.2007.06.020 |
Mackenzie DI, Nichols JD, Sutton N, et al. Improving inferences in population studies of rare species that are detected imperfectly. Ecology, 2005, 86(5): 1101-1113 DOI:10.1890/04-1060 |
Majaneva M, Diserud OH, Eagle SHC, et al. Environmental DNA filtration techniques affect recovered biodiversity. Scientific Reports, 2018, 8(1): 4682 DOI:10.1038/s41598-018-23052-8 |
Minamoto T, Fukuda M, Katsuhara KR, et al. Environmental DNA reflects spatial and temporal jellyfish distribution. PLoS One, 2017, 12(2): e0173073 DOI:10.1371/journal.pone.0173073 |
Pillioddavid S, Goldbergcaren S, Arklerobert S, et al. Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples. Canadian Journal of Fisheries and Aquatic Sciences, 2013, 70(8): 1123-1130 DOI:10.1139/cjfas-2013-0047 |
Pilliod DS, Goldberg CS, Arkle RS, et al. Factors influencing detection of eDNA from a stream-dwelling amphibian. Molecular Ecology Resources, 2013, 14(1): 109-116 |
Renshaw MA, Olds BP, Jerde CL, et al. The room temperature preservation of filtered environmental DNA samples and assimilation into a phenol-chloroform-isoamyl alcohol DNA extraction. Molecular Ecology Resources, 2015, 15(1): 168-176 DOI:10.1111/1755-0998.12281 |
Shan XJ, Li M, Wang WJ. Application of environmental DNA technology in aquatic ecosystem. Progress in Fishery Sciences, 2018, 39(3): 23-29 [单秀娟, 李苗, 王伟继. 环境DNA (eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29] |
Sigsgaard EE, Nielsen IB, Bach SS, et al. Population characteristics of a large whale shark aggregation inferred from seawater environmental DNA. Nature Ecology and Evolution, 2016, 1(1): 4 |
Sigsgaard EE, Nielsen IB, Carl H, et al. Seawater environmental DNA reflects seasonality of a coastal fish community. Marine Biology, 2017, 164(6): 128 DOI:10.1007/s00227-017-3147-4 |
Strickler KM, Fremier AK, Goldberg CS. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms. Biological Conservation, 2015, 183: 85-92 DOI:10.1016/j.biocon.2014.11.038 |
Thomsen PF, Kielgast J, Iversen LL, et al. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 2012, 21(11): 2565-2573 DOI:10.1111/j.1365-294X.2011.05418.x |
Thomsen PF, Willerslev E. Environmental DNA-An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183: 4-18 DOI:10.1016/j.biocon.2014.11.019 |
Tillotson MD, Kelly RP, Duda JJ, et al. Concentrations of environmental DNA (eDNA) reflect spawning salmon abundance at fine spatial and temporal scales. Biological Conservation, 2018, 220: 1-11 DOI:10.1016/j.biocon.2018.01.030 |
Tsuji S, Yamanaka H, Minamoto T. Effects of water pH and proteinase K treatment on the yield of environmental DNA from water samples. Limnology, 2016, 18(1): 1-7 |
Wickham H. ggplot2. Springer New York, 2009
|
Yamanaka H, Minamoto T. The use of environmental DNA of fishes as an efficient method of determining habitat connectivity. Ecological Indicators, 2016, 62(1): 147-153 |
Zhu B. Degradation of plasmid and plant DNA in water microcosms monitored by natural transformation and real-time polymerase chain reaction (PCR). Water Research, 2006, 40(17): 3231-3238 DOI:10.1016/j.watres.2006.06.040 |



