2. 上海海洋大学水产与生命学院上海 201306;
3. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室 青岛 266071
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306;
3. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071
大菱鲆(Scophthalmus maximus)为我国北方沿海工厂化养殖业的主导品种之一(雷霁霖等, 2002)。大菱鲆为冷温性鱼类,对温度等环境指标要求较严,适宜生长水温为12℃~19℃;通常在良好的充气和流水条件下,耐受高温可达25℃~26℃(马爱军等, 2012),但对夏季高温耐受能力较差。因受水温条件限制,我国北方主要采用“养殖大棚+深井海水”模式进行养殖,但近年来,随着地下海水的匮乏,南北接力养殖模式的出现,养殖生产中对大菱鲆耐高温新品种的需求日益增加(关健等, 2013)。另外,应用分子生物学手段,加快开展大菱鲆耐热性状的分子机制研究,对于培育耐高温的养殖品种具有较大意义。
转录组是指生物体的细胞或组织在特定的状态下基因组所转录的全部mRNA,反映了基因在不同生命阶段、生理状态、组织类型以及环境条件下表达的情况(罗辉等, 2015)。生物抗逆性受多个信号转导途径的调控,需要多基因协同表达(崔文晓等, 2017),因此,开展转录组测序分析,筛选关键信号通路及主效基因、转录因子等,并利用关键转录因子的调控作用促使多个功能基因表达来提高鱼类机体的抗逆性,是一种非常有效的方法和途径。近年来,从转录水平分析生物对环境胁迫的分子响应机制的相关研究屡见不鲜。如在水生生物中,Bilyk等(2014)利用转录组测序分析,探索了博氏南冰鰧(Pagothenia borchgrevinki)应对温度胁迫的分子机制,并首次推测性别会影响鱼类应对温度胁迫时基因的转录;Huang等(2018)在热应激条件下对虹鳟鱼(Oncorhynchus mykiss)头肾进行了转录组表达谱研究,但仅开展了常温组(18)、高温组(24) 2个温度点的比较分析。
本研究为了尽可能完整、系统地获得热应激对大菱鲆肾脏影响的数据信息,通过增加温度点,对头肾组织进行转录组测序和生物信息学分析,包括基因功能注释、SSR(简单重复序列)分析等,探讨差异基因的表达情况,为建立大菱鲆热应激条件下的物质代谢途径,构建信号转导通路模型,及发掘新的耐温基因提供一定的数据支撑。
1 材料与方法 1.1 养殖实验及样品采集实验用大菱鲆养殖于山东省烟台市开发区天源水产有限公司,均为人工繁育的健康幼鱼。幼鱼为随机挑选,平均体重为(100±12.3) g,于实验室玻璃水缸(1 m直径, 1 m深)暂养7 d。根据实验经验,共设计5个实验温度:(13±1)℃(常温CT)、20℃(T1)、23℃(T2)、25℃(T3)和28℃(T4)。升温方法参照Diegane等(2007)并优化:从常温(13±1)℃按每12 h增加1℃的速度升高至实验水温,每个处理组设3个平行组,每组投放15尾大菱鲆幼鱼。驯养与实验期间,每日早晚各投喂1次自制人工饵料,并换水1次。采用静水法,自动恒温加热器控温;到达实验温度并维持12 h后,每个平行组取3尾鱼体肾脏组织,即每个处理组共取9尾幼鱼,将9个样本放入1.5 ml EP管中用液氮速冻,置于–80℃保存备用。
1.2 RNA提取将对照组及处理组中的每个平行组的3个样本混合均匀后,提取样品总RNA,即每组处理3份生物学重复。提取方法:取肾脏组织在液氮中研磨成粉末,用动物组织总RNA提取试剂盒(天根生化科技有限公司, 北京)提取总RNA。用NanoDrop 2000核酸检测仪(Thermo公司, 美国)检测RNA纯度,用核酸分析仪Agilent2100 (安捷伦科技公司, 美国)检测总RNA浓度及RNA质量。
1.3 肾脏转录组文库构建及测序RNA-Seq文库制备及测序由广州基迪奥生物科技有限公司完成,测序平台为Illumina HiSeqTM2500,采用双末端测序方法。
1.4 序列拼接与功能注释、富集去除原始序列数据中含有接头及低质量的序列后,得到Clean reads,并进行De novo组装。使用短reads组装软件Trinity将转录组从头组装成Unigenes数据集。通过blastx将Unigenes序列比对到Nr、SwissProt、KEGG和COG,得到Unigenes的蛋白功能注释信息。对所有Unigenes进行GO功能分类统计、KEGG Pathway注释及通路富集分析。
1.5 预测编码蛋白框和转录因子按Nr、Swiss-Prot、KEGG和COG/KOG的优先级顺序将Unigenes序列与以上蛋白库做blastx比对(E值< 1×10–5),利用Hmmsearch将Unigenes的ORF比对到TF数据库(Animal TFdb)预测TF,并将预测的TF按照家族类型分类。
1.6 微卫星(SSR)分析简单重复序列(Simple sequence repeat, SSR)又称微卫星DNA,利用MISA软件对转录组的所有Unigenes进行搜索,查找SSR。搜索标准为:单、一、二、三、四、五、六核苷酸基序(Motif)至少重复次数分别为10、8、5、4、3、3,对查找的SSR类型进行特征分析(贾新平等, 2014)。
1.7 基因表达差异分析Unigene表达量的计算使用RPKM法(Reads Per kb per Million reads)计算,RPKM法能消除基因长度和测序量差异对计算基因表达的影响,计算得到的基因表达量可直接用于比较不同样品间的基因表达差异。分组之间的差异基因统计,利用FDR与log2FC筛选,筛选条件为FDR < 0.05且|log2FC| > 1,其中,FC (Fold Change, FC)标示两样品间表达量的比值。得到差异表达基因之后,对差异表达基因进行GO功能富集分析,KEGG Pathway注释及通路富集分析。
2 结果与分析 2.1 转录组数据组装利用Illumina Hiseq 2500高通量测序技术对大菱鲆不同温度胁迫条件下肾脏组织开展转录组测序分析,1个对照组与4个实验温度组共获得Raw reads及去除杂质后的Clean reads (表 1)。由表 1可以看出,共得到224, 473, 610个Clean reads片段,包含了99.7 Gb的序列信息,GC%含量平均值为51.05%。在测序质量值统计评估方面,碱基Q30为88.65%,由以上数据得出,该转录组测序数据量和质量都较高,可为后续的组装提供可靠的原始数据。
|
|
表 1 大菱鲆高温胁迫转录组测序数据统计 |
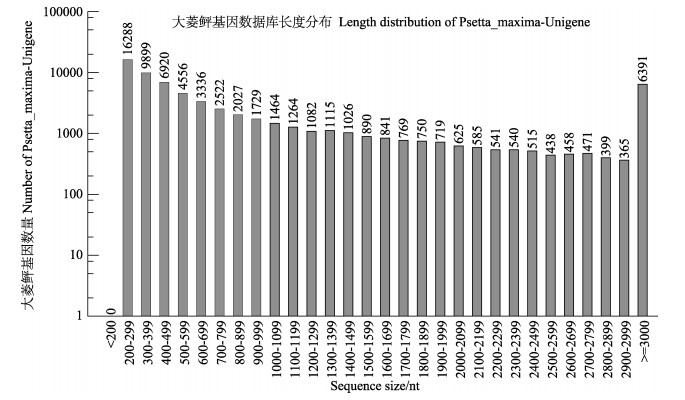
采用Trintiy软件对Clean reads组装,共获得Unigenes总数目为68525,长度范围为201~23456 bp,平均长度1124 bp,N50长度2316 bp。组装数据统计见表 2。Unigenes长度分布图见图 1,结果显示,组装完整性较高,从而为后续信息学注释及差异基因的筛选奠定了基础。
|
|
表 2 大菱鲆高温胁迫转录组组装数据统计 Tab.2 Statistics of transcriptome assembly data of heat stress of turbot |
|
图 1 Unigene长度分布 Fig.1 The statistics of sequence length of unigenes |
将Unigenes分别在Nr、Swissprot、KEGG、KOG、GO数据库进行序列比对及功能注释,共注释25498条Unigenes。其中,Nr数据库注释到的Unigenes数量最多,具体结果分别为:Nr库25442条,Swissprot库21179条,KEGG库15488条,KOG库17111条,Go库12760条(表 3)。
|
|
表 3 Unigene的注释结果 Tab.3 The result of Unigenexs annotation |
利用Blastx将组装出来的Unigene序列与Nr数据库进行比对,结果显示,功能注释匹配的物种中,大黄鱼(Larimichthys crocea)所占的比例最高(29.2%),随后依次是雀鲷(Stegastes partitus) (23.1%)、罗非鱼(Oreochromis niloticus) (5.3%)、革首南极鱼(Notothenia coriiceps) (4.7%)、半滑舌鳎(Cynoglossus semilaevis) (4.7%)等物种。
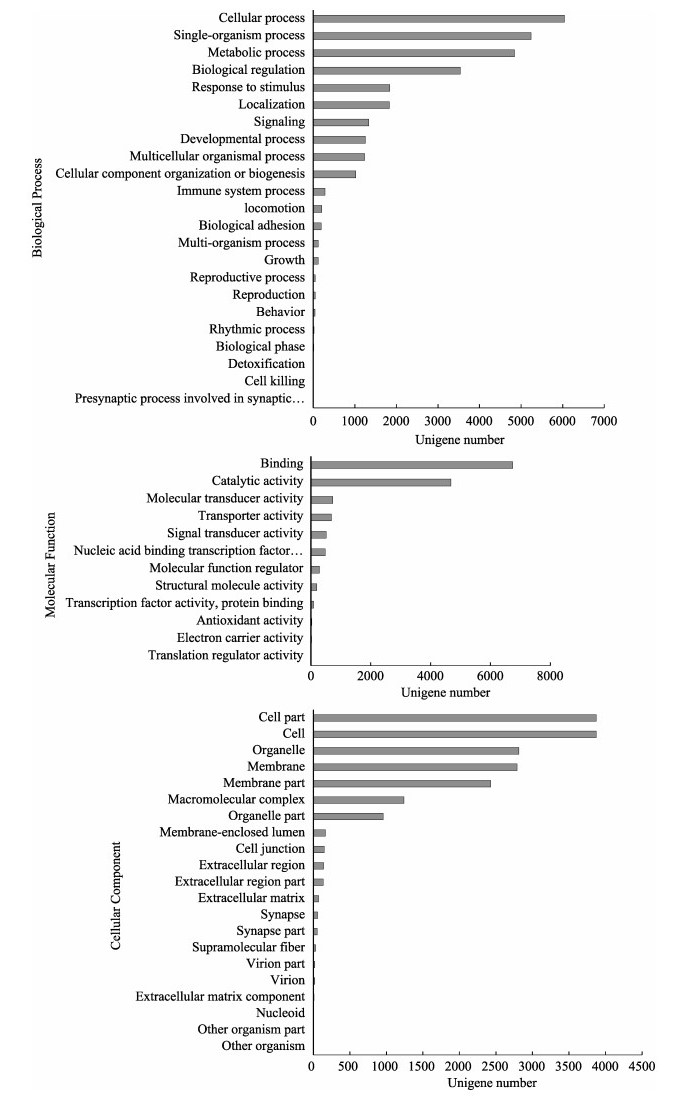
2.2.2 Unigenes的GO分类采用Blast2Go软件,结合GO数据库对大菱鲆的Unigene进行功能分类,从宏观上分析大菱鲆热应激胁迫后表达基因的功能分布特征。在Go分类体系中,共有3个Ontology,分别描述基因的生物学过程(Biological process)、细胞组分(Cellular component)和分子功能(Molecular function)。结果表明,共有12760条Unigenes获得GO注释,划分为56个功能组,并对每个功能组涉及的Unigene进行统计分析(图 2)。注释到Molecular function上的基因数目最多,为10053个,其次是Biological process 9644个,Cellular component 6270个。其中,细胞进程6041个,单生物代谢过程5240个,代谢过程4842个,催化活性4681个,细胞3874个,生物调节3535个,应激反应1840个和信号转导1334个功能组中涉及的Unigenes较多。这一分类结果显示了大菱鲆热应激过程中基因表达谱的总体情况。
|
图 2 Unigenes的GO功能分类图 Fig.2 GO functional categories of Unigenes |
蛋白质直系同源数据库(COG/KOG)对基因产物进行直系同源分类。研究显示,大菱鲆肾脏转录组共获得17111个注释信息,可归属于25类,并对每类Unigenes进行统计分析。其中,信号转导机制为8661个,一般功能预测为6528个,翻译后修饰、蛋白质折叠和分子伴侣为3346个,转录为2435个,细胞内转运、分泌和小泡运输为2174个,细胞构架为1925个,其他类别的基因表达丰度均各不相同(图 3)。

|
图 3 Unigenes的KOG功能分类 Fig.3 KOG function classification of Unigenes |
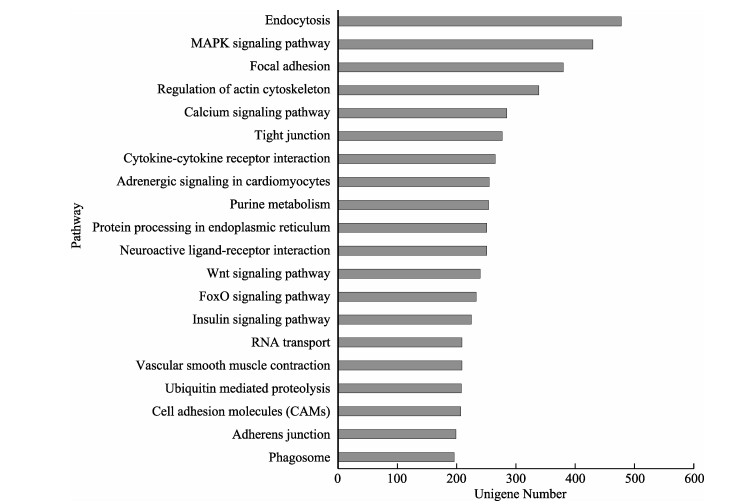
结合KEGG数据库注释信息,进一步将大菱鲆肾脏Unigenes进行Pathway注释,其中,有15488个Unigenes获得对应的Ko编号,归属于218条代谢通路,分类为5类KEGG途径:代谢途径、遗传信息处理、细胞过程、环境信息和生物系统。其中,代谢途径主要包括碳水化合物代谢通路、氨基酸代谢通路、能量代谢通路、脂类代谢通路、核苷酸代谢通路等,占据KO注释Unigenes的25.4%(3941条);遗传信息处理主要包括翻译,折叠、分选和相互作用,转录,复制和修复等,共注释到1907条Unigenes,占12.3%;细胞过程主要由转运和代谢作用,细胞生长与死亡,运输分解代谢,细胞连接等组成,共注释到2927条Unigenes,占18.9%;环境信息主要包括信号转导,信号分子与相互作用,共注释3230条Unigenes,占20.8%;此外2161条Unigenes被注释到生物系统中。Unigenes数量最多的20个代谢途径见图 4,涉及细胞内吞、信号转导、能量代谢、循环系统、细胞连接等。
|
图 4 Unigenes的KEGG功能分类 Fig.4 KEGG function classification of Unigenes |
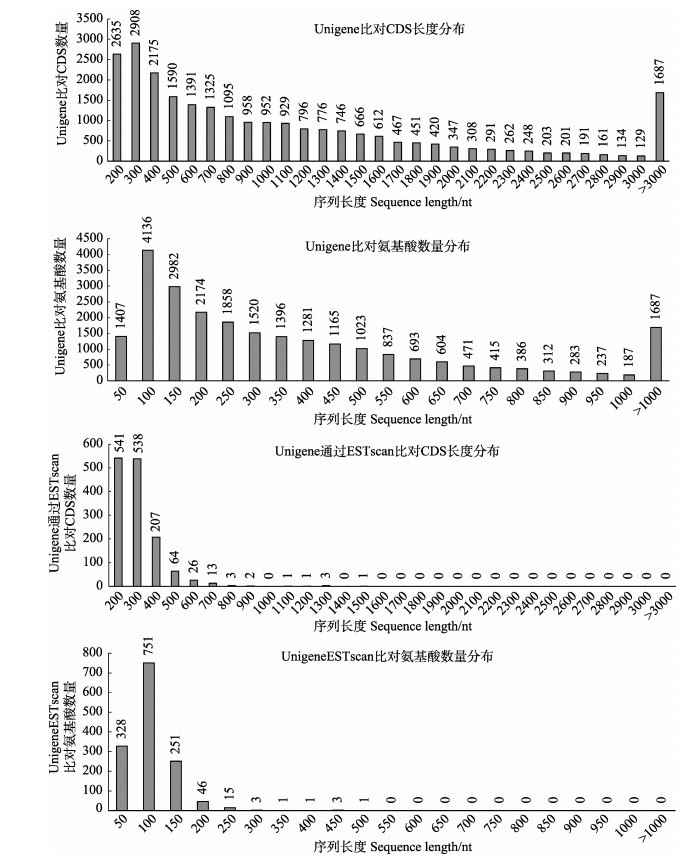
通过Blast比对得到CDS核酸和氨基酸序列分布,ESTscan预测编码区的核酸和氨基酸序列的长度分布(图 5)。
|
图 5 CDS分析 Fig.5 CDS analysis |
转录因子是能够结合在基因上游特异的核苷酸序列上的蛋白质,能调控其靶基因的转录。对大菱鲆肾脏Unigenes进行转录因子分析,共检测到65类转录因子。其中,C2H2锌指蛋白家族的基因数目最大,达到429个Unigenes;调节动物生长发育、响应逆境胁迫相关的Homeobox同源异位基因,有101个Unigenes;与Bhlh家族相关的有86个Unigenes;参与抗逆响应的TF-Bzip家族相关的Unigenes有83个;参与组织损伤修复、细胞迁移、再生等生命进程的HMG基因有55个Unigenes;MYB家族42个,ZBTB家族38个;Fork-head家族34个,THR-like家族33个;其余家族基因数目相对较少。
2.5 SSR分析对大菱鲆肾脏转录组的68525条Unigenes进行SSR位点搜索,共检测到21177个SSR位点。SSR类型丰富,其中,包括二碱基、三碱基、四碱基、五碱基和六碱基重复,各碱基重复数量分别为1100、7604、1941、393和231个。
2.6 基因在大菱鲆不同温度胁迫中的表达分析对5组不同高温胁迫环境(CT:常温, T1: 20℃, T2: 23℃, T3: 25℃, T4: 28℃)的基因进行表达比较分析,结果显示(图 6),CT组同4个高温组均表现出较大的差异,而且上调基因占较大比重;T1组与T4组存在较大的差异,差异基因达到4734个,其中,上调基因3386个,下调基因1348个;T1与T3组差异表达基因163个,其中,上调基因131个,下调基因32个;T2与T4组差异基因4157个,其中,上调基因2691个,下调基因1466个;T3与T4组差异表达基因2501个,其中,上调基因1673个,下调基因828个;而T1与T2组、T2与T3组的差异表达基因较少,分别为25和36。
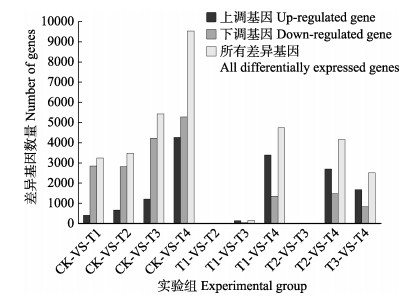
|
图 6 差异基因统计 Fig.6 Statistics of different expression genes |
作为变温性动物,鱼类对环境温度等指标要求较严,其中,热应激是影响鱼体生长发育最严重的环境胁迫(周朝伟等, 2018),鱼类在长期进化过程中对热应激的响应形成了一套完善的应答协调机制,因此,揭示其热应激胁迫的分子机制,培育出耐高温的新品种,在目前全球气候变暖的背景下显得尤为迫切。
转录组高通量测序是功能基因组研究的一个重要技术手段,通过数字基因表达谱技术,可以将不同组织、各个发育阶段以及逆境胁迫下的差异表达基因快速检测出来,在功能基因组学研究方面正在发挥着重要的作用(王申昌等, 2016)。该技术目前在水产动物中的应用显著增加,对于鱼类疾病、免疫、生长、抗逆、系统进化和生物毒理过程及机理等方面的研究都有极大帮助(陈琼等, 2014;陈之航等, 2017)。为尽可能完整、系统地获得热应激对大菱鲆肾脏影响的转录组数据信息,本研究选取5个不同的温度梯度处理,开展了大菱鲆高温胁迫转录组分析。
通过对不同温度胁迫下基因表达谱结果进行分析,不同温度组之间存在较显著的差异,尤其是CT、T1、T3和T4组,4个温度组的差异表达基因较多,而T2与T1、T3组之间的差异基因个数较少。我们推测,随着温度的升高,鱼体的生理机能发生不断的变化从而适应胁迫,当到达某个温度区域时,机体将处于一个平衡态,机体变化相对较小,但如果温度继续升高,则打破这一平衡,继续调整,从而适应胁迫。
逆境条件下,许多胁迫相关基因在转录水平都会有所增加,其主要原因是转录因子对目标基因启动子的顺式作用元件的特异结合(赵丽娟等, 2016)。本研究对大菱鲆肾脏热应激转录因子进行了分析,共检测到65类转录因子,为深入研究大菱鲆热应激适应机制提供分子基础和依据。其中,C2H2锌指蛋白家族的基因数目最多,其又称为TFIIIA类型的锌指蛋白(TFIIIA因子是RNA聚合酶Ⅲ转录5S rRNA基因所必需),在真核细胞中广泛富集的锌指蛋白之一(Laity et al, 2001)。在植物中研究较多,主要参与胁迫应答信号转导和细胞过程,如ZAT7基因在盐胁迫下的表达主要在根中有所增高(Yilmaz et al, 2007); TFⅢA型锌指蛋白SCOF-1,作为冷调控基因(Cold-regulated, COR)的正调节物,通过与脱落酸效应元件相互作用来调控COR,增强植物的耐寒能力(Kim et al, 2001);从牵牛花中得到的锌指蛋白ZPT2-3,其在植物体内的过表达可以增强耐寒能力(Sugano et al, 2003)。在人类疾病方面也有相关的研究报道,其对信号通路的下游基因表达有着明显的调控作用,可以选择性结合特异的靶结构,调控细胞的生长、增殖、分化和凋亡等(雷陈, 2010)。但在水生生物中的研究却极少,本文研究结果显示,高温胁迫条件下,C2H2锌指蛋白家族Unigene数目远远高于其他转录因子,其中,差异表达基因数为108个,表明C2H2锌指蛋白家族的部分基因在大菱鲆热胁迫条件下发生表达水平的变化,哪些基因与热应激相关?这些基因是如何参与胁迫应答信号转导过程的?目前,在海水鱼类未见报道,本课题组将在今后的研究中继续深入开展。
本研究首次利用Illumina Hiseq高通量测序技术对大菱鲆肾脏组织在5个不同温度胁迫条件下的cDNA文库进行测序,建立了大菱鲆热应激肾脏转录组数据库,相关数据更加完整、系统,为今后开展海水鱼类热应激胁迫分子调控机制提供数据支撑,也为耐高温选育工作提供更多新的功能基因和分子标记。
Bilyk KT, Cheng CHC. RNA-seq analyses of cellular responses to elevated body temperature in the high Antarctic Cryopelagic nototheniid fish Pagothenia borchgrevinki. Marine Genomics, 2014, 18: 163-171 DOI:10.1016/j.margen.2014.06.006 |
Chen Q, Gu ZF, Wang AM, et al. Data reassembling reveals more information of transcriptome for Pinctada fucata martensii Dunker. Progress in Fishery Sciences, 2014, 35(6): 97-102 [陈琼, 顾志峰, 王爱民, 等. 马氏珠母贝转录组数据的重新组装. 渔业科学进展, 2014, 35(6): 97-102] |
Chen ZH, Dong JJ, Sun CF, et al. cDNA cloning and analyses of two myosin heavy chain isoforms of mandarin fish (Siniperca chuatsi) based on transcriptome sequencing. Progress in Fishery Sciences, 2017, 38(3): 51-61 [陈之航, 董浚键, 孙成飞, 等. 基于转录组测序对翘嘴鳜(Siniperca chuatsi) 2种肌球蛋白重链基因的克隆与分析. 渔业科学进展, 2017, 38(3): 51-61] |
Cui WX, Ma AJ, Huang ZH, et al. Effects of salinity stress on the mRNA expression of PRL and Na+-K+-ATPase α1 of turbot (Scophthalmus maximus). Progress in Fishery Sciences, 2017, 38(6): 32-40 [崔文晓, 马爱军, 黄智慧, 等. 大菱鲆(Scophthalmus maximus) PRL基因、Na+-K+-ATPase α1基因对盐度胁迫的响应. 渔业科学进展, 2017, 38(6): 32-40] |
Diegane N, Chen YY, Lin YH, et al. The immune response of tilapia Oreochromis mossambicus and its susceptibility to Streptococcus iniae under stress in low and high temperatures. Fish and Shellfish Immunology, 2007, 22: 686-694 DOI:10.1016/j.fsi.2006.08.015 |
Guan J, Liu HJ, Guan SG, et al. Selection of high temperature broodstocks of turbot by using population selection method. Scientific Fish Farming, 2013(3): 50-51 [关健, 刘洪军, 官曙光, 等. 使用群体选育法选择大菱鲆耐高温亲鱼群体. 科学养鱼, 2013(3): 50-51] |
Huang JQ, Li YJ, Liu Z, et al. Transcriptomic responses to heat stress in rainbow trout Oncorhynchus mykiss head kidney head kidney. Fish and Shellfish Immunology, 2018, 82: 32-40 DOI:10.1016/j.fsi.2018.08.002 |
Jia XP, Sun XB, Deng YM, et al. High-throughput sequencing and analysis of the transcriptome of the bird's nest fern. Journal of Horticulture, 2014, 41(11): 2329-2341 [贾新平, 孙晓波, 邓衍明, 等. 鸟巢蕨转录组高通量测序及分析. 园艺学报, 2014, 41(11): 2329-2341] |
Kim JC, Lee SH, Cheong YH, et al. A novel cold-inducible zinc finger protein from soybean, SCOF-1, enhances cold tolerance in transgenic plants. Plant Journal, 2001, 25(3): 247-259 DOI:10.1046/j.1365-313x.2001.00947.x |
Laity JH, Lee BM, Wright PE. Zinc finger proteins: New insights into structural and functional diversity. Current Opinion in Structural Biology, 2001, 11(1): 39-46 DOI:10.1016/S0959-440X(00)00167-6 |
Lei C. Initial exploration of the function of ZNFD-a C2H2-type zinc finger protein. Master's Thesis of Suzhou University, 2010 [雷陈. C2H2型锌指蛋白——ZNFD的生物学功能初探.苏州大学硕士研究生学位论文, 2010]
|
Lei JL, Men Q, Wang YG, et al. The factory farming mode of Dalingyu "greenhouse shed + deep well seawater". Marine Aquatic Research, 2002, 23(4): 1-7 [雷霁霖, 门强, 王印庚, 等. 大菱鲆"温室大棚+深井海水"工厂化养殖模式. 海洋水产研究, 2002, 23(4): 1-7] |
Luo H, Ye H, Xiao SJ, et al. Application of transcriptomics technology in aquatic animal research. Journal of Fisheries of China, 2015, 39(4): 598-607 [罗辉, 叶华, 肖世俊, 等. 转录组学技术在水产动物研究中的运用. 水产学报, 2015, 39(4): 598-607] |
Ma AJ, Huang ZH, Wang XA, et al. Breeding and temperature resistance performance evaluation of high temperature resistant line of turbot. Oceanologia et Limnologia Sinica, 2012, 43(4): 797-804 [马爱军, 黄智慧, 王新安, 等. 大菱鲆耐高温品系选育及耐温性能评估. 海洋与湖沼, 2012, 43(4): 797-804] |
Sugano S, Kaminaka H, Rybka Z, et al. Stress-responsive zinc finger gene ZPT2-3 plays a role in drought tolerance in petunia. Plant Journal, 2003, 36(6): 830-841 DOI:10.1046/j.1365-313X.2003.01924.x |
Wang SC, Hu SL, Cao Y, et al. High-throughput transcriptome sequencing and differentially expressed gene analysis of Liangshan Cizhu. North China Agricultural Journal, 2016, 31(3): 65-71 [王身昌, 胡尚连, 曹颖, 等. 梁山慈竹高通量转录组测序及差异表达基因分析. 华北农学报, 2016, 31(3): 65-71] |
Yilmaz SC, Morsy MR, Song LH, et al. The EAR-motif of the Cys2/His2-type zinc finger protein Zat7 plays a key role in the defense response of Arabidopsis to salinity stress. Journal of Biological Chemistry, 2007, 282(12): 9260-9268 DOI:10.1074/jbc.M611093200 |
Zhao LJ, Yi XY, Zeng YL. Research progress on plant stress related C2H2 zinc finger protein. Molecular plant breeding., 2016, 14(3): 578-585 [赵丽娟, 易小娅, 曾幼玲. 植物逆境相关C2H2型锌指蛋白的研究进展. 分子植物育种, 2016, 14(3): 578-585] |
Zhou CW, Hu XW, Lei L, et al. Effects of heat stress on biochemical indices and HSP70 mRNA expression in gibel carp (Carassius auratus gibelio). Progress in Fishery Sciences, 2018, 39(6): 65-71 [周朝伟, 胡续雯, 雷骆, 等. 热应激对银鲫生化指标和HSP70 mRNA表达的影响. 渔业科学进展, 2018, 39(6): 65-71] |



