2. 海南热带海洋学院 水产与生命学院 海南 三亚 572022;
3. 浙江海洋大学水产学院 浙江 舟山 316022
2. College of Fisheries and Life Science, Hainan Tropical Ocean University, Sanya 572022, China;
3. College of Fisheries, Zhejiang Ocean University, Zhoushan 316022, China
全球生物多样性丧失是20世纪和21世纪最严重的环境危机之一,对生态系统的平衡和可持续发展有极大的负面影响(Pimm et al, 2014)。鱼类多样性作为生物多样性的一个重要组成部分也呈现衰退之势(Reid et al, 2018)。作为水生生态系统的核心,鱼类不仅在生态系统中扮演着重要的角色,也可以反映水生生态系统的环境状态(Park et al, 2006; Guo et al, 2015)。因此,保护鱼类多样性对生态系统科学管理和资源可持续利用至关重要。而准确掌握鱼类在水环境中的生物量及其时空分布动态是开展鱼类多样性保护的重要前提和基础(高天翔等, 2018)。
舟山群岛是中国最大的群岛,位于浙江东北部海域,由2 085个岛屿组成,总面积为22 216 km2 (Huang et al, 2020)。由于其地处长江、钱塘江、甬江和台湾暖流的交汇区,从而形成了独特复杂的水文环境,为鱼类提供了丰富的营养来源和适宜的栖息环境(Wang et al, 2020)。近年来,由于海洋资源的大规模开发以及海洋污染,使得舟山群岛附近的鱼类多样性丧失严重(Xu et al, 2019; 许永久等, 2019)。为了恢复鱼类多样性,政府和研究人员开展了诸多保护工作,包括实施更严格的禁渔制度和重要鱼种增殖放流,以期改善鱼类区系组成、保护鱼类多样性、发展可持续的生态渔业。然而,缺乏了解物种和评估生物多样性的可靠工具是开展保护工作的主要问题(Lodge et al, 2012)。诸多学者对舟山海域的渔业资源进行了调查,调查方法主要为拖网采样后通过形态学鉴定进行分类统计(张龙, 2011; 于南京等, 2020、2021; 唐嘉威等, 2021; 陆延等, 2022),然而,这种方式效率较低、费时费力且具有选择性和破坏性(Valentini et al, 2016; 陈炼等, 2016; 姜维等, 2016; 马鸿娟等, 2016)。此外,这些调查大多是在大面积海域进行的,对于舟山近海岛礁区的小规模鱼类资源调查较少。
环境DNA (environmental DNA, eDNA)技术的出现和应用为水生生物的调查与监测带来了创新。与传统的调查方法相比,环境DNA技术具有更高的监测能力和成本效益以及环境友好和非侵入性等诸多优势,克服了传统调查手段的诸多不足(Valentini et al, 2016; 姜维等, 2016; 马鸿娟等, 2016; 单秀娟等, 2018)。环境DNA技术实现的方式主要有两种:第一种通过设计特异性引物和探针,使用定量PCR对环境中特定目标物种的存在、生物量及时空分布进行监测(Wang et al, 2022)。另外一种是针对目标类群选择合适的通用引物,通过PCR扩增结合高通量测序(high-throughput sequencing, HTS),对环境样本中存在的多个目标物种进行识别,称为环境DNA metabarcoding (eDNA metabarcoding),常用于生物多样性的快速评估(Lodge et al, 2012; Aylagas et al, 2016)。总体而言,环境DNA方法更适合了解海洋生物的分布和动态。
近年来,环境DNA metabarcoding技术已在水族馆、河流、湖泊和海洋等水环境中得到广泛应用(Kelly et al, 2014; Miya et al, 2015; Stat et al, 2017; Drummond et al, 2021)。国内对于环境DNA metabarcoding技术的研究区域主要集中于河流和湖泊,对于开放海域和渔场等方面的相关应用较少。由于舟山群岛多岛礁的海域特征,传统的渔业资源调查方法难以开展。因此,建立简单、准确的物种多样性调查新方法,是更好地在舟山近海岛礁区进行鱼类多样性监测工作的重点和难点。本研究以舟山近海岛礁区——西轩岛为例,探讨环境DNA metabarcoding技术在监测舟山近海鱼类多样性的可行性,同时,评估不同时空鱼类生物多样性的差异,以期为舟山近海的渔业资源调查提供有效的补充,并将为后续鱼类多样性调查评估及其保护管理提供技术支撑。
1 材料与方法 1.1 水样采集本研究4个水样采样点位于浙江省舟山市西轩岛沿岸海域,分别为S01 (122°30′49″E, 29°89′09″N)、S02(122°30′40″E, 29°89′38″N)、S03 (122°30′73″E, 29°89′42″N)和S04 (122°31′23″E, 29°89′04″N) (图 1)。样品分别采集于2019年2月(冬季)、5月(春季)和11月(秋季),使用手提式采水器在表层(0~1 m)采集水样,每个采样点采集1个样品,同时使用等量蒸馏水作为阴性对照。

|
图 1 西轩岛采样位点 Fig.1 Sampling sites around Xixuan Island in Zhoushan |
使用孔径为0.45 μm的玻璃纤维滤膜(上海兴亚)对采集的海水样品及阴性对照样品进行真空抽滤,每个水样抽滤1.5 L。将滤膜用铝箔纸包裹,保存在2 mL冻存管中并放置于液氮速冻,然后储存于实验室–80 ℃冰箱,直到进一步提取环境DNA。为防止样品间交叉污染,所有的过滤设备在每次使用前后在10%的次氯酸钠溶液中浸泡15 min以去除残留的DNA,然后用自来水彻底冲洗,最后用市售的纯净水冲洗。抽滤每个样品时均使用全新的一次性手套。环境DNA的提取参照之前制定的方法(Wang et al, 2022)。使用DNeasy Blood & Tissue试剂盒(Qiagen, 德国)从滤膜中提取总环境DNA。总环境DNA提取后用100 μL AE Buffer (Qiagen, 德国)进行洗脱并存放于–20 ℃冰箱。
1.3 扩增子文库构建及测序使用MiFish-U/E (Miya et al, 2015)通用引物制备鱼类eDNA metabarcoding 12S扩增子文库,共进行2次PCR,参照Wu等(2021)的研究方法进行环境DNA浓度测定、扩增子文库构建和高通量测序。PCR体系12 µL:6.0 μL 2×KAPA HiFi Hot Start Ready Mix (KAPA Biosystems, 美国),正、反向引物各1.0 µL,模板1.0 µL,ddH2O补齐至12 µL。扩增条件:95 ℃预变性3 min,98 ℃变性20 s,65 ℃退火15 s,72 ℃延伸15 s,共40次循环,72 ℃终延伸5 min。将第1次PCR的产物纯化、稀释后作为后续PCR的模板。第2次PCR总体系与第1次相同,热循环条件为95 ℃ 3 min,98 ℃ 20 s,72 ℃ 20 s,共计12次循环,72 ℃孵育5 min。每个样品设置3个生物学重复,并用超纯水作为阴性对照。采用2.0%的琼脂糖凝胶电泳对扩增产物进行质量检测及纯化,使用Qubit dsDNA HS assay kit和Qubit fluorometer 2 (Thermo Fisher Scientific, 美国)对文库浓度进行量化,之后使用灭菌蒸馏水将文库的浓度调整到4 nmol/L。最后在iSeq 100平台(Illumina, 美国)上使用iSeq 100 i1试剂盒(Illumina, 美国)对文库进行2×150 bp双端测序。
1.4 数据分析使用USEARCH软件对下机数据进行预处理和分析,包括数据组装、删除两端引物序列,以及去除低质量(预期错误率 > 1%)、嵌合体和短于140 bp的噪音序列。将序列以97%的相似性聚类,得到可操作分类单元(Operational Taxonomic Units, OTUs)以及OTU代表序列。使用OTU代表序列与Nt数据库(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome)比对进行物种注释。结合舟山近海海域水生生物分布信息对所注释的物种进行人工校对并手动去除非鱼类信息。此外,阴性对照中监测到的reads可能已被污染,因此,将其从对应的环境样本中剔除(Wu et al, 2021)。
使用R语言(4.1.2版本)进行统计分析并将结果可视化。使用ggplot2、vegan和ggpubr包进行α多样性分析。主坐标分析(Principal Co-ordinates Analysis, PCoA)是一种非约束性的数据降维分析方法,可用于研究数据的相似性或差异性。PCoA分析使用ape包。使用metacoder包绘制进化树,分析不同季节鱼种的富集情况。通过metacoder包可以将鱼种的分类关系以树状图形式展示,并使用进化树中节点与边的颜色和大小定量描述不同样本间的差异情况。
2 结果与分析 2.1 物种组成分析在3个季节的4个采样点共监测到鱼类33种,隶属于12目26科32属。图 2展示了各采样点在不同季节的鱼种组成情况(鱼种种类和序列相对丰度)。其中,鲻(Mugil cephalus)检出率最高,为3季共有鱼种。花鲈(Lateolabrax maculatus)次之,在S02 (2月和5月)、S03 (2月和5月)和S04 (2月和5月)采样点被检出。此外,日本䲢(Uranoscopus japonicus)、断纹紫胸鱼(Stethojulis terina)、浪花银汉鱼(Iso flosmaris)、日本鲐(Scomber japonicus)、褐菖鲉(Sebastiscus marmoratus)、矶鳚(Parablennius yatabei)、黑鲷(Acanthopagrus schlegelii)、大泷六线鱼(Hexagrammos otakii)、日本白姑鱼(Argyrosomus japonicus)、日本鳀(Engraulis japonicus)、赖氏犁齿鳚(Entomacrodus lighti)、龙头鱼(Harpadon nehereus)、海鳗(Muraenesox cinereus)和髭缟虾虎鱼(Tridentiger barbatus)均在多个采样点的不同采样季节被检出。28个鱼种只在1个季节出现,占所有物种的84.9%,表明调查海域多数为洄游性鱼类并以季节性分布为主。
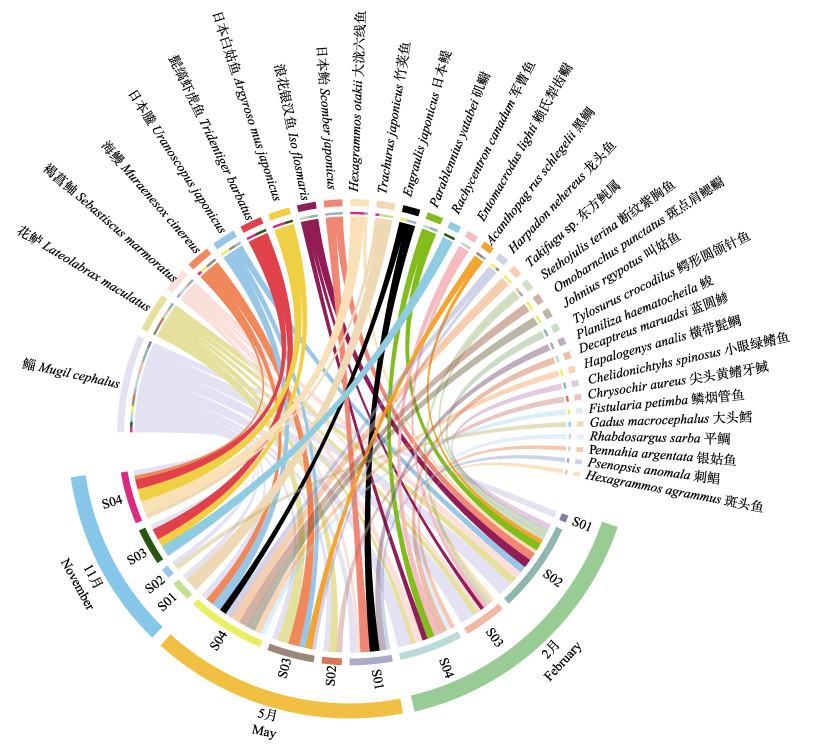
|
图 2 各站位物种组成 Fig.2 Composition of species at each station 不用颜色的弧线代表不同鱼种;弧线宽度代表鱼种丰度;外侧圆环代表不同采样时间。 Arcs of different colors represent different fish species, widths of arcs represent species abundance, and outer circles represent different sampling times. |
在本调查的4个站位中,采样点S04累计监测鱼种数最多,为22种。其次为S03,共监测到14种鱼类。S01和S02采样点检出鱼种较少,分别为7种和1种。物种多样性指数可以作为环境变化对鱼类群落结构影响的指标。西轩岛不同采样点鱼种α多样性状况如图 3a所示。在各项指数中,所有采样点之间均无显著性差异。具体指数如下:Chao1丰富度指数平均为5.67 (变幅为3.00~8.33),Shannon多样性指数平均为0.63 (变幅为0.38~0.94),Simpson多样性指数平均值为0.33 (变幅为0.20~0.44),Pielou均匀度指数平均值为1.89 (变幅为1.52~2.59)。S04的丰富度指数和多样性指数均为最高(Chao1=8.33, Shannon=0.95, Simpson=0.44),表明S04采样点鱼种数量多且较为丰富。S02采样点的Pielou均匀度指数最高(0.62),表明在该采样点各鱼种数目接近。

|
图 3 西轩岛近海不同采样点(a)和不同采样季节(b)的α多样性 Fig.3 α diversity at different sampling sites (a) and in different sampling seasons (b) of Xixuan Island coastal waters |
对4个采样点(每个样本的所有季节结果均展示)在OTU水平进行PCoA分析(图 4a)。横纵坐标轴PCoA1对样本组成差异的解释度为24.4%,PCoA2为14.1%。由图 4可以看出,S01和S02与其他站位距离较远,表明这2个站位的鱼种组成与其他站位具有一定的差异。S03和S04采样点在3个季节的鱼类多样性相似。
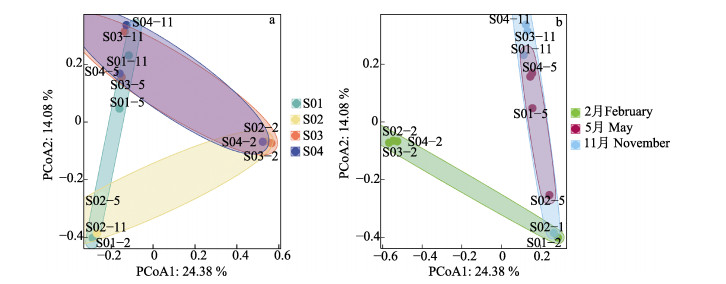
|
图 4 基于Bray-Curtis距离矩阵的鱼类群落的主坐标分析 Fig.4 PCoA of fish community based on the Bray-Curtis distance matrix a:不同采样站位;b:不同采样季节 a: Different sampling stations; b: Different sampling seasons |
将本次调查采集的样品按照采样时间分为2月、5月和11月共3个月份。根据检出鱼种数由多至少排序,依次为2月、5月和11月,检出鱼种数分别为16种、14种和8种。西轩岛不同季节鱼种α多样性情况如图 3b所示。各项指数如下:Chao1丰富度指数平均为5.67 (变幅为3.50~7.25),Shannon多样性指数平均值为0.63 (变幅为0.16~1.16),Simpson多样性指数平均值为0.34 (变幅为0.05~0.60),Pielou均匀度指数平均值为0.41 (变幅为0.07~0.66)。2月的丰富度指数最高(Chao1=8.33),5月的物种多样性指数和均匀度指数均为最高(Shannon=0.945, Simpson= 0.439, Pielou=0.66),表明春季鱼种较为丰富且各鱼种个体数目接近。此外,2月和5月的多样性指数和均匀度指数均存在显著差异(Shannon: P=0.018; Simpson: P=0.002; Pielou: P=0.001)。基于不同采样季节分组的PCoA分析发现(图 4b),2月与5月和11月鱼种差异较大,5月和11月样本点基本重叠,表明这2个季节的鱼类群落组成相似。
为了比较不同时间鱼种的差异性,绘制了分类学分级结构和不同采样时间两两比较的进化树(图 5),节点的大小和颜色与采样时间的reads丰度相关。2月与5月相比,褐菖鲉、浪花银汉鱼、断纹紫胸鱼、赖氏犁齿鳚和矶鳚在2月被显著富集,表明这些物种在2月较丰富;5月显著富集的鱼种有花鲈、日本䲢、龙头鱼、日本鳀和海鳗。相比11月,2月的丰富鱼种为浪花银汉鱼、鲻、褐菖鲉、花鲈、矶鳚和赖氏犁齿鳚;髭缟虾虎鱼、竹荚鱼(Trachurus japonicus)和日本白姑鱼在11月显著富集。5月与11月相比,5月的丰富鱼种为海鳗、龙头鱼、日本鳀、日本䲢和花鲈;11月丰富鱼种为髭缟虾虎鱼、竹荚鱼和日本白姑鱼。总体来说,赖氏犁齿鳚、浪花银汉鱼、褐菖鲉和矶鳚在2月更为丰富。在5月显著富集的鱼种包括海鳗、龙头鱼、日本鳀、日本䲢和花鲈。11月的丰富鱼种为髭缟虾虎鱼、竹荚鱼和日本白姑鱼。

|
图 5 西轩岛近海水域不同季节鱼种进化树 Fig.5 Phylogenetic tree of fish species in Xixuan Island coastal waters at different sampling seasons |
分子方法为识别、评估和量化生物多样性提供了便捷手段,特别是针对生物多样性丰富以及缺乏研究的生态系统和地区(Schwartz et al, 2007)。本研究使用环境DNA metabarcoding技术分析了西轩岛近海的鱼类多样性,共监测到33种鱼类。由于缺少同步开展的网具调查结果(渔获物),无法同时验证环境DNA与鱼类资源之间的相关性。为了佐证环境DNA分析结果的可靠性,通过查阅先前使用传统底拖网的调查结果并与之对比分析。关于西轩岛近海的相关研究仅见Wang等(2019),其调查方式为刺网。本研究使用eDNA metabarcoding技术监测得到的结果与刺网的渔获物种类存在差异,但均为舟山海域常见鱼种。可能有以下几点原因:首先由于本研究采样点设置在岸边,采集水样深度较浅,均为表层水(0~1 m),无法监测到在中下层栖息的鱼类;其次,所参考研究的采样时间为2016年12月和2017年1月、4月和5月,主要集中在冬季和春季,而环境DNA结果会因采样时间和采样点的不同而产生差异,造成这种差异的原因一方面可能是由于本次调查设计的采样位点较少,采集水样无法完全代表西轩岛近海;另一方面可能由于近岸水体多泥沙,含有大量的PCR抑制剂(腐殖酸等),使得结果产生偏差。鱼类在不同季节的体型、年龄和生命阶段各不相同,同一个体的不同生命阶段会产生不同量的环境DNA,会影响最终的分析结果。最后,刺网采集得到的渔获组成信息可能有限,渔获物的数量与作业时间不成正比,当渔获达到一定限度的时候,渔获率相对降低(徐胜南等, 2016)。此外,刺网对小个体鱼的网获量相对较低,渔获率还与天气、水透明度、调查持续时间及鱼类自身的游泳、视觉能力及体态等因素有关(Olin et al, 2003; Hamley et al, 1975; Minns et al, 1998; Buijse et al, 1992)。
对于环境DNA技术与传统调查结果的差异在其他研究中也有所体现。例如,对南大洋进行拖网和环境DNA作业所获得的样本的分析结果显示,eDNA技术所检测物种覆盖了44.45%的拖网捕获物种(曹帅, 2022);李晓玲等(2022)基于环境DNA技术对夏季东海鱼类物种多样性进行研究,发现环境DNA分析结果与东海传统渔业资源调查相比,存在一些未曾报道或报道较少的鱼类;蒋佩文等(2022)使用环境DNA和底拖网结合的方法对珠江河口鱼类多样性开展调查,发现34%的物种没有出现在历史统计数据中。综上所述,不论是环境DNA技术还是传统调查方式,均存在一定的缺陷,这些问题很有可能影响结果的准确性。因此,应当将环境DNA方法与传统的调查方法相结合,以确保监测结果的准确性和可靠性。
为了进一步验证eDNA metabarcoding监测结果,统计了西轩岛及其邻近海域其他渔业资源调查研究结果(张龙, 2011; 于南京等, 2020; 唐嘉威等, 2021; 陆延等, 2022; 汪振华等, 2012; 樊紫薇, 2020; 王业辉, 2021)。如图 6所示,图中深色色块代表有数据记录,浅色色块代表无数据记录。环境DNA检出的75.76%的鱼类在舟山海域报道结果中均有出现,表明环境DNA技术进行鱼类物种多样性研究的可行性。
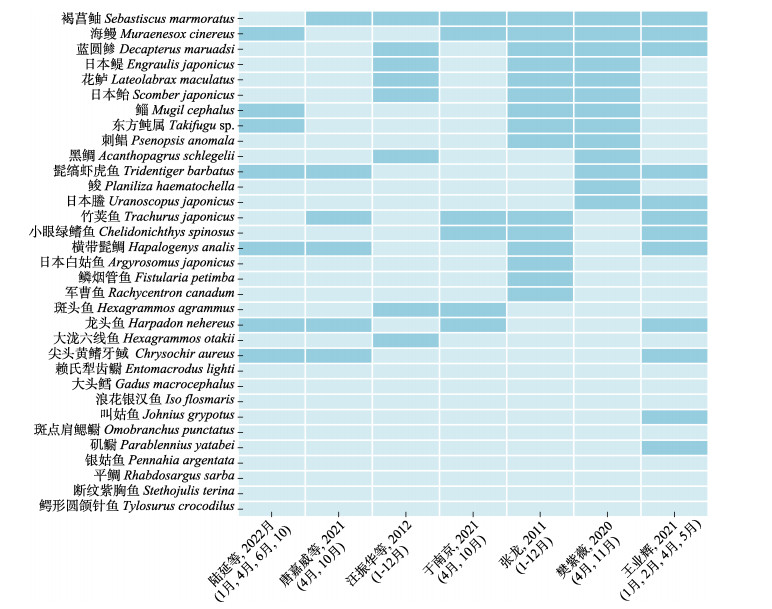
|
图 6 鱼种在传统调查方法中的检出情况 Fig.6 Detection of fish species by traditional survey methods 深色色块代表有数据记录,浅色色块代表无数据记录。 Dark blocks mean that data is recorded, and light blocks mean that no data is recorded. |
本研究中的4个采样点的α多样性各项指数并无差异,然而这些采样点无论是在物种种类水平还是在序列丰度水平上仍然揭示了鱼类空间分布的微小差异。研究表明,鱼类多样性受经度变化的影响(Yemane et al, 2015)。本研究中,采样点S04和S03鱼种检出数分别为22种和14种,相比于低经度的S01和S02站位检出鱼种数更多,这可能是因为S04和S03距离舟山本岛更远,更容易受到各种洋流的影响,因此鱼类多样性也较高,也可能与海底地形、水深不同有关,仍需要广泛的空间采样才能够揭示更细致的鱼类空间分布模式和群落结构。
对于不同采样时间,5月的多样性指数和均匀度指数最高,其次是11月,2月各项指数均为最低。之前的研究结果也显示出相似的变化趋势,即春季和夏季多样性指数较冬季、秋季大(张龙, 2011)。2月和5月检出鱼种数均约为11月检出鱼种数的2倍,这种差异可能与鱼类繁殖活动有关。一般来说,位于温带和亚热带的海域,鱼类丰度会在春季和夏季达到峰值,这是由于多数鱼类的繁殖期为春季和夏季(Young et al, 2003)。在本调查检出的鱼种中,大多数繁殖期均在2—5月,在此时间段检出的鱼种较为丰富。对特定目标物种的研究中也报告了环境DNA浓度或监测频率在繁殖季节有所增加(Fukumoto et al, 2015; Spear et al, 2015; Bylemans et al, 2017)。因此,使用环境DNA metabarcoding也被认为在繁殖季节更容易捕获环境DNA,从而导致监测到的物种数量增加(Hill, 1973)。与春季相比,冬季监测到环境DNA的概率往往较低(Turner et al, 2014; Buxton et al, 2018)。本研究也观察到同样的趋势,在2月和5月监测出的鱼种在多样性和均匀度上均有差异。此外,过去基于拖网调查结果的研究也证实了这种差异(张龙, 2011; 于南京, 2021)。最近的一项使用环境DNA metabarcoding对舟山近海鱼类多样性的研究报道了夏季的生物多样性指数显著高于冬季(Zhou et al, 2022),因此,温度可能是影响舟山近海多样性的重要因素。从不同采样时间和位点的结果可以得出结论,在繁殖季节且远离舟山本岛侧的采样点监测到的鱼种数量更多。
通过进化树富集各季节的优势鱼种与传统调查手段的结果有较大差异。例如,龙头鱼、黄
环境DNA metabarcoding的发展与应用为鱼类多样性的调查研究和生态保护提供了便捷。相比于传统渔业资源的监测手段,环境DNA方法往往可以捕捉到更多的信息,特别是对于隐蔽种、稀有种以及早期入侵种的发现(Song et al, 2017; Balasingham et al, 2018; Takeuchi et al, 2019)。然而,在实际应用中,使用环境DNA metabarcoding监测鱼类多样性还存在诸多问题。数据库的准确性和完整性是保证环境DNA metabarcoding结果的必要条件,目前仍存在数据库的错误和缺失,可能会导致假阴性监测(Rey et al, 2019; Duarte et al, 2021)。此外,环境DNA监测结果无法提供鱼类个体的具体细节,如长度、重量、生长阶段和生存情况(Buxton et al, 2017; Deutschmann et al, 2019; Kasai et al, 2021)。因此,无法确定这些阳性信号是来自幼鱼还是成鱼以及存活与否。而鱼类在不同季节的体型、年龄和生命阶段各不相同,同一个体的不同生命阶段会产生不同量的环境DNA,可能会影响后续分析结果。不同采样点的水环境也有所差异,对于各种海流如何影响环境DNA在开放海域中的运输、降解和稀释,以及监测概率如何随环境DNA释放源的距离而变化,仍然存在知识盲区(Jane et al, 2015; Hansen et al, 2018; Duda et al, 2020; Spence et al, 2020)。由于这些原因,在具体应用中仔细考虑环境DNA的生态细节是至关重要的(Rees et al, 2014; Thomsen et al, 2015),例如采样点、采样深度、采样季节、水化学以及人类活动等因素的影响(单秀娟等, 2018; 李苗等, 2020)。最近诸多研究通过使用多种方法的交叉应用以提高环境DNA监测的准确性,例如水动力模型和占用模型(Duda et al, 2020; Brys et al, 2021; Fukaya et al, 2021)。
环境DNA metabarcoding技术越来越多地被用于海洋生物多样性研究。然而,目前该技术仍不能完全替代传统调查方法,尚需深入研究以提高环境DNA的检测效率及准确性,但可以将环境DNA方法与传统的调查方法相结合,以确保监测结果的准确性和可靠性。未来可以将环境DNA metabarcoding技术作为一种辅助手段应用于渔业资源监测,提高监测效率并减少对生态系统的干扰。
致谢: 感谢日本神户大学人间发达环境学研究科源利文教授实验室邬倩倩博士和吴卢汉博士帮助获得实验数据。
AYLAGAS E, BORJA Á, IRIGOIEN X, et al. Benchmarking DNA metabarcoding for biodiversity-based monitoring and assessment. Frontiers in Marine Science, 2016, 3: 96 |
BALASINGHAM K D, WALTER R P, MANDRAK N E, et al. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries. Molecular Ecology, 2018, 27(1): 112-127 DOI:10.1111/mec.14395 |
BRYS R, HAEGEMAN A, HALFMAERTEN D, et al. Monitoring of spatiotemporal occupancy patterns of fish and amphibian species in a lentic aquatic system using environmental DNA. Molecular Ecology, 2021, 30(13): 3097-3110 DOI:10.1111/mec.15742 |
BUIJSE A D, SCHAAP L A, BUST T P. Influence of water clarity on the catchability of six freshwater fish species in bottom trawls. Canadian Journal of Fisheries and Aquatic Sciences, 1992, 49(5): 885-893 DOI:10.1139/f92-099 |
BUXTON A S, GROOMBRIDGE J J, GRIFFITHS R A. Seasonal variation in environmental DNA detection in sediment and water samples. PLoS One, 2018, 13(1): e0191737 DOI:10.1371/journal.pone.0191737 |
BUXTON A S, GROOMBRIDGE J J, ZAKARIA N B, et al. Seasonal variation in environmental DNA in relation to population size and environmental factors. Scientific Reports, 2017, 7: 46294 DOI:10.1038/srep46294 |
BYLEMANS J, FURLAN E M, HARDY C M, et al. An environmental DNA-based method for monitoring spawning activity: A case study, using the endangered Macquarie perch (Macquaria australasica). Methods in Ecology and Evolution, 2017, 8(5): 646-655 DOI:10.1111/2041-210X.12709 |
CAO S. Antarctic fish diversity based on DNA barcoding and eDNA metabarcoding. Master´s Thesis of Shanghai Ocean University, 2022 [曹帅. 基于DNA条形码和环境DNA的南极鱼类多样性研究. 上海海洋大学硕士研究生学位论文, 2022]
|
CHEN L, WU L, LIU Y, et al. Application of environmental DNA metabarcoding in ecology. Acta Ecologica Sinica, 2016, 36(15): 4573-4582 [陈炼, 吴琳, 刘燕, 等. 环境DNA metabarcoding及其在生态学研究中的应用. 生态学报, 2016, 36(15): 4573-4582] |
DEUTSCHMANN B, MULLER A K, HOLLERT H, et al. Assessing the fate of brown trout (Salmo trutta) environmental DNA in a natural stream using a sensitive and specific dual-labelled probe. Science of the Total Environment, 2019, 655: 321-327 DOI:10.1016/j.scitotenv.2018.11.247 |
DRUMMOND J A, LARSON E R, LI Y, et al. Diversity metrics are robust to differences in sampling location and depth for environmental DNA of plants in small temperate lakes. Frontiers in Environmental Science, 2021, 9: 617924 DOI:10.3389/fenvs.2021.617924 |
DUARTE S, VIEIRA P E, LAVRADOR A S, et al. Status and prospects of marine NIS detection and monitoring through (e) DNA metabarcoding. Science of the Total Environment, 2021, 751: 141729 DOI:10.1016/j.scitotenv.2020.141729 |
DUDA J J, HOY M S, CHASE D M, et al. Environmental DNA is an effective tool to track recolonizing migratory fish following large-scale dam removal. Environmental DNA, 2020, 3(1): 121-141 |
FAN Z W. A study on the structural characteristics of ichthyoplankton in the middle of Zhejiang Province coastal waters. Master´s Thesis of Zhejiang Ocean University, 2020 [樊紫薇. 浙江中部近岸海域鱼卵、仔稚鱼群落结构特征研究. 浙江海洋大学硕士研究生学位论文, 2020]
|
FUKAYA K, MURAKAMI H, YOON S, et al. Estimating fish population abundance by integrating quantitative data on environmental DNA and hydrodynamic modelling. Molecular Ecology, 2021, 30(13): 3057-3067 DOI:10.1111/mec.15530 |
FUKUMOTO S, USHIMARU A, MINAMOTO T. A basin-scale application of environmental DNA assessment for rare endemic species and closely related exotic species in rivers: A case study of giant salamanders in Japan. Journal of Applied Ecology, 2015, 52(2): 358-365 DOI:10.1111/1365-2664.12392 |
GAO T X, CHEN Z, WANG X Y. Environmental DNA, a new method for fish diversity investigation in the coastal waters. Journal of Zhejiang Ocean University (Natural Science), 2018, 37(1): 1-7 [高天翔, 陈治, 王晓艳. 近海鱼类多样性调查新方法——环境DNA分析技术. 浙江海洋大学学报(自然科学版), 2018, 37(1): 1-7] |
GUO C B, LEK S, YE S, et al. Predicting fish species richness and assemblages with climatic, geographic and morphometric factors: A broad-scale study in Chinese lakes. Limnologica, 2015, 54: 66-74 DOI:10.1016/j.limno.2015.08.002 |
HAMLEY J M. Review of gillnet selectivity. Journal of the Fisheries Research Board of Canada, 1975, 32(11): 1943-1969 DOI:10.1139/f75-233 |
HANSEN B K, BEKKEVOLD D, CLAUSEN L W, et al. The sceptical optimist: Challenges and perspectives for the application of environmental DNA in marine fisheries. Fish and Fisheries, 2018, 19(5): 751-768 DOI:10.1111/faf.12286 |
HILL M O. Diversity and evenness: A unifying notation and its consequences. Ecology, 1973, 54(2): 427-432 DOI:10.2307/1934352 |
HUANG H, SUN Y, YU W, et al. Analysis of the characteristics of eco-exergy-based indices and diversity indices in the Zhoushan archipelago, China. Environmental Science and Pollution Research International, 2020, 27(30): 37278-37285 DOI:10.1007/s11356-019-07544-2 |
JANE S F, WILCOX T M, MCKELVEY K S, et al. Distance, flow and PCR inhibition: eDNA dynamics in two headwater streams. Molecular Ecology Resources, 2015, 15(1): 216-227 DOI:10.1111/1755-0998.12285 |
JIANG P W, LI M, ZHANG S, et al. Investigating the fish diversity in Pearl River estuary based on environmental DNA matebarcoding and bottom trawling. Acta Hydrobiologica Sinica, 2022, 46(11): 1701-1711 [蒋佩文, 李敏, 张帅, 等. 基于环境DNA宏条码和底拖网的珠江河口鱼类多样性. 水生生物学报, 2022, 46(11): 1701-1711 DOI:10.7541/2022.2021.0265] |
JIANG W, ZHAO H, DENG J, et al. Detection of aquatic species using environmental DNA. Journal of Hydroecology, 2016, 37(5): 1-7 [姜维, 赵虎, 邓捷, 等. 环境DNA分析技术——一种水生生物调查新方法. 水生态学杂志, 2016, 37(5): 1-7] |
KASAI A, YAMAZAKI A, AHN H, et al. Distribution of Japanese eel Anguilla japonica revealed by environmental DNA. Frontiers in Ecology and Evolution, 2021, 9: 621461 DOI:10.3389/fevo.2021.621461 |
KELLY R P, PORT J A, YAMAHARA K M, et al. Using environmental DNA to census marine fishes in a large mesocosm. PLoS One, 2014, 9(1): e86175 DOI:10.1371/journal.pone.0086175 |
LI M, SHAN X J, WANG W J, et al. Studying the retention time of Fenneropenaeus chinensis eDNA in water. Progress in Fishery Sciences, 2020, 41(1): 51-57 [李苗, 单秀娟, 王伟继, 等. 环境DNA在水体中存留时间的检测研究——以中国对虾为例. 渔业科学进展, 2020, 41(1): 51-57] |
LI X L, LIU Y, WANG C C, et al. Study on fish species diversity in the East China Sea in summer based on environmental DNA technology. Haiyang Xuebao, 2022, 44(4): 78-84 [李晓玲, 刘洋, 王丛丛, 等. 基于环境DNA技术的夏季东海鱼类物种多样性研究. 海洋学报, 2022, 44(4): 78-84] |
LODGE D M, TURNER C R, JERDE C L, et al. Conservation in a cup of water: Estimating biodiversity and population abundance from environmental DNA. Molecular Ecology, 2012, 21(11): 2555-2558 DOI:10.1111/j.1365-294X.2012.05600.x |
LU Y, NIU W Z, CHEN A Y, et al. Fish community structure and biodiversity in the northern sea area of Zhoushan Island. Journal of Dalian Ocean University, 2022, 37(6): 1022-1031 [陆延, 牛威震, 程爱勇, 等. 舟山岛北部海域鱼类群落结构及其生物多样性. 大连海洋大学学报, 2022, 37(6): 1022-1031] |
MA H J, KATHRYN S, MA L M, et al. Environmental DNA and its application in protecting aquatic ecosystems. Chinese Journal of Ecology, 2016, 35(2): 516-523 [马鸿娟, Kathryn S, 马利民, 等. 环境DNA及其在水生生态系统保护中的应用. 生态学杂志, 2016, 35(2): 516-523] |
MINNS C K, HURLEY D A. Effects of net length and set time on fish catches in gill nets. North American Journal of Fisheries Management, 1988, 8(2): 216-223 DOI:10.1577/1548-8675(1988)008<0216:EONLAS>2.3.CO;2 |
MIYA M, SATO Y, FUKUNAGA T, et al. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: Detection of more than 230 subtropical marine species. Royal Society Open Science, 2015, 2(7): 150088 DOI:10.1098/rsos.150088 |
OLIN M, MALINEN T. Comparison of gillnet and trawl in diurnal fish community sampling. Hydrobiologia, 2003, 506/507/508/509(1/2/3): 443-449 |
PARK Y S, GRENOUILLET G, ESPERANCE B, et al. Stream fish assemblages and basin land cover in a river network. Science of the Total Environment, 2006, 365(1/2/3): 140-153 |
PIMM S L, JENKINS C N, ABELL R, et al. The biodiversity of species and their rates of extinction, distribution, and protection. Science, 2014, 344(6187): 1246752 DOI:10.1126/science.1246752 |
REES H C, MADDISON B C, MIDDLEDITCH D J, et al. Review: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology, 2014, 51(5): 1450-1459 DOI:10.1111/1365-2664.12306 |
REID A J, CARLSON A K, CREED I F, et al. Emerging threats and persistent conservation challenges for freshwater biodiversity. Biological Reviews, 2018, 94(3): 849-873 |
REY A, CARNEY K J, QUINONES L E, et al. Environmental DNA metabarcoding: A promising tool for ballast water monitoring. Environmental Science and Technology, 2019, 53(20): 11849-11859 DOI:10.1021/acs.est.9b01855 |
SCHWARTZ M K, LUIKART G, WAPLES R S. Genetic monitoring as a promising tool for conservation and management. Trends in Ecology and Evolution, 2007, 22(1): 25-33 DOI:10.1016/j.tree.2006.08.009 |
SHAN X J, LI M, WANG W J. Application of environmental DNA technology in aquatic ecosystem. Progress in Fishery Sciences, 2018, 39(3): 23-29 [单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29] |
SONG J W, SMALL M J, CASMAN E A. Making sense of the noise: The effect of hydrology on silver carp eDNA detection in the Chicago area waterway system. Science of the Total Environment, 2017, 605/606: 713-720 DOI:10.1016/j.scitotenv.2017.06.255 |
SPEAR S F, GROVES J D, WILLIAMS L A, et al. Using environmental DNA methods to improve detectability in a hellbender (Cryptobranchus alleganiensis) monitoring program. Biological Conservation, 2015, 183: 38-45 DOI:10.1016/j.biocon.2014.11.016 |
SPENCE B C, RUNDIO D E, DEMETRAS N J, et al. Efficacy of environmental DNA sampling to detect the occurrence of endangered coho salmon (Oncorhynchus kisutch) in Mediterranean‐climate streams of California's central coast. Environmental DNA, 2020, 3(4): 727-744 |
STAT M, HUGGETT M J, BERNASCONI R, et al. Ecosystem biomonitoring with eDNA: Metabarcoding across the tree of life in a tropical marine environment. Scientific Reports, 2017, 7(1): 12240 DOI:10.1038/s41598-017-12501-5 |
TAKEUCHI, A, SADO T, GOTOH R O, et al. New PCR primers for metabarcoding environmental DNA from freshwater eels, genus Anguilla. Scientific Reports, 2019, 9(1): 7977 DOI:10.1038/s41598-019-44402-0 |
TANG J W, YU N J, ZHEN J, et al. Fish community structure and diversity in the Zhoushan fishing ground sea near Daxiao Yushan. Journal of Zhejiang Ocean University (Natural Science), 2021, 40(3): 209–217, 233 [唐嘉威, 于南京, 郑基, 等. 舟山渔场大小鱼山附近海域鱼类群落结构及生物多样性. 浙江海洋大学学报(自然科学版), 2021, 40(3): 209–217, 233 DOI:10.3969/j.issn.1008-830X.2021.03.003] |
THOMSEN P F, WILLERSLEV E. Environmental DNA –an emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183: 4-18 DOI:10.1016/j.biocon.2014.11.019 |
TURNER C R, BARNES M A, XU C C, et al. Particle size distribution and optimal capture of aqueous macrobial eDNA. Methods in Ecology and Evolution, 2014, 5(7): 676-684 DOI:10.1111/2041-210X.12206 |
VALENTINI A, TABERLET P, MIAUD C, et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942 DOI:10.1111/mec.13428 |
WANG S, LIU J, LI J C, et al. Environmental magnetic parameter characteristics as indicators of heavy metal pollution in the surface sediments off the Zhoushan Islands in the East China Sea. Marine Pollution Bulletin, 2020, 150: 110642 DOI:10.1016/j.marpolbul.2019.110642 |
WANG X Y, ZHANG H B, LU G Q, et al. Detection of an invasive species through an environmental DNA approach: The example of the red drum Sciaenops ocellatus in the East China Sea. Science of the Total Environment, 2022, 815: 152865 DOI:10.1016/j.scitotenv.2021.152865 |
WANG Y H, DUAN J N, SHI H L, et al. Species identification of small fish in Xixuan Island coastal waters of Zhoushan using DNA barcoding. Journal of Applied Ichthyology, 2019, 36(1): 75-84 |
WANG Y H, HU C Y, SONG N, et al. Taxonomic diversity of common fish species in Zhoushan coastal waters. Journal of Dalian Ocean University, 2021, 36(3): 488-494 [王业辉, 胡成业, 宋娜, 等. 舟山近海常见鱼类分类多样性研究. 大连海洋大学学报, 2021, 36(3): 488-494] |
WANG Z H, ZHANG S Y, CHEN Q M, et al. Fish community ecology in rocky reef habitat of Ma´an Archipelago. Ⅰ. Species composition and diversity. Biodiversity Science, 2012, 20(1): 41–50, 117–119 [汪振华, 章守宇, 陈清满, 等. 马鞍列岛岩礁生境鱼类群落生态学. Ⅰ. 种类组成和多样性. 生物多样性, 2012, 20(1): 41–50, 117–119] |
WU Q Q, SAKATA M K, WU D Y, et al. Application of environmental DNA metabarcoding in a lake with extensive algal blooms. Limnology, 2021, 22(3): 363-370 DOI:10.1007/s10201-021-00663-1 |
XU S N, WANG Z H, LIANG J L, et al. Use of different sampling tools for comparison of fish-aggregating effects along horizontal transect at two artificial reef sites in Shengsi. Journal of Fisheries of China, 2016, 40(5): 820-831 [徐胜南, 汪振华, 梁金玲, 等. 不同网具在人工鱼礁水平方向上诱集效果的比较. 水产学报, 2016, 40(5): 820-831] |
XU Y J, YU C G, ZHANG P, et al. Spring nekton community structure and its relationship with environmental variables in Hangzhou Bay-Zhoushan inshore waters. Journal of Fisheries of China, 2019, 43(3): 605-617 [许永久, 俞存根, 张平, 等. 杭州湾—舟山近海春季游泳动物群落结构及与环境因子的关系. 水产学报, 2019, 43(3): 605-617] |
XU Y, MA L, SUN Y, et al. Spatial variation of demersal fish diversity and distribution in the East China Sea: Impact of the bottom branches of the Kuroshio Current. Journal of Sea Research, 2019, 144: 22-32 DOI:10.1016/j.seares.2018.11.003 |
YEMANE D, MAFWILA S K, KATHENA J, et al. Spatio-temporal trends in diversity of demersal fish species in the Benguela current large marine ecosystem region. Fisheries Oceanography, 2015, 24: 102-121 DOI:10.1111/fog.12075 |
YOUNG G C, POTTER I C. Do the characteristics of the ichthyoplankton in an artificial and a natural entrance channel of a large estuary differ?. Estuarine, Coastal and Shelf Science, 2003, 56(3/4): 765-779 |
YU N J, YU C G, JIAN K K, et al. Analysis of fish species composition and diversity in the sea area around Shengsi Archipelago. Journal of Fisheries Research, 2020, 42(4): 293-301 [于南京, 俞存根, 菅康康, 等. 嵊泗列岛邻近海域鱼类种类组成及多样性分析. 渔业研究, 2020, 42(4): 293-301] |
YU N J, YU C G, XU Y J, et al. Fish community structure and biodiversity in the offshore waters of Zhoushan Islands in spring and autumn. Journal of Fisheries of China, 2021, 45(8): 1374-1383 [于南京, 俞存根, 许永久, 等. 舟山群岛外海域春秋季鱼类群落结构及生物多样性. 水产学报, 2021, 45(8): 1374-1383] |
ZHANG L. The fish community structure and diversity in north of Zhejiang set-net fishing ground. Master´s Thesis of Zhejiang Ocean University, 2011 [张龙. 浙江北部沿岸张网作业区鱼类群落结构与多样性的研究. 浙江海洋大学硕士研究生学位论文, 2011]
|
ZHOU S, FAN C R, XIA H M, et al. Combined use of eDNA metabarcoding and bottom trawling for the assessment of fish biodiversity in the Zhoushan sea. Frontiers in Marine Science, 2022, 8: 809703 |



